
А не замахнуться ли нам на... изменение генома?
05 августа 2013
А не замахнуться ли нам на... изменение генома?
- 3855
- 0
- 11
Ученые научились редактировать молекулу ДНК в интересующем месте
-
Автор
-
Редакторы
Статья на конкурс «био/мол/текст»: Человек стремится покорить природу, и ключ для этого — ДНК. Но чтобы подобраться к молекуле ДНК и повлиять на нее, необходимо использовать точнейшие молекулярные инструменты — белки или РНК. В этой статье рассказывается о новейшем инструменте для внесения мутаций в геном in vivo — сиквенс-cпецифичных нуклеазах.
Конкурс «био/мол/текст»-2013
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2013 в номинации «Лучший обзор».
Спонсор конкурса — дальновидная компания Thermo Fisher Scientific. Спонсор приза зрительских симпатий — фирма Helicon.
История изучения ДНК непроста, как и любое значительное исследование. Открыта она была еще в 1868 году Иоганном Фридрихом Мишером, который выделил некое вещество, содержащее азот и фосфор, из остатков клеток в гное [1]. После того, как Мишер обнаружил, что оно обладает кислотными свойствами, вещество назвали нуклеиновой кислотой. Однако долгое время функция ДНК была неясной: ее считали всего лишь способом запасания фосфора в организме. Данная теория считалась правильной почти до середины XX века; в качестве косвенного доказательства ученые ссылались на однообразное, по их мнению, строение молекулы ДНК (состоящей исключительно из четырех различных видов нуклеотидов: дезоксиаденозина — А, дезоксигуанозина — G, дезокситимидина — Т, дезоксицитидина — С).
Носителями генетической информации первоначально предполагались белки. Потребовались опыты нескольких групп ученых, чтобы опровергнуть эту теорию. В частности, эксперименты Освальда Эвери, Колина Мак-Леода и Маклин Мак-Карти в 1944 году [2] показали, что для трансформации бактерий — приобретения болезнетворных свойств ранее безвредной культурой — необходима именно ДНК, выделенная из мертвых болезнетворных бактерий. В 1952 году были проведены опыты Альфреда Херши и Марты Чейз [3], благодаря которым выяснили, что при заражении фагами бактерий внутрь последних впрыскивается только ДНК первых, в то время как белок остается снаружи. Новое поколение фагов, однако, полностью повторяет родительское как по белку, так и по содержащейся ДНК.
Функцию ДНК, естественно, открыли раньше, чем ее вторичную структуру, которая оставалась неизвестной вплоть до 1953 года. Соответственно, из-за этого не могли установить, как проходят такие процессы как передача наследственной информации, как белки распознают определенные последовательности в ДНК и т.п.
В 1949–1951 годах Эрвин Чаргафф сформулировал свои знаменитые правила, которые говорят о строгом соотношении количества азотистых оснований внутри каждой молекулы ДНК [4] (в частности, что количество аденина равняется количеству тимина, а количество цитозина — количеству гуанина); затем Морисом Уилкинсом и Розалиндой Франклин были получены рентгеноструктурные данные, несущие структурную информацию о ДНК. На основании этих двух фактов Френсис Крик и Джеймс Уотсон предложили структуру двойной спирали ДНК [5], которая была доказана позднее в опытах Мэтью Мезельсона и Франклина Сталя [6].
Как только была открыта вторичная структура ДНК, стало легче предсказывать, а затем доказывать экспериментами механизмы различных процессов, происходящих с ДНК. Репликация (удвоение молекулы ДНК), транскрипция (перенос генетической информации с ДНК на РНК), различная регуляция экспрессии генов стали главными проблемами, интересующими молекулярных биологов.
Очевидно, что для осуществления этих процессов необходимы определенные белки, которые, в свою очередь, взаимодействуют как друг с другом, так и с ДНК. И если некоторые стадии протекают неспецифично (например, у эукариот репликация ДНК может начаться во множестве участков ДНК [7]), то, например, регуляция экспрессии генов происходит с помощью взаимодействия белков с довольно консервативными, т.е. строгими последовательностями ДНК.
Эндонуклеазы рестрикции: «Режем ДНК в нужном вам месте!»
Одними из первых открытых белков, специфически связывающих ДНК, являются эндонуклеазы рестрикции. Это ферменты, которые разрезают ДНК по специфическим нуклеотидным последовательностям, называемым сайтами рестрикции (рис. 1) [8].
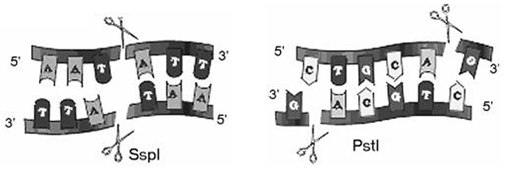
Рисунок 1. Рестрикция с образованием а) «тупых» концов; б) «липких» концов
Сам термин «рестрикция» появился благодаря исследованиям фага λ и феномена контролируемой рестрикции и модификации бактериальных вирусов клетками-хозяевами. Этот процесс был впервые обнаружен в результате работ Сальвадора Лурии [9] и Джузеппе Бертани [10] в 1950-х годах. Ученые обнаружили, что если фаг λ хорошо растет в одном штамме Escherichia coli, то в другом штамме его рост падает на несколько порядков, т.е. клетка-хозяин может снижать его биологическую активность. Оказалось, что запрет роста — «рестрикция» — вызывается ферментативным расщеплением ДНК фага. Соответственно, и фермент, отвечающий за этот процесс, назвали ферментом рестрикции.
Сами ферменты были обнаружены в лабораториях Вернера Арбера и Мэттью Мезельсона [11]; причем эндонуклеазы, которые исследовались в их работах, принадлежали, как определили позже, к I классу эндонуклеаз рестрикции. Они разрезают ДНК в случайном месте снаружи от места распознавания, причем как минимум на расстоянии 1000 нуклеотидов.
В 1970-х годах Гамильтон Смит, Томас Келли и Кент Велкокс выделили и охарактеризовали первую эндонуклеазу рестрикции II класса. Этот тип ферментов рестрикции более удобен для лабораторного использования, поскольку расщепляет ДНК внутри своего сайта распознавания, который, как правило, представляет собой палиндром из 4–8 нуклеотидов. Кроме того, эндонуклеазы этого класса не нуждаются в энергетической молекуле АТФ для активности, — только в ионах магния в качестве кофактора.
В 1990-х и ранних 2000-х годах были открыты другие представители данного семейства, которые не соответствовали всем критериям этого класса ферментов [12]. На данный момент уже открыты эндонуклеазы типов IIB, IIE, IIF, IIG, IIM, IIS, IIT... Возможно, в будущем этот список еще значительно расширится.
Существует также III класс эндонуклеаз рестрикции. Они были также открыты как компоненты прокариотической защитной системы против чужеродной ДНК. Эндонуклеазы III типа распознают две отдельных последовательности ДНК длиной 5–6 нуклеотидов и разрезают её примерно через 20–30 пар после сайта распознавания. Для их работы необходимы молекулы АТФ.
Эндонуклеазы рестрикции используют для обработки ДНК в различных лабораторных экспериментах. Обычно их применяют для вставки генов в процессах генного клонирования и экспериментах по исследованию белков.
Кроме того, эндонуклеазы могут быть использованы для выявления различий в аллелях гена [13]. Это возможно только в случае, если мутация произойдет в сайте узнавания, содержащемся внутри аллеля. Таким образом, отпадает нужда в дорогостоящем секвенировании гена; в частности, до изобретения секвенирования обработка эндонуклеазами рестрикции использовалась для картирования геномов.
Одно из новейших применений нуклеаз — внесение с помощью них различных изменений в геном клетки, таких как мутации уже имеющихся генов, вставка новых генов или вырезание исходных. Это очень перспективное направление генной инженерии с широчайшими возможностями практического применения! О нем будет рассказано в следующем разделе.
Эндонуклеазы — это эффекторы, т.е. белки действия. Если они окажутся у цели, то точно ее разрежут. Но природные белки распознают лишь небольшое число последовательностей ДНК, что накладывает огромные ограничения на использование этого уникального инструмента. Ах, если бы можно было доставить эндонуклеазу к любой желаемой последовательности ДНК!..
Революция в области ДНК-связывающих белков: «Доставим ваш белок по любому адресу ДНК, используя TAL-эффекторы или CRISPR/Cas9-систему»
Одной из наиболее заманчивых идей для ученых является возможность напрямую точечно влиять на работу клетки. Для этого необходимо прицельно воздействовать на ДНК именно в области интересующего гена. Воздействие может быть разного характера: включение/выключение гена, внесение мутации и др. Для этого потребовалось уметь синтезировать белки, узнающие конкретную последовательность ДНК («следопыты»), к которым можно было бы пришить белки «действия».
Недавно открытые белки TAL-эффекторы и система Cas9 произвели настоящую революцию в нацеливании различных эффекторов на специфическую последовательность ДНК!
TAL-эффекторы: «Используем паразитов во благо»
TAL-эффекторы (Transcription Activator Like Effectors) — белки, синтезируемые многочисленным видами бактерий рода Xanthomonas — паразитами растений. Они были открыты в 2009 году практически одновременно несколькими группами ученых. TAL-эффекторы активируют считывание нужных паразитам генов в растениях-хозяевах (крайне специфично связываясь с хозяйской ДНК), что облегчает бактериальную колонизацию этого растения и выживание бактерий внутри него [14].
TAL-эффектор можно мысленно поделить на три части (рис. 2); наибольший интерес представляет средняя, крайне консервативная центральная область белка, состоящая из повторяющихся блоков размером 34 аминокислоты. В 2009 же году была показана простая и однозначная связь между повторами и узнаваемыми нуклеотидами. Блоки отличаются друг от друга лишь аминокислотами № 12 и № 13, которые и определяют вид нуклеотида, распознаваемый данным мономером. На каждый из четырех нуклеотидов ДНК приходится определенное сочетание этих аминокислот TAL-эффектора [15]; т.е. можно представить себе этот белок как паровозик, состоящий из одинаковых вагонов, центральное окошко которых покрашено в один из четырех цветов.

Рисунок 2. Строение TAL-эффектора на примере avrBs3
Простота «TALE-кода» открывает многочисленные возможности для биологического применения этих белков. Недавние исследования показали, что можно специально подобрать «вагоны» для искусственного «паровозика» и синтезировать ранее не существовавшие в природе белки для узнавания конкретных последовательностей ДНК в различных типах клеток — как растений, так и млекопитающих. Многие группы ученых уже используют искусственные TAL-эффекторы, узнающие новые последовательности ДНК, например, в томатах, Arabidopsis thaliana [16] и клетках человека [17].
Синтезированные TAL-эффекторы также сшивают с каталитическим доменом нуклеазы (см. раздел «Эндонуклеазы рестрикции») для создания TAL-эффекторных нуклеаз. Эти нуклеазы могут быть полезны для генной инженерии и генной терапии [18].
TAL-нуклеазы используют для создания двуцепочечных разрывов в строго определенном месте ДНК. Известно, что нарушенные таким образом гены клетка может чинить двумя путями: либо просто соединением концов (что может привести к случайным мутациям), либо заново синтезируя поврежденную цепь по комплементарной ей цепи ДНК (т.н. гомологичная рекомбинация).
Во втором случае вырезается значительный по длине участок ДНК, а вместо него по комплементарной (на краях разрыва) матрице синтезируется вставка, закрывающая брешь [19]. Если в клетку поместить ДНК специально мутированного гена, то он будет использован в качестве донорной матрицы, и в геном встроится желаемая мутация. Можно таким методом даже вырезать отдельные гены — или вставлять новые! Таким образом, благодаря разработанной технике сборки ДНК-узнающих повторов, TAL-эффекторы в составе гибридных белков предоставляют возможность направить желаемый эффектор к любой специфической последовательности ДНК — и добиться самых разных воздействий на ДНК: от регуляции транскрипции до внесений мутаций.
CRISPR/Cas9 система: «Неожиданное альтернативное применение защитного комплекса бактерий»
В нашем организме, после того, как мы переболели какой-то болезнью, остаются клетки памяти иммунной системы, которые при повторном попадании в организм вредителей активизируются и помогают ускоренно справиться с ними.
Бактерии справляются со своими патогенами с помощью аналогичной «системы памяти»: CRISPR/Cas-системы обеспечивают иммунитет бактерий и архей против вирусов и плазмид. Этот защитный комплекс состоит из:
- «блокнотика памяти» — кассеты CRISPR, куда скопированы кусочки уникальных последовательностей ДНК поверженных вредителей, разделенные идентичными повторами;
- двух РНК, которые находят жертву — последовательность ДНК повторно вторгшегося патогена;
- белка, приводящего к подавлению чужеродных нуклеиновых кислот.
Этот комплекс был открыт в 2012 году при совместной работе ученых из США, Швейцарии, Австрии и Швеции [20], [21].
Иммунитет, обусловленный CRISPR/Cas, возникает за три стадии (рис. 3). В первой фазе бактерии или археи, у которых в геноме есть «блокнотик для записи» — CRISPR-кассета, — отвечают на появление вируса включением в конец кассеты коротких фрагментов чужеродной ДНК. На следующей стадии происходит транскрипция — считывание этой свежевключенной последовательности (образование описанных выше двух РНК) и обработка их нуклеазами. Теперь эти РНК могут специфично связаться с вирусной ДНК, и это взаимодействие через цепь событий приводит к ее расщеплению и «смерти» вредителя.
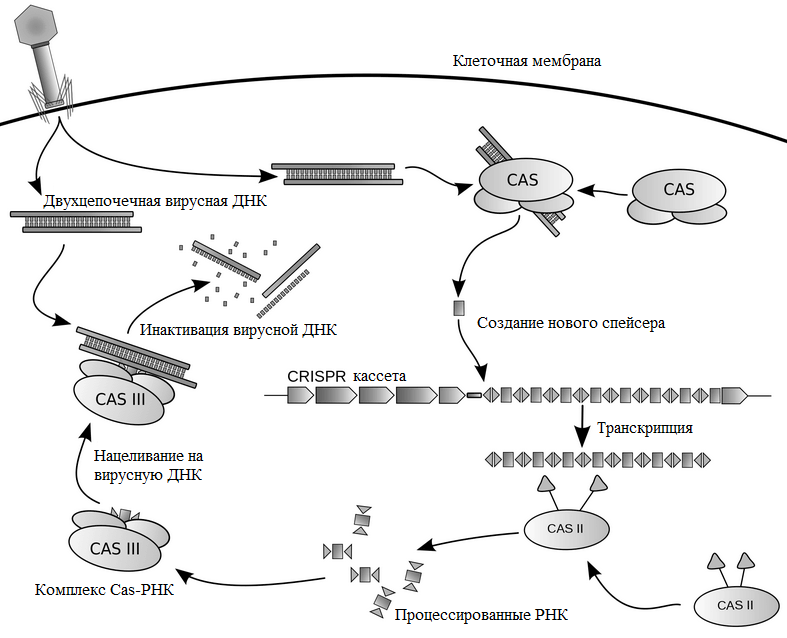
Рисунок 3. Схема работы комплекса Cas—РНК и CRISPR-кассеты
Грубо говоря, ситуация напоминает сказку про репку: первая РНК связывается с «репкой» — чужеродной ДНК — и «зовёт» вторую, вторая — специальный белок Cas9, который уже осуществляет двухцепочечный разрыв цепи чужой ДНК [22].
Несмотря на то, что в природе две РНК системы Cas9 разделены, учёные показали, что химерная РНК, представляющая собой сшитые в одну цепь вышеуказанные РНК, так же способна направлять Cas9 к ДНК-мишени. Более того, при соблюдении ряда условий можно менять последовательность первой внутри химерной РНК так, чтобы нацеливать Cas9 на желаемые гены.
Эта система даже проще TAL-эффекторов, поскольку для распознавания одного дезоксирибонуклеотида ДНК не нужен громоздкий «вагон» белка: один рибонуклеотид РНК легко и точно узнаёт комплементарный ему дезоксирибонуклеотид ДНК мишени! Экономия как в размере инструмента, так и в легкости его синтеза.
К настоящему времени показано, что эта система работает не только в археях и бактериях, но и в организмах рыб (Danio rerio), а также в клетках млекопитающих [23]. Уже установлено, что, как и в случае TAL-эффекторных нуклеаз, обработка ДНК Cas9-системой приводит к эффективному (10–30% случаев) возникновению мутаций.
Человек стремится покорить природу, и ключ для этого — ДНК. Но чтобы подобраться к ДНК и повлиять на нее, необходимо спуститься на молекулярный уровень и использовать белки или РНК. Сейчас развитие данных направлений идет стремительно, каждый месяц случаются значительные прорывы в этой области. Кто знает, какие чудеса ожидают нас через год? Возможно, будет найдена новая система, максимально упрощающая исходно сложный и кропотливый процесс вмешательства человека в работу природы.
Литература
- Ralf Dahm. (2005). Friedrich Miescher and the discovery of DNA. Developmental Biology. 278, 274-288;
- O. T. Avery. (1944). STUDIES ON THE CHEMICAL NATURE OF THE SUBSTANCE INDUCING TRANSFORMATION OF PNEUMOCOCCAL TYPES: INDUCTION OF TRANSFORMATION BY A DESOXYRIBONUCLEIC ACID FRACTION ISOLATED FROM PNEUMOCOCCUS TYPE III. Journal of Experimental Medicine. 79, 137-158;
- A. D. Hershey. (1952). INDEPENDENT FUNCTIONS OF VIRAL PROTEIN AND NUCLEIC ACID IN GROWTH OF BACTERIOPHAGE. The Journal of General Physiology. 36, 39-56;
- ERWIN CHARGAFF, STEPHEN ZAMENHOF, CHARLOTTE GREEN. (1950). Human Desoxypentose Nucleic Acid: Composition of Human Desoxypentose Nucleic Acid. Nature. 165, 756-757;
- J. D. WATSON, F. H. C. CRICK. (1974). Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid. Nature. 248, 765-765;
- M. Meselson, F. W. Stahl. (1958). The replication of DNA in Escherichia coli. Proceedings of the National Academy of Sciences. 44, 671-682;
- Heinz-Peter Nasheuer, Richard Smith, Christina Bauerschmidt, Frank Grosse, Klaus Weisshart. (2002). Initiation of eukaryotic DNA replication: Regulation and mechanisms. Progress in Nucleic Acid Research and Molecular Biology. 41-94;
- Richard J. Roberts, Kenneth Murray. (1976). Restriction Endonuclease. CRC Critical Reviews in Biochemistry. 4, 123-164;
- Luria S.E. and Human M.L. (1952). A nonhereditary, host-induced variation of bacterial viruses. J. Bacteriol. 64, 557–569;
- Bertani G. and Weigle J.J. (1953). Host controlled variation in bacterial viruses. J. Bacteriol. 65, 113–121;
- Seymour Lederberg, Matthew Meselson. (1964). Degradation of non-replicating bacteriophage DNA in non-accepting cells. Journal of Molecular Biology. 8, 623-628;
- A. Pingoud. (2001). Structure and function of type II restriction endonucleases. Nucleic Acids Research. 29, 3705-3727;
- R. Zhang, Z. Zhu, H. Zhu, T. Nguyen, F. Yao, et. al.. (2005). SNP Cutter: a comprehensive tool for SNP PCR-RFLP assay design. Nucleic Acids Research. 33, W489-W492;
- Jens Boch, Ulla Bonas. (2010). XanthomonasAvrBs3 Family-Type III Effectors: Discovery and Function. Annu. Rev. Phytopathol.. 48, 419-436;
- M. J. Moscou, A. J. Bogdanove. (2009). A Simple Cipher Governs DNA Recognition by TAL Effectors. Science. 326, 1501-1501;
- R. Morbitzer, P. Romer, J. Boch, T. Lahaye. (2010). Regulation of selected genome loci using de novo-engineered transcription activator-like effector (TALE)-type transcription factors. Proceedings of the National Academy of Sciences. 107, 21617-21622;
- Jeffrey C Miller, Siyuan Tan, Guijuan Qiao, Kyle A Barlow, Jianbin Wang, et. al.. (2011). A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29, 143-148;
- De Francesco L. (2011). Move over ZFNs. Nat Biotechnol. 29, 681-684;
- Alberts B. et al. Chapter 5: DNA Replication, Repair, and Recombination. In: Molecular Biology of the Cell (4th Edition). New York: Garland Science, 2002. 845 p.;
- M. Jinek, K. Chylinski, I. Fonfara, M. Hauer, J. A. Doudna, E. Charpentier. (2012). A Programmable Dual-RNA-Guided DNA Endonuclease in Adaptive Bacterial Immunity. Science. 337, 816-821;
- Элементы: «Прокариотическая система иммунитета поможет редактировать геном»;
- Woong Y Hwang, Yanfang Fu, Deepak Reyon, Morgan L Maeder, Shengdar Q Tsai, et. al.. (2013). Efficient genome editing in zebrafish using a CRISPR-Cas system. Nat Biotechnol. 31, 227-229;
- Seung Woo Cho, Sojung Kim, Jong Min Kim, Jin-Soo Kim. (2013). Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nat Biotechnol. 31, 230-232.








