
Беспечный фактор транскрипции: что он скрывает от системы репарации
10 июня 2021
Беспечный фактор транскрипции: что он скрывает от системы репарации
- 946
- 0
- 0
В клетках млекопитающих, включая соматические стволовые клетки и клетки опухолей, метилированный цитозин в динуклеотидах CpG часто мутирует в тимин. Неожиданно часто это происходит в участках связывания транскрипционных факторов семейства C/EBP. Устроено это так: происходит дезаминирование 5-метилцитозина, которое, превращает его в тимин и нарушает спаривание со стоящим в другой цепи гуанином. Такие неверные спаривания должна устранять система репарации. Но в CpG-динуклеотиде (обозначен как дорожный знак), находящемся в участке связывания белка C/EBP, правильное спаривание нуклеотидов не может быть восстановлено системой репарации (на рисунке ее представляет команда мастеров-сантехников), потому что место, куда должен был бы сесть занимающийся репарацией ферментный комплекс, прочно занято транскрипционным фактором C/EBP (на рисунке роль транскрипционного фактора выполняет довольная белка, купающаяся в луже — участке связывания).
иллюстрация Татьяны Русситы
-
Авторы
-
Редакторы
Темы
Транскрипционные факторы не только регулируют работу генов, но могут обладать и другими, порой неожиданными и даже нежелательными функциями. В этой статье — рассказ о такой функции, когда транскрипционный фактор C/EBP крепко связывается с частично поврежденной ДНК и не позволяет системе репарации починить спрятанный под собой участок ДНК. А что он спрятал и почему — сейчас и расскажем!
Долгое время было принято считать, что самую важную роль в геноме, то есть в ДНК, играют только белок-кодирующие участки — гены, кодирующие последовательности белков. Которые, к слову, у человека занимают лишь 1–2% от длины генома [1], [2]. Остальную часть генома называли темной материей, или мусорной ДНК, — разумеется, ошибочно. Но сегодня научные исследования, посвященные некодирующим участкам ДНК и их функциональной роли, расцветают все ярче и ярче.
Действительно, некодирующая ДНК способна на многое.
Первое, о чем обычно вспоминают, — повторяющиеся последовательности [3]. Правда, полезных их функций нам известно маловато.
Затем — гены некодирующих РНК, которые самостоятельно или при участии белков выполняют различную работу в клетке [4]. Систематическим изучением таких РНК занимается, например, международный консорциум FANTOM6.
Но и помимо повторов и РНК-генов, некодирующие участки генома содержат много интересного. Например, в них находятся регуляторные элементы (промоторы и энхансеры), которые определяют активность генов — как кодирующих, так и не кодирующих белки, — в различных типах клеток и в разных условиях. Работают они за счет привлечения специальных белков — факторов транскрипции, — которые способны распознать в ДНК сравнительно короткие, но характерные, паттерны (мотивы). В пределах регуляторных районов генома эти факторы связывают подходящие участки ДНК, а затем образуют белок-белковые комплексы друг с другом и с компонентами молекулярной машины, осуществляющей транскрипцию генов, — то есть непосредственно синтез РНК. Именно участками посадки факторов транскрипции (так называемыми ДНК-сайтами связывания, или просто сайтами связывания) мы, то есть наш дружный коллектив соавторов теперь уже опубликованной статьи [5], занимаемся уже больше 10 лет. Кстати, о компьютерном предсказании сайтов связывания в масштабе полного генома (на основе нашей работы) можно прочитать в статье «Биомолекулы» «Мечту вызывали?» [6].
Участки ДНК, непосредственно взаимодействующие с факторами транскрипции в геноме человека, сравнительно коротки — 10–20 нуклеотидов. Сила взаимодействия фактора транскрипции с ДНК (и как следствие, активность зависимого от него гена) может существенно меняться даже при замене всего одного нуклеотида-«буквы» (например, это приводит к аллель-специфичному связыванию участков [7], различающихся между материнской и отцовской хромосомами). То есть даже однонуклеотидные мутации могут вызывать серьезные нарушения генной регуляции: влиять на судьбу клеток и осложнять течение заболеваний. В частности, такие мутации способны превращать стволовые клетки человека в опухолевые или приводить к болезням, связанным с неправильной дифференцировкой клеток. Во многих опухолях часто находят специфичные мутации в промоторе гена теломеразы TERT [8]. Этот фермент необходим для репликации концов хромосом во время деления клеток. Логично, что в соматических клетках, которые не делятся, ген теломеразы находится в неактивном состоянии. Но если в промоторе этого гена происходят указанные выше специфичные мутации, то факторы транскрипции вновь начинают связываться с промотором, тем самым активируя ген теломеразы. Это позволяет клетке активно делиться, а эта способность, как мы знаем, — важное свойство опухолевых клеток [9].
Мутации, происходящие в соматических клетках (то есть во всех, кроме половых), называют соматическими. Обычно их изучают в опухолевых клетках, где часто затруднена работа систем репарации, а потому мутации накапливаются с высокой скоростью. На возникновение и частоту мутаций в популяции раковых клеток влияет сразу множество факторов: и отбор (как положительный, так и отрицательный), и активность процессов, напрямую вызывающих повреждения ДНК, и различная эффективность систем репарации. В общем, целый клубок, который достаточно сложно расплести, а результаты анализа, в итоге, сложно однозначно интерпретировать. Мы решили зайти с другой стороны и использовали опубликованные данные по мутагенезу соматических стволовых клеток взрослых и здоровых людей, где основной вклад в накопление мутаций вносят непосредственно мутационные процессы, без поломки репарационных систем и следов естественного отбора.
Для начала мы разделили мутации на группы, соответствующие известным мутационным процессам: например, вызванные влиянием активных форм кислорода или дезаминированием 5-метилцитозина. Затем мы наложили координаты мутаций на геномную карту всех известных участков связывания транскрипционных факторов. Оказалось, что как минимум треть мутаций расположена недалеко от участков связывания. Теперь имело смысл внимательно посмотреть на районы работы конкретных факторов транскрипции по отдельности.
Мы сопоставили паттерны в ДНК, узнаваемые определенными факторами транскрипции, с точным положением мутаций в пределах небольших «окошек» в геноме (не далее 50 нуклеотидов от мутации). Выяснилось, что мутации CpG-динуклеотидов вида [C>T]G, вызываемые дезаминированием 5-метилцитозина, неожиданно часто попадают точно в центр участков связывания белков C/EBP. Интересно, что этот эффект ранее обнаруживали для опухолевых клеток [10], но не смогли интерпретировать.
C/EPB: что за зверь?
Факторы транскрипции семейства C/EBP (CCAAT/enhancer-binding proteins) необходимы для нормальной дифференцировки различных типов клеток. Они активируют различные гены, взаимодействуя с их промоторами, а также могут взаимодействовать с белками клеточного цикла. Интересно, что белки этого семейства могут как подавлять переход соматических клеток в опухолевые, так и активировать его [11].
Чаще всего метилирование ДНК осуществляется по цитозину в динуклеотидах CpG. Это она из наиболее распространенных эпигенетических модификаций . Повышенная частота мутаций метилированных CpG-динуклеотидов за счет дезаминирования метилцитозина известна очень давно, но почему же участки связывания C/EBP мутируют еще в 2–4 раза чаще, чем ожидается? Могут ли транскрипционные факторы напрямую провоцировать дезаминирование цитозина в метилированных CpG в пределах своих сайтов? Эта гипотеза первой приходит в голову, но, сразу скажем, она слишком фантастическая. Ведь прочное связывание фактора транскрипции, наоборот, должно бы приводить к дополнительной защите ДНК от изменений.
Более обстоятельно с эпигенетикой вы можете познакомиться в одноименном спецпроекте «Биомолекулы», — в частности, в статье «Молекулы и эпигеном» [12].
Второй вариант, который мы рассматривали, был такой: может ли быть так, что транскрипционный фактор не дает возможности репарационной машине добраться до неспаренного основания и починить его? Например, из-за избыточно крепкой связи с таким «несовершенным» участком двойной спирали. Эта возможность — прочное связывание конкретных вариантов сайтов с неспаренными основаниями — недавно была показана для многих транскрипционных факторов. Но не для белков C/EBP, что тоже наталкивало на сомнения в «работоспособности» предположения, но мы решили его проверить.
Беспечный ездок
Один из широкого изучаемых белков семейства C/EBP — C/EBPβ. Для него в банке данных PDB доступны закристаллизованные комплексы ДНК-связывающего домена с каноническим (наилучшим возможным) ДНК-сайтом. За помощью мы обратились к коллегам (а они, забегая вперед в нашем рассказе, стали соавторами этой статьи), которые на основе этих структур провели моделирование комплекса белка C/EBPβ с различными вариантами участков ДНК. В одном случае, вместо цитозина в CpG-пару по одной из цепей вводили тимин, оставляя гуанин на противоположной цепи (тем самым образуя неверно спаренные нуклеотиды; см. рис. 1 для подробностей). А в другом случае вместе с заменой цитозина на тимин поменяли еще и гуанин на аденин, чтобы была сформирована каноническая пара — такая, какой она будет в одной из дочерних клеток после репликации ДНК и деления клетки с мутацией. И вот что интересно: наибольшее сродство к белку было у фрагмента ДНК, содержащего неканоническое спаривание нуклеотидов!
![Предложенная модель фиксации мутаций ([C>T]G) Предложенная модель фиксации мутаций ([C>T]G)](/img/content/3187/3187-01.model-fiksacii-mutacij.png)
Рисунок 1. Предложенная модель фиксации мутаций ([C>T]G), вызванных дезаминированием метилцитозина и повышенным сродством белков C/EBP к «поврежденным» сайтам в ДНК. А сродство белка к ДНК повышается благодаря одноцепочечной замене в центре сайта связывания.
Выходит, идея о том, что транскрипционный фактор мешает системе репарации, действительно может работать! Но расскажем о структурном моделировании до конца: если ранжировать по силе связи, то после этой последовательности (с неидеальной спиралью и наибольшим сродством к C/EBPβ-белку) шла каноническая форма, и только затем — та, где цитозин мутировал в тимин, а гуанин — в аденин. То есть и каноничный комплекс со спариванием G·C, и комплекс после мутации и репликации с A·T, не могут тягаться по силе с тем, как тяготеет транскрипционный фактор к неспаренным основаниям G·T. Здесь интересно, что мутировавший сайт c TG-, вместо CG-динуклеотида, существенно хуже связывает C/EBP. То есть в отсутствие давления естественного отбора геном мог бы постепенно лишиться всех сильных сайтов C/EBP. Судя по всему, сайты действительно постепенно теряются, но отбор «старается» сохранить полноценный сайт хотя бы на одной из парных хромосом, ведь гетерозиготные сайты C/EBP часто являются именно парой из CG- и TG-сайтов, как мы видели в работе [5].
Такая неожиданная согласованность результатов, пришедших и из статистического анализа мутаций, и из структурного моделирования, мотивировала нас проверить, как поведет себя комплекс в реальном мире. Ведь хотелось не только моделей, но и эксперимента.
Поэтому мы провели анализ электрофоретической подвижности в геле методом EMSA для точно таких же фрагментов ДНК, ровно как и в структурном моделировании (канонический участок связывания, фрагмент с неверно спаренными G·T-нуклеотидами и фрагмент «после репликации» со спариванием A·T). И эксперимент EMSA подтвердил высокое сродство комплекса именно с вариантом с неверно спаренными нуклеотидами.
В этом эксперименте мы «следили» за радиоактивно меченым фрагментом ДНК, в котором участок связывания был каноническим. Когда формируется комплекс с белком, подвижность ДНК в геле замедляется. Чтобы узнать о том, как мутации влияют на связывание C/EBPβ, и как при этом изменяется подвижность в геле, была устроена конкуренция между радиоактивно меченым каноническим (видимым) и немеченными (невидимыми) участками связывания ДНК. То есть мы устроили соревнование между каноническим участком связывания, фрагментом с неверно спаренными G·T-нуклеотидами и фрагментом «после репликации» со спариванием A·T. И что же получилось? А получилось, что победил в соревновании фрагмент ДНК с мутацией [C>T]pG! Он вытеснял белок C/EBPβ из комплекса даже лучше, чем канонический участок связывания!
Таким образом, мы сначала предсказали, а затем подтвердили — C/EBP чрезвычайно прочно связывает сайты ДНК после спонтанного дезаминирования метилцитозина в тимин. И, соответственно, система репарации не может добраться до замены и восстановить правильное их спаривание. А модель закрепления мутаций и общее описание проделанной работы можно изучить на рис. 1 и 2.
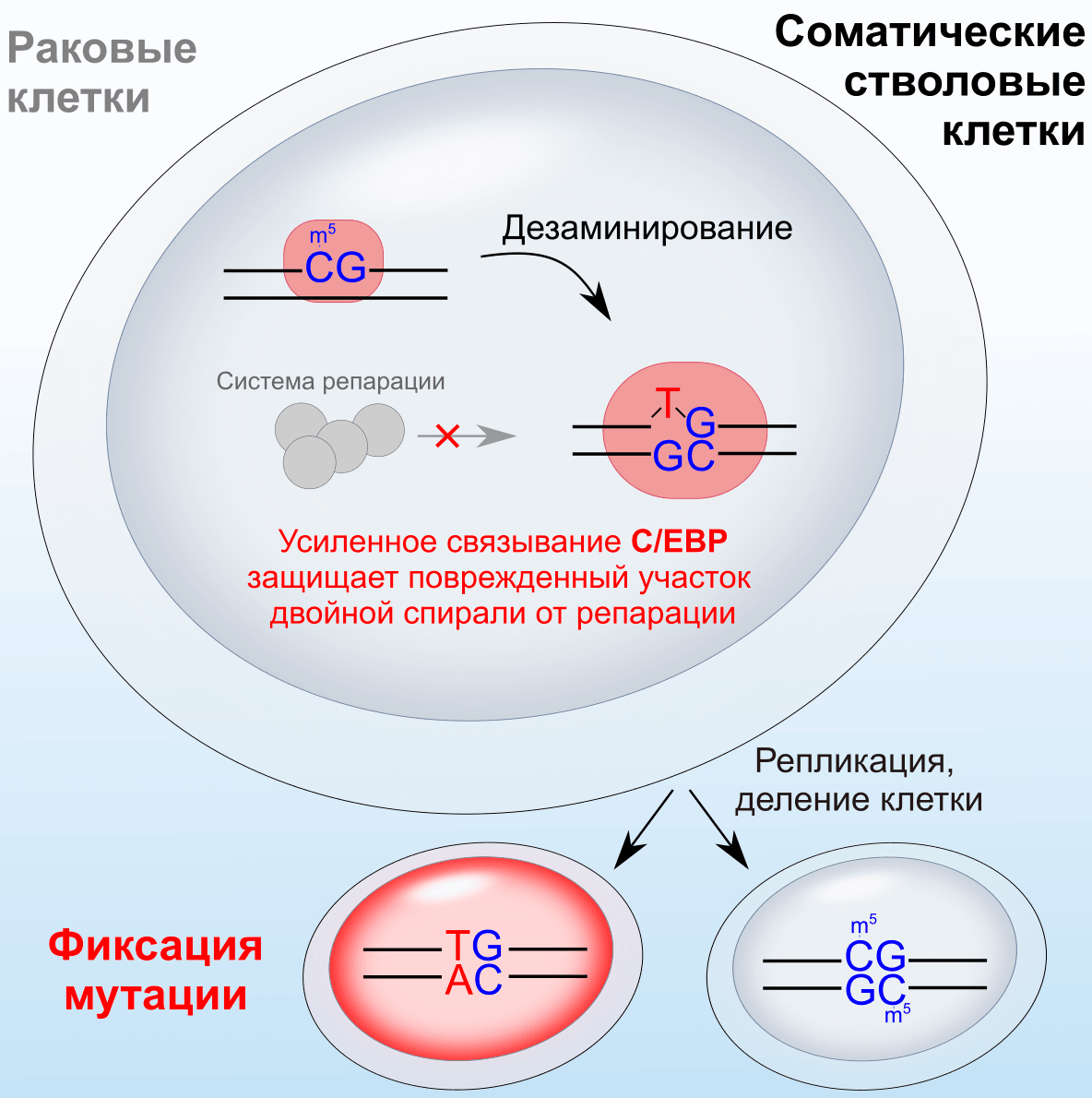
Рисунок 2. Краткое визуальное описание результата исследования
Печальная и поучительная история о конкуренции в науке
И, в завершение, короткая и немного грустная история о первенстве и конкуренции в науке. Год назад мы опубликовали препринт этой работы [13] на сервере bioRxiv. Затем статья некоторое время искала «свой журнал», а потом довольно долго находилась на рецензии в Cell Reports. И вроде бы, по меркам нашей науки, не так много времени ушло на рецензирование и ответы редактора, но пару месяцев назад, уже на финальной стадии, мы неожиданно обнаружили — вышла аналогичная работа в журнале Nucleic Acids Research, в которой показано примерно то же самое [14]. Причем на сложный путь от первоначальной подачи статьи в журнал до итоговой публикации авторам потребовалось всего лишь полтора месяца!
Совпадение? В научном мире в популярной теме, можно сказать, находящейся «на передовой», это не редкость. Может быть, авторы прочитали, но почему-то не стали цитировать наш прошлогодний препринт? Может быть, статью опубликовали так быстро, потому что один из авторов еще и редактор этого журнала? Можем ли мы вообще называть себя «первооткрывателями» интересного феномена? Ведь до нас успели опубликовать практически идентичные результаты, а наша статья все еще находилась на рассмотрении. К счастью, после связи с редакцией Cell Reports процесс публикации нашей статьи ускорился, и вот, 8 июня 2021 года, и наша статья [5], наконец, вышла.
Стоит ли возмущаться такой ситуацией, стоит ли винить рецензентов, которые незлонамеренно затягивали рецензирование? Является ли опубликованный в открытом доступе препринт гарантией научного первенства? Мы не знаем ответов на эти вопросы, но сама история может быть любопытной для тех, кому интересны разные грани работы ученых в современном мире, даже разочаровывающие. Но в науке, как и везде, где есть конкуренция, такие истории всегда будут случаться, и самое главное — не переживать и продолжать копать что-нибудь интересное.
Авторы выражают благодарность Марине Фридман и Алле Федоровой за помощь в работе над текстом.
Литература
- Геном человека: полезная книга, или глянцевый журнал?;
- Сколько сора в нашей ДНК;
- Повтор, еще повтор!;
- Обо всех РНК на свете, больших и малых;
- Anna S. Ershova, Irina A. Eliseeva, Oleg S. Nikonov, Alla D. Fedorova, Ilya E. Vorontsov, et. al.. (2021). Enhanced C/EBP binding to G·T mismatches facilitates fixation of CpG mutations in cancer and adult stem cells. Cell Reports. 35, 109221;
- Мечту вызывали?;
- Мухина В. (2021). Аллели регуляторных областей ДНК повлияли на поведение транскрипционных факторов. N+1;
- Rebecca C Hennessey, Kevin M Brown. (2021). Cancer regulatory variation. Current Opinion in Genetics & Development. 66, 41-49;
- François Hafezi, Danielle Perez Bercoff. (2020). The Solo Play of TERT Promoter Mutations. Cells. 9, 749;
- Collin Melton, Jason A Reuter, Damek V Spacek, Michael Snyder. (2015). Recurrent somatic mutations in regulatory regions of human cancer genomes. Nat Genet. 47, 710-716;
- Manlio Tolomeo, Stefania Grimaudo. (2020). The “Janus” Role of C/EBPs Family Members in Cancer Progression. IJMS. 21, 4308;
- Молекулы и эпигеном ;
- Ershova A.S., Eliseeva I.A., Nikonov O.S., Fedorova A.D. et al. (2020). Enhanced C/EBPs binding to C>T mismatches facilitates fixation of CpG mutations. bioRxiv;
- Jie Yang, John R Horton, Kadir C Akdemir, Jia Li, Yun Huang, et. al.. (2021). Preferential CEBP binding to T:G mismatches and increased C-to-T human somatic mutations. Nucleic Acids Research. 49, 5084-5094.






