Биоинформатика в мире РНК-структур
25 ноября 2014
Биоинформатика в мире РНК-структур
- 4858
- 0
- 5
Понимание, как формируется пространственная конфигурация РНК, необходимо для разработки методов предсказания вторичных структур молекул и определения выполняемых ими функций в клетке живых организмов
рисунок А.В. Головина
-
Автор
-
Редакторы
Статья на конкурс «био/мол/текст»: Амбициозный проект «Геном человека», завершившийся в 2003 году, был призван расшифровать всю ДНК, содержащуюся в человеческом геноме. Считалось, что проект поможет понять, как функционируют гены, каким образом они определяют состояние клетки и организма человека в целом, а также даст толчок развитию медицины и биологических наук. Однако исследования показали, что вся сложность клетки как целостной системы не сводится к одному лишь геному. Функционирование и роль молекулы РНК в регуляции клеточных процессов во многом зависят от ее пространственной организации, изучение которой — сложная задача, справиться с которой под силу только биоинформатике.
Конкурс «био/мол/текст»-2014
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2014 в номинации «Биоинформатика и молекулярная эволюция».
Главный спонсор конкурса — дальновидная компания «Генотек».
Конкурс поддержан ОАО «РВК».
Спонсором номинации «Биоинформатика» является Институт биоинформатики.
Спонсором приза зрительских симпатий выступила фирма Helicon.
Свой приз также вручает Фонд поддержки передовых биотехнологий.
Вторичная структура РНК
Проект «Геном человека» [1], [2] показал, что последовательность ДНК — это еще не всё. РНК, как и ДНК, имеет огромное значение для жизни. Внутри клеток существует целый «зоопарк» РНК [3]. К примеру, матричная РНК (мРНК) является важным посредником между ДНК и белками. Она синтезируется на основе ДНК, после чего в эукариотической клетке отправляется из ядра в цитоплазму, где используется в качестве шаблона для синтеза белков на рибосоме . Рибосомные РНК (рРНК) представляют собой основной компонент рибосом, в частности, рРНК большой субъединицы выполняет синтез полипептидной цепи, рРНК малой — прочтение мРНК в соответствии с генетическим кодом. Транспортные РНК (тРНК) переносят к аминокислоты к рибосомам, где происходит синтез белков. Другие виды РНК, такие как микроРНК или интерферирующие РНК, длинные некодирующие РНК, РНК-губки и циклические РНК, участвуют в клеточной регуляции. Всё разнообразие функций, выполняемых РНК, нельзя объяснить только на основании данных о последовательности нуклеотидов. Очевидно, в РНК есть что-то еще.
У прокариот процесс транскрипции и трансляции обычно сопряжен в пространстве и во времени. — Ред.
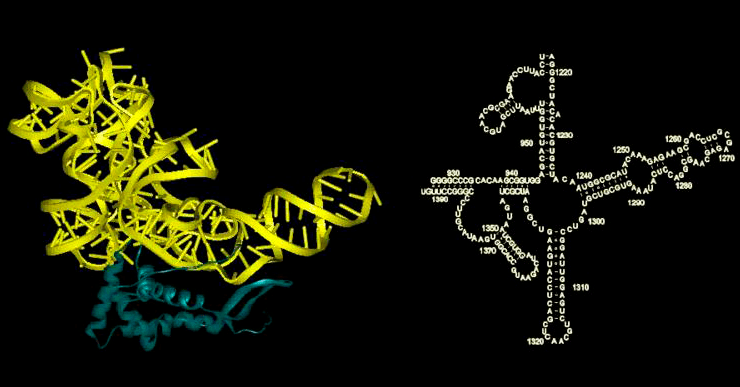
Дело в том, что РНК, подобно белковым молекулам [4], не существуют в виде простой цепочки. Нуклеотиды РНК взаимодействуют друг с другом, объединяясь в пары по принципу комплементарности, в результате чего молекула нуклеиновой кислоты приобретает причудливую конфигурацию: возникают петли, шпильки, псевдоузлы и другие формы (рис. 1). Если первичная структура — это просто последовательность нуклеотидов, то подобные образования относятся к вторичной структуре. И этот уровень организации молекулы также несет информацию, которая используется в регуляции таких процессов, как транскрипция ДНК, сплайсинг РНК, изменение активности генов с помощью микроРНК, а также транспорт транскриптов в определенные области клетки [5].
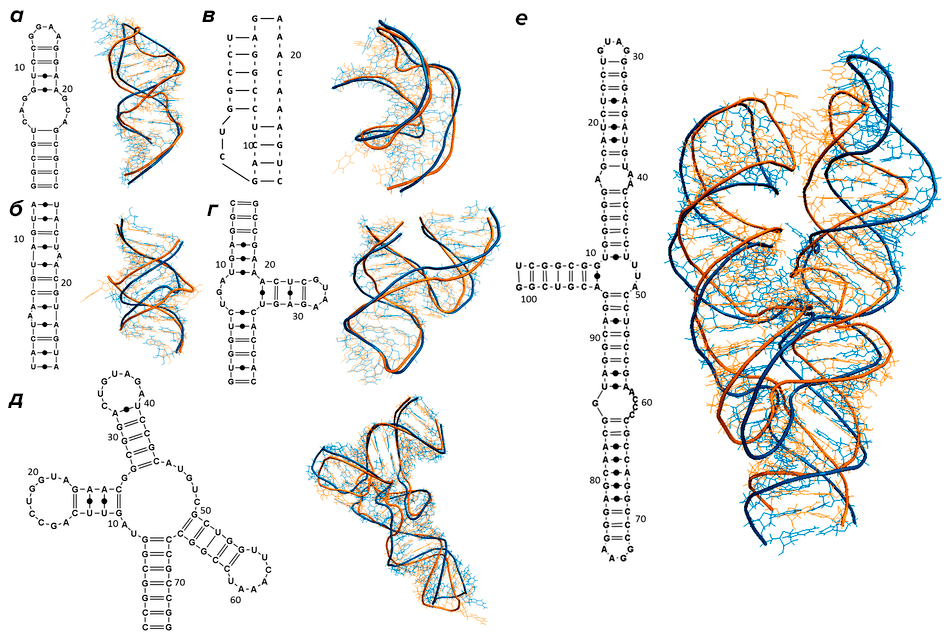
Рисунок 1. Разнообразие вторичных структур РНК: а — шпилька с внутренней петлей; б — дуплекс с двумя выступами; в — псевдоузел; г — молотовидный рибозим (hammerhead ribozyme) PDB: 1NYI; д — тРНК PDB: 1J1U; е — РНК из белок-РНКовго комплекса, узнающего и осуществляющего доставку на мембрану синтезируемых на рибосоме мембранных белков (SRP RNA) PDB: 1Z43. Справа от каждой структуры показана ее 3D-модель: предсказанные с помощью вычислительных методов (синий цвет) и полученные экспериментально (желтый цвет).
Вторичная структура также влияет на взаимодействие РНК с молекулами, например, со специфическими белками RBP (RNA binding proteins). Они связываются с РНК с образованием рибонуклеопротеиновых комплексов и регулируют сплайсинг, трансляцию и другие процессы. Если участок связывания «закрыт» петлей, белок не сможет провзаимодействовать с молекулой [6]. Роль вторичной структуры можно также проиллюстрировать воздействием мутаций. В работе Мэтью Халворсена (Matthew Halvorsen), опубликованной в журнале PLoS Genetics в 2010 году, изучались мутации, связанные с заболеваниями человека и находящиеся в регуляторных участках РНК, называемых 5′- и 3′-нетранслируемыми областями. Эти области отвечают за регуляцию процесса трансляции, деградацию РНК, участвуют в РНК-интерференции и при этом сильно структурированы. Мутации могут вызвать серьезные структурные перестройки, что становится причиной таких заболеваний, как наследственный синдром гиперферритинемии-катаракты, ретинобластома или гипертензия [7].
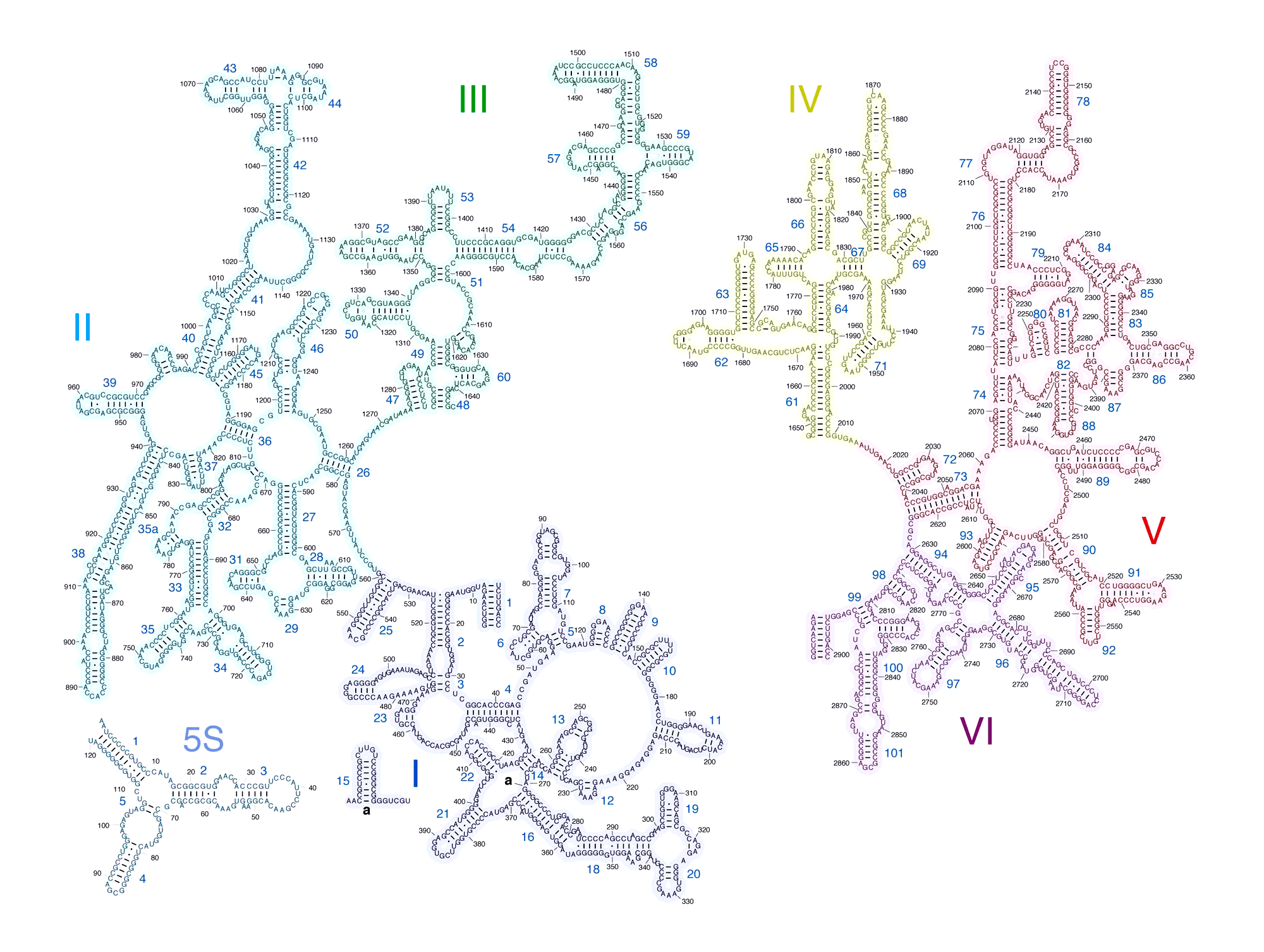
Рисунок 2. Вторичная структура РНК большой рибосомной субъединицы Escherichia coli
При наличии определенных факторов конфигурация РНК может изменяться, что, в свою очередь, влияет на регуляцию генов. Один из лучших примеров специфичности и динамичности вторичной структуры — РНК-переключатели (riboswitch), имеющиеся в клетках бактерий, дрожжей, водорослей и высших растений. Это участки мРНК, которые способны получать информацию о внутриклеточных условиях, связываясь с лигандами. Обычно РНК-переключатели состоят из двух доменов — аптамера, который взаимодействует с лигандами, и домена экспрессии. Как только лиганд (аминокислота, нуклеотиды, ионы металлов) связывается с аптамером, происходят изменения во вторичной структуре примыкающего к аптамеру домена экспрессии, что имеет значение для дальнейшей судьбы мРНК. Например, возникновение шпилек может воспрепятствовать транскрипции или синтезу белка в рибосомах [5]. Структура аптамера, подобно устройству дверного замка, подходит только определенному лиганду, который играет роль ключа
РНК-структуромика
Весь клеточный набор вторичных РНК-структур называется «РНК-струтурóмом» (по аналогии с генóмом или транскриптóмом) [2]. Исследование структурома позволяет ученым понять, как определенные разновидности (мотивы) укладки молекул РНК связаны с участием в различных клеточных процессах, таких как транскрипция, сплайсинг, локализация в клетке, трансляция и регуляция транскриптов. Здесь ученые сталкиваются с определенными проблемами. Недостаточно высокая эффективность методов изучения структуры РНК и сложность получения длинных фрагментов РНК — существенные препятствия на пути к полному описанию структурома. Однако буквально за последние годы технологии секвенирования совершили огромный скачок в развитии. Появились инструменты секвенирования нового поколения, которые позволяют с высокой точностью и относительно быстро определить последовательности ДНК и РНК. Очень важно также использование вычислительных методов, которые сильно продвинулись в своей способности точно предсказывать структуру РНК. При этом все же золотым стандартом определения структуры РНК был и остается эксперимент [5].
Исследования с использованием компьютерных методов привели ко многим открытиям. Например, транспортные РНК обладают структурными особенностями, которые соответствуют их функциям. Для поиска тРНК существуют различные инструменты, например, программа tRNAscan-SE, написанная на популярном среди биоинформатиков языке Perl. Она занимается поиском «подозрительных» участков в геноме, которые могут кодировать тРНК. После тщательного «просеивания» этих участков, остаются гены, которые являются истинными тРНК с вероятностью в 99–100%. Программа ориентируется не только на последовательность нуклеотидов, которая может различаться у разных тРНК, но также на вторичную структуру, являющейся общей для всех тРНК и напоминающей лист клевера. Кроме этого, tRNAscan-SE сравнивает гены-кандидатов с последовательностью, структура которой известна. Если гены-кандидаты формируют такую же структуру, значит они с высокой вероятностью являются тРНК [8].
Естественный отбор имеет значение
Структуры РНК, как и гены, в процессе эволюции могут подвергаться мощному отсекающему отбору, иными словами, большая часть изменений отбраковывается. Ковариационный метод предсказания структуры основан на том простом факте, что множество известных структур РНК консервативны, то есть сохраняются в процессе эволюции, поскольку любое отклонение от этой структуры может повлечь за собой серьезные нарушения и смерть организма. Все тРНК имеют вид листа клевера, поскольку это важно для их участия в процессе синтеза белка. В ковариационном методе производится поиск консервативных пар нуклеотидов среди гомологичных генов (эволюционировавших от общего предкового гена).
Как узнать, что на данной последовательности образуется структура? Для этого нужно, чтобы замена нуклеотида на одном участке последовательности обязательно сопровождалась компенсаторной заменой нуклеотида на другом участке последовательности [9]. Это значит, что оба нуклеотида участвуют в формировании структуры, к примеру, образуя пару G—C или A—U. Структура сохранится, если пара G—C заменится на пару A—U или наоборот. Когда в распоряжении есть достаточное количество гомологов, то, исходя из расположения многих консервативных пар, можно вывести вторичную структуру РНК (рис. 3).
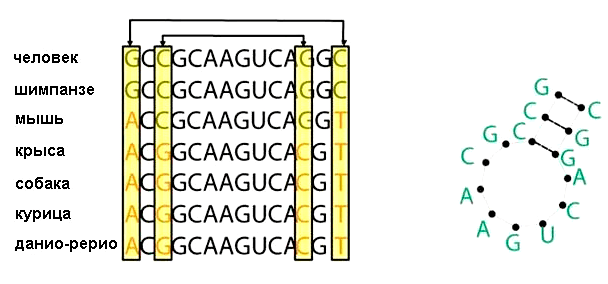
Рисунок 3. Консервативность пар оснований в гомологичных последовательностях является основой для предсказания вторичной структуры. Стрелочками указаны взаимодействующие друг с другом нуклеотиды, которые могут быть различными в ряду последовательностей, однако сохраняющие конфигурацию молекулы (справа).
Вездесущая термодинамика
Когда в распоряжении исследователя имеется не ряд гомологов, а только одна последовательность, имеет смысл воспользоваться термодинамическим моделированием. Предполагается, что молекула РНК правильной структуры находится в состоянии термодинамического равновесия [10]. На основе энергетических параметров данной молекулы, полученных экспериментально, строятся всевозможные конфигурации вторичной структуры. Среди них ищут наиболее устойчивую, которая и будет правильным решением. Для коротких цепочек, длиной менее 700 пар оснований, данный метод корректно определяет структуру для 70% пар. Однако если цепочка длиннее, точность падает вплоть до 20%. Альтернативный подход — использование алгоритмов, основанных на вероятностном моделировании, — к сожалению, сильно уступает термодинамическим методам. Другая возможная стратегия — объединение термодинамического моделирования и машинного обучения [11]. Также с помощью PARS-метода можно выяснить, какие нуклеотиды являются непарными, чтобы исключить их из алгоритма и улучшить точность предсказания.
В методах предсказания вторичной структуры существуют существенные ограничения. Конформационные изменения РНК-переключателей в результате взаимодействия с лигандом настолько сложно смоделировать, что существующие алгоритмы оказываются бесполезными. Также большие трудности вызывает предсказание псевдоузлов, состоящих из двух совмещенных особым образом шпилек (рис. 1). Псевдоузлы встречаются в рРНК, тРНК, а также в геномах вирусных РНК, где они участвуют в процессах трансляции [12]. Для их поиска разрабатывают специальные методы, которые ориентированы на отдельные типы псевдоузлов. Однако с увеличением длины последовательности экспоненциально растет время, затрачиваемое алгоритмом на решение данной задачи. В этих условиях эксперимент становится необходим, позволяя ученым улучшить существующие алгоритмы и разработать новые стратегии поиска.
В игру вступает параллельное секвенирование
В параллельном анализе структуры РНК (или PARS-методе) применяются инструменты секвенирования нового поколения, которые позволяют получить миллионы расшифрованных последовательностей за один единственный эксперимент. Сначала образцам РНК позволяют свернуться с образованием вторичной структуры, затем их подвергают воздействию рибонуклеаз — ферментов, катализирующих расщепление связи между нуклеотидами. В PARS-методе используют два типа рибонуклеаз — V1 и S1. Первая расщепляет спаренные участки, вторая — простую цепь. Полученные фрагменты конвертируют в ДНК и секвенируют. Те фрагменты, которые были обработаны V1, в большинстве случаев обрываются на том нуклеотиде, который участвовал в образовании вторичной структуры. Сравнение большого числа копий одной РНК, позволяет судить об интенсивности расщепления на определенных участках молекулы и определить, формируется ли на этом участке вторичная структура [5].
С помощью PARS в 2010 году Кертес и его коллеги изучили вторичную структуру транскриптома дрожжей, проанализировав приблизительно 4,2 миллиона нуклеотидов в 3 тысячах транскриптов. Ими были обнаружены регуляторные мотивы, например, участок внутренней посадки рибосомы URE2, который позволяет начать синтез белка не с одного из концов мРНК, а с середины молекулы. Это исследование, опубликованное в журнале Nature [13], показало, что PARS-метод способен пролить свет на глобальную структурную организацию мРНК. Было открыто преимущественное расположение вторичных структур в кодирующих участках по сравнению с нетранслируемыми областями, а также их роль в регуляции процесса трансляции, который протекает тем активнее, чем менее структурирован сайт инициации трансляции. Объединение параллельного анализа с программными средствами предсказания конфигурации, а также термодинамическим моделированием, дает в руки ученых мощный инструмент для более глубокого постижения мира РНК-структур, что способствует появлению новых гипотез и открытий, касающихся регуляции клеточных процессов.
Стóит отметить, что данный метод дает весьма приблизительную и косвенную информацию о вторичной структуре РНК. На данный момент разработаны более «продвинутые» экспериментальные методики, основанные на химической модификации неспаренных нуклеотидов и позволяющие получать данные более высокого разрешения (напр. SHAPE). — Ред.
Самая большая РНК-лаборатория в мире
Моделирование вторичной структуры РНК, несмотря на большое количество методов, до сих пор остается сложной задачей. Не все принципы известны, а экспериментальные данные настолько обширны, что на анализ, который проводят небольшие группы ученых, уходит много труда и времени. Чтобы решить эту проблему, в университете Карнеги-Меллон и Стэнфордском университете разработали краудсорсинговый проект EteRNA.
EteRNA объединяет 37 тысяч интернет-пользователей, которые занимаются созданием библиотеки РНК-структур в режиме онлайн-игры . Большинство энтузиастов не обладают никакими специальными биологическими знаниями. Участники проекта, в число которых может попасть любой желающий, подбирают последовательность нуклеотидов РНК, пытаясь получить искомую структуру. Лучшие решения проверяются экспериментально и публикуются на сайте проекта для окончательной проверки. Если результаты будут признаны удовлетворительными, то они становятся основой для новых термодинамических правил построения конфигурации молекул, использующихся в алгоритме EteRNABot. К 2014 году накопилось свыше 100 тысяч решений, и большинство из них ранее не применялись в моделировании вторичной структуры. За все время проекта EteRNABot стал более точным, чем другие алгоритмы предсказания. В ближайшем будущем, как надеются авторы проекта, их «Большая Открытая Лаборатория» (Massive Open Laboratory) будет способна решить широкий спектр практических задач, начиная от моделирования псевдоузлов и разработки синтетических РНК-переключателей и заканчивая моделированием РНК-наномашин.
В последние годы все больше серьезных научных проектов идут «в народ» в форме онлайн-игр. Такое явление получило название «гражданской науки». Примером аналогичной инициативы служит аркадная игра FoldIt, нацеленная на предсказание строения белковых молекул: «Тетрис XXI века» [14]. — Ред.
Прошло около 10 лет с окончания проекта «Геном человека». С тех пор было совершено много открытий. Сравнительно недавно стало известно, что транскрибируется 95% человеческого генома , [15]. Это означает, что большая часть мутаций переходит в транскриптом, влияя на вторичную структуру молекул, которая регулирует многие клеточные процессы. Ранее ускользающий от внимания РНК-структуром стал объектом интенсивного изучения не только отдельных групп биоинформатиков, но и «гражданских ученых» из числа обычных людей. Каждый год публикуется несколько сотен статей, посвященных исследованиям структуры РНК, совершенствуются методы, разрабатываются новые алгоритмы. Мы проникаем все глубже в мир информации, организованной в виде биологических макромолекул, постигаем основные принципы и законы, чтобы когда-нибудь воспользоваться ими во благо всех нас.
Концепция «мусорной ДНК» неоднократно сильно менялась: лет 10 назад бытовало мнение, что ДНК «активна лишь на 10%», а всё остальное является бесполезным кладбищем генов. В последнее время эти воззрения сильно модифицировались, в который раз подтверждая, что не все так просто: «Сколько сора в нашей ДНК» [16]. — Ред.
Литература
- Геном человека: как это было и как это будет;
- «Омики» — эпоха большой биологии;
- Обо всех РНК на свете, больших и малых;
- Торжество компьютерных методов: предсказание строения белков;
- Yue Wan, Michael Kertesz, Robert C. Spitale, Eran Segal, Howard Y. Chang. (2011). Understanding the transcriptome through RNA structure. Nat Rev Genet. 12, 641-655;
- X. Li, G. Quon, H. D. Lipshitz, Q. Morris. (2010). Predicting in vivo binding sites of RNA-binding proteins using mRNA secondary structure. RNA. 16, 1096-1107;
- Matthew Halvorsen, Joshua S. Martin, Sam Broadaway, Alain Laederach. (2010). Disease-Associated Mutations That Alter the RNA Structural Ensemble. PLoS Genet. 6, e1001074;
- Todd M. Lowe, Sean R. Eddy. (1997). tRNAscan-SE: A Program for Improved Detection of Transfer RNA Genes in Genomic Sequence. Nucleic Acids Research. 25, 955-964;
- Sean R. Eddy, Richard Durbin. (1994). RNA sequence analysis using covariance models. Nucl Acids Res. 22, 2079-2088;
- Michael Zuker, Patrick Stiegler. (1981). Optimal computer folding of large RNA sequences using thermodynamics and auxiliary information. Nucl Acids Res. 9, 133-148;
- C. B. Do, D. A. Woods, S. Batzoglou. (2006). CONTRAfold: RNA secondary structure prediction without physics-based models. Bioinformatics. 22, e90-e98;
- F. H. D. van Batenburg. (2001). PseudoBase: structural information on RNA pseudoknots. Nucleic Acids Research. 29, 194-195;
- Michael Kertesz, Yue Wan, Elad Mazor, John L. Rinn, Robert C. Nutter, et. al.. (2010). Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107;
- Тетрис XXI века;
- Newton E. Morton. (2008). Into the Post‐HapMap Era. Genetic Dissection of Complex Traits. 727-742;
- Сколько сора в нашей ДНК.