
Ядро и эпигеном
02 июля 2021
Ядро и эпигеном
- 3060
- 0
- 15
Взаимодействие репрессивных комплексов Polycomb (Polycomb repressive complex, PRC), которые определяют распространение определенного типа гетерохроматина
иллюстрация Михаила Гурьева по структурам PRC1 [119] и PRC2 [120]
-
Автор
-
Редакторы
-
Иллюстратор
В первой статье спецпроекта «Эпигенетика» мы рассказали, как далеко эта наука шагнула по сравнению с генетикой из школьного учебника, а также познакомили вас с основными молекулами, образующими хроматин. Здесь же мы опишем основные хроматиновые домены и формирующие их молекулы, а также силы, которые эти домены формируют. Вы узнаете, что ядро клетки далеко не однородно, как о нем принято думать.
Эпигенетика
Эпигенетика — с одной стороны, молодая и бурно развивающаяся наука. С другой стороны, в этой науке уже сложился фундамент основных понятий и концепций. Цель спецпроекта — рассказать об основных концепциях эпигенетики просто, но в то же время со всех сторон. Читатель узнает, какие молекулы и структуры стоят за передачей наследственной информацией и какими механизмами осуществляется эта передача. Часть контента позаимствована из блога автора Friends of Chromatin.

Генеральный партнер спецпроекта — компания «Диаэм».
Это крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
Хроматиновые домены и их типы
Хроматиновые домены (рис. 1) — это фрагменты хроматина, собранные в одной области ядра и отличающиеся от остального ядра составом молекул и функцией [2]. Такие структуры могут включать в себя участки как одной, так и нескольких хромосом. Например, инактивированная Х-хромосома образует отдельный домен в ядре [3], а гетерохроматин, содержащий белок HP1a, собирается в несколько доменов, близких по строению и включающих в себя хроматин сразу из нескольких хромосом (рис. 2) [4–6]. Можно сказать, что, например, протяженный метилированный участок ДНК или участок хроматина с особо модифицированным гистоном H3 тоже образуют домены. Или участки ДНК, контактирующие с липидно-белковой оболочкой ядра, образуют домены (домены, ассоциированные с ламиной, ЛАДы). И «сложенные» из ДНК глобулы — ТАДы — тоже образуют.
Чтобы удовольствие от прочтения этой статьи было полным, мы рекомендуем сначала ознакомиться с материалами «Молекулы и эпигеном» [7] и «Новый взгляд на геном: не просто цепочка генов, а трехмерная сеть, интегрирующая функциональные домены ядра» [8], где объясняется, например, что такое ТАДы (топологически-ассоциированные домены) и почему они важны для понимания строения ядра и хроматина. — Ред.
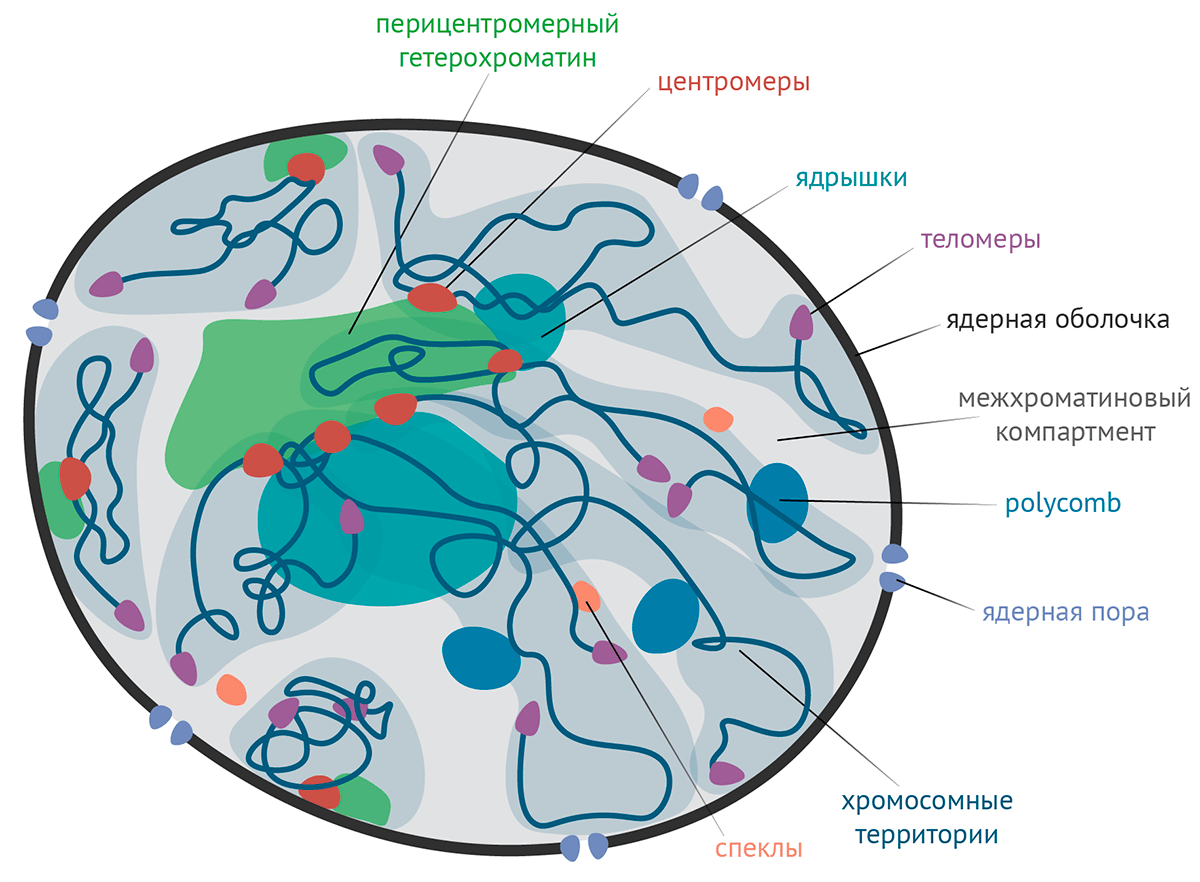
Рисунок 1. Схема клеточного ядра с хроматиновыми доменами. Многие домены (например, функциональные элементы хромосом) можно пометить по специфическим белкам, которые входят преимущественно в них. «По классике», пара митотических хромосом соединена перетяжкой и предстает перед нами в виде буквы «Х». Место перетяжки называется центромерой. Во время деления клетки (в митозе) к центромере крепятся цитоскелетные волокна — микротрубочки, — которые растаскивают сестринские хроматиды к противоположным полюсам делящейся клетки. Таким образом каждая из дочерних клеток наследует одинарный набор хромосом, который уже в ней удвоится во время репликации перед очередным делением.
- Оказывается, центромера как домен со своими специфическими молекулами сохраняется и тогда, когда клетка не делится [9], [10]. Она окружена перицентромерным гетерохроматином, ключевой особенностью которого является уже знакомый нам белок HP1a (рис. 2).
- Концы хромосом тоже образуют домены — теломеры, — которые, как и центромеры, сохраняются на протяжении всего клеточного цикла, независимо от того, делится клетка или нет.
- ДНК, кодирующая рибосомную РНК, собрана в ядре в отдельный домен (пожалуй, самый известный) — ядрышко [11]. Там рРНК транскрибируется и собирается вместе с белками в комплексы — предшественники рибосом [12]. Они потом переносятся из ядра в цитоплазму, где и достраиваются до конца.
- Белки группы Polycomb [13], [14] (считываются с генов Polycomb) (от «много щетинок» — у мух-мутантов по этим белкам много половых щетинок на ногах), которые участвуют в «замалчивании» генов, необходимых для развития организма, также собираются в отдельные тельца в ядре.
- Белки группы PML (promyelotic leukemia, промиелотическая лейкемия) тоже образуют тельца (правда, их точная функция пока не очень понятна) [15].
И так далее.
иллюстрация Михаила Гурьева
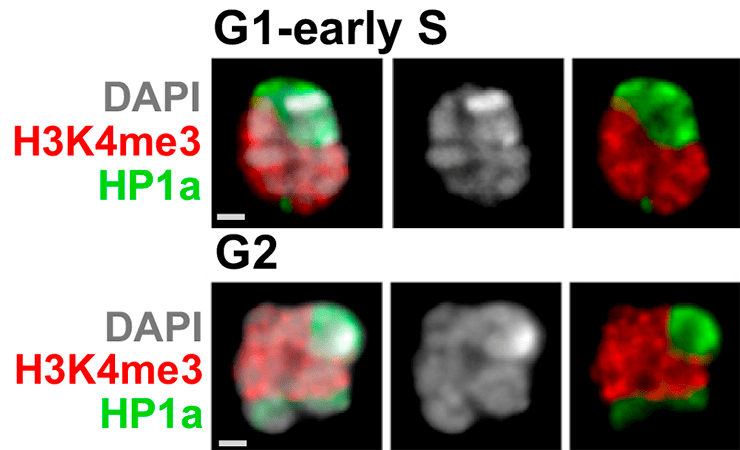
Рисунок 2. Хроматиновые домены в ядре клеток дрозофилы. На фото можно различить гетерохроматин (помечен белком HP1a) и активный хроматин (помечен гистоновой модификацией H3K4me3), а также покрашенную ДНК (DAPI). Верхняя панель — сразу после деления клетки; нижняя — прямо перед ним (разные стадии клеточного цикла). Интересно, что число доменов HP1a в разных фазах клеточного цикла разное.
Получается, что многие белки (а следовательно, и их функции), собраны в ядре в одном месте. Почему так сложилось в ходе эволюции, мы поговорим дальше. Многим доменам в этой статье будет посвящен отдельный параграф.
Как увидеть хроматиновый домен?
В первой статье спецпроекта мы уже обсудили методы ChIP-секвенирования и Dam-метилирования, которые позволяют воссоздать профиль связывания белка с ДНК [7]. Теперь давайте обсудим метод, который позволяет увидеть хроматиновый домен, отмеченный каким-нибудь специфическим белком, под флуоресцентным микроскопом. Метод называют иммунофлуоресценцией (рис. 3) [16].
Иногда у белка в ядре есть две фракции: плотно связанная с хроматином и «свободно плавающая» по ядру (такая ситуация с уже известным нам белком HP1a в ядре клеток дрозофилы). Чтобы четче увидеть хроматиновые домены, при иммунофлуоресценции перед фиксацией клеток добавляют липидный детергент, «дырявящий» мембрану: так неплотно связанные с хроматином молекулы уплывут из клетки через поры в мембране, а связанные останутся. Так, например, можно четко увидеть гетерохроматиновые домены у мушек, помеченные HP1a (рис. 2).
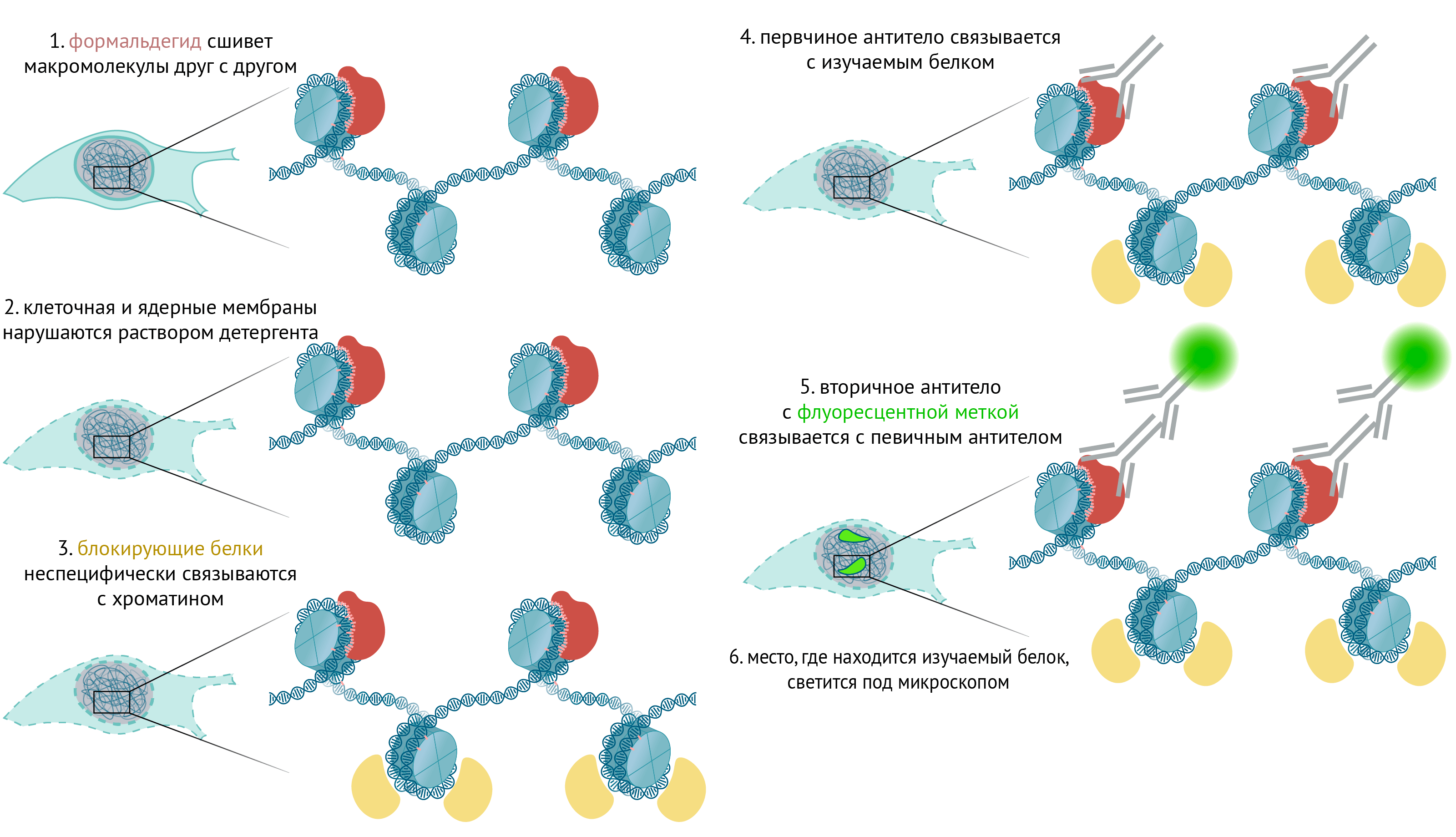
Рисунок 3. Схема иммунофлуоресценции. 1 — Сначала клетки фиксируют формальдегидом, и соседние молекулы ковалентно сшиваются вместе. 2 — Затем мембраны клеток «дырявят» каким-нибудь детергентом, позволяя веществам проходить сквозь нее. 3 — Затем, чтобы уменьшить неспецифическое связывание антител с белками, добавляют специальный блокирующий раствор, молекулы которого связываются со всеми белками и уменьшают подобные взаимодействия антител. 4 — Затем клетки инкубируют с антителом против интересующего нас белка, а затем (5) со вторым антителом, которое связывается с первым и несет флуоресцентную метку. Вуаля (6): наш хроматиновый домен можно увидеть под флуоресцентным микроскопом!
иллюстрация Михаила Гурьева
Как светятся флуоресцентные метки? Они могут поглощать свет определенной длины волны и переизлучать более длинноволновый свет (это и называется флуоресценцией). Интересно, что некоторые белки тоже обладают свойствами флуорофоров: самые известные из них — это зеленый, красный и другие флуоресцентные белки (GFP, RFP и т.д.), выделенные в свое время из медуз и кораллов и усовершенствованные биотехнологически [17–22]. Ген GFP (или RFP) часто привязывают к гену изучаемого белка: таким образом место расположения изучаемого белка будет светиться под флуоресцентным микроскопом зеленым или красным светом.
Иерархичность и динамичность хроматиновых доменов
Хроматиновые домены устроены иерархически. Например, ТАДы побольше могут содержать в себе субТАДы [23]. Одна из моделей укладки ДНК в ядре — фрактальная глобула [24]: маленькие глобулы (скомканные участки ДНК) складываются в глобулы побольше, а те, в свою очередь, — в глобулы еще большего размера; и так далее, пока не будет достигнут уровень целой хромосомы. Еще один пример. Помните, в предыдущей статье [7] мы обсуждали «цвета» хроматина — домены, выделенные по специфическим комбинациям профилей белков на ДНК? Сначала таких цветов выделили всего пять (по профилям связывания 53-х белков) [25], а потом это число было доведено до девяти (там в дело пустили разные модификации гистонов) [26]. Что интересно, большинство этих цветов оказались подтипами пяти предыдущих [2], [27], что дополнительно подчеркивает иерархичность.
Хроматиновые домены динамичны. Например, гетерохроматин плодовой мушки дрозофилы формирует от одного до трех доменов в неделящемся ядре в зависимости от того, произошла в клетке репликация или нет (рис.3) [5]. Некоторые домены, связанные с ядерной ламиной, перемещаются и после деления оказываются уже возле ядрышка [28]. ТАДы исчезают в процессе подготовки клетки к делению (когда хромосомы становятся очень компактными), и появляются вновь уже после окончания деления [29–31].
Центромеры
Давайте рассмотрим, как определенные молекулярные события участвуют в формировании и поддержании клеточного домена на примере центромеры. Как уже упоминалось, центромера — участок, за который хромосома притягивается к одному из полюсов клетки во время деления. Это место сохраняется на каждой хромосоме даже когда клетка не делится, а хромосома развернута [9], [10].
Клетке, чтобы «запомнить», в каком месте хромосомы находится центромера, нужно, во-первых, ее «пометить», а во-вторых — чтобы эта «метка» сохранялась на протяжении клеточных делений. Перед каждым клеточным делением двуцепочечная ДНК расплетается, и к каждой одиночной цепочке достраивается парная. Однако ДНК-то удваивается, а белки с исходной хромосомы распределяются по дочерним хромосомам, в результате чего их количество на каждой хромосоме оказывается в два раза меньше исходного (за исключением канонических гистонов, которые активно синтезируются прямо во время репликации). И нужно, чтобы вновь синтезированные «центромерные» белки могли «найти» это место, переместиться туда и восполнить этот недостаток.
Какие же молекулы определяют центромеру? У разных организмов центромеры устроены немного по-разному, однако у большинства, во-первых, они строятся на повторяющихся последовательностях ДНК, а во-вторых — у них есть конкретная «метка», определяющая этот хроматиновый домен. Это центромерный вариант гистона H3, называемый CenpA (centromeric protein A) и входящий в центромерные нуклеосомы. Структура таких нуклеосом отличается от обычных, а следовательно, и центромерный хроматин отличается от остального [32]. Если убрать из клетки CenpA, функциональной центромеры не образуется, и клетка нормально делиться не может [33], [34].
Есть и другие белки, которые находятся на центромерах. У человека, включая CenpA, их 16 типов, и они связаны с центромерой на протяжении всего клеточного цикла. Открыли их, сделав иммунопреципитацию CenpA и определив взаимодействующие с ним белки масс-спектрометрией [35–37]. Среди этих 16 белков есть те, которые напрямую взаимодействуют с ДНК, а есть и образующие «надстройку» над первыми (то есть они связаны уже не с ДНК, а с взаимодействующими с ней белками). Ко всему этому присоединяется другая белковая структура, соединяющая центромеру с микротрубочками — внешний кинетохор. Но он, в отличие от центромеры, образуется только при делении клетки.
Иногда CenpA может встраиваться и в те места хромосом, где его обычно нет, что приводит к возникновению неоцентромеры. Поскольку на одной хромосоме при этом оказывается больше одной центромеры, она может просто порваться при делении клетки из-за тянущих ее в разные стороны микротрубочек [39].
Интересно, что, например, у плодовой мушки дрозофилы минимально функционирующая центромера устроена намного проще, чем у человека. И центромеру плодовой мушки можно реконструировать в человеческих клетках, поместив туда всего три белка: CenpA, шаперон CenpA и CenpC (centromeric protein C) [40]. Только CenpA и CenpC у мушки имеют гомологи у человека. Остальные 14 из 16 центромерных белков человека у мушки отсутствуют [41].
Близкий к центромерам гетерохроматин
Перицентромерный гетерохроматин, расположенный вблизи от центромер и полный повторяющихся последовательностей, обозначили у дрозофилы как «зеленый» по профилям разных белков на ДНК [25]. Это, пожалуй, самый неактивный тип хроматина: большинство находящихся там генов «замалчивается». Самый изученный белок такого гетерохроматина — HP1a (heterochromatic protein 1a). Он есть и у человека, и у дрожжей, и у плодовой мушки дрозофилы. Как уже упоминалось, если покрасить клетку на этот белок и взглянуть на нее во флуоресцентный микроскоп, можно увидеть несколько доменов. Они, во-первых, (частично) совпадают с участками повышенной плотности ДНК, поскольку такой вид хроматина самый компактный. Во-вторых, они располагаются рядом с центромерами — и когда клетка делится, и когда нет [5], [42–44].
Надо сказать, что гетерохроматин неоднороден. В одном исследовании с помощью иммунопреципитации и масс-спектрометрии выделили белки, взаимодействующие с HP1a плодовой мушки дрозофилы. Потом покрасили клетки на эти белки и проверили, совпадают ли покрашенные области для этих белков и для HP1a? Оказалось, что совпадают лишь частично: во многих случаях зона этих белков находится внутри «территории» HP1а, но эта картина меняется в зависимости от стадии клеточного цикла: собирается ли клетка удваивать свою ДНК или делиться. Например, участок белка HP2 (heterochromatic protein 2) совпадает с участком HP1a и даже немножко выходит за его границы перед удвоением ДНК, а во время удвоения образует небольшие пятнышки внутри и вне участка HP1a [44].
Но есть и молекулярные особенности, очень тесно связанные с HP1a. Это, в первую очередь, двойное и тройное метилирование гистона H3 по девятому лизину (H3K9me2/3), которые распознает HP1a и таким образом заякоривается на нуклеосоме. Две молекулы HP1a на соседних нуклеосомах могут также взаимодействовать друг с другом, сближая нуклеосомы между собой и делая хроматин компактнее (рис. 4) [42], [45]. Навешиванием на гистоны H3 двойной и тройной метильных меток занимается специальный белок-«писатель» Su(var)3-9 (происхождение этого названия объясняется на рис. 5). У плодовой мушки дрозофилы белки HP1a и Su(var)3-9 не могут найти гетерохроматин при отсутствии друг друга [46].
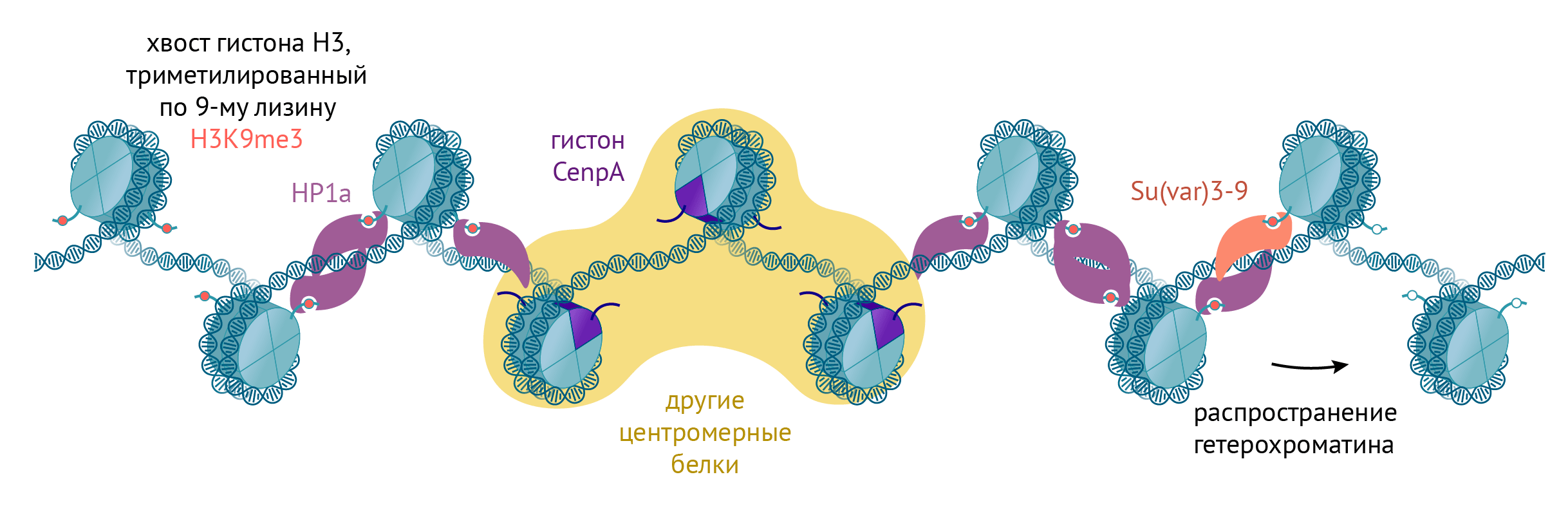
Рисунок 4. Центромера и перицентромерный хроматин. Центромера — место расположения CenpA-нуклеосом и других центромерных белков.
иллюстрация Михаила Гурьева
Интересно, что у Su(var)3-9 есть часть, похожая на часть HP1a и способная распознавать H3K9me2/3. Таким образом, Su(var)3-9 может заякориваться на нуклеосомах с H3K9me2/3 (самостоятельно или через HP1a) и модифицировать близко расположенные нуклеосомы. Это основной механизм распространения гетерохроматина.
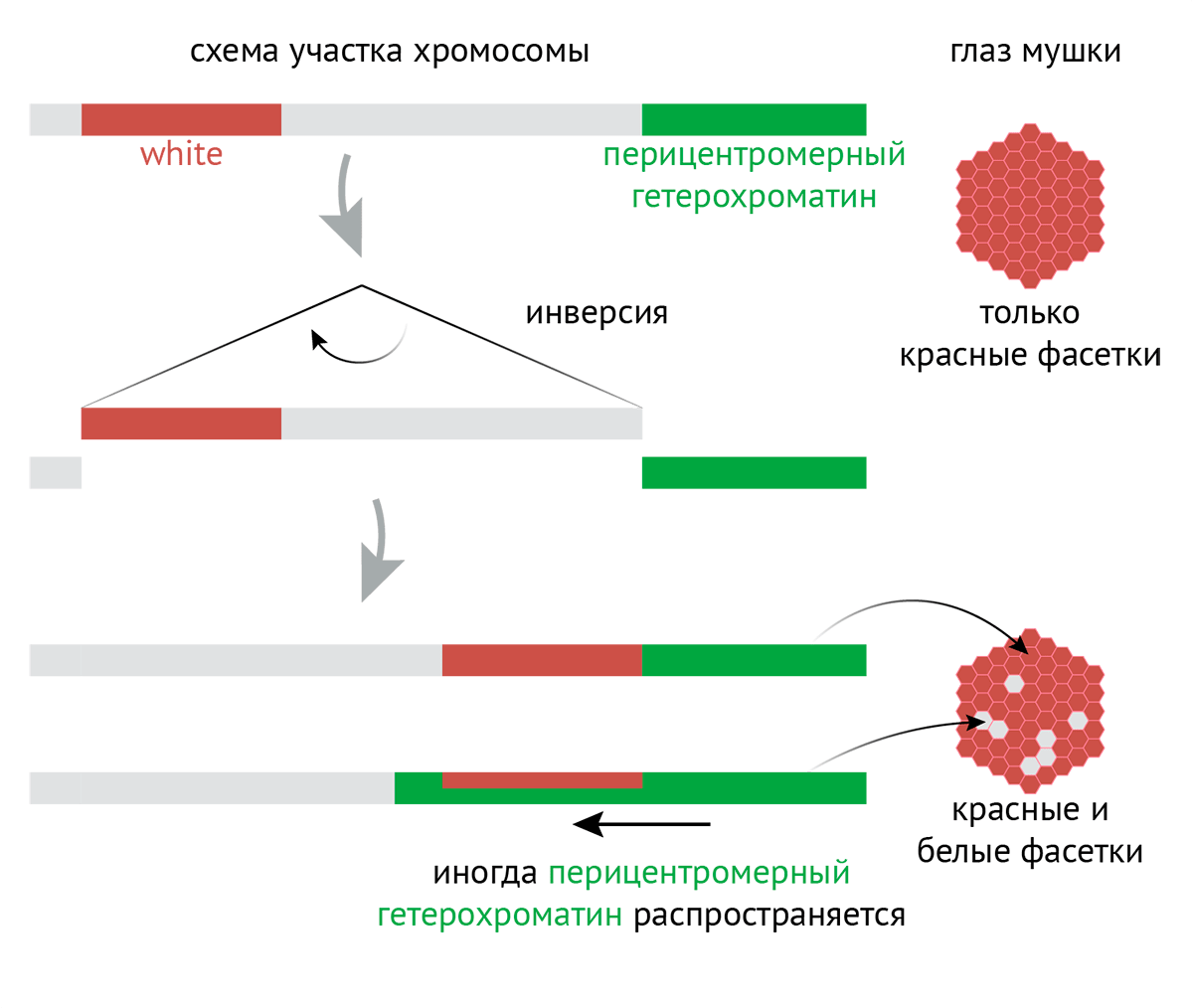
Рисунок 5. Мозаичная окраска, зависящая от позиции гена. Почему Su(var)3-9 назвали именно так? Еще на границе 1920–1930-x годов немецкий ученый Герман Мюллер обнаружил, что если облучить плодовую мушку рентгеновским излучением, то у ее потомков будут глаза довольно необычного цвета. В норме они красные, а у некоторых мутантов оказались красно-белые: некоторые фасетки красного, а некоторые белого цвета. За красный цвет глаз у плодовой мушки отвечает ген white (белый), кодирующий белок, делающий глаза красными. При его мутации глаза теряют окраску и становятся белыми. Так вот, позже выяснилось, что в некоторых фасетках на хромосоме, содержащей ген white, произошла так называемая инверсия: кусочек хромосомы вырезался, перевернулся на 180° и вставился обратно. Причем в итоге ген white оказался рядом с перицентромерным гетерохроматином, который распространился на ген и привел к тому, что тот перестал «читаться». Это и привело к белым фасеткам. Эффект получил название «разнообразная окраска, зависящая от позиции [гена]» (position-effect variegation, PEV). Гены, мутация которых подавляет распространение перицентромерного гетерохроматина и ведет к красным глазам, получили название «подавляет разнообразную окраску» (suppressor of variegation, Su(var)), а гены, мутация которых усиливает вышеописанные эффекты и ведет к белым глазам, — «усиливает разнообразную окраску» (enhancer of variegation, E(var)) [47], [48].
иллюстрация Михаила Гурьева
Пожалуй, стоит упомянуть еще один механизм формирования гетерохроматина. Без него гетерохроматин не может образовываться у обыкновенных делящихся дрожжей (рис. 6).
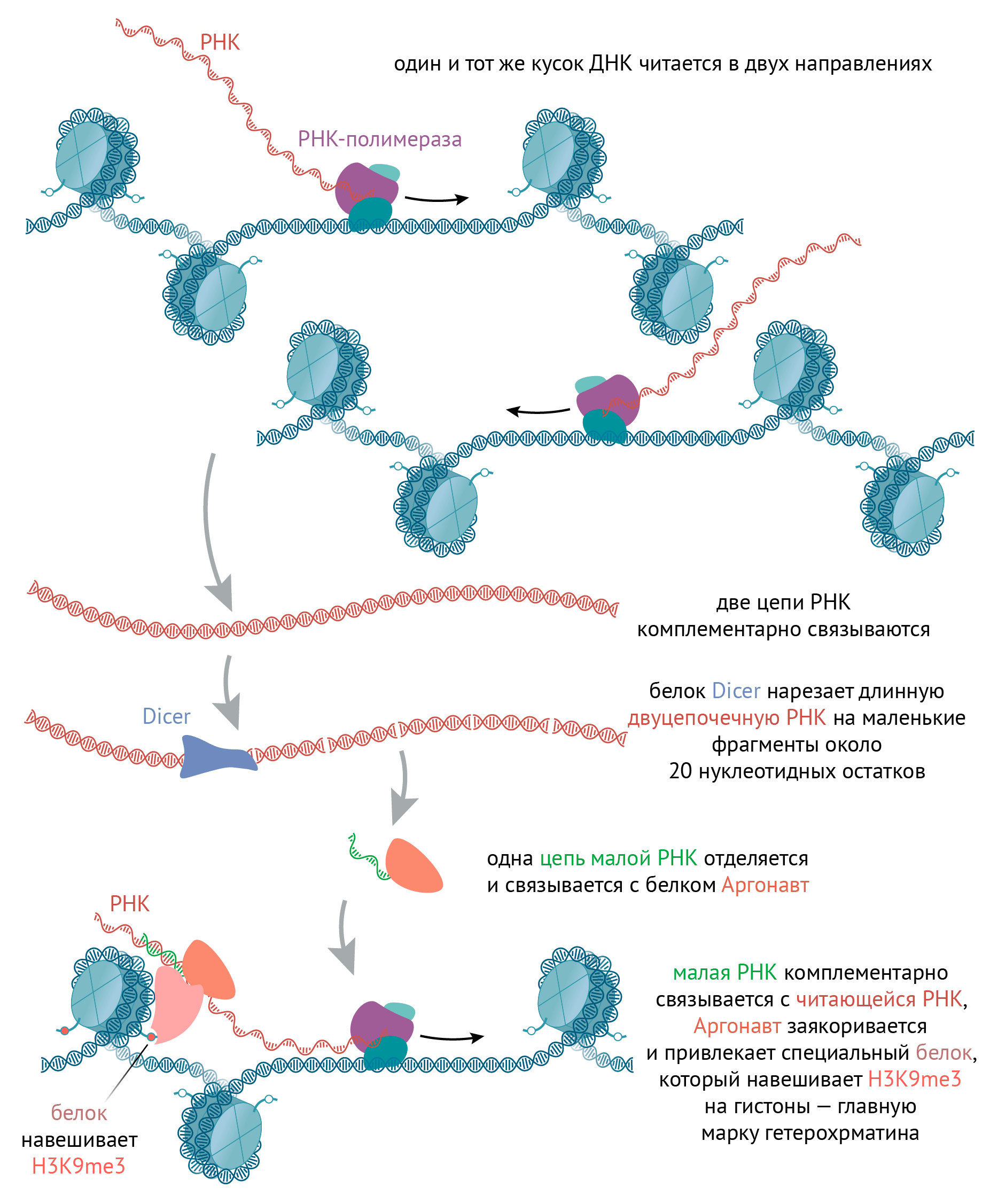
Рисунок 6. Образование гетерохроматина у дрожжей. Как уже упоминалось, гены и повторяющиеся последовательности внутри гетерохроматина преимущественно замалчиваются. Но иногда такие повторяющиеся последовательности могут читаться в обоих направлениях на очень низком уровне: назад и вперед. Образующиеся двухцепочечные участки РНК нарезаются специальным белком на маленькие фрагменты около 20 нуклеотидных остатков. Они «передаются» в другой белковый комплекс RISC (RNA-induced silencing complex, замалчивающий комплекс, индуцированный РНК), одна цепочка такой маленькой РНК отделяется, а через вторую вышеупомянутый белковый комплекс комплементарно находит РНК, которая читается с гетерохроматина и всё еще находится близко к этому гетерохроматину в процессе транскрипции. Этот комплекс взаимодействует с белками, которые навешивают на гистон H3K9me2/3, и заякоривает их на гетерохроматине. Кроме того, он взаимодействует с РНК-зависимой РНК полимеразой, которая комплементарно считывает молекулы РНК с малых молекул РНК. Таким образом, двуцепочечных РНК становится больше, и механизм усиливает сам себя.
иллюстрация Михаила Гурьева по [121]
Теломеры
Теломеры — это концы хромосом. Они тоже состоят из повторяющейся ДНК (правда, эти повторы иные, чем на центромерах и перицентромерном гетерохроматине), белкового комплекса, защищающего концы хромосом (шелтерина), и комплекса из белка теломеразы и РНК, который может достраивать хромосомные концы после удвоения ДНК [49]. Как и центромеры, теломеры часто кластеризуются вместе. Но обо всем по порядку.
Давайте сначала разберемся — а зачем защищать концы хромосом? Дело в том, что в клетке периодически происходят двухцепочечные разрывы ДНК, и чтобы их чинить, в ядре есть специальные белки, узнающие такие разрывы и сшивающие их. И если клетка примет концы своих хромосом за двухцепочечные разрывы, то она сошьет концы соседних хромосом, и потом у нее будут проблемы с расхождением этих хромосом при делении клетки. Следовательно, нужно сделать так, чтобы белки, чинящие двухцепочечные разрывы ДНК в ядре, не начали «чинить» и хромосомные концы.
А почему концы хромосом нужно достраивать после репликации? Для ответа на этот вопрос давайте в общих чертах рассмотрим, как происходит удвоение ДНК. Как уже упоминалось, ДНК находится в двухцепочечной форме. Концы каждой цепочки неодинаковы: один носит название 3′, а другой 5′. Белок, удваивающий ДНК (ДНК-полимераза), нуждается в 3′-конце, чтобы продолжить его, и может синтезировать ДНК только в направлении 5′→3′ . Специальный комплекс белков движется по двухцепочечной ДНК, расплетая ее и достраивая каждую из цепочек. Но как начать синтезировать цепь 5′→3′? И что делать с цепью 3′→5′? Чтобы создать ДНК-полимеразе условия для продолжения синтеза, специальный белок строит короткие праймеры — последовательности из РНК на одноцепочечной ДНК. Для цепи 5′→3′ такая затравка нужна лишь одна и в самом начале, а для другой цепи таких затравок много: они появляются регулярно недалеко от удваивающего ДНК комплекса белков, как бы опережая место, где удваивается цепь 5′→3′, и достраиваясь на цепи 3′→5′, но обратно — в направлении 5′→3′. РНК этих праймеров потом разрушается, а на место, где была РНК, ставит ДНК-«заплатки» специальный белок [50].
И тут-то и возникает проблема с удвоением ДНК на конце хромосом. Чтобы удвоить конец цепи 3′→5′, нужно синтезировать затравку РНК на самом конце. Но дело в том, что когда эта затравка разрушается, на ее место невозможно поставить «заплатку» ДНК, потому что она находится на самом конце хромосомы (и белок, строящий ДНК, не может сесть на пустое место рядом с концом), и это место остается пустым. Вдобавок к этому, цепь 5′→3′ сначала удваивается до конца, а потом одна из дочерних цепей укорачивается специальным белком. Получается, во-первых, что на конце хромосом 3′-конец длиннее 5′-конца, и часть цепи ДНК существует в одноцепочечной форме (рис. 7). Во-вторых, выходит, что при каждом раунде удвоения концы хромосомы чуть-чуть укорачиваются. Сделать так, чтобы они не укорачивались, может специальный комплекс из белка теломеразы и специальной РНК. Теломераза достраивает цепь ДНК комплементарно к цепи этой РНК как раз на концах хромосом [51].
Интересно, что в нормальных клетках ген теломеразы не экспрессируется (и ее самой в них нет). И клетки могут удваивать свою ДНК и делиться только ограниченное число раз (это называют пределом Хейфлика). Но если заставить клетку производить теломеразу, то этот предел снимается, и клетка может делиться гораздо большее, в теории бесконечное число раз [52]. Звучит заманчиво, но такая способность характерна в том числе и для раковых клеток...
Как теломерные белки узнают, в каком месте хромосомы находятся теломеры? Всё дело в специальных последовательностях ДНК, из которых и состоят теломеры. У позвоночных это 3′-TTAGGG-5′. Она много раз повторяется на концах хромосом и изредка на внутренних участках. Преимущественно именно с ней связывается специальный белок — TRF1 (TTAGGG DNA repeat proteins 1, белок повтора ДНК TTAGGG 1) [53]. Он регулирует привлечение на теломеры большинства компонентов комплекса шелтерина [54–57]. Теломераза же оказывается на теломерах как раз благодаря взаимодействию с одним из компонентов шелтерина [58].
Интересно, что на концах хромосом все вышеописанные теломерные белки сочетаются с гетерохроматином, похожим на перицентромерный, с H3K9-метилированием и белком HP1a. Правда, этот хроматин отличается еще одной метильной модификацией: H3K79me2 [59].
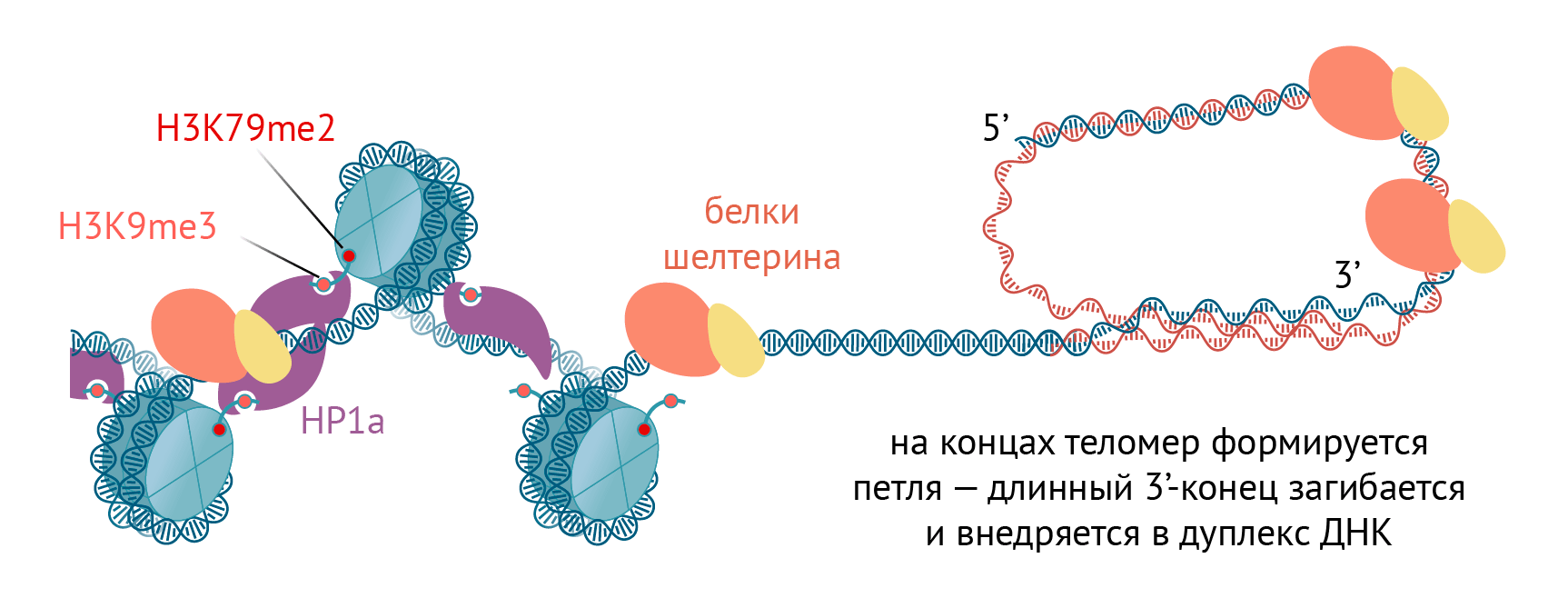
Рисунок 7. Схема теломеры. Теломера состоит из белков шелтерина, защищающих концы хромосом, теломеразы (не показана) и гетерохроматина с H3K9me3 и H3K79me2. На конце хромосомы удлиненный 3′-конец загибается, внедряется в дуплекс ДНК и образует петлю.
иллюстрация Михаила Гурьева
Ядрышко
Пожалуй, самая изученная функция ядрышка — сборка предшественников рибосом: больших комплексов из РНК и белка, занимающихся производством белка в цитоплазме. Для этого нужно, во-первых, считать рибосомную РНК с ДНК, правильным образом обработать ее и модифицировать. Во-вторых, нужно синтезировать в цитоплазме белки, входящие в состав рибосом, и переместить их в ядрышко. Именно по первой причине ядрышко организовано вокруг последовательностей ДНК, кодирующих рРНК.
рРНК считывается с рибосомной ДНК в виде одной длинной молекулы белком РНК-полимеразой I (в отличие от большинства генов, транскрипция которых осуществляется РНК-полимеразой II), затем разрезается на три молекулы, а вслед происходит модификация этой РНК с участием особых коротких РНК, одной своей частью комплементарных рРНК, а другой — взаимодействующих с белками-модификаторами рРНК [12], [62].
Интересно, что ядрышко неоднородно по структуре: там есть несколько компартментов, различимых на электронно-микроскопических фотографиях — в центре ядрышка находится фибриллярный центр. Он окружен плотным фибриллярным компонентом, а тот в свою очередь — гранулярным компонентом. Этапы формирования прерибосомных частиц разнесены по этим компартментам.
Во время деления клетки ядрышко разбирается, а после — собирается обратно. Интересно, что многие белки ядрышка распределяются по дочерним клеткам с помощью хромосом: во время деления клетки плотно упакованная ДНК митотических хромосом покрыта РНК и белками, в том числе и ядрышковыми. Получается, что многие ядрышковые белки используют митотические хромосомы в качестве транспортного средства для равномерного распределения по дочерним клеткам. В дополнение к этому, когда клетка собирается делиться, ядерная оболочка разбирается, а собирается уже после деления клетки вокруг разворачивающихся хромосом. Если бы вышеупомянутые ядрышковые белки оказались в цитоплазме, им бы пришлось перемещаться в ядро через ядерную оболочку с ядерными порами, а так они уже оказываются внутри формирующегося ядра [63].
Как устроен хроматин ядрышка? Его там целых три типа. Первый — неактивный. Второй — активный. И еще есть смешанный третий тип хроматина, содержащий как неактивную polycomb-метку H3K27me3, так и активную метку H3K4me3. С активного хроматина транскрибируются предшественники рРНК. С неактивного хроматина ничего не читается: все гены там «замалчиваются». Смешанный тип хроматина тоже не читается; однако похоже, что его можно быстро переделать в активный хроматин и прочитать, если понадобится. Доля этого хроматина повышена в клетках, рост которых приостановлен, но они готовятся к быстрому прочтению рибосомных генов, чтобы сделать много рибосом, синтезировать много белка и возобновить рост [64–66].
Polycomb, или «Синий» хроматин
Как уже упоминалось, хроматин polycomb («много щетинок») назвали так потому, что у мух, мутантных по некоторым белкам Polycomb, много половых щетинок на ногах (рис. 8). Пожалуй, это одна из самых сложных тем в эпигенетике из-за большого количества ключевых белков и не до конца понятной их взаимной регуляции.
Рисунок 8. Половые щетинки на ногах мух
Этот тип хроматина «отмечен» специальными гистоновыми метками: триметилированием 27-го лизина гистона H3 (H3K27me3) и убиквитинилированием (когда к гистону пришивается белок убиквитин) гистона H2A по 119 лизину (H2AK119ub). Также на этом хроматине присутствуют два главных комплекса Polycomb, которые связываются с этими метками: PRC1 и PRC2 (Polycomb repressive complexes 1 и 2, репрессивные комплексы Polycomb 1 и 2) [67]. Как и гетерохроматин, помеченный белком HP1a, polycomb-хроматин неоднороден. Например, профили ChIP-секвенирования PRC1 и PRC2 совпадают не до конца [68]; есть и другие особенности.
PRC1 и PRC2 «притягиваются» на хроматин в том числе специальными последовательностями ДНК: у млекопитающих для PRC2 это участки, богатые чередующимися нуклеотидными остатками C и G, неметилированные. У мушки дрозофилы аналогичные, но отличные по последовательности фрагменты ДНК называются PRE (Polycomb-response elements, polycomb-отвечающие элементы) [69], [70].
Как распространяются гистоновые модификации, характерные для связывания комплекса Polycomb? Для ответа на этот вопрос давайте сначала рассмотрим, как эти комплексы устроены. PRC1 состоит из белка, навешивающего убиквитиновые метки на H2AK119; белка, одним из своих доменов распознающего H3K27me3; и еще нескольких белков. PRC2, в свою очередь, может одним из белков комплекса навешивать метильные группы на H3K27, а другим — узнавать эту модификацию. Есть в PRC2 и другие белки [67], [70].
PRC2 навешивает на гистон H3 метку H3K27me, сам же с ней связывается и модифицирует следующую нуклеосому [70–72]. Механизм напоминает распространение H3K9me2/3 по HP1a-хроматину. У млекопитающих комплекс PRC2 через H3K27me3 способен заякоривать на хроматине PRC1 [69]. Кроме того, по одной из моделей, комплекс PRC1 может привлекать комплекс PRC2 через белки-адаптеры, которые связываются как с H2A119ub, так и с PRC2 [70], [73], [74]. Правда, взаимное привлечение PRC1 и PRC2 пока не до конца изучено и доказано (рис. 9).
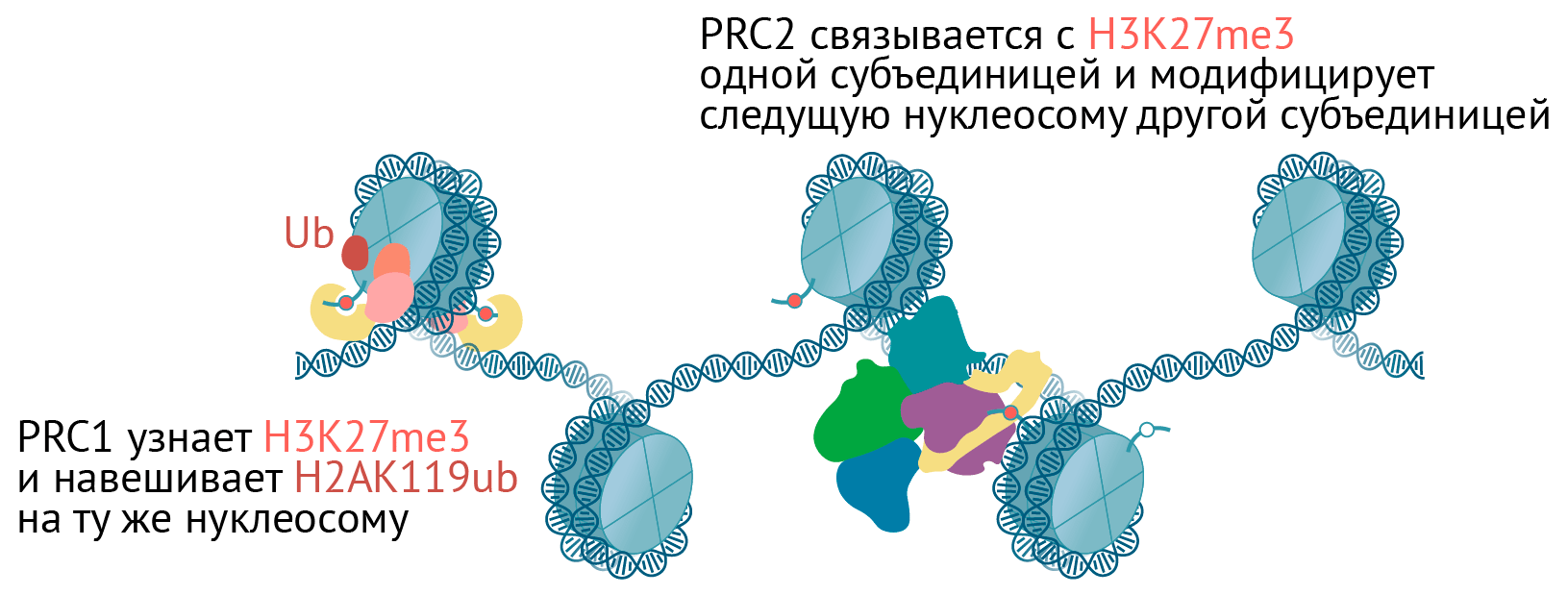
Рисунок 9. Схема комплексов Polycomb. Комплекс PRC2 навешивает H3K27me3, комплекс PRC1 узнает эту метку и навешивает H2AK119Ub.
иллюстрация Михаила Гурьева по структурам PRC1 [119] и PRC2 [120]
Хроматин, который создают polycomb-белки, неактивен: гены в нем не транскрибируются. Эти белки через модификации H3K27me3 «замалчивают» определенные гены в ходе развития организма, что необходимо для нормального развития. Но почему в ходе эволюции регуляция polycomb-белков так усложнилась? Для развития эмбриона белки должны выключить определенные гены быстро и точно. Многие из polycomb-белков регулируют активность белка из комплекса PRC2, который навешивает метильные метки на H3K27. Возможно, регулируя эту активность многими способами, можно очень точно настроить степень распространения H3K27me3. Кроме того, вероятно, через сложные регуляторные сети можно очень точно определять места образования H3K27me3.
В ядре polycomb-белки собраны в специальные кластеры, которые получили название polycomb-телец [14]. При помощи обычной флуоресцентной микроскопии в ядре можно заметить лишь несколько таких телец. Однако микроскопия высокого разрешения выявляет присутствие большого количества маленьких телец, часто и образующих большие polycomb-тельца [14], [75], [76].
Как и многие другие домены, polycomb-тельца динамичны. Например, в недифференцированных клетках (которые еще могут стать клетками многих типов) количество телец меньше, и они больше в размерах, чем в дифференцированных. Также, если заставить эмбрион плодовой мушки дрозофилы производить слитые polycomb- и GFP-белки, то можно увидеть, что polycomb-тельца по мере развития эмбриона становятся всё многочисленнее и ярче.
Интересно, что polycomb-белки регулируют не только гены развития организма, но и гены Х-хромосомы у млекопитающих. Но об этом позже.
Активный хроматин (эухроматин)
Он отмечен специальными гистоновыми метками. Их много, поэтому давайте обсудим только основные. В отличие от неактивного, в активном хроматине гены транскрибируются. Чтобы на матрице ДНК начала комплементарно синтезироваться РНК, фермент РНК-полимераза должен связаться с промотором — областью ДНК прямо перед геном. Гистон H3 на промоторе модифицирован специальным образом — триметилирован по четвертому лизину (H3K4me3). На самом же гене гистон H3 триметилирован по 36 лизину (H3K36me3) (рис. 10). Наконец, еще две важные модификации: как мы уже обсуждали в предыдущей статье [7], есть последовательности ДНК, способные усиливать или ослаблять (зависит от последовательности) транскрипцию гена, находясь от него на значительном удалении в последовательности, но взаимодействуя с ним в пространстве. Они называются энхансерами [77] и сайленсерами соответственно. Так вот, гистон H3 на энхансерах монометилирован по четвертому лизину (H3K4me1) и ацетилирован по 27 лизину (H3K27ac). Есть также пара гистоновых меток, которые присутствуют в конце гена, возле терминатора — области, нужной для того, чтобы РНК-полимераза отсоединилась, и транскрипция закончилась. Но эти метки присутствуют в конце гена не так четко и последовательно, как упомянутые метки на других частях гена [78].
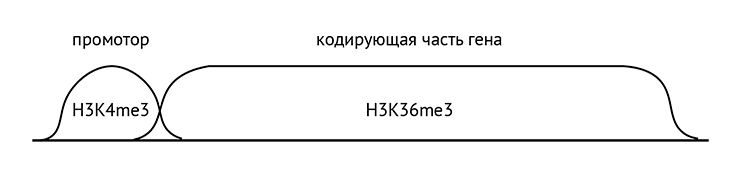
Рисунок 10. Распределение основных гистоновых модификаций по гену
иллюстрация Михаила Гурьева
Какие белки присутствуют на активном хроматине, кроме гистонов и модифицирующих их белков? Например, это транскрипционные факторы, которые помогают РНК-полимеразе связаться с промотором и начать транскрипцию гена. Это и ремоделеры хроматина. Они, во-первых, устанавливают нуклеосомы вокруг места начала транскрипции строго определенным образом, и это важно для транскрипции РНК с ДНК. Во-вторых, когда РНК-полимераза комплементарно синтезирует РНК с ДНК, нужно частично разобрать нуклеосомы, которые попадаются РНК-полимеразе на пути, а потом собрать их обратно [79]. Этим тоже занимаются ремоделеры.
Нужно понимать, что РНК-полимераза — это не один-единственный белок, а целое нагромождение из многих субъединиц. У комплекса РНК-полимеразы есть и много вспомогательных белков, которые поэтапно формируют комплексы перед началом, во время и в конце транскрипции.
После этого с полученной в результате транскрипции РНК происходят три важные вещи:
- Кэпирование РНК — на 5‘-конец молекулы навешивается метилированный нуклеотид гуанозин.
- Сплайсинг РНК: из нее вырезаются участки, называемые интронами, а участки, называемые экзонами, сшиваются между собой.
- На РНК может навешиваться поли(А)-хвост из многих адениновых нуклеотидных остатков — этот процесс называется полиаденилированием [80].
Зачем РНК кэп и поли(А)-хвост? Дело в том, что они защищают молекулу от деградации — разборки на нуклеотиды. Белки, начинающие разборку РНК с какого-то из ее концов, не могут работать, пока на концах РНК есть кэп и поли(А)-хвост.
Спеклы и параспеклы
Спеклы — это ядерные домены, играющие роль центров сплайсинга. Поскольку сплайсинг тесно связан с другими процессами в ядре (например, с модификацией 3′-конца матричной РНК и с переносом готовой матричной РНК из ядра в цитоплазму), то белки, собранные в спеклах, непосредственно влияют и на эти процессы [81–83]. Спеклы образуются в местах скопления эухроматина, и интенсивно транскрибируемые гены часто находятся на границе спекл.
Параспеклы похожи на спеклы в том смысле, что в них тоже накапливаются РНК-связывающие белки. Вот только формируются они совершенно особенным путем: вокруг длинной-предлинной (около 23 000 нуклеотидных остатков) молекулы некодирующей РНК [84]. Она называется NEAT1 (nuclear paraspeckle assembly transcript, ядерный транскрипт сборки параспекл). Если повысить уровень этой РНК в клетках, параспеклы становятся больше в размерах [84], [85]. К NEAT1 присоединяется несколько РНК-связывающих белков, а к ним в свою очередь — другие белки.
Под микроскопом высокого разрешения домены параспекл, как и крупные тельца Polycomb, выглядят как собрание доменов поменьше. Вероятно, то же самое выяснится для некоторых других компартментов в ядре. Разные части NEAT1 находятся в разных частях параспекл: в середине РНК находится в центре, а 3′- и 5′-концы располагаются по краям. Следовательно, и белки, которые связываются либо с серединой, либо с концами РНК, находятся либо в центре, либо на краях параспекл, соответственно [84], [86].
Какова функция параспекл? Например, они могут задерживать молекулы РНК и РНК-связывающих белков. Еще они важны для некоторых процессов модификации разных молекул РНК [84]. Хотя, по большому счету, их роль еще предстоит установить.
Х-хромосома
Как уже упоминалось, у многих организмов есть половые хромосомы, набор которых неодинаков у разных полов. Например, женщины имеют две Х-хромосомы, а мужчины — одну Х и одну Y. Удивительным образом, у дрозофил это устроено так же. Но в связи с этим у одного пола какие-то гены появляются в двойном количестве, а значит, нужно уравнять их транскрипцию. Компенсаторные механизмы могут быть разными. Например, у человека одна из Х-хромосом в женских клетках замалчивается, и гены с нее не транскрибируются (рис. 11). У мушек баланс достигается иначе: у самцов гены с единственной Х-хромосомы транскрибируются в два раза активнее, чем с каждой Х-хромосомы у самок. Белки, которые обеспечивают равную транскрипцию генов Х-хромосом, называются системой дозовой компенсации.
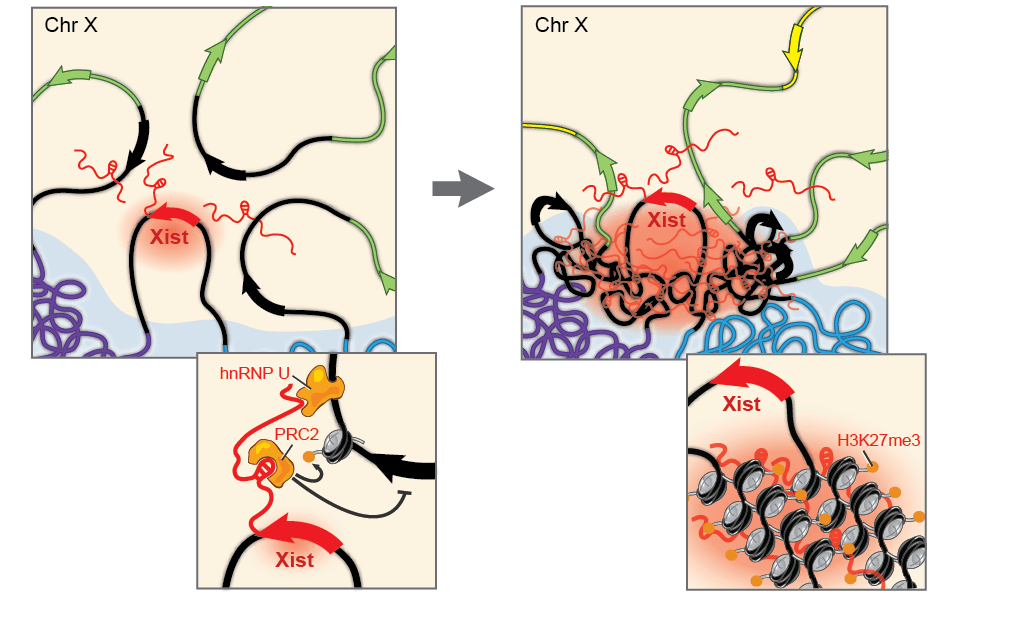
Рисунок 11. Дозовая компенсация у человека — выключение Х-хромосомы. В системе задействована очень важная молекула РНК, которая не кодирует никакого белка и называется Xist (X-inactive specific transcript, Х-неактивный специфический транскрипт). Эта молекула целых 17 тысяч нуклеотидных остатков в длину. Одним из своих участков Xist взаимодействует с комплексом PRC2, и, распространяясь от места своей транскрипции по всей Х-хромосоме, подтаскивает PRC2 ко всем генам хромосомы, на которые комплекс навешивает H3K27me3. Соответственно, эти гены замалчиваются.
У самцов плодовой мушки дрозофилы гены Х-хромосомы транскрибируются в два раза активнее благодаря комплексу дозовой компенсации, состоящему из:
- белка MSL2 (male specific lethal 2, самцы летальны 2, см. предыдущую статью [7]);
- двух белков MSL; белка MLE (maleless, без самцов), умеющего переплетать РНК;
- белка MOF (males absent on the first, самцы отсутствуют в первом [поколении], см. предыдущую статью [7]), навешивающего ацетильные метки на гистоны;
- двух некодирующих РНК roX1 и roX2 (RNA on the X, РНК на Х-хромосоме) [87].
Белок MSL2 связывается с ДНК и собирает остальной комплекс. Он производится только в клетках самцов, но если запустить его синтез и у самок, у них тоже будет собираться и функционировать комплекс дозовой компенсации [88]. В комплексе, который собирает MSL2, ключевую роль играет навешивающий ацетильные метки на гистоны белок MOF: именно благодаря ему транскрипция Х-хромосомных генов повышается.
Как MSL2 узнает Х-хромосому? Во-первых, есть специальные последовательности ДНК, привлекающие MSL2, и если смешать только очищенный белок MSL2 и ДНК в пробирке, то MSL2 будет связываться с некоторыми их этих последовательностей. Интересно, что именно с этими фрагментами ДНК комплекс дозовой компенсации связывается в первую очередь, когда только начинает распространяться по Х-хромосоме [89]. Во-вторых, есть еще один белок Clamp, помогающий комплексу связываться с ДНК. Похоже, для того чтобы комплекс облепил значительную часть Х-хромосомы, нужно, чтобы с ДНК связались одновременно Clamp и MSL2 [90], [91].
У самцов круглого червя C. elegans одна Х-хромосома, а у гермафродитов две. И дозовая компенсация работает таким образом, что транскрипция генов в гермафродитах снижается вдвое на каждой хромосоме. В этом участвует комплекс белков, очень похожий на конденсин, упаковывающий хромосомы перед делением клетки. Есть и еще несколько важных белков. Один из них — SDC-2 (sex determination and dosage compensation protein 2, белок определяющий пол и дозовую компенсацию 2). Он синтезируется только во время эмбрионального развития у гермафродитов и собирает на Х-хромосоме весь комплекс [92], [93]. SDC-2 узнает Х-хромосому по специальным последовательностям ДНК [93], [94]. Детали того, как происходит полузамалчивание генов на Х-хромосомах гермафродитов, пока не очень понятны, но вероятно, что похожий на конденсин комплекс слегка упаковывает хромосомы и делает хроматин менее доступным для белков транскрипции.
Механизмы формирования доменов
Для того чтобы домен в ядре сформировался, нужно несколько вещей:
- Чтобы что-то инициировало его формирование, будь это последовательность ДНК PRE, способная инициировать сборку «синего» хроматина [69], [70], или нуклеосомы с гистоном CenpA, которые запускают сборку центромер [95].
- Чтобы работали молекулярные каскады, формирующие этот домен после каждого удвоения ДНК и деления клетки. Примером этого может быть уже упомянутое привлечение CenpA туда, где уже есть центромерные нуклеосомы, или распространение гетерохроматина.
- Чтобы постоянно были доступны молекулы белков — «запчастей» этого домена [96].
И похоже, что специфическое (то есть, чаще всего, самое сильное) взаимодействие двух отдельных молекул и молекулярные каскады, строящиеся из таких бинарных специфических взаимодействий — только один из механизмов, участвующий в постройке домена. Эта область еще не до конца изучена, но похоже, что есть механизмы, позволяющие концентрировать определенные молекулы в определенной области ядра. Один из этих механизмов называется разделением фаз [97].
Что будет, если смешать масло и воду? Конечно же, наивно ожидать от них образования истинного раствора, потому что молекулы масла и воды «отталкиваются» друг от друга, образуя две несмешивающиеся фазы — это называется гидрофобным эффектом [98]. Нечто похожее происходит и в живой клетке. Некоторые домены (например ядрышко и гетерохроматин [99–101]) образуют фазу, отличающуюся по свойствам от остального вещества ядра и несмешивающуюся с ним. Причем в ядрышке есть внутренняя (плотный фибриллярный компонент) и внешняя (гранулярный компонент) фазы, которые также не смешиваются между собой [99].
Интересно, что по крайней мере некоторые такие фазы в ядре могут накапливать одни молекулы и выталкивать другие [100], [102], [103]. Вполне возможно, что за счет этого эффекта находят друг друга те молекулы, которым необходимо встретиться, чтобы сыграть свои роли. Нужно отметить, что тема фазового разделения в ядре стремительно набирает популярность, и в очень многих работах заявляется, что тот или иной белок стимулирует разделение фаз. Но на деле инструментарий для изучения таких воистину наноскопических явлений довольно скуден, а потому в точности подтвердить, что это так, а помимо этого выявить роль фазовых явлений — довольно трудно. Кроме разделения фаз, есть и другие механизмы, концентрирующие молекулы в определенном месте ядра, а потому читать статьи о фазовых эффектах нужно с осторожностью [104].
Какие еще могут существовать механизмы концентрирования определенных молекул в нужных участках ядра? Хорошим примером могут быть компартменты, которые формируются в ядре при инфекции вирусом герпеса. В этих доменах вирус удваивает свою ДНК. Она свободна от нуклеосом, а поэтому основной белок транскрипции — РНК-полимераза II — и некоторые другие ДНК-связывающие белки накапливаются в этих «вирусных доменах», эффективнее взаимодействуя с «чистой» вирусной ДНК, чем с хозяйским хроматином, покрытым нуклеосомами. Это и впрямь напоминает разделение фаз, но механизм в данном случае другой [105].
Еще один важный механизм формирования доменов — это образование петель. Рассмотреть этот механизм нам поможет ответ на вопрос: как хроматин упаковывается в митотические хромосомы или ядра? Как уже упоминалось, этим занимаются белки конденсины и когезины. Они выпетливают ДНК, пока не встречают инсуляторные белки (это справедливо по крайней мере для когезинов, с конденсинами все может быть сложнее) (рис. 12).
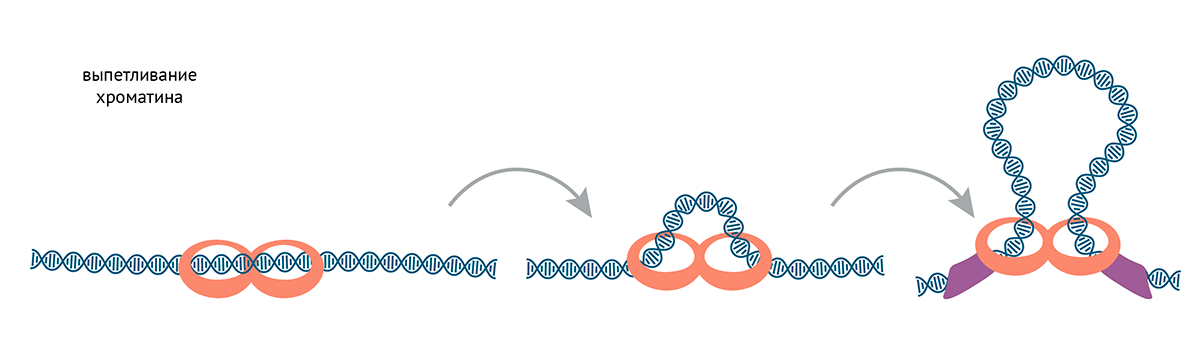
Рисунок 12. Выпетливание хроматина. Когезины и конденсины напоминают кольцо с регулируемым отверстием, которое можно надеть на ДНК и защелкнуть. Кольцо выпетливает ДНК, пока не встречает белки, заякоренные на специальных «блокирующих» последовательностях — инсуляторах. Последние могут взаимодействовать друг с другом, как бы поддерживая основание петли [106]. Одно кольцо когезина/конденсина выпетливает хроматин, связываясь с одной двойной спиралью ДНК специальным ДНК-связывающим доменом, а другую помещая внутри кольца. Так и формируются петли [107], [108].
иллюстрация Михаила Гурьева по [123]
Расположение доменов в ядре
Как уже описывалось выше, в ядре находится несколько типов хроматиновых доменов (рис. 1). Исходя из того, что они расположены на определенных последовательностях ДНК, а ДНК уложена в ядре определенным образом, по крайне мере некоторые домены позиционированы в ядре относительно друг друга. Но возможно, всё наоборот: домены любят быть в определенных местах ядра, и следовательно, ДНК распределена по ядру определенным образом. В любом случае, распределение доменов связано с белками и РНК, которые связываются с хроматином и характерны для определенных доменов. Например, в ядре четко позиционирована ядерная ламина — слой белков, выстилающих внутреннюю оболочку ядра и связывающих хроматин.
Как уже кратко упоминалось, если в человеческих клетках пометить прилегающие к ядерной ламине домены хроматина флуоресцентной меткой и понаблюдать, что с ними происходит после деления в дочерних клетках, окажется, что часть из них остается у ядерной ламины, а часть перемещается к ядрышку [28] (которое практически не соприкасается с ламиной [109]). Следовательно, одни и те же домены хроматина контактируют с оболочкой ядра и с ядрышком.
Интересно, что если удалить из ядра все белки ядерной ламины, домены хроматина по-прежнему сохраняются, однако гетерохроматин становится больше похож на эухроматин [110].
Как в конденсированных хромосомах во время деления клетки, так и вне процесса деления, перицентромерный гетерохроматин прилегает к центромерам, и вместе они образуют кластеры на периферии ядра либо ядрышка [111], [112]. У червячка C. elegans гетерохроматин заякорен на ядерную ламину с помощью связанного с ней белка CEC-4 (С. elegans chromodomain protein 4), распознающего модификацию гетерохроматинового гистона H3K9me2/3 [113].
Теломеры в человеческих клетках и в пекарских дрожжах расположены возле оболочки ядра сразу после деления клетки, когда клеточное ядро еще только собирается заново, и перемещаются во внутреннюю часть ядра по ходу клеточного цикла [114]. Как и центромеры, теломеры собраны в кластеры [115], [116] с той лишь разницей, что они находятся внутри ядра, а не на периферии.
Бывает так, что у одного и того же организма ядро организовано по-разному в зависимости от типа клетки. Например, во всех клетках млекопитающих гетерохроматин находится на периферии ядра, а эухроматин — в центре. В палочках сетчатки ночных животных всё наоборот: гетерохроматин сконцентрирован в середине ядра, а эухроматин вынесен на периферию [117]. Такая организация достигается взаимодействиями внутри гетерохроматина, и отсутствием взаимодействий гетерохроматина с ядерной ламиной [118].
Иногда часть хроматина может отделяться от основной массы домена и перемещаться по ядру. Например, в плодовой мушке дрозофиле, если в гетерохроматине сделать двухцепочечные разрывы ДНК, то часть починки этих двухцепочечных разрывов происходит на периферии ядра. Поломанный участок ДНК при этом перемещается туда, отделяясь от основной массы гетерохроматина [5].
Заключение
Домены в ядре образуются координированными взаимодействиями определенных наборов молекул — игроков эпигенетики [7] (при этом разные домены могут формироваться под влиянием одних и тех же сил). Домены организованы иерархически. Они позиционированы в ядре относительно друг друга, и по крайней мере часть из них ведет себя динамически — перемещается по ядру и обменивается хроматиновыми молекулами (но «знаковые» для данного домена молекулы всегда остаются в избытке).
Хроматиновые домены важно изучать, поскольку их нарушенная регуляция может вести к заболеваниям. Подробнее про это будет написано в шестой статье цикла — «Медицина и эпигеном».
В следующей же статье — «Наследование и эпигеном» — мы рассмотрим, как отдельные эпигенетические молекулы, а также целые хроматиновые домены наследуются в ходе репликации и деления клетки на протяжении поколений.
Литература
- Kochanova N. Intricate Structure of the interphase chromocenter revealed by proximity-based biotinylation: LMU Doktorarbeit. München, 2020;
- Wendy A. Bickmore, Bas van Steensel. (2013). Genome Architecture: Domain Organization of Interphase Chromosomes. Cell. 152, 1270-1284;
- Tobias Straub, Martin F. Neumann, Matthias Prestel, Elisabeth Kremmer, Christoph Kaether, et. al.. (2005). Stable chromosomal association of MSL2 defines a dosage-compensated nuclear compartment. Chromosoma. 114, 352-364;
- S. W. Brown. (1966). Heterochromatin. Science. 151, 417-425;
- Irene Chiolo, Aki Minoda, Serafin U. Colmenares, Aris Polyzos, Sylvain V. Costes, Gary H. Karpen. (2011). Double-Strand Breaks in Heterochromatin Move Outside of a Dynamic HP1a Domain to Complete Recombinational Repair. Cell. 144, 732-744;
- Joel C. Eissenberg, Gunter Reuter. (2009). Chapter 1 Cellular Mechanism for Targeting Heterochromatin Formation in Drosophila. International Review of Cell and Molecular Biology. 1-47;
- Молекулы и эпигеном ;
- Новый взгляд на геном: не просто цепочка генов, а трехмерная сеть, интегрирующая функциональные домены ядра;
- William C. Earnshaw, Barbara R. Migeon. (1985). Three related centromere proteins are absent from the inactive centromere of a stable isodicentric chromosome. Chromosoma. 92, 290-296;
- DK Palmer, K O'Day, MH Wener, BS Andrews, RL Margolis. (1987). A 17-kD centromere protein (CENP-A) copurifies with nucleosome core particles and with histones. The Journal of Cell Biology. 104, 805-815;
- Размер имеет значение;
- François-Michel Boisvert, Silvana van Koningsbruggen, Joaquín Navascués, Angus I. Lamond. (2007). The multifunctional nucleolus. Nat Rev Mol Cell Biol. 8, 574-585;
- Vincenzo Pirrotta, Hua-Bing Li. (2012). A view of nuclear Polycomb bodies. Current Opinion in Genetics & Development. 22, 101-109;
- Matheson L. and Elderkin S. Polycomb bodies. In: Nuclear architechture and dynamics / ed. by Lavelle C., Jean-Marc V. Academic Press, 2018. P. 297–320;
- V. Lallemand-Breitenbach, H. de The. (2010). PML Nuclear Bodies. Cold Spring Harbor Perspectives in Biology. 2, a000661-a000661;
- Julie G. Donaldson. (1998). Immunofluorescence Staining. Current Protocols in Cell Biology. 00;
- Dmitriy M. Chudakov, Sergey Lukyanov, Konstantin A. Lukyanov. (2005). Fluorescent proteins as a toolkit for in vivo imaging. Trends in Biotechnology. 23, 605-613;
- Arkady F. Fradkov, Ying Chen, Li Ding, Ekaterina V. Barsova, Mikhail V. Matz, Sergey A. Lukyanov. (2000). Novel fluorescent protein from Discosoma coral and its mutants possesses a unique far-red fluorescence. FEBS Letters. 479, 127-130;
- Osamu Shimomura, Frank H. Johnson, Yo Saiga. (1962). Extraction, Purification and Properties of Aequorin, a Bioluminescent Protein from the Luminous Hydromedusan,Aequorea. J. Cell. Comp. Physiol.. 59, 223-239;
- Флуоресцирующая Нобелевская премия по химии;
- Флуоресцентные белки: разнообразнее, чем вы думали!;
- Флуоресцентные репортеры и их молекулярные репортажи;
- Jesse R. Dixon, David U. Gorkin, Bing Ren. (2016). Chromatin Domains: The Unit of Chromosome Organization. Molecular Cell. 62, 668-680;
- E. Lieberman-Aiden, N. L. van Berkum, L. Williams, M. Imakaev, T. Ragoczy, et. al.. (2009). Comprehensive Mapping of Long-Range Interactions Reveals Folding Principles of the Human Genome. Science. 326, 289-293;
- Guillaume J. Filion, Joke G. van Bemmel, Ulrich Braunschweig, Wendy Talhout, Jop Kind, et. al.. (2010). Systematic Protein Location Mapping Reveals Five Principal Chromatin Types in Drosophila Cells. Cell. 143, 212-224;
- Peter V. Kharchenko, Artyom A. Alekseyenko, Yuri B. Schwartz, Aki Minoda, Nicole C. Riddle, et. al.. (2011). Comprehensive analysis of the chromatin landscape in Drosophila melanogaster. Nature. 471, 480-485;
- Bas van Steensel. (2011). Chromatin: constructing the big picture. The EMBO Journal. 30, 1885-1895;
- Jop Kind, Ludo Pagie, Havva Ortabozkoyun, Shelagh Boyle, Sandra S. de Vries, et. al.. (2013). Single-Cell Dynamics of Genome-Nuclear Lamina Interactions. Cell. 153, 178-192;
- Johan H. Gibcus, Kumiko Samejima, Anton Goloborodko, Itaru Samejima, Natalia Naumova, et. al.. (2018). A pathway for mitotic chromosome formation. Science. 359, eaao6135;
- N. Naumova, M. Imakaev, G. Fudenberg, Y. Zhan, B. R. Lajoie, et. al.. (2013). Organization of the Mitotic Chromosome. Science. 342, 948-953;
- Kristin Abramo, Anne-Laure Valton, Sergey V. Venev, Hakan Ozadam, A. Nicole Fox, Job Dekker. (2019). A chromosome folding intermediate at the condensin-to-cohesin transition during telophase. Nat Cell Biol. 21, 1393-1402;
- Florian A Steiner, Steven Henikoff. (2015). Diversity in the organization of centromeric chromatin. Current Opinion in Genetics & Development. 31, 28-35;
- Michael D. Blower, Gary H. Karpen. (2001). The role of Drosophila CID in kinetochore formation, cell-cycle progression and heterochromatin interactions. Nat Cell Biol. 3, 730-739;
- Vinciane Régnier, Paola Vagnarelli, Tatsuo Fukagawa, Tatiana Zerjal, Elizabeth Burns, et. al.. (2005). CENP-A Is Required for Accurate Chromosome Segregation and Sustained Kinetochore Association of BubR1. Mol Cell Biol. 25, 3967-3981;
- Daniel R. Foltz, Lars E. T. Jansen, Ben E. Black, Aaron O. Bailey, John R. Yates, Don W. Cleveland. (2006). The human CENP-A centromeric nucleosome-associated complex. Nat Cell Biol. 8, 458-469;
- Hiroshi Izuta, Masashi Ikeno, Nobutaka Suzuki, Takeshi Tomonaga, Naohito Nozaki, et. al.. (2006). Comprehensive analysis of the ICEN (Interphase Centromere Complex) components enriched in the CENP-A chromatin of human cells. Genes to Cells. 11, 673-684;
- Chikashi Obuse, Hua Yang, Naohito Nozaki, Shouhei Goto, Tuneko Okazaki, Kinya Yoda. (2004). Proteomics analysis of the centromere complex from HeLa interphase cells: UV-damaged DNA binding protein 1 (DDB-1) is a component of the CEN-complex, while BMI-1 is transiently co-localized with the centromeric region in interphase. Genes to Cells. 9, 105-120;
- Dongqing Pan, Kai Walstein, Annika Take, David Bier, Nadine Kaiser, Andrea Musacchio. (2019). Mechanism of centromere recruitment of the CENP-A chaperone HJURP and its implications for centromere licensing. Nat Commun. 10;
- The structure and function of specific chromosome regions. I. Centromeres. (1998). Chromosome Res. 6, 65–71;
- Virginie Roure, Bethan Medina-Pritchard, Vasiliki Lazou, Luciano Rago, Eduard Anselm, et. al.. (2019). Reconstituting Drosophila Centromere Identity in Human Cells. Cell Reports. 29, 464-479.e5;
- Teresa K. Barth, Georg O. M. Schade, Andreas Schmidt, Irene Vetter, Marc Wirth, et. al.. (2014). Identification of novel Drosophila centromere-associated proteins. Proteomics. 14, 2167-2178;
- Joel C. Eissenberg, Sarah C.R. Elgin. (2014). HP1a: a structural chromosomal protein regulating transcription. Trends in Genetics. 30, 103-110;
- Tomohiro Hayakawa, Tokuko Haraguchi, Hiroshi Masumoto, Yasushi Hiraoka. (2003). Cell cycle behavior of human HP1 subtypes: distinct molecular domains of HP1 are required for their centromeric localization during interphase and metaphase. Journal of Cell Science. 116, 3327-3338;
- Joel M Swenson, Serafin U Colmenares, Amy R Strom, Sylvain V Costes, Gary H Karpen. (2016). The composition and organization of Drosophila heterochromatin are heterogeneous and dynamic. eLife. 5;
- Daniele Canzio, Evelyn Y. Chang, Smita Shankar, Kristopher M. Kuchenbecker, Matthew D. Simon, et. al.. (2011). Chromodomain-Mediated Oligomerization of HP1 Suggests a Nucleosome-Bridging Mechanism for Heterochromatin Assembly. Molecular Cell. 41, 67-81;
- G. Schotta. (2002). Central role of Drosophila SU(VAR)3-9 in histone H3-K9 methylation and heterochromatic gene silencing. The EMBO Journal. 21, 1121-1131;
- S. C. R. Elgin, G. Reuter. (2013). Position-Effect Variegation, Heterochromatin Formation, and Gene Silencing in Drosophila. Cold Spring Harbor Perspectives in Biology. 5, a017780-a017780;
- Grigliatti T. and Mottus R. Position effects. In: Encyclopedia of genetics / ed. by Brenner S., Miller J.H. Academic Press, 2001. P. 1523–1530;
- John Maciejowski, Titia de Lange. (2017). Telomeres in cancer: tumour suppression and genome instability. Nat Rev Mol Cell Biol. 18, 175-186;
- M. O'Donnell, L. Langston, B. Stillman. (2013). Principles and Concepts of DNA Replication in Bacteria, Archaea, and Eukarya. Cold Spring Harbor Perspectives in Biology. 5, a010108-a010108;
- V. Pfeiffer, J. Lingner. (2013). Replication of Telomeres and the Regulation of Telomerase. Cold Spring Harbor Perspectives in Biology. 5, a010405-a010405;
- A. G. Bodnar. (1998). Extension of Life-Span by Introduction of Telomerase into Normal Human Cells. Science. 279, 349-352;
- Thomas Simonet, Laure-Emmanuelle Zaragosi, Claude Philippe, Kevin Lebrigand, Clémentine Schouteden, et. al.. (2011). The human TTAGGG repeat factors 1 and 2 bind to a subset of interstitial telomeric sequences and satellite repeats. Cell Res. 21, 1028-1038;
- Tomohiko Iwano, Makoto Tachibana, Michael Reth, Yoichi Shinkai. (2004). Importance of TRF1 for Functional Telomere Structure. Journal of Biological Chemistry. 279, 1442-1448;
- Diego Loayza, Titia de Lange. (2003). POT1 as a terminal transducer of TRF1 telomere length control. Nature. 423, 1013-1018;
- Keiji Okamoto, Tomohiko Iwano, Makoto Tachibana, Yoichi Shinkai. (2008). Distinct Roles of TRF1 in the Regulation of Telomere Structure and Lengthening. Journal of Biological Chemistry. 283, 23981-23988;
- Jeffrey Zheng-Sheng Ye, Jill R. Donigian, Megan van Overbeek, Diego Loayza, Yan Luo, et. al.. (2004). TIN2 Binds TRF1 and TRF2 Simultaneously and Stabilizes the TRF2 Complex on Telomeres. Journal of Biological Chemistry. 279, 47264-47271;
- Eladio Abreu, Elena Aritonovska, Patrick Reichenbach, Gaël Cristofari, Brad Culp, et. al.. (2010). TIN2-Tethered TPP1 Recruits Human Telomerase to Telomeres In Vivo. Mol Cell Biol. 30, 2971-2982;
- Stefan Schoeftner, Maria A Blasco. (2009). A ‘higher order’ of telomere regulation: telomere heterochromatin and telomeric RNAs. EMBO J. 28, 2323-2336;
- Мода на ретро. Где встречается обратная транскрипция, и как она эволюционировала;
- Elena Casacuberta. (2017). Drosophila: Retrotransposons Making up Telomeres. Viruses. 9, 192;
- Arnold M. Kiss, Beáta E. Jády, Edouard Bertrand, Tamás Kiss. (2004). Human Box H/ACA Pseudouridylation Guide RNA Machinery. Mol Cell Biol. 24, 5797-5807;
- Daniel G. Booth, Alison J. Beckett, Oscar Molina, Itaru Samejima, Hiroshi Masumoto, et. al.. (2016). 3D-CLEM Reveals that a Major Portion of Mitotic Chromosomes Is Not Chromatin. Molecular Cell. 64, 790-802;
- Raffaella Santoro, Junwei Li, Ingrid Grummt. (2002). The nucleolar remodeling complex NoRC mediates heterochromatin formation and silencing of ribosomal gene transcription. Nat Genet. 32, 393-396;
- Christian Schöfer, Klara Weipoltshammer. (2018). Nucleolus and chromatin. Histochem Cell Biol. 150, 209-225;
- W. Xie, T. Ling, Y. Zhou, W. Feng, Q. Zhu, et. al.. (2012). The chromatin remodeling complex NuRD establishes the poised state of rRNA genes characterized by bivalent histone modifications and altered nucleosome positions. Proceedings of the National Academy of Sciences. 109, 8161-8166;
- Bernd Schuettengruber, Henri-Marc Bourbon, Luciano Di Croce, Giacomo Cavalli. (2017). Genome Regulation by Polycomb and Trithorax: 70 Years and Counting. Cell. 171, 34-57;
- Manching Ku, Richard P. Koche, Esther Rheinbay, Eric M. Mendenhall, Mitsuhiro Endoh, et. al.. (2008). Genomewide Analysis of PRC1 and PRC2 Occupancy Identifies Two Classes of Bivalent Domains. PLoS Genet. 4, e1000242;
- Eshagh Dorafshan, Tatyana G. Kahn, Yuri B. Schwartz. (2017). Hierarchical recruitment of Polycomb complexes revisited. Nucleus. 8, 496-505;
- Guido van Mierlo, Gert Jan C. Veenstra, Michiel Vermeulen, Hendrik Marks. (2019). The Complexity of PRC2 Subcomplexes. Trends in Cell Biology. 29, 660-671;
- Raphael Margueron, Neil Justin, Katsuhito Ohno, Miriam L. Sharpe, Jinsook Son, et. al.. (2009). Role of the polycomb protein EED in the propagation of repressive histone marks. Nature. 461, 762-767;
- Ozgur Oksuz, Varun Narendra, Chul-Hwan Lee, Nicolas Descostes, Gary LeRoy, et. al.. (2018). Capturing the Onset of PRC2-Mediated Repressive Domain Formation. Molecular Cell. 70, 1149-1162.e5;
- Neil P. Blackledge, Anca M. Farcas, Takashi Kondo, Hamish W. King, Joanna F. McGouran, et. al.. (2014). Variant PRC1 Complex-Dependent H2A Ubiquitylation Drives PRC2 Recruitment and Polycomb Domain Formation. Cell. 157, 1445-1459;
- Sarah Cooper, Anne Grijzenhout, Elizabeth Underwood, Katia Ancelin, Tianyi Zhang, et. al.. (2016). Jarid2 binds mono-ubiquitylated H2A lysine 119 to mediate crosstalk between Polycomb complexes PRC1 and PRC2. Nat Commun. 7;
- Hongjae Sunwoo, John Y. Wu, Jeannie T. Lee. (2015). The Xist RNA-PRC2 complex at 20-nm resolution reveals a low Xist stoichiometry and suggests a hit-and-run mechanism in mouse cells. Proc Natl Acad Sci USA. 112, E4216-E4225;
- Ajazul H. Wani, Alistair N. Boettiger, Patrick Schorderet, Ayla Ergun, Christine Münger, et. al.. (2016). Chromatin topology is coupled to Polycomb group protein subnuclear organization. Nat Commun. 7;
- Энхансеры и промоторы, регулирующие формирование коры головного мозга, у человека активнее, чем у других животных;
- Leah A. Gates, Charles E. Foulds, Bert W. O’Malley. (2017). Histone Marks in the ‘Driver’s Seat’: Functional Roles in Steering the Transcription Cycle. Trends in Biochemical Sciences. 42, 977-989;
- Транскрипция в хроматине: как проходить сквозь стены;
- A Uzman. (2001). Molecular Cell Biology (4th edition) Harvey Lodish, Arnold Berk, S. Lawrence Zipursky, Paul Matsudaira, David Baltimore and James Darnell; Freeman & Co., New York, NY, 2000, 1084 pp., list price $102.25, ISBN 0-7167-3136-3. Biochemistry and Molecular Biology Education. 29, 126-128;
- Anusha P. Dias, Kobina Dufu, Haixin Lei, Robin Reed. (2010). A role for TREX components in the release of spliced mRNA from nuclear speckle domains. Nat Commun. 1;
- Lukasz Galganski, Martyna O. Urbanek, Wlodzimierz J. Krzyzosiak. (2017). Nuclear speckles: molecular organization, biological function and role in disease. Nucleic Acids Research. 45, 10350-10368;
- Cyrille Girard, Cindy L. Will, Jianhe Peng, Evgeny M. Makarov, Berthold Kastner, et. al.. (2012). Post-transcriptional spliceosomes are retained in nuclear speckles until splicing completion. Nat Commun. 3;
- Archa H. Fox, Shinichi Nakagawa, Tetsuro Hirose, Charles S. Bond. (2018). Paraspeckles: Where Long Noncoding RNA Meets Phase Separation. Trends in Biochemical Sciences. 43, 124-135;
- Tetsuro Hirose, Giorgio Virnicchi, Akie Tanigawa, Takao Naganuma, Ruohan Li, et. al.. (2014). NEAT1 long noncoding RNA regulates transcription via protein sequestration within subnuclear bodies. MBoC. 25, 169-183;
- Jason A. West, Mari Mito, Satoshi Kurosaka, Toru Takumi, Chiharu Tanegashima, et. al.. (2016). Structural, super-resolution microscopy analysis of paraspeckle nuclear body organization. Journal of Cell Biology. 214, 817-830;
- Tobias Straub, Peter B. Becker. (2007). Dosage compensation: the beginning and end of generalization. Nat Rev Genet. 8, 47-57;
- Richard L Kelley, Irina Solovyeva, Laura M Lyman, Ron Richman, Victor Solovyev, Mitzi I Kuroda. (1995). Expression of Msl-2 causes assembly of dosage compensation regulators on the X chromosomes and female lethality in Drosophila. Cell. 81, 867-877;
- Raffaella Villa, Tamas Schauer, Pawel Smialowski, Tobias Straub, Peter B. Becker. (2016). PionX sites mark the X chromosome for dosage compensation. Nature. 537, 244-248;
- Christian Albig, Evgeniya Tikhonova, Silke Krause, Oksana Maksimenko, Catherine Regnard, Peter B Becker. (2019). Factor cooperation for chromosome discrimination inDrosophila. Nucleic Acids Research. 47, 1706-1724;
- M. M. L. Soruco, J. Chery, E. P. Bishop, T. Siggers, M. Y. Tolstorukov, et. al.. (2013). The CLAMP protein links the MSL complex to the X chromosome during Drosophila dosage compensation. Genes & Development. 27, 1551-1556;
- H. E. Dawes. (1999). Dosage Compensation Proteins Targeted to X Chromosomes by a Determinant of Hermaphrodite Fate. Science. 284, 1800-1804;
- Sevinç Ercan. (2015). Mechanisms of X Chromosome Dosage Compensation. J. Genomics. 3, 1-19;
- G. Csankovszki. (2004). Recruitment and Spreading of the C. elegans Dosage Compensation Complex Along X Chromosomes. Science. 303, 1182-1185;
- Sebastian Müller, Geneviève Almouzni. (2017). Chromatin dynamics during the cell cycle at centromeres. Nat Rev Genet. 18, 192-208;
- Robin C. Allshire, Hiten D. Madhani. (2018). Ten principles of heterochromatin formation and function. Nat Rev Mol Cell Biol. 19, 229-244;
- Разделяй и властвуй: роль разделения фаз в жизни клетки;
- Физическая водобоязнь;
- Marina Feric, Nilesh Vaidya, Tyler S. Harmon, Diana M. Mitrea, Lian Zhu, et. al.. (2016). Coexisting Liquid Phases Underlie Nucleolar Subcompartments. Cell. 165, 1686-1697;
- Adam G. Larson, Daniel Elnatan, Madeline M. Keenen, Michael J. Trnka, Jonathan B. Johnston, et. al.. (2017). Liquid droplet formation by HP1α suggests a role for phase separation in heterochromatin. Nature. 547, 236-240;
- Amy R. Strom, Alexander V. Emelyanov, Mustafa Mir, Dmitry V. Fyodorov, Xavier Darzacq, Gary H. Karpen. (2017). Phase separation drives heterochromatin domain formation. Nature. 547, 241-245;
- Huasong Lu, Dan Yu, Anders S. Hansen, Sourav Ganguly, Rongdiao Liu, et. al.. (2018). Phase-separation mechanism for C-terminal hyperphosphorylation of RNA polymerase II. Nature. 558, 318-323;
- Benjamin R. Sabari, Alessandra Dall’Agnese, Ann Boija, Isaac A. Klein, Eliot L. Coffey, et. al.. (2018). Coactivator condensation at super-enhancers links phase separation and gene control. Science. 361, eaar3958;
- David T. McSwiggen, Mustafa Mir, Xavier Darzacq, Robert Tjian. (2019). Evaluating phase separation in live cells: diagnosis, caveats, and functional consequences. Genes Dev.. 33, 1619-1634;
- David Trombley McSwiggen, Anders S Hansen, Sheila S Teves, Hervé Marie-Nelly, Yvonne Hao, et. al.. (2019). Evidence for DNA-mediated nuclear compartmentalization distinct from phase separation. eLife. 8;
- Oksana Maksimenko, Pavel Georgiev. (2014). Mechanisms and proteins involved in long-distance interactions. Front. Genet.. 5;
- Новый взгляд на геном: не просто цепочка генов, а трехмерная сеть, интегрирующая функциональные домены ядра;
- Организовать геном: запутанная история гипотез и экспериментов;
- Jan Padeken, María José Mendiburo, Sarantis Chlamydas, Hans-Jürgen Schwarz, Elisabeth Kremmer, Patrick Heun. (2013). The Nucleoplasmin Homolog NLP Mediates Centromere Clustering and Anchoring to the Nucleolus. Molecular Cell. 50, 236-249;
- Sergey V. Ulianov, Semen A. Doronin, Ekaterina E. Khrameeva, Pavel I. Kos, Artem V. Luzhin, et. al.. (2019). Nuclear lamina integrity is required for proper spatial organization of chromatin in Drosophila. Nat Commun. 10;
- Geneviève Almouzni, Aline V. Probst. (2011). Heterochromatin maintenance and establishment: Lessons from the mouse pericentromere. Nucleus. 2, 332-338;
- Claudia Weierich, Alessandro Brero, Stefan Stein, Johann von Hase, Christoph Cremer, et. al.. (2003). . Chromosome Research. 11, 485-502;
- Adriana Gonzalez-Sandoval, Benjamin D. Towbin, Veronique Kalck, Daphne S. Cabianca, Dimos Gaidatzis, et. al.. (2015). Perinuclear Anchoring of H3K9-Methylated Chromatin Stabilizes Induced Cell Fate in C. elegans Embryos. Cell. 163, 1333-1347;
- Laure Crabbe, Anthony J. Cesare, James M. Kasuboski, James A.J. Fitzpatrick, Jan Karlseder. (2012). Human Telomeres Are Tethered to the Nuclear Envelope during Postmitotic Nuclear Assembly. Cell Reports. 2, 1521-1529;
- Nancy Adam, Erin Degelman, Sophie Briggs, Rima-Marie Wazen, Pina Colarusso, et. al.. (2019). Telomere analysis using 3D fluorescence microscopy suggests mammalian telomere clustering in hTERT-immortalized Hs68 fibroblasts. Commun Biol. 2;
- C. Molenaar. (2003). Visualizing telomere dynamics in living mammalian cells using PNA probes. The EMBO Journal. 22, 6631-6641;
- Irina Solovei, Moritz Kreysing, Christian Lanctôt, Süleyman Kösem, Leo Peichl, et. al.. (2009). Nuclear Architecture of Rod Photoreceptor Cells Adapts to Vision in Mammalian Evolution. Cell. 137, 356-368;
- Martin Falk, Yana Feodorova, Natalia Naumova, Maxim Imakaev, Bryan R. Lajoie, et. al.. (2019). Heterochromatin drives compartmentalization of inverted and conventional nuclei. Nature. 570, 395-399;
- Robert K. McGinty, Ryan C. Henrici, Song Tan. (2014). Crystal structure of the PRC1 ubiquitylation module bound to the nucleosome. Nature. 514, 591-596;
- Simon Poepsel, Vignesh Kasinath, Eva Nogales. (2018). Cryo-EM structures of PRC2 simultaneously engaged with two functionally distinct nucleosomes. Nat Struct Mol Biol. 25, 154-162;
- T. Volpe, R. A. Martienssen. (2011). RNA Interference and Heterochromatin Assembly. Cold Spring Harbor Perspectives in Biology. 3, a003731-a003731;
- J. M. Engreitz, A. Pandya-Jones, P. McDonel, A. Shishkin, K. Sirokman, et. al.. (2013). The Xist lncRNA Exploits Three-Dimensional Genome Architecture to Spread Across the X Chromosome. Science. 341, 1237973-1237973;
- Je-Kyung Ryu, Allard J. Katan, Eli O. van der Sluis, Thomas Wisse, Ralph de Groot, et. al.. (2020). The condensin holocomplex cycles dynamically between open and collapsed states. Nat Struct Mol Biol. 27, 1134-1141.







