Alu: история одной последовательности
31 октября 2012
Alu: история одной последовательности
- 5516
- 0
- 10
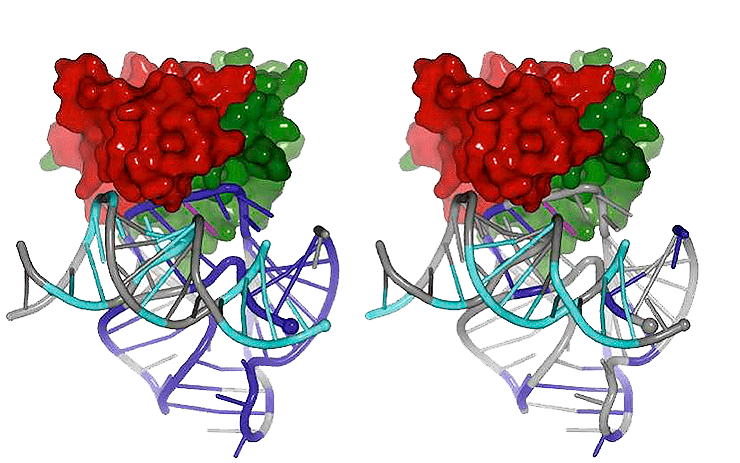
Модель связывания РНК Alu (показаны левый и правый мономеры) с белком SRP9/14. Cубъединица SRP9 показана красным, SRP14 — зелёным.
-
Автор
-
Редакторы
Статья на конкурс «био/мол/текст»: Мобильные элементы, или транспозоны, — это последовательности ДНК, способные перемещаться по геному. С помощью ферментов одни мобильные элементы вырезаются из ДНК и встраиваются в другое место двойной спирали, другие — копируются и встраивают в геном свои копии. Последние получили название «ретроэлементов», или «ретротранспозонов». Именно об одном из таких элементов, относящемся к группе SINE (short interspersed nucleotide elements), — Alu — и пойдёт речь в этой статье.
Конкурс «био/мол/текст»-2012
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2012 в номинации «Лучший обзор».
Спонсор конкурса — дальновидная компания Thermo Fisher Scientific.
Немного молекулярной анатомии
Alu — один из самых многочисленных ретроэлементов в геноме человека. Больше миллиона копий одной последовательности разбросано по нашим хромосомам, составляя почти 11% нашей ДНК [1]. Бóльшая часть этих копий уже давно не «прыгает» по геному, и только недавно появившиеся, молодые Alu-элементы способны к самокопированию-встраиванию. Все копии Alu в нашем геноме образуют целое семейство [2].
Своё название Alu позаимствовал у фермента рестриктазы (молекула, которая режет нить ДНК). Рестриктазу когда-то выделили из бактерии Arthrobacter luteus и назвали по первым буквам латинского наименования микроорганизма. Однажды, когда взяли и обработали этой рестриктазой ДНК человека (порезали на фрагменты), Alu и был открыт [27].
Итак, семейство Alu относится к группе мобильных элементов SINE (от англ. short interspersed nucleotide elements), дословно — «короткие разбросанные нуклеотидные элементы». Давайте посмотрим, как устроен типичный элемент Alu (рис. 1). Он представляет собой последовательность длиной около 300 нуклеотидных остатков.

Рисунок 1. Схема Alu. ЛМ — левый мономер; ПМ — правый мономер.
Последовательность эта состоит из двух мономеров: правого и левого. Ещё у Alu есть так называемая А-богатая область («А-богатая» — значит, из четырёх оснований в её нуклеотидах больше всего аденина), которая важна для того, чтобы Alu распространялся по нашим геномам [3], [4], [24].
Элементы SINE не содержат последовательностей, кодирующих белки (так называемых «открытых рамок считывания»). Значит, у них нет и ферментов, которые могут скопировать их и вставить в другое место ДНК. Именно поэтому SINE используют ферменты, которые закодированы в ретроэлементах другой группы — LINE (от англ. long interspersed nucleotide elements), дословно — «длинные разбросанные нуклеотидные элементы». Партнёр Alu — длинный элемент в 6000 нуклеотидных остатков, которого зовут LINE-1 (рис. 2) .
На конкурс «био/мол/текст»-2012 подана еще одна статья, в которой говорится про ретротранспозон LINE-1: «Разнообразия много не бывает: чем занимаются мобильные элементы генома в мозге» [14]. Кроме того, мы писали о нем и ранее: «Тайны „молекулярных паразитов“, или Как путешествовать по геному» [26]. — Ред.
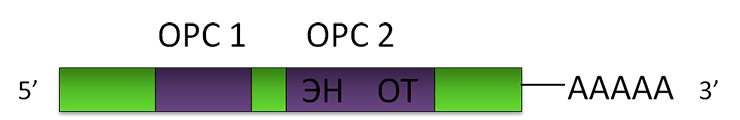
Рисунок 2. Схема LINE-1. ОРС — открытая рамка считывания; ЭН — эндонуклеаза; ОТ — обратная транскриптаза.
Элемент LINE-1 содержит две открытые рамки считывания (ОРС) — последовательности, каждая из которых кодирует одну полипептидную (состоящую из аминокислот) цепь. Первая рамка считывания кодирует белок, который нужен для ретропозиции самому LINE-1, вторая — белок-фермент, обладающий двумя функциями: эндонуклеазы (ЭН) и обратной транскриптазы (ОТ). Эндонуклеаза — это молекулярные «ножницы», которые могут разрезать нить ДНК. Обратная транскриптаза может синтезировать (химически «строить») нить ДНК на матрице РНК как на шаблоне, другими словами — осуществлять процесс обратной транскрипции. Поли(А)-хвост LINE-1, в отличие от Alu, достраивается уже после окончания транскрипции предназначенным для этого клеточным ферментом: полиА-полимеразой.
«Как вы думаете, кто украл транскриптазу, Холмс?» — «Элементарно, Ватсон! Это был Alu»
Модель того, как Alu распространяется по геному, представляют себе следующим образом. Представим себе копию Alu — часть двуцепочечной ДНК. Клеточный фермент РНК-полимераза комплементарно достраивает РНК по одной из цепей ДНК. Эта стадия называется транскрипцией. Дальше РНК Alu выходит из клеточного ядра, где происходила транскрипция, в цитоплазму, где связывается с ферментом, обладающим функциями обратной транскриптазы и эндонуклеазы (для краткости буду дальше называть его просто обратной транскриптазой). Вместе с ферментом РНК Alu идет обратно в ядро, где фермент вносит разрыв в одну цепь двуцепочечной геномной ДНК. Затем РНК Alu комплементарно «цепляется» за фрагмент геномной ДНК в месте разрыва, и обратная транскриптаза LINE-1 осуществляет обратную транскрипцию: синтезирует одну цепь ДНК Alu на матрице РНК. При этом фрагмент геномной ДНК, который «зацепился» за РНК Alu, она использует в качестве затравки (то есть на него она должна «сесть» и продолжить синтез уже существующей цепи). Потом фермент достраивает вторую цепь, а другие клеточные ферменты достраивают ДНК по бокам от Alu и «зашивают» все разрывы (осуществляют «репарацию» геномной ДНК); см. рис. 3, 4. Следует заметить, что такой цикл копирования-встраивания свойственен не только Alu, но и другим элементам SINE.
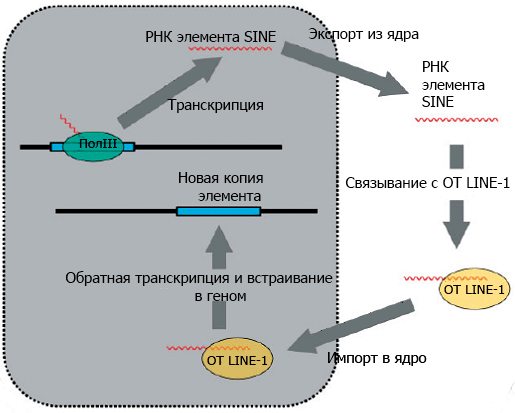
Рисунок 3. Цикл транспозиции элементов SINE, в том числе и Аlu. Элемент транскрибируется клеточной РНК полимеразой III (ПолIII), его РНК выходит в цитоплазму, связывается с обратной транскриптазой LINE-1 (ОТ LINE-1) и импортируется в ядро, где происходит обратная транскрипция и встраивание в геном.
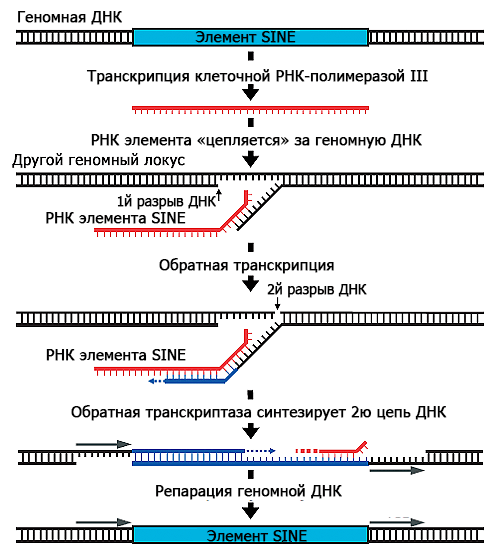
Рисунок 4. Схема копирования и встраивания элемента SINE (таким элементом может быть и Alu). Клеточная РНК-полимераза III считывает РНК с геномной ДНК. Обратная транскриптаза LINE-1 вносит в геномную ДНК одноцепочечный разрыв, РНК SINE-элемента «цепляется» за выступающую нить геномной ДНК, и обратная транскриптаза синтезирует сначала одну нить ДНК на матрице РНК, а потом достраивает вторую нить ДНК, комплементарную первой. Клеточные ферменты достраивают нужные нуклеотиды и «зашивают» разрывы.
Как же РНК Alu, находясь в цитоплазме клетки, умудряется «переманить» на себя обратную транскриптазу, полноправный хозяин которой — LINE-1? Дело тут вот в чём: РНК Alu может связываться с двумя важными белками. Один — это так называемый поли(А)-связывающий белок (связывается с А-богатой областью Alu), другой носит название SRP9/14 (Signal Recognition Particle 9/14). РНК Alu с поли(А)-связывающим белком и SRP 9/14 вместе могут образовывать рибонуклеопротеид, то есть соединение из белка и нуклеиновой кислоты (см. заглавную иллюстрацию).
Этот рибонуклеопротеид может «заякориваться» на синтезирующей (химически «строящей») белок рибосоме, и, в то время как матричная РНК LINE-1 транслируется, Alu и LINE-1 конкурируют за взаимодействие с вновь образующимся белком — ферментом обратной транскриптазой LINE-1. Это вероятностный процесс, и если Alu оказывается достаточно удачлив, он коварно «крадёт» транскриптазу у элемента LINE и направляется в клеточное ядро, чтобы пройти обратную транскрипцию и встроиться в геном. Для того, чтобы «украсть» фермент, РНК Alu необходима А-богатая область, поскольку обратная транскриптаза садится именно на неё. Этот белок химически взаимодействует с поли(А)-областью и не знает, за чей хвост «схватился» (рис. 5). Alu пользуется этим и «выдаёт» свой поли(А)-хвост за поли(А)-хвост LINE-1, забирая транскриптазу на себя [25].
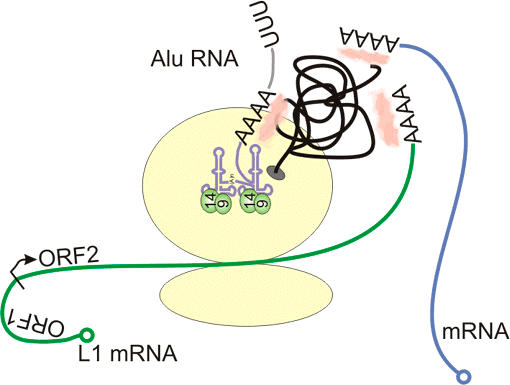
Рисунок 5. Alu собирается забрать транскриптазу на рибосоме. Рибосома на рисунке — две большие бежевые субъединицы. РНК Alu показана фиолетовым, мРНК LINE-1 — зелёным. Чёрная линия — синтезирующаяся на рибосоме обратная транскриптаза, поли(А)-связывающий белок показан розовым, SRP9/14 — салатовым. Синим показана подошедшая к рибосоме клеточная мРНК.
И что дальше?
Копии мобильных элементов и Alu в частности в разных местах нашей ДНК могут стать причиной перегруппировки генома путём разрыва и соединения молекул ДНК — рекомбинации. Гомологичная рекомбинация осуществляется на основе гомологии между рекомбинирующими последовательностями: они должны быть очень сходными. Вследствие гомологичной рекомбинации разных участков ДНК, в том числе, и элементов SINE, внутри одной хромосомы отдельные участки последовательности могут, например, выпадать, или переворачиваться на 180° (рис. 6). Соответствующие мутации носят названия делеций и инверсий. Помимо мутаций, затрагивающих одну хромосому, могут происходить межхромосомные перегруппировки.
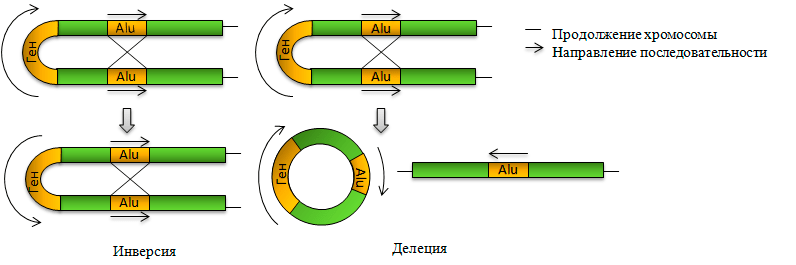
Рисунок 6. Внутрихромосомные перегруппировки, которые могут быть вызваны копиями Alu и других мобильных элементов.
В клеточном ядре хромосомы хоть и не сильно отделены друг от друга в пространстве, но обмен фрагментами между ними блокируется целым рядом клеточных механизмов. Обмен генетическим материалом между хромосомами может происходить только при делении клеток, когда на некоторых стадиях клеточного цикла гомологичные (сходные) хромосомы оказываются рядом друг с другом. При мейозе происходит кроссинговер — обмен генетическим материалом между гомологичными хромосомами. Разрывы и соединения обменивающихся хромосом происходят по специальным точкам, причем хромосомы строго «выровнены» относительно друг друга.
Но могут происходить и ошибки: смещение рекомбинирующих хромосом относительно друг друга или рекомбинация негомологичных хромосом. В обоих случаях перегруппировка происходит по отдельным гомологичным последовательностям, в роли которых могут выступать и копии Alu. Такая несбалансированная рекомбинация может привести к делеции гена на одной хромосоме, появлению дополнительной его копии на другой, к инверсии гена. Если ген на хромосоме удвоился, то через некоторое число поколений его копия может измениться и, с некоторой вероятностью, приобрести новую функцию, или, наоборот, деградировать и превратиться в «псевдоген» — ген, который свою кодирующую функцию потерял. Поскольку такие перегруппировки осуществляются случайно, вовсе необязательно, что мутации окажутся полезными. Наоборот, довольно часто они приводят к генетическим заболеваниям [11]. Как после перетасовки карт в игре: у вас на руках может оказаться как выигрышная комбинация, так и нет.
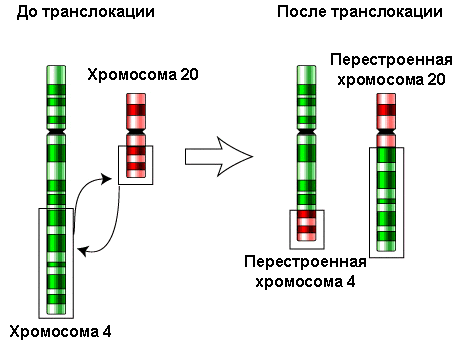
Рисунок 7. Схема транслокации
Если происходит рекомбинация негомологичных хромосом — транслокация (рис. 7), — то путём такой «перетасовки» могут возникать хромосомы новые. Транслокации могут возникать как при ошибках в мейозе, так и при проблемах с расхождением хромосом в митозе — делением клеток соматических.
Транслокации, с одной стороны, очень важны для эволюции и образования новых видов, а с другой — опасны, поскольку могут нарушать работу клеточного генетического аппарата и приводить к наследственным или онкологическим заболеваниям. Наследственные заболевания в этом случае будут следствием транслокаций во время мейоза. Если сбой осуществляется в митозе, то мутация проявит себя только в соматическом потомстве клеток, в которых она произошла. В таком случае мутация может привести к раковому заболеванию [12], [13].
Alu может вызывать болезни и без рекомбинации, напрямую вставляясь в последовательность ДНК (ген), кодирующую белок или регуляторную РНК , нарушая, таким образом, его — гена — работу. Например, вставка копии Alu в кодирующую часть гена рецептора фактора роста фибробластов — причина синдрома Аперта, а встраивание в кодирующую часть гена фактора свёртывания крови VIII — причина гемофилии А [13], [22].
От гена к РНК: влияние Alu
Механизмов и молекулярных путей, которыми Alu может изменять экспрессию генов — «перевод» кода ДНК в функциональный белок или действующую РНК, — довольно много. Я затрону только некоторые из них и постараюсь проиллюстрировать, насколько разнообразным может быть влияние этого элемента.
Копии элементов SINE, в том числе, и Alu, могут встраиваться непосредственно перед геном или на некотором расстоянии от гена. При встраивании непосредственно перед кодирующей последовательностью гена элемент может работать промотором — областью, с которой связывается РНК-полимераза перед началом транскрипции. Копии элемента на некотором расстоянии от гена могут выступать в роли энхансеров или сайленсеров: последовательностей, которые через взаимодействие с белками усиливают или ослабляют транскрипцию гена, соответственно [11].
Многие гены не являются непрерывными, а состоят из интронов и экзонов. После того, как с ДНК считывается матричная РНК, из неё вырезаются интроны, а экзоны сшиваются. С такой, уже зрелой, мРНК может транслироваться белок. Удаление интронов и сшивание экзонов называют сплайсингом: молекула разрезается в точках, которые находятся внутри определённых последовательностей в несколько нуклеотидов. Такие последовательности называют сайтами сплайсинга. За счёт использования разных сайтов из одной незрелой мРНК можно сделать много разных зрелых молекул («сплайс-изоформ») мРНК, которые кодируют разные изоформы белка. Этот процесс носит название альтернативного сплайсинга (рис. 8). Alu, встраиваясь в ген, может вносить в него дополнительные сайты сплайсинга, увеличивая разнообразие считываемых с гена изоформ мРНК. По некоторым подсчётам, больше 5% альтернативных экзонов — производные Alu [16].
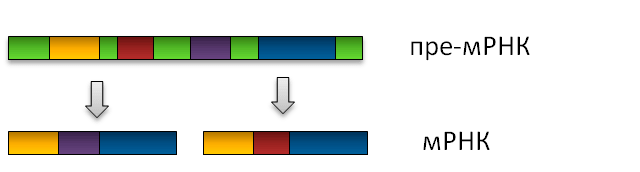
Рисунок 8. Схема альтернативного сплайсинга. Зелёным цветом обозначены интроны, другими цветами — разные экзоны.
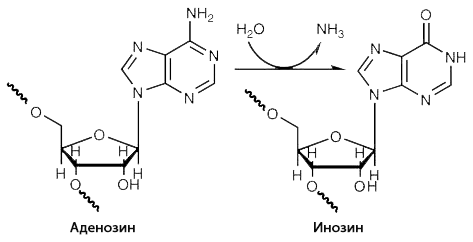
Рисунок 9. РНК-редактирование: замена аденозина (нуклеозида, содержащего аденин) на инозин (нуклеозид, содержащий гипоксантин).
Ещё один механизм, благодаря которому разных мРНК с одного гена становится больше, — это РНК-редактирование. Один из вариантов процесса — модификация рибонуклеотидных остатков мРНК с помощью ферментов. Например, замена азотистого основания аденина (через удаление аминогруппы и замещение её другой химической группой) на другое основание — гипоксантин (рис. 9). Последнее может происходить в двуцепочечных участках мРНК с последовательностью Alu. При трансляции аденозин (нуклеозид, содержащий аденин) распознаётся как гуанозин (нуклеозид, содержащий гуанин), что может изменить аминокислоту, считываемую с данного участка генетического кода и, как следствие, изменить структуру получившегося белка. В одном Alu несколько десятков потенциальных мишеней для замены. Тысячи миллионов разных комбинаций замен становятся возможными! Конечно, далеко не все они реализуются на практике. Но учитывая то, сколько генов содержат последовательность Alu, а некоторые из них — и не одну, можно предположить, что механизм редактирования гораздо мощнее альтернативного сплайсинга [17].
Действовать Alu может не только в форме ДНК-последовательности в геноме, но и в форме РНК в цитоплазме клетки. Некодирующая РНК, комплементарная Alu (антисмысловая РНК), может связываться с мРНК, в которой есть последовательность этого элемента, и тогда две РНК формируют двуцепочечную структуру. Структура узнаётся белком STAU1, который инициирует ускоренный распад мРНК [18].
Метильный запрет
Как я уже упоминала вначале, Alu составляет 11% нашего генома. И хоть к транспозиции способны самые молодые копии, если бы они постоянно копировались и вставлялись в случайные места нашей ДНК, то это был бы хаос: большая часть наших генов быстро перестала бы работать.
К счастью, законом клетки беспорядочная ретротранспозиция запрещена. Наряду с другими мобильными элементами и некоторыми другими последовательностями, ДНК Alu в клетках метилирована. К цитозинам присоединены химические группы, называемые метильными, и к транскрипции метилированные участки генома не способны. Это основной механизм подавления мобильных элементов в клетке, и с недостаточным метилированием ДНК связаны некоторые заболевания — в том числе, и онкологические [15].
Интересно, что на определённых стадиях развития половых клеток происходит повальное деметилирование генов: метильные метки «стираются» с нашей ДНК. На короткий период мобильные элементы, в том числе и Alu, свободно копируются и вставляются в геном. Скорее всего, это нужно для обеспечения плюрипотентности этих клеток [19].
Точка отсчёта
Когда-то давным-давно, ещё до разделения эволюционных ветвей, которые привели к приматам и грызунам, когда по земле бегали большие тираннозавры, а высоко в небе парили птеродактили, в верхнем Меловом периоде ген 7SL-РНК эволюционировал в предка Alu. Постепенно в геномах ветви приматов появился левый мономер Alu, а в геномах ветви грызунов — «двоюродный брат» Alu — B1 (он есть в геномах грызунов и сейчас). Вслед за левым мономером элемента у приматов появился правый, а 65 миллионов лет назад, на заре мезозойской эры произошло «рождение» Alu — как он есть сегодня. С этого момента Alu начал распространяться по геномам [7].
Как уже было сказано в начале, за прошедшие 65 миллионов лет Alu успел скопироваться и вставиться в нашу ДНК больше миллиона раз. Поскольку во вставившихся последовательностях периодически происходят мутации, эти последовательности изменяются независимо в разных геномах. Зная это, учёные, которые занимаются молекулярной систематикой, по последовательностям Alu в геномах разных приматов строят филогенетические деревья (рис. 10).
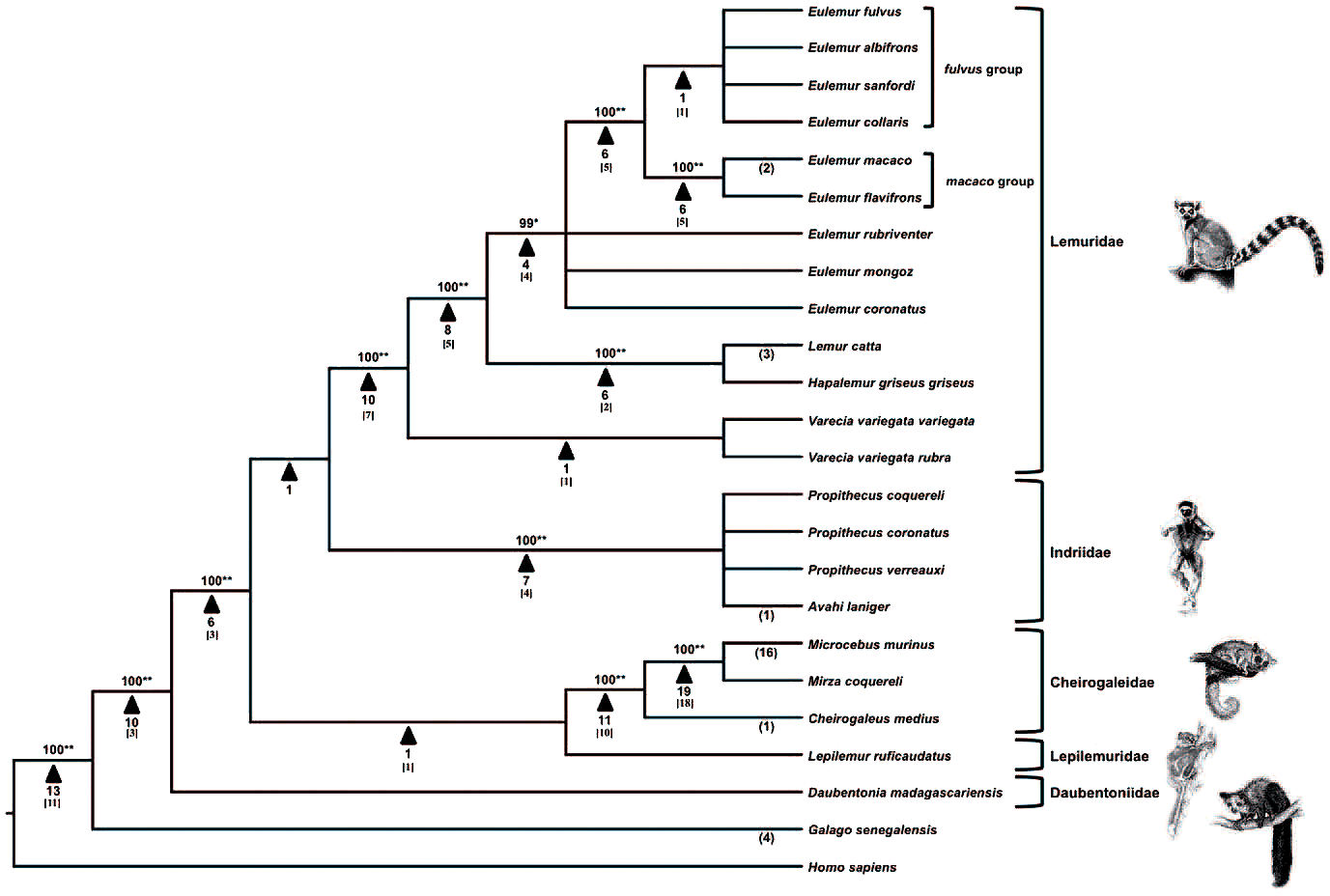
Рисунок 10. Филогенетическое древо лемуров, построенное на основании анализа 138 вставок Alu
За всё время нашей эволюции активность Alu сильно снизилась. На ранних стадиях эволюции приматов в среднем на одно рождение приходилась одна новая копия Alu. Сейчас новая вставка элемента осуществляется примерно раз в 20 поколений [9]. Почему так произошло? Во-первых, возможно, чисто по техническим причинам: Alu вставляется в геном только по определённым сайтам — местам в ДНК с определённой последовательностью. Эта последовательность — несколько тиминов: за них РНК Alu комплементарно «цепляется» поли(А)-богатой областью (см. выше механизм встраивания в геном). По мере того, как Alu копировался в нашем геноме, таких сайтов становилось всё меньше, и вероятность встраивания уменьшалась. Во-вторых, естественный отбор мог действовать на уменьшение скорости ретропозиции: как-никак, это довольно опасно (см. выше про мутации), хоть и вносит в наш геном структурное разнообразие и с каждой новой копией расширяет наше геномное пространство [7], [11].
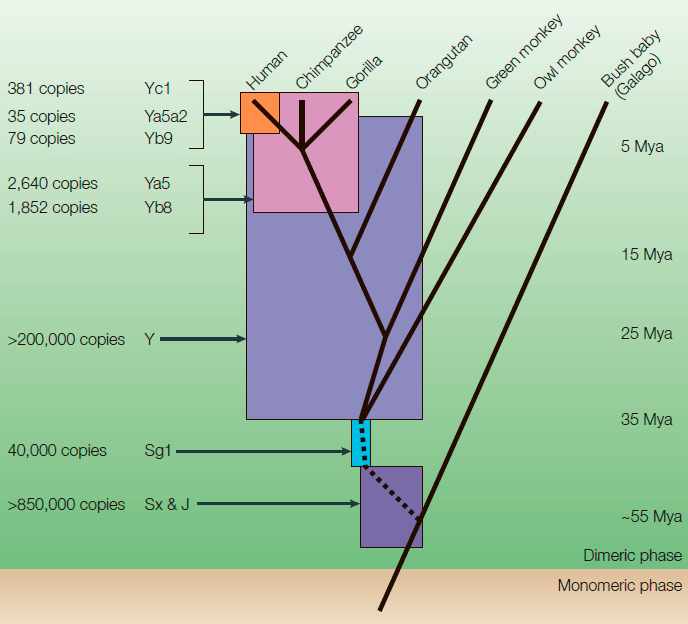
Рисунок 11. Распространение элементов Alu среди приматов. Monomeric phase — время, когда Alu существовал только в виде мономера. Dimeric phase — время существования полноценного, димерного Alu. Распространение подсемейств Alu (Yc1, Ya5a2, Yb9, Yb8, Y, Sg1, Sx и J) наложено на древо эволюции приматов. Слева указано примерное количество копий Alu в каждом подсемействе. Mya — миллионов лет назад. Цветные квадраты соответствуют пикам активности Alu.
Как бы часто ни появлялись сейчас новые копии, за долгий период эволюции Alu успел вставиться во многие наши гены, так или иначе влияя на их функционирование через механизмы, которые мы обсуждали выше. Поразительно, но из 165 генов со вставкой Alu, общих для человека и шимпанзе, 115 работают главным образом в нейронах (рис. 12). Кроме того, было показано, что Alu вместе со своим партнёром LINE-1 значительно экспрессируются в мозге и играют роль в создании генетического разнообразия нервных клеток (см. «Разнообразия много не бывает: чем занимаются мобильные элементы генома в мозге» [14]). Таким образом, Alu сыграл и продолжает играть не последнюю роль в эволюции нашего мозга и развитии высших когнитивных функций [6], [21].
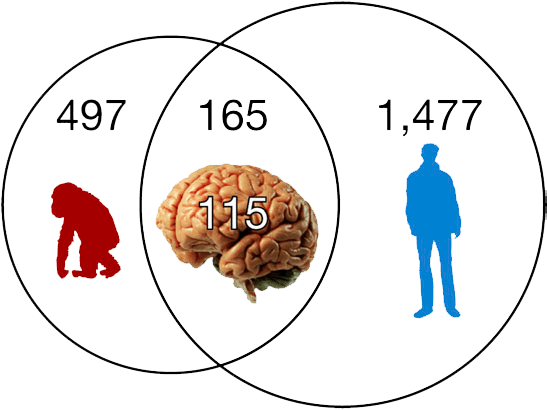
Рисунок 12. Копии Alu есть в 497 генах шимпанзе и в 1477 наших генах. 165 из этих генов общие, и 115 из этих 165 задействованы непосредственно в работе нашего мозга
История одной последовательности
Я сижу за столом и дописываю эту статью. Каждый миг в моих клетках считываются огромные количества матричной РНК с регулируемых последовательностью Alu генов. Каждый миг из фрагментов незрелых РНК по Alu-сайтам комбинируются зрелые молекулы, редактируются, транслируются, и вот уже новые белки синтезировались и готовы к работе. Миллионы молекул взаимодействуют между собой в единой невообразимой паутине.
Вот транскрипт Alu переманил транскриптазу LINE-1 и направился с ней в ядро. В какую часть ДНК он встроится? Повлияет ли на экспрессию гена, или через ряд поколений образует новый ген?
Конечно, не каждая последовательность нашей ДНК настолько многочисленна и влиятельна. Но о скольких можно рассказать изученную часть их истории, и сколько молекулярных историй ещё предстоит изучить...
Литература
- Eric S. Lander, Lauren M. Linton, Bruce Birren, Chad Nusbaum, Michael C. Zody, et. al.. (2001). Initial sequencing and analysis of the human genome. Nature. 409, 860-921;
- M. S. Comeaux, A. M. Roy-Engel, D. J. Hedges, P. L. Deininger. (2009). Diverse cis factors controlling Alu retrotransposition: What causes Alu elements to die?. Genome Research. 19, 545-555;
- E. A. Bennett, H. Keller, R. E. Mills, S. Schmidt, J. V. Moran, et. al.. (2008). Active Alu retrotransposons in the human genome. Genome Research. 18, 1875-1883;
- Graeme I. Bell, Raymond Pictet, William J. Rutter. (1980). Analysis of the regions flanking the human insulin gene and sequence of an Alu family member. Nucl Acids Res. 8, 4091-4110;
- Dmitri A. Kramerov, Nikita S. Vassetzky. (2011). SINEs. WIREs RNA. 2, 772-786;
- Eviatar Nevo. (2011). Evolution Under Environmental Stress at Macro- and Microscales. Genome Biology and Evolution. 3, 1039-1052;
- Mark A. Batzer, Prescott L. Deininger. (2002). Alu repeats and human genomic diversity. Nat Rev Genet. 3, 370-379;
- Adam T. McLain, Thomas J. Meyer, Christopher Faulk, Scott W. Herke, J. Michael Oldenburg, et. al.. (2012). An Alu-Based Phylogeny of Lemurs (Infraorder: Lemuriformes). PLoS ONE. 7, e44035;
- Richard Cordaux, Dale J. Hedges, Scott W. Herke, Mark A. Batzer. (2006). Estimating the retrotransposition rate of human Alu elements. Gene. 373, 134-137;
- N. Paz-Yaacov, E. Y. Levanon, E. Nevo, Y. Kinar, A. Harmelin, et. al.. (2010). Adenosine-to-inosine RNA editing shapes transcriptome diversity in primates. Proceedings of the National Academy of Sciences. 107, 12174-12179;
- J. Schmitz. (2012). SINEs as Driving Forces in Genome Evolution. Genome Dynamics. 92-107;
- Elena Kolomietz, M. Stephen Meyn, Ajay Pandita, Jeremy A. Squire. (2002). The role ofAlu repeat clusters as mediators of recurrent chromosomal aberrations in tumors. Genes Chromosom. Cancer. 35, 97-112;
- P.A. Callinan, M.A. Batzer. (2006). Retrotransposable Elements and Human Disease. Genome and Disease. 104-115;
- Разнообразия много не бывает: чем занимаются мобильные элементы генома в мозге;
- Yoshihisa Watanabe, Masato Maekawa. (2010). Methylation of DNA in Cancer. Advances in Clinical Chemistry. 145-167;
- R. Sorek. (2002). Alu-Containing Exons are Alternatively Spliced. Genome Research. 12, 1060-1067;
- Michal Barak, Erez Y. Levanon, Eli Eisenberg, Nurit Paz, Gideon Rechavi, et. al.. (2009). Evidence for large diversity in the human transcriptome created by Alu RNA editing. Nucleic Acids Research. 37, 6905-6915;
- Chenguang Gong, Lynne E. Maquat. (2011). lncRNAs transactivate STAU1-mediated mRNA decay by duplexing with 3′ UTRs via Alu elements. Nature. 470, 284-288;
- W. Reik. (2001). Epigenetic Reprogramming in Mammalian Development. Science. 293, 1089-1093;
- A. M. Kallman. (2003). ADAR2 A->I editing: site selectivity and editing efficiency are separate events. Nucleic Acids Research. 31, 4874-4881;
- J. Kenneth Baillie, Mark W. Barnett, Kyle R. Upton, Daniel J. Gerhardt, Todd A. Richmond, et. al.. (2011). Somatic retrotransposition alters the genetic landscape of the human brain. Nature. 479, 534-537;
- Kathleen H. Burns, Jef D. Boeke. (2012). Human Transposon Tectonics. Cell. 149, 740-752;
- Junk DNA — repetitive sequences. Current genetics;
- Martin Muñoz-Lopez, Angela Macia, Marta Garcia-Cañadas, Richard M. Badge, Jose L. Garcia-Perez. (2011). An epi [c] genetic battle. Mobile Genetic Elements. 1, 122-127;
- Marie Dewannieux, Cécile Esnault, Thierry Heidmann. (2003). LINE-mediated retrotransposition of marked Alu sequences. Nat Genet. 35, 41-48;
- Тайны «молекулярных паразитов», или Как путешествовать по геному;
- Robert Che-An YANG, Andre VOORDE, Walter FIERS. (1976). Specific Cleavage and Physical Mapping of Simian-Virus-40 DNA by the Restriction Endonuclease of Arthrobacter luteus. Eur J Biochem. 61, 119-138.