ДНК-оригами: путь от гравюры до нанороботов длиной в 30 лет
21 апреля 2014
ДНК-оригами: путь от гравюры до нанороботов длиной в 30 лет
- 4593
- 7
- 5
Журавлик из бумаги — один из самых известных образов в оригами
-
Автор
-
Редакторы
Сложить журавлика из бумаги — легко! Сложить журавлика из молекулы ДНК... тоже легко! Немного усидчивости и мастерства позволяют своими руками создавать из бумаги настоящие произведения искусства. Молекулы ДНК, в свою очередь, не требуют специальных навыков и собираются в красивые структуры на подобие оригами легко и непринужденно! Звучит как бред сумасшедшего, скажете вы. Отнюдь! Из этой статьи вы узнаете, как создать свою собственную фигурку оригами из ДНК, как похитить золото с помощью роботов, и кто победит в схватке между тараканом и ДНК-машиной.
«Биология — наука 21 века»
Эта работа публикуется в рамках конкурса научно-популярных статей, проведенного на конференции «Биология — наука 21 века» в 2014 году.
ДНК-оригами и связанные с этим ДНК-нанотехнологии сформировали в последнее десятилетие отдельное научное направление и получили стремительное развитие в работах нескольких научных групп по всему миру. В общем случае, за термином «ДНК-оригами» скрывается технология направленного конструировании молекул ДНК, способных к самосборке в заранее рассчитанные и смоделированные объекты. Такие конструкции могут быть как плоскими, так и объемными, довольно простыми и чрезвычайно замысловатыми. Все так же, как в японском искусстве складывания бумажного листа, только здесь вместо листа бумаги выступает нить ДНК!
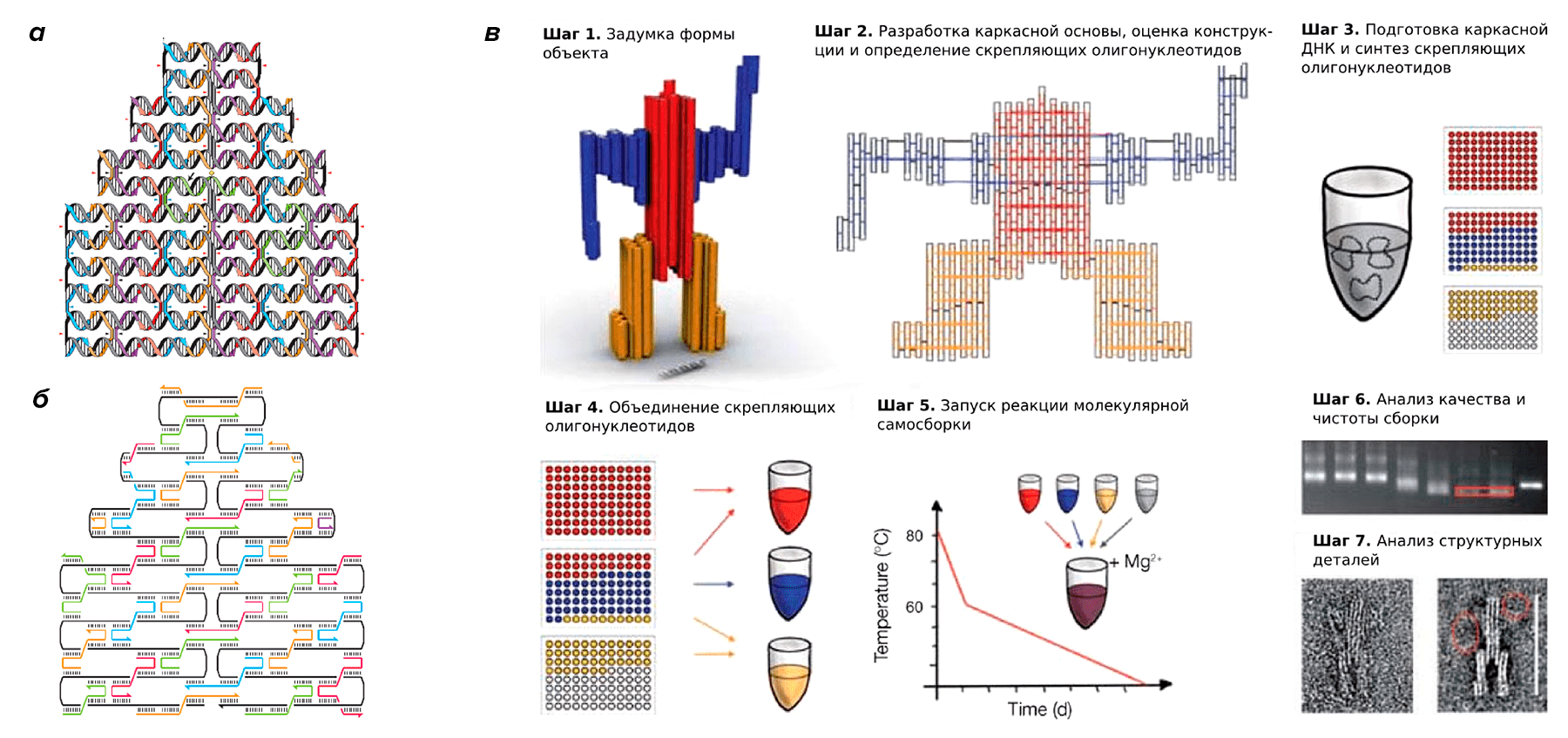
Как и многие научные открытия и разработки, это направление возникло, в некоторым смысле, случайно и неожиданно. Впервые о конструировании и использовании 3D-структур из ДНК всерьез заговорил американский ученый Нэд Симан (Ned Seeman) в начале 1980-х гг. Исследователь указывал на одну из главных сложностей метода рентгеновской кристаллографии (используемого тогда и по сей день для определения структуры белковых молекул), а именно необходимость подбора точных условий для получения «чистого» кристалла, по которым можно судить о структуре белка, и ставил своей целью разработку вспомогательной технологии фиксации белковых образцов (рис. 1). Для решения поставленных задач нужно было для начала разобраться с тем, как по собственному желанию и разумению собирать молекулы ДНК в необходимые конструкции.
Рисунок 1. а — Гравюра на дереве «Глубина», созданная Маурицем Корнелисом Эшером в 1955 году. Поговаривают, что, глядя на это произведение искусства в университетской столовой, Нэд Симан вдохновился на создание новой технологии, упрощающей кристаллизацию полипептидов и, следовательно, структурные исследования белков. С определением пространственной организации белков что-то не заладилось, но зато идеи Симана были подхвачены другими исследователями и привели к возникновению ДНК-оригами [1], [2]. б — Схема процесса кристаллизации белков, нарисованная Симаном. в — Идея ДНК-структур для правильной ориентации молекул в пространстве, изображенная Симаном.
«Глубина», Current Crystallization Protocol и DNA Cages Containing Oriented Guests; рисунки адаптированы
Поиск и описание различных свойств элементарных ДНК-конструктов длились несколько лет. В 1991 году Нэд Симан представил нанометровый куб, ребра которого представляли собой молекулы ДНК [3]. Спустя некоторое время, несмотря на скептическое отношение некоторых ученых, работа была признана выдающейся. За неё Нэд Симан был удостоен Фейнмановской премии по нанотехнологиям в 1995 году и навсегда вошел в историю науки как создатель первых ДНК-нанотехнологий.
Результаты Нэда Симана и его лаборатории послужили фундаментом для идей другого блистательного исследователя и, без преувеличения, крупной фигуры в области ДНК-оригами — американца Пола Ротемунда. В 2006 году он опубликовал статью в авторитетнейшем научном издании Nature [4], в которой был описан метод получения точных ДНК-структур с заданной формой, а также были представлены детальные результаты и анализ такого направленного конструирования. В отличие от других исследователей, ему удалось строить не решетки из отдельных молекул, а настоящие плоские фигуры шириной в несколько цепочек ДНК (рис. 2). Эта статья сразу разлетелась по научно-популярным журналам, новостям и блогам, ведь представленные структуры и изображения впечатляли даже неподготовленного с научной точки зрения читателя. Не удивительно, что иллюстрации эксперимента красовались на обложке выпуска журнала.

Рисунок 2. Некоторые структуры, построенные при помощи ДНК-оригами и представленные в статье Пола Ротемунда
В последующие годы вышло несколько десятков статей, посвященных технологии ДНК-оригами. Росло число полученных форм, размеров конструкций и их сложности. Некоторые из результатов были экспериментально опробованы на реальных биологических объектах для решения прикладных биотехнологических и медицинских задач.
Двумерное ДНК-оригами: от простого к сложному
Как же ученые складывают ДНК-оригами? Разберемся в деталях данного метода. Для начала нам потребуется длинная одноцепочечная молекула ДНК, которая будет играть роль каркаса и основы нашего будущего объекта. В первых экспериментах использовалась ДНК фага M13 длиной 7249 нуклеотидов, однако сейчас с усовершенствованием ряда технологий стали использовать другие последовательности ДНК. Затем нам понадобятся заранее синтезированные короткие комплементарные цепочки ДНК (также называемые «скрепляющими цепочками» или «ДНК-скрепками», обычно 30-40 нуклеотидов в длину), последовательность которых необходимо подобрать при помощи компьютерного моделирования и анализа структур. Теперь смешаем растворы с длинной молекулой и короткими «скрепками» и нагреем смесь до температуры 95 °C, чтобы случайные и ненужные молекулярные связи распались. В процессе остывания до комнатной температуры (эта процедура называется отжигом) молекулы ДНК сами соберутся вместе, образуя нужную нам структуру. Проще простого — они всё делают за нас сами!
В результате эксперимента получается раствор, содержащий желаемые ДНК-конструкции. В одной-единственной капле раствора скрываются миллиарды крошечных объектов, которые, в отличие от бумажных фигурок оригами, нельзя потрогать, повертеть в руках и рассмотреть. Для оценки результата нам потребуется прибор со сверхвысоким разрешением — атомно-силовой микроскоп (АСМ) [6] или электронный микроскоп. Ведь рассмотреть фигурки размером 50-100 нм так непросто!
Для создания плоских структур ДНК-оригами смежные двухцепочечные молекулы должны быть соединены друг с другом кроссовером — особым типом переплетения нитей ДНК. Такое переплетение «склеивает» соседние цепочки посредством уотсон-криковского комплементарного спаривания и не дает всей структуре рассыпаться. Учитывая большое количество скрепляющих цепочек, требуются алгоритмы для расчета вероятности их точной посадки на основную цепь. Если ДНК-скрепка сядет не в том месте, то это может повлечь за собой как дефект структуры, так и полную путаницу в посадке всех остальных скрепок. В худшем случае это может привести к тому, что структура не соберется вовсе. Все-таки самосборка молекул в идеально плоскую структуру — это не такая уж и легкая задача.
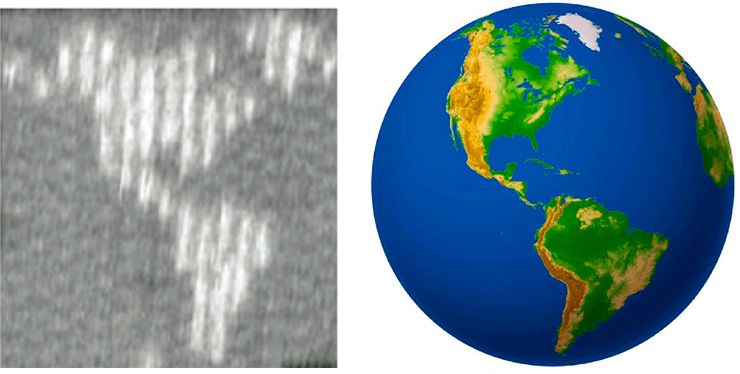
Рисунок 4. Точность собранного рисунка может быть довольно высока и находиться буквально на грани разрешения современных приборов. Можно добиться того, чтобы на ровном плоском «ДНК-полотне» в заранее предусмотренных местах будут выбиваться ДНК-шпильки. Это выглядит так, как если бы на кусочке ткани сделали рисунок узелками. Именно так была собрана карта западного полушария Земли, которую можно было увидеть исключительно при помощи АСМ.
Двумерные структуры на основе ДНК-оригами позволяют достичь не только большого многообразия форм — с помощью этой техники можно добиться невиданной до этого точности в размещении требуемых функциональных групп и молекул. Связанные с ДНК-скрепками молекулы могут быть размещены с точностью до нескольких нанометров и даже ангстрем (при условии правильной сборки)!
Если требуется собрать структуру побольше, нужно всего лишь соединить несколько длинных цепочек в одну составную конструкцию, как в конструкторе или крупных оригами-фигурах. На практике это можно осуществить так же, как было описано для одной единственной каркасной молекулы ДНК — нужно смешать все ингредиенты будущего объекта в одной пробирке, нагреть и ждать чуда, или собрать каждую деталь по отдельности, после чего объединить уже готовые элементы для окончательной сборки при менее интенсивном нагреве. В первом подходе нам приходится работать с достаточно большим количеством компонентов, ввиду чего увеличивается вероятность неправильной сборки молекул. При сборке деталей по отдельности необходимо провести несколько независимых экспериментов и совершить дополнительный шаг — повторный отжиг малых структур при нагреве до температуры 50 °C. При такой температуре детали еще не разваливаются на части, но уже более охотно связываются с друг с другом [7], [8].
Трехмерное ДНК-оригами
При определенных модификациях подход, который применяется для конструирования плоских структур, может быть обобщен до более сложного объемного случая. При конструировании 3D-структур можно, как и раньше, использовать кроссоверы, учитывая дополнительное третье измерение, и собирать все за один эксперимент, либо нужно начинать с собранных по отдельности плоских ДНК-объектов и лишь потом объединять их в конечную конструкцию. Выбор правильной последовательности действий в случае трехмерного ДНК-оригами чрезвычайно важен из-за значительно большего числа используемых молекул. Для особо сложных конструкций (особенно, при выборе первой стратегии сборки за один эксперимент) самосборка объекта может занимать несколько дней.
Несмотря на все сложности, которые могут возникнуть, объемные конструкции так привлекательны для исследователей! Ведь объемные объекты, ввиду многообразия возможных форм, могут быть использованы в широком круге самых разных прикладных задач.
Так, используя несколько одинаковых квадратов, ученым удалось собрать полый куб [9] (правда, немного деформированный). Для устранения недостатков конструкции исследователи приделали к этому кубу крышку, которая запиралась на замок нанометровых размеров. Открытием крышки можно было управлять при помощи изменения конформации замка за счет спаривания с небольшими «ДНК-ключами» (рис. 5). Убедиться в том, что куб надежно закрывается на замок и открывается лишь определенным ключом, помог эффект FRET [17]. При этом данная конструкция стала одним из первых в своем роде контейнером для адресной доставки лекарств. Пока только в перспективе, конечно же.
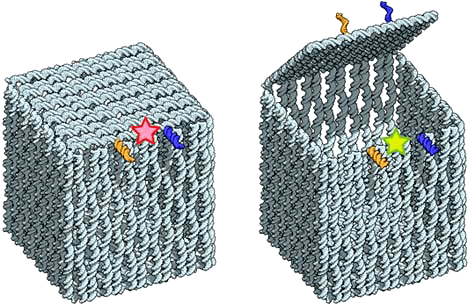
Рисунок 5. ДНК-«коробочка» с открывающейся крышкой и молекулярным «замком». Получена в Датском центре ДНК-нанотехнологий в 2009 году. Предполагается, что в будущем такая конструкция будет использоваться для адресной доставки лекарств к определенным клеткам, где она будет открыта при помощи молекулярного «ключа».
Следующим этапом конструирования 3D объектов стала сборка строительных блоков, которые в дальнейшем скреплялись между собой, как детали конструктора (подробнее об этом можно прочесть в [10]).
Применение ДНК-оригами: ДНК-чипы, молекулярные машины и нанороботы
Пока мы затрагивали в основном процесс конструирования и сборки ДНК-оригами, и практически никак не упоминали о том, зачем все это нужно. И действительно, ведь ДНК-структуры разрабатываются не для того чтобы ими любоваться и получать эстетическое удовольствие! Современные ДНК-нанотехнологии направлены на решение нескольких прикладных задач, связанных с медициной, биотехнологией и программированием.
ДНК-конструкции могут нести на поверхности несколько строго ориентированных функциональных групп, специфически связывающих ту или иную молекулу, и, таким образом, регистрировать их присутствие. В самых простых случаях синтезируется специальная ДНК-скрепка с последовательностью, комплементарной молекуле РНК или ДНК в растворе. При использовании АСМ мы можем зафиксировать даже акт единичного связывания такой молекулы, так как при возникновении связи между структурой ДНК-оригами и целевой молекулой, последняя начинает сильно «выпирать» [11]. Это сразу бросается в глаза при анализе изображения.
Использование лигандов или аптамеров позволяет создавать настоящие сенсорные чипы. С их помощью можно регистрировать наличие не только одноцепочечных молекул нуклеиновых кислот, но и интересующих нас молекул белков и других соединений. При удачном стечении обстоятельств, речь может идти об обнаружении даже единичных молекул.
Способность к регистрации можно улучшить, фиксируя структуры ДНК-оригами на поверхности подложки. Подложка при этом заранее размечается методами литографии и травления, после чего обрабатывается специальными химическими соединениями. При правильной подготовке «плацдарма» для посадки, ДНК-структуры выстраиваются точно по порядку в интересующих нас местах и даже в нужной ориентации [12]. В совокупности, последовательность таких операций дает довольно точное размещение на подложке конструкций ДНК-оригами, которые, в свою очередь, служат подложкой для еще более точного размещения исследуемых молекул самой разной природы. Чип для широкого круга регистрируемых химических соединений готов к использованию!
Одним из интереснейших направлений ДНК-нанотехнологий является создание молекулярных машин, которые могли бы проводить разнообразные операции при минимальном участии человека. Например, Нэд Симан с коллегами собрал шагающую ДНК-машину с двумя ногами [13], [14]. На заранее сконструированной подложке (тоже собранной из ДНК) они разместили несколько других простых ДНК-машин, которые держали золотые наночастицы и могли их высвобождать при изменении конформации. Наш «молекулярный пешеход» ходил по подложке (по заранее известной дороге, которую тоже надо было собрать) и, когда оказывался вблизи носителей золота, отбирал у них золотую наночастицу! Заполучив немного золота, наш герой не успокаивался и шел за следующей порцией золотой добычи. По окончанию экспериментов жадный ДНК-пешеход должен был неплохо обогатиться!
Для того, чтобы продемонстрировать возможности программируемого перемещения молекулярных машин, другая группа исследователей собрала ДНК-«паука» с тремя ногами и одним хвостом [15]. (Странный, конечно, паук получился, но мы закроем на это глаза.) К ногам ДНК-«паука» были прикреплены функциональные молекулярные группы, которые позволяли перемещаться по специально созданной для этого трассе. Паук был привязан молекулой-замком за хвост в самом начале своего пути; затем, после связывания молекулы-замка с молекулой-ключом, его отпускали на свободу, и он убегал исследовать мир! Передвижение ДНК-паука было заснято в реальном времени при помощи микроскопии полного внутреннего отражения — его средняя скорость составила 3 нм/мин. Видимо, он не убегал, а скорее с наслаждением прогуливался по своей дорожке.
Большие надежды возлагаются на ДНК-оригами и другие ДНК-нанотехнологии в связи с вопросом адресной доставки лекарственных средств нуждающимся клеткам. К сожалению, это направление не проработано так хорошо, как другие, и всё ещё находится на стадии интенсивных исследований. Остается верить, что открытия, связанные с ДНК-роботами, служащими на благо здравоохранения и человечества в целом, ещё впереди!
Вместо заключения
К настоящему моменту учеными из разных стран собран большой объем экспериментальных данных и описано большое число механизмов на основе ДНК-технологий, которые ещё только предстоит полностью осмыслить и оценить. Уже сейчас подробно описать каждую из полученных структур и её преимущества над другими не представляется возможным. Ведь если только 10 лет назад исследованиями такого рода занималось всего несколько лабораторий во всем мире, сейчас их количество исчисляется несколькими десятками. Относительно будущего данной области науки сказать определенно можно только одно — дальше будет еще интереснее! Чтобы убедить вас в этом, приведем заголовок статьи, которая вышла в апреле 2014 года — Universal computing by DNA origami robots in a living animal, в которой описано использование ДНК-нанороботов в живых тараканах [16]. Уверяем вас, удивительное будущее не за горами!
Литература
- Nadrian C. Seeman. (1982). Nucleic acid junctions and lattices. Journal of Theoretical Biology. 99, 237-247;
- Nadrian C. Seeman. (2003). DNA in a material world. Nature. 421, 427-431;
- Junghuei Chen, Nadrian C. Seeman. (1991). The electrophoretic properties of a DNA cube and its substructure catenanes. Electrophoresis. 12, 607-611;
- Paul W. K. Rothemund. (2006). Folding DNA to create nanoscale shapes and patterns. Nature. 440, 297-302;
- Carlos Ernesto Castro, Fabian Kilchherr, Do-Nyun Kim, Enrique Lin Shiao, Tobias Wauer, et. al.. (2011). A primer to scaffolded DNA origami. Nat Methods. 8, 221-229;
- Атомно-силовая микроскопия: увидеть, прикоснувшись;
- Arivazhagan Rajendran, Masayuki Endo, Yousuke Katsuda, Kumi Hidaka, Hiroshi Sugiyama. (2011). Programmed Two-Dimensional Self-Assembly of Multiple DNA Origami Jigsaw Pieces. ACS Nano. 5, 665-671;
- Zhao Zhao, Yan Liu, Hao Yan. (2011). Organizing DNA Origami Tiles into Larger Structures Using Preformed Scaffold Frames. Nano Lett.. 11, 2997-3002;
- Ebbe S. Andersen, Mingdong Dong, Morten M. Nielsen, Kasper Jahn, Ramesh Subramani, et. al.. (2009). Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459, 73-76;
- Элементы: «Наноструктуры из ДНК можно собирать по принципу конструктора „Лего“»;
- Y. Ke, S. Lindsay, Y. Chang, Y. Liu, H. Yan. (2008). Self-Assembled Water-Soluble Nucleic Acid Probe Tiles for Label-Free RNA Hybridization Assays. Science. 319, 180-183;
- Ryan J. Kershner, Luisa D. Bozano, Christine M. Micheel, Albert M. Hung, Ann R. Fornof, et. al.. (2009). Placement and orientation of individual DNA shapes on lithographically patterned surfaces. Nature Nanotech. 4, 557-561;
- Tosan Omabegho, Ruojie Sha, Nadrian C. Seeman. (2009). A Bipedal DNA Brownian Motor with Coordinated Legs. Science. 324, 67-71;
- Hongzhou Gu, Jie Chao, Shou-Jun Xiao, Nadrian C. Seeman. (2010). A proximity-based programmable DNA nanoscale assembly line. Nature. 465, 202-205;
- Kyle Lund, Anthony J. Manzo, Nadine Dabby, Nicole Michelotti, Alexander Johnson-Buck, et. al.. (2010). Molecular robots guided by prescriptive landscapes. Nature. 465, 206-210;
- Yaniv Amir, Eldad Ben-Ishay, Daniel Levner, Shmulik Ittah, Almogit Abu-Horowitz, Ido Bachelet. (2014). Universal computing by DNA origami robots in a living animal. Nature Nanotech. 9, 353-357;
- Рулетка для спектроскописта.