Биоразнообразие и молекулярная биология помогут друг другу?
08 сентября 2014
Биоразнообразие и молекулярная биология помогут друг другу?
- 900
- 3
- 1
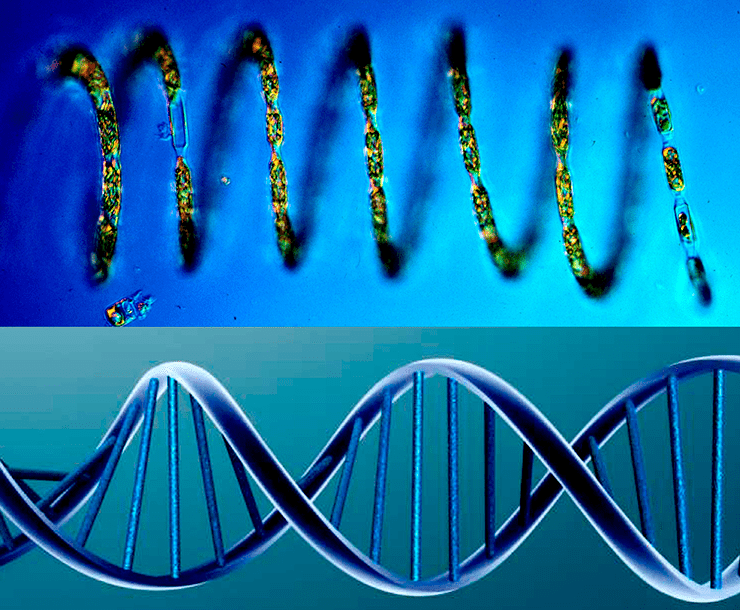
Что скрывается за этой спиралью? Природа любит демонстрировать единство и, одновременно, остается очень разной. Вверху — широко распространенная диатомовая водоросль Eucampia zodiacus; внизу — схематическое изображение молекулы ДНК.
-
Автор
-
Редакторы
Статья на конкурс «био/мол/текст»: Природа многообразна на самых разных уровнях: от отдельных внутриклеточных процессов до целых организмов. Животные и растения составляют лишь две небольшие группы в царстве эукариот, но на все другие группы человек редко обращает внимание, так как они крайне малы и относятся к так называемому микромиру. В этом году произошло событие, которое открывает перед исследователями возможность заглянуть гораздо глубже в мир простейших (одноклеточных эукариот). Объявлено о расшифровке более 650 транскриптомов этих организмов, и все данные уже находятся в свободном онлайн доступе. Транскриптомы дают информацию о всех генах, которые в данном организме в данный момент работают. Таким образом можно говорить о начале качественно нового уровня в понимании редко доступного глазу, но ключевого элемента биосферы.
Конкурс «био/мол/текст»-2014
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2014 в номинации «Лучшее новостное сообщение».
Главный спонсор конкурса — дальновидная компания Генотек.
Конкурс поддержан ОАО «РВК».
Спонсором номинации «Биоинформатика» является Институт биоинформатики.
Спонсором приза зрительских симпатий выступила фирма Helicon.
Свой приз также вручает Фонд поддержки передовых биотехнологий.
Существует несколько вариантов научных новостей. Это может быть сообщение об открытии чего-то, ранее совершенно неизвестного. А может быть рассказ о публикации труда, в котором наконец-то детально разобран некий известный процесс. Но есть еще и третий вариант, который я назвала бы «приглашение в будущее»: когда сделан первый шаг или придуман новый метод, или поднято со дна морского чудище невиданное и непонятное. Само по себе это еще не описание нового биологического закона, но это то, без чего не сделать новых исследований. В данной заметке я хочу рассказать именно о таком событии, которое открывает новые возможности. Возможности лучше понять окружающую нас природу и найти новые биохимические головоломки, решение которых — кто знает? — может нам однажды очень пригодиться.
Не бактерии, но и не многоклеточные
Мы с вами — это колонии клеток. Когда же клетка живет сама по себе, её называют одноклеточным организмом. При этом когда она имеет в целом такое же строение, что и наши клетки (в отличие от бактерий, которые устроены намного проще), то её называют простейшим (или микроэукариотом) и изучают в школе как амебу. Или инфузорию. Или как диатомовую водоросль. Но, конечно, вариантов простейших гораздо больше (рис. 1). И далеко не все они друг другу родственники — некоторые, например, ближе к человеку, чем к другому одноклеточному [1]. Они много чего умеют и многое изобрели: как избегать того, как сделать это, как выжить. Все мы — живые организмы — придумываем, как же нам остаться живыми. И простейшие, будучи очень разнообразными, придумали массу способов. Только мы мало знаем об этом (гораздо меньше, чем о тех же бактериях). Изучен вот тот, который вызывает болезнь, и немного вон тот, из-за которого массово отравились рыбы, и, может, вот этот, так как он особенно хорошо размножается в лаборатории. За кадром остаются многие миры, которые изменяли и изменяют облик нашей планеты.

Рисунок 1. Некоторые свободноживущие микроэукариоты (сканирующая электронная микроскопия)
Colomban de Vargas и Noan Le Bescot с использованием фотографий Margaux Carmichael (CNRS/EPEP, биологическая станция Роскофф, Франция)
В последнее время, чтобы хотя бы оценить разнообразие простейших плюс в целом подглядеть, какие специальные функции может выполнять сообщество микроорганизмов, делают следующее: выделяют всю ДНК из, допустим, воды и всю её расшифровывают. Информации получается с избытком, правда, многое в ней непонятно, а главное, неизвестно, кому конкретно принадлежала та или иная молекула ДНК [2], [3]. В июне этого года в журнале PLoS Biology вышла статья, в которой использован другой подход для получения большого количества информации о микробах [4]. Многочисленные авторы статьи приняли участие в программе по расшифровке более 650 транскриптомов различных простейших. На самом деле, участников проекта гораздо больше, чем авторов данной статьи (посмотрите на список благодарностей в конце публикации). Работа проделана, действительно, большая. Стоит только сказать, что просто вырастить в лаборатории тот или иной вид одноклеточного — часто нетривиальная задача, требующая много месяцев труда, а ведь это лишь начальная стадия исследования.
Больше разных транскриптомов
Что же означает «расшифровали транскриптомы»? Геном — это вся ДНК, какая есть в организме, но не вся эта ДНК «дает жизнь» белкам. Часть ДНК выполняет регуляторную функцию, часть просто неактивна, и только часть — это именно те гены, на основе которых синтезируется РНК, а затем белок (рис. 2). Транскриптóмом и называют ту РНК, которая на данный момент синтезировалась с активных генов и несёт информацию о будущих белках [5]. Расшифровывая транскриптом, изучают не длинную цепь генома, а набор этих РНК. Соответственно, транскриптом меньше генома и анализировать его легче: не надо воссоздавать длинную цепь, в которой важно, где что находится; собираешь только отдельные куски, соответствующие тому или иному белку. Это особенно важно для простейших, у которых геномы могут быть очень запутанными (например, в них много повторов), содержать странные элементы и быть просто слишком большими (например, геномы многих динофлагеллят в десятки раз больше человеческого [6]!). В итоге исследователь имеет большой массив данных, который, хотя и не соответствует всей ДНК клетки, но отражает её функционально активную на данный момент часть. Вместе с тем, это не значит, что расшифровка транскриптома — это альтернатива расшифровке генома. Нет, это два разных подхода, и оба они нужны. Но пока и тот, и другой слабо использовались. До сих пор такая мега-расшифровка велась для единичных видов, разными учеными, с помощью разных подходов.
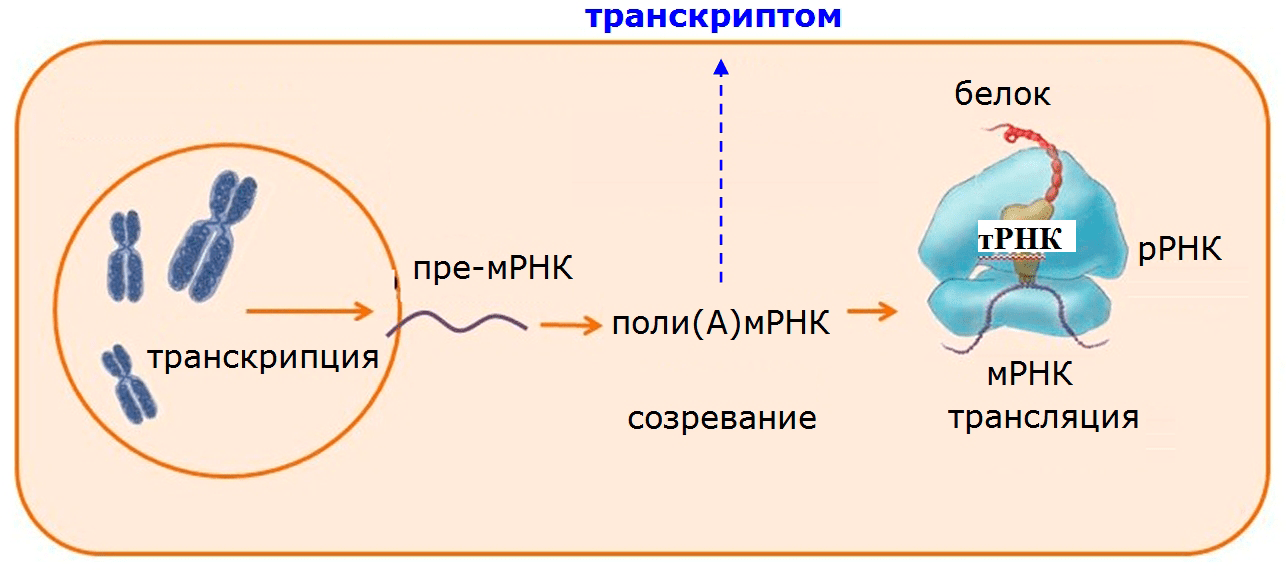
Рисунок 2. Схема генной экспрессии. Для расшифровки транскриптома из организма выделяют РНК. При этом у эукариот часто для того, чтобы не анализировать также и РНК, не несущую информацию о белках (например, рибосомную РНК), из всей РНК выделяют ту, которая имеет так называемый поли(А)-хвост, а значит является белок-кодирующей.
Статья Килинга с соавт. [4] принципиально иного вида:
- она охватывает действительно большое число видов, причем из самых разных таксономических групп (рис. 3), большинство из которых — не «модельные организмы», а, следовательно, плохо изучены. Среди них есть принадлежащие таким группам простейших, о геномах которых мы вообще ничего не знаем. С другой стороны, есть те, кто относится в целом к более изученным таксонам (инфузории, диатомовые), и значит в их случае исследование может идти «в глубину». В наборе присутствуют как очень далекие друг от друга организмы, так и штаммы одного вида.
- Большинство взятых на рассмотрение простейших более или менее растут в лаборатории, находятся в специальных коллекциях, а значит имеют хорошее морфологическое описание, то есть мы точно знаем, кто это и откуда.
- Все транскриптомы были не просто механически «прочитаны» — их аккуратно расшифровали, отсортировали, нашли соответствие белкам, — причем всё это по единой схеме сделала одна команда Национального Центра Геномных ресурсов США. Таким образом, во-первых, все данные прошли одну и ту же биоинформатическую обработку, и это облегчает их сравнение между собой. Во-вторых, уже аннотированные транскриптомы проще использовать большому кругу исследователей. Данные по 678 транскриптомам уже выложены в интернет в открытый доступ: camera.calit2.net/mmetsp.
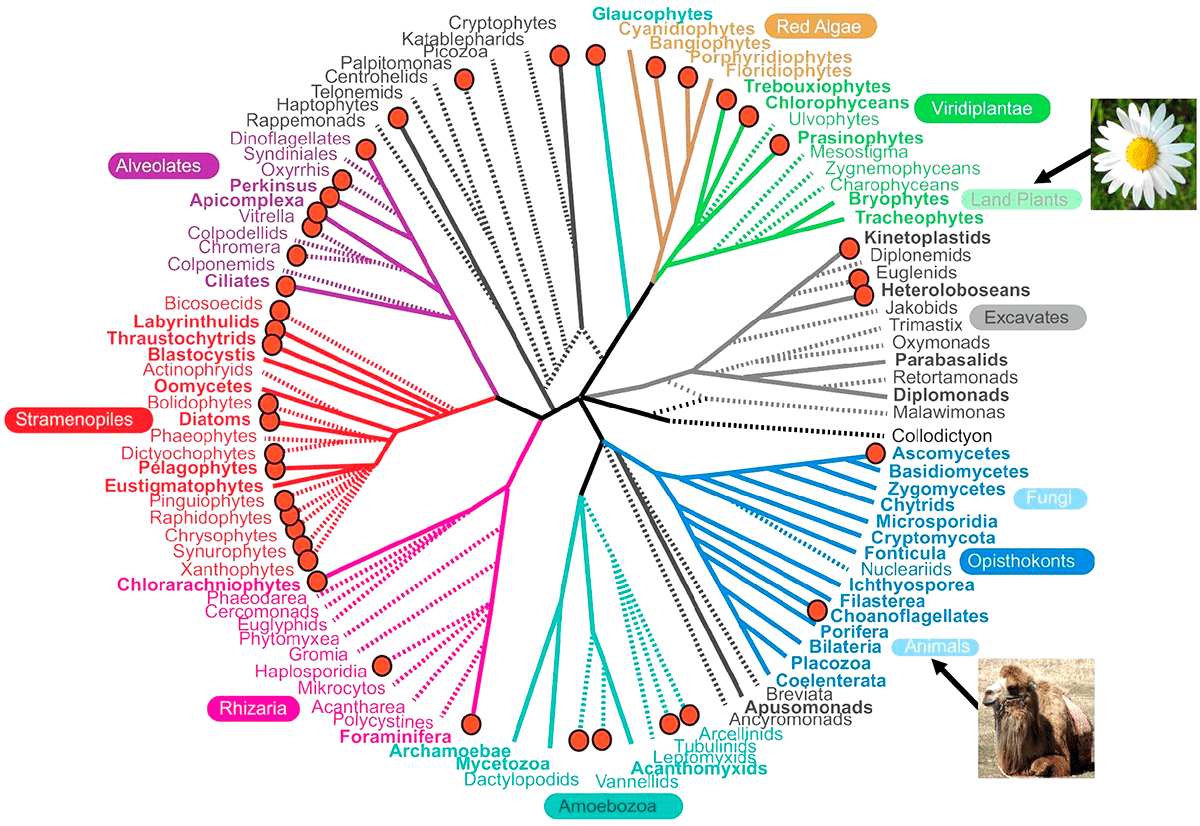
Рисунок 3. Схематическое изображение «дерева жизни», включающее основные группы эукариот. Животные и растения отмечены стрелками. Сплошная линия означает группы с уже расшифрованным геномом хотя бы одного представителя; пунктирная — группы, в которых пока нет ни одного расшифрованного генома. Красными точками указаны группы, для которых в ходе обсуждаемого проекта расшифрован по крайней мере один транскриптом.
Зачем это нужно и что дальше?
Статья в PLoS Biology — по сути лишь пресс релиз, сообщающий о факте получения данных и их первичного анализа. В целом, в программе участвовало порядка 80 ведущих исследователей из разных стран (в т.ч. и из СпбГУ). Набор видов, вошедших в статью, был отнюдь не случайным — каждый из ученых делает свой проект, со своими задачами и целью, под который и заказывал расшифровку транскриптомов. А это означает, что совсем скоро мы увидим ряд конкретных статей по каждому проекту внутри этого большого. Кто-то изучал один и тот же вид, но развивающийся при разных условиях, чтобы понять, какие гены активны в одной ситуации, а какие — в другой. Кто-то сравнивал близкородственные виды, чтобы узнать, что же отдалило вчерашних братьев друг от друга. Кто-то... мало ли что ещё, будем ждать публикаций! Главное же, это только начало и у него может быть — просто обязано быть! — интересное продолжение. Какие же возможности открывает полученная информация? Назову лишь несколько:
- Это база данных. Сейчас в биологии используются достаточно сложные математические методы, но так или иначе в основе многих лежит банальное сравнение. И надо иметь в наличии то, с чем сравнивать [7]. Например, это необходимо для того, о чем я упомянула в начале — для анализа общей ДНК из образцов воды. Теперь легче будет понять, кому принадлежит эта ДНК, и почему она оказалась в этой пробе.
- Изучение эволюционных взаимоотношений. Раньше эти выводы делали на основе сравнения определенных, но коротких фрагментов ДНК — выводы часто были правильны, но в целом ряде случаев информации было просто недостаточно, чтобы сказать что-то конкретное. Были проблемы и иного рода. Например, когда история одного изученного гена не совпадала с историей вида в целом [8]. Транскриптомы дают возможность сделать эволюционный анализ, взяв за основу под тысячу генов. Это особенно полезно, когда есть группа разных, но близких видов, и стоит вопрос — кто же там кого породил. Сейчас простейшие только вчерне «раскиданы» по Древу жизни, а заглянешь внутрь каждой группы — что за чехарда?! Наведение порядка периодически выявляет неожиданные, но с момента открытия такие простые и понятные линии развития и, соответственно, роли в экосистеме.
- Изучение функций тех или иных генов. На какие именно гены влияет данная окружающая среда? Какие гены «горизонтально» перенеслись от кого-то другого микроба к этому? И почему популяция микроорганизмов так светится (рис. 4)?

Рисунок 4. Развитие динофлагелляты Noctiluca scintillans в Гипслендских озерах (Австралия). Это морское простейшее распространено практически по всему миру и способно к биолюминесценции (днем такая вода чаще всего кажется красноватой). Безвредно для человека.
© Фил Харт
Закончу своё сообщение традиционными словами о такого рода новостях: это лишь первый шаг. И неправда, что только он труден — все последующие будут не легче. Понадобится много времени и внимания, чтобы интерпретировать полученные данные. И идти дальше. Ведь многие из изученных простейших — фотосинтетики (поскольку их легче культивировать), а гетеротрофы остались менее охваченными. Программа была посвящена морским видам, так что пресноводные и почвенные остались не у дел.
В наши души вселяет надежду тот факт, что внимания в этот раз удостоились малозаметные, на первый взгляд, организмы, а не более распространенные «фигуранты» новостных заголовков — такие как гены болезней, вирусы или сельскохозяйственные генно-модифицированные организмы. Для того, чтобы понимать биологию организмов, необходимо рассматривать картину под названием жизнь целиком, потому что каждый фрагмент мозаики важен, и никогда не знаешь, какой именно откроет столь нужное знание.
Литература
- Sina M. Adl, Alastair G. B. Simpson, Christopher E. Lane, Julius Lukeš, David Bass, et. al.. (2012). The Revised Classification of Eukaryotes. J. Eukaryot. Microbiol.. 59, 429-514;
- C. Lepere, I. Domaizon, D. Debroas. (2008). Unexpected Importance of Potential Parasites in the Composition of the Freshwater Small-Eukaryote Community. Applied and Environmental Microbiology. 74, 2940-2949;
- Virginia Edgcomb, William Orsi, John Bunge, Sunok Jeon, Richard Christen, et. al.. (2011). Protistan microbial observatory in the Cariaco Basin, Caribbean. I. Pyrosequencing vs Sanger insights into species richness. ISME J. 5, 1344-1356;
- Patrick J. Keeling, Fabien Burki, Heather M. Wilcox, Bassem Allam, Eric E. Allen, et. al.. (2014). The Marine Microbial Eukaryote Transcriptome Sequencing Project (MMETSP): Illuminating the Functional Diversity of Eukaryotic Life in the Oceans through Transcriptome Sequencing. PLoS Biol. 12, e1001889;
- «Омики» — эпоха большой биологии;
- Yubo Hou, Senjie Lin. (2009). Distinct Gene Number-Genome Size Relationships for Eukaryotes and Non-Eukaryotes: Gene Content Estimation for Dinoflagellate Genomes. PLoS ONE. 4, e6978;
- Alexandra Z Worden, Andrew E Allen. (2010). The voyage of the microbial eukaryote. Current Opinion in Microbiology. 13, 652-660;
- Gregory D.D Hurst, Francis M Jiggins. (2005). Problems with mitochondrial DNA as a marker in population, phylogeographic and phylogenetic studies: the effects of inherited symbionts. Proc. R. Soc. B. 272, 1525-1534.