
Поиск иголки в стоге сена за 10 минут — подсвети себе LAMPой
12 ноября 2014
Поиск иголки в стоге сена за 10 минут — подсвети себе LAMPой
- 7495
- 5
- 9
Многие молекулярно-биологические операции напоминают поиск иголки в стоге сена
-
Автор
-
Редакторы
Статья на конкурс «био/мол/текст»: Современные биологи в большинстве своём работают с генами. Ген — участок молекулы ДНК, кодирующий белок или РНК. Изучая активность гена и изменения в его работе, чаще всего пользуются методом полимеразной цепной реакции (ПЦР) и различными её модификациями. Метод позволяет найти ген и сделать множество его копий. Однако прибор и реактивы для проведения ПЦР дóроги, а время, необходимое на реакцию, составляет около двух часов. В данной статье описывается аналог полимеразной реакции — LAMP (loop-mediated isothermal amplification), позволяющий провести то же исследование в 10 раз быстрее, дешевле и, что крайне важно, более специфично. Также рассмотрены перспективы применения LAMP в фундаментальных и клинических исследованиях.
Конкурс «био/мол/текст»-2014
 Эта работа представлена в номинации «Лучший обзор» и заслужила приз зрительских симпатий конкурса «био/мол/текст»-2014.
Эта работа представлена в номинации «Лучший обзор» и заслужила приз зрительских симпатий конкурса «био/мол/текст»-2014.
Главный спонсор конкурса — дальновидная компания «Генотек».
Конкурс поддержан ОАО «РВК».
Спонсором номинации «Биоинформатика» является Институт биоинформатики.
Спонсором приза зрительских симпатий выступила фирма Helicon.
Свой приз также вручает Фонд поддержки передовых биотехнологий.
Как это здорово звучит: я занимаюсь генетикой, работаю с ДНК, РНК, получаю ГМО... Как это странно выглядит: я добавляю прозрачные растворы к прозрачным растворам, ставлю в сложный прибор, который показывает, сколько у меня ДНК, или из каких нуклеотидов она состоит. Конечно, целая молекула ДНК, особенно в связи с белками, поддерживающими её структуру (aka хромосома), ещё видна в микроскоп, но представьте себе один ген, один небольшой участок хромосомы. Чтобы работать с одним геном, его нужно сначала найти. Представьте, найти определённый участок, состоящий из 10000 букв (нуклеотидов) среди 3 миллиардов таких же букв! Разница в пять порядков. Это действительно сравнимо с поиском иголки в стоге сена.
Первым механизм такого поиска в 1983 году запатентовал Кэри Мюллис (Kary Mullis) [1]. Он предложил наработать множество копий исследуемой ДНК. Чтобы понять метод Мюллиса, широко известный как полимеразная цепная реакция (ПЦР) [11] и используемый уже более 30 лет практически в каждой лаборатории мира, обратимся к истокам. Как происходит увеличение количества ДНК в живом организме в естественных условиях.
Репликация ДНК
Природный механизм удвоения ДНК, который происходит перед делением каждой клетки, называется репликация (aka редупликация). Надо сказать, что до сих пор не все белки-участники данного процесса изучены, что говорит о его сложности. Главное, что происходит при репликации — сборка дочерней цепи ДНК на половине материнской. Напомню, что ДНК представляет собой двойную спираль, а информативная часть — азотистые основания — скрыта внутри спирали. Задача удвоения ДНК состоит в том, чтобы правильно собрать нуклеотиды в новую цепь и получить две идентичные молекулы. Собирает нуклеотиды фермент ДНК-полимераза. Но как ей добраться до азотистых оснований, скрытых в материнской цепи? Ответ прост — расплести цепи. Исполнение же трудно и требует участия целого ряда ферментов — гираз, лигаз, топоизомераз, белков, связывающих одиночную нить ДНК и препятствующих её повторному воссоединению (SSB белков). Тем не менее, ансамбль белков справляется с этой задачей (рис. 1). Итак, есть ДНК-полимераза, ей доступны азотистые основания матрицы, но что-то синтез не идёт...
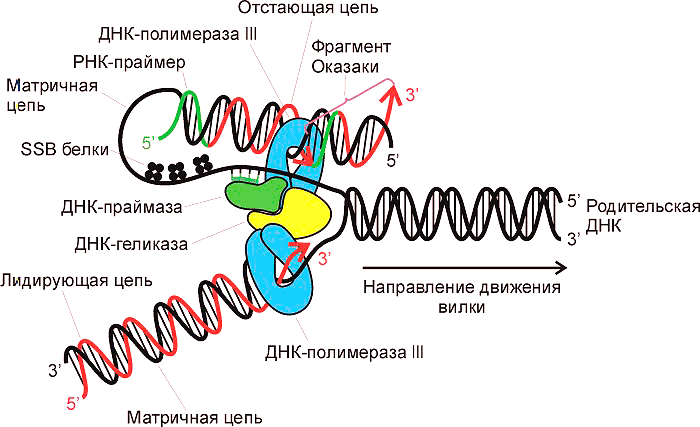
Рисунок 1. Репликация ДНК. Обратите внимание, как много белков участвует в этом процессе.
Дело в том, что полимераза — фермент привередливый, синтез ДНК «с нуля» и не пойдёт: необходима затравка (праймер) — свободная OH-группа предыдущего нуклеотида или какого-либо белка. Теперь, присоединяя нуклеотиды к праймеру, ДНК-полимераза может копировать материнскую цепь.
Амплификация ДНК (ПЦР)
Амплификация — это искусственная многократная реплицкация. Итак, процесс репликации ДНК в клетке даже на настоящий момент изучен не до конца. Что же придумал Мюллис 30 лет назад? Как он смог скоординировать, по меньшей мере, десяток ферментов в один момент, о существовании части которых он даже и не знал? Представьте себе, никак. Он взял всего один фермент — ДНК-полимеразу. Доступ к азотистым основаниям обеспечил не белками, а плавлением. («Плавление» ДНК — неферментативное расхождение цепей при нагревании до 92–95 °C.)
Мы помним, что нужен праймер, чтобы «привередливая» полимераза начала работать. Добавляем 18—30-нуклеотидный фрагмент ДНК, комплементарный границе интересующей нас последовательности. Однако больших успехов все равно не получаем. Реакция идёт, но мы не можем сказать, что удваивается именно наш ген, потому что будут получаться фрагменты разной длины, зависящие от времени работы полимеразы и её активности. Как же понять, какой из образующихся продуктов — искомый?
В ответе на этот вопрос и появилась гениальная идея Мюллиса — надо брать не один праймер, а два: прямой (F) и обратный (R). Тогда мы будем знать длину нашего фрагмента и быстро увеличим его количество. Также Мюллис предложил проводить реакцию не один раз, а циклом 30–45 повторов. Математический подсчёт показывает, что количество продукта будет расти как 2n (рис. 2), где n — число циклов. (А если бы использовался один праймер, то число фрагментов увеличивалось бы просто как n.) То есть через 30 циклов мы получаем в 109 (в миллиард) раз больше продукта, чем содержалось в исходном образце (а не в 30 раз больше, как было бы при использовании одного праймера). Почему же такая огромная разница? Дело в том, что при использовании двух праймеров каждый образующийся фрагмент служит матрицей в ходе следующих циклов. В случае одного праймера продукт будет ему не комплементарен и не будет использоваться как матрица в дальнейшем.
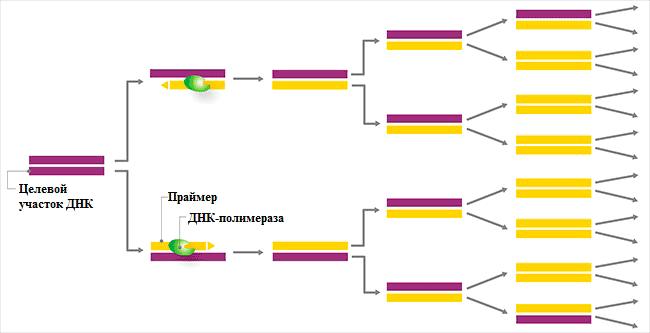
Рисунок 2. Схематическое изображение процесса полимеразной цепной реакции. Фиолетовым цветом показана исходная молекула ДНК, жёлтым — образующиеся в ходе реакции молекулы ДНК, идентичные исходной. Видно, что уже после третьего цикла число молекул увеличивается в 8 раз.
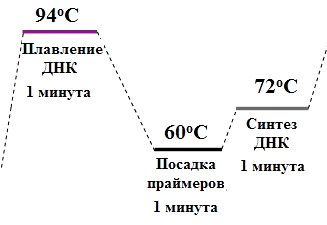
Рисунок 3. Цикл амплификации. Каждый цикл состоит из трёх ступеней: расхождение цепей ДНК, посадка праймеров и синтез новых цепей. Каждая ступень длится около 1 минуты. Таким образом, программа из 30 циклов будет длиться около 90 минут.
Сейчас проведение ПЦР занимает около 2 часов, благодаря использованию особой ДНК-полимеразы [2]. Дело в том, что каждый цикл ПЦР начинается с плавления ДНК на 95 °C (рис. 3), а «простая» полимераза — обычный белок — не выдерживала такой температуры и теряла функциональность, свернувшись, как белок варёного яйца. Поэтому по Мюллису после каждого цикла надо было добавлять новую порцию полимеразы. Рэнди Сайки (Randy Saiki) в 1986 году предложил использовать полимеразу из организма, живущего в термальных источниках — бактерии Thermophilus aquaticus (Taq-полимеразу), выдерживающую такой перегрев и работающую оптимально при 72 °C [3].
Как же теперь доказать, что, переливая прозрачные растворы, мы всё-таки не потеряли ДНК? Это можно увидеть глазами после 40-минутнтого разделения продуктов ПЦР в геле с помощью электрофореза. Гель — это, своего рода, молекулярное сито, сквозь которое под действием электрического тока идут молекулы: мелкие проходят быстро, крупные — медленно. Кроме того, фрагменты одинаковой длины концентрируются на одном уровне (а мы, следуя рекомендации Мюллиса, специально подбирали праймеры так, чтобы фрагменты были заданной длины). Всё это рассматривается в ультрафиолетовом (УФ) свете после добавления красителей, связывающихся с ДНК. В итоге мы увидим наш один ген или его фрагмент (или ничего не увидим, если искомой ДНК не было) (рис. 4).
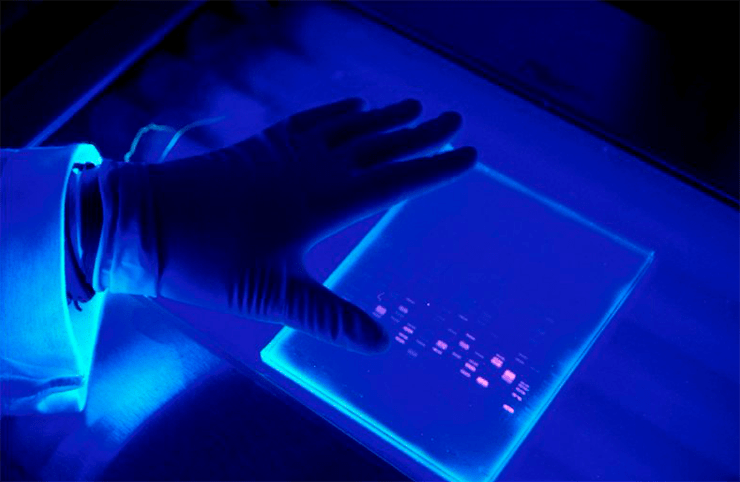
Рисунок 4. Результат разделения продуктов ПЦР в агарозном геле. В качестве красителя, связывающего ДНК, использован бромистый этидий. Съёмка сделана в УФ-свете.
Итак, Мюллис предложил увеличить число иголок в разы, чтобы их блеск без труда можно было различить в сене не интересующей нас ДНК. В настоящее время существует множество модификаций ПЦР, применяемых для самых различных целей. Однако у этой реакции есть существенный минус — потребность в специализированном лабораторном оборудовании: термоциклере-амплификаторе (приборе, способном быстро менять температуру раствора столько раз, сколько нам нужно), камере для электрофореза и т.д. А если этого нет? Можно ли всё провести при одинаковой температуре — например, на водяной бане или в простом термостате? Оказалось, что это возможно, и есть целый ряд методов изотермической амплификации. Слово изотермическая означает, что реакция идёт при постоянной температуре. Наиболее быстрым, специфичным, дешёвым и часто используемым методом является опосредованная образованием петель изотермическая амплификация (loop-mediated isothermal amplification, LAMP). Итак, прольём свет этих LAMP.
LAMP
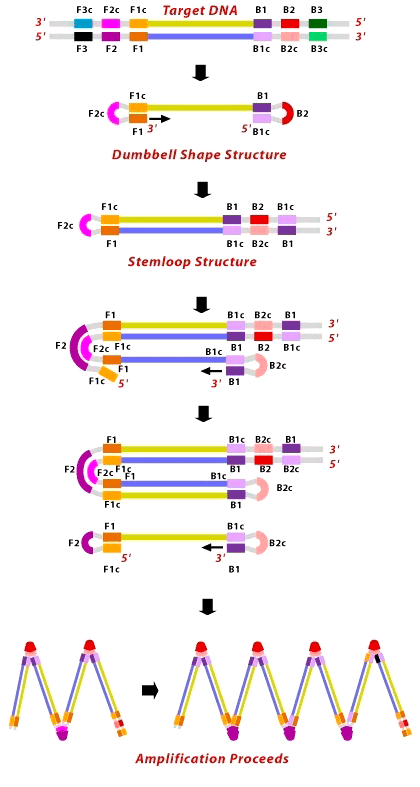
Рисунок 5. Схематическое изображение метода LAMP. Для понимания метода вспомните, что синтез идёт от 3′- к 5′-концу (маленькие горизонтальные стрелочки), а обратно — никогда.
Метод LAMP, как и ПЦР, использует термостабильную полимеразу. Примечательно, что, когда искали полимеразу для реакции Мюллиса, нашли две — Taq, которая используется и по сей день для ПЦР, и Bst (из Bacillus stearothermophilus). Было показано, что Bst-полимераза нестабильна и быстро выходит из строя при 95 °C, да и документация к Taq была лучше. Однако у Bst есть преимущество перед Taq: она вымещает вторую цепь ДНК сама, без участия ферментов или использования высоких температур. Использование Bst в методе LAMP позволило проводить реакцию на 30–40 минут быстрее, поскольку исчезает потребность в первом шаге цикла, но это ещё далеко не всё.
В названии метода, кроме постоянства температуры, упоминаются ещё некие петли. Давайте разберёмся. Метод LAMP, описанный японским учёным Цугунори Нотоми (Tsugunori Notomi) в 2000 году [4], подразумевал использование четырех праймеров (пары внутренних и пары внешних), узнающих шесть различных участков искомой ДНК. Внутренние праймеры подобраны таким образом, чтобы сформировать те самые петли на концах искомого фрагмента (рис. 5). Чтобы это удалось, к 5′-концу праймера F2 прикреплена вторая часть, комплементарная F1 части матрицы, — F1c, то есть фрагмент F1c—F2 — это и есть внешний праймер, длинной до 50 нуклеотидов (F от forward, «прямой»). В результате, как только новая цепь ДНК останется одна, из-за вымещающей активности Bst-полимеразы её конец тут же замкнётся в петлю. То же происходит и с праймерами, садящимися на противоположный конец матрицы (B от backward, «обратный»). В конечном итоге, появится одноцепочечный фрагмент ДНК с петлями с обеих сторон — гантелевидная структура (dumbbell structure). На этом завершается первый шаг LAMP.
После получения «гантельки» с одного из её концов (3′) полимераза продолжает синтез к другому (5′), образуя «рукоятку» (stemloop). Концы «рукоятки», оказавшись в одноцепочечном состоянии, замыкаются в петли, продолжая синтез. Так постепенно формируются загзагообразные продукты.
Внешние праймеры (F3 и B3) необходимы лишь в самом начале реакции для разделения двух материнских цепей.
Такой метод позволяет быстрее нарабатывать продукт и длится от часа до получаса. Однако уже через два года этим же учёным удалось усовершенствовать свой метод, добавив ещё одну пару праймеров — петлевые праймеры [5]. При их использовании, предположительно, идёт наработка продукта с петель в обе стороны, а не в одну, как в оригинальном методе. Как показывает график из этой работы (рис. 6), уже через 10–20 минут можно было регистрировать продукт в достаточном количестве.
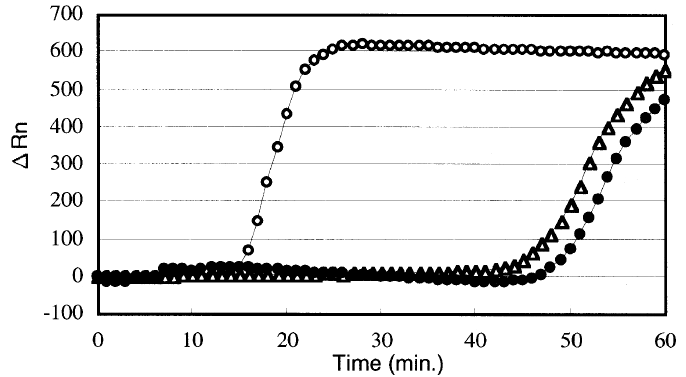
Рисунок 6. Сравнение эффективности LAMP с добавлением петлевых праймеров (пустые кружки) с классической LAMP (зачернённые кружки). По оси Х отложено время, по оси Y — изменение интенсивности флуоресценции (по нему можно судить об изменение количества ДНК в образце).
Да, как же регистрировать продукт в этой реакции? Представляете, просто взглянув на пробирку (не нужно ждать 40 минут), можно увидеть помутнение — это выпадает осадок пирофосфата магния. Ионы магния входят в состав реакционной среды, помогая работать Bst-полимеразе, а пирофосфат-ионы высвобождаются из нуклеотидов при синтезе цепи. Для верности можно также добавить краситель, связывающий ДНК и осветить УФ (рис. 7).
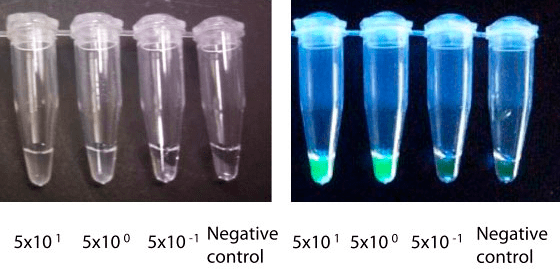
Рисунок 7. Визуализация результатов LAMP. Это одинаковые пробирки в естественном (слева) и УФ-свете (справа). В подписях указано исходное число молекул ДНК в пробе. Интересно, что даже 5 молекул ДНК в 25 мкл детектируются этим методом.
Сравнение ПЦР и LAMP
Таким образом, используя метод LAMP, можно ответить на вопрос «Присутствует ли интересующий нас ген в пробе?» за 5–20 минут [7], в то время как при использовании ПЦР ответа придётся ждать около двух часов, не считая времени на электрофоретическое разделение продуктов. Но, как известно, спешка хороша при ловле блох, а для научного исследования куда важнее точность и специфичность (то есть амплификация всегда и только той последовательности ДНК, которую мы задали, а не просто похожей).
Однако и тут LAMP опережает ПЦР. LAMP более специфичен, потому что ему необходимо узнать целых шесть участков в искомой молекуле ДНК, а ПЦР — только два (которые находятся непосредственно под праймерами). Но, что даже более важно, на LAMP не оказывает влияния присутствие биологических компонентов, зачастую не позволяющих провести ПЦР [8]. Исследуемый образец (например, слюну) можно заносить в реакционную смесь без очистки и искать гены вируса [6]. Благодаря этому свойству, LAMP используется в клинической практике для выявления патогенов человека и животных [9], определения пола коров до имплантации зародыша и даже нахождения метастазов прямо во время хирургической операции [10]. Метод прост в исполнении, дёшев (в отличие от других методов изотермической амплификации) и не требует ни сложной техники, ни обязательных биологических знаний.
| Критерий | ПЦР | LAMP |
|---|---|---|
| Необходимость плавления ДНК | + | − |
| Температура (°C) | 94; 55–60; 72 | 60–65 |
| Устойчивость к биологическим компонентам | − | + |
| Продолжительность (минут) | 90–120 | 5–20 |
| Регистрация продукта | электрофорез | невооружённым глазом |
Какой из методов лучше применять, зависит от эксперимента. Если исследователь постоянно меняет праймеры, условия или изучаемый ген, то ПЦР будет удобнее. Несмотря на то, что она занимает больше времени, на деле два часа едва хватает, чтобы приготовить смесь для следующей реакции и занести пробы в биохимический планшет. Если же речь идёт о предотвращении пандемии, и необходимо как можно быстрее определить присутствие известного вирусного агента, то, конечно, LAMP. Его можно применять, например, для мониторинга здоровья пассажиров в аэропортах стран, из которых возможно занесение вируса.
В заключение хочу отметить интересную тенденцию научных исследований, проявившуюся в эволюции методов амплификации нуклеиновых кислот. Как указано выше, во время создания ПЦР был выбор между двумя полимеразами: Taq и Bst. Мюллис выбрал Taq и построил свой метод на ней. А Нотоми спустя двадцать лет вернулся к Bst и придумал ещё более мощный метод LAMP.
Литература
- K. Mullis, F. Faloona, S. Scharf, R. Saiki, G. Horn, H. Erlich. (1986). Specific Enzymatic Amplification of DNA In Vitro: The Polymerase Chain Reaction. Cold Spring Harbor Symposia on Quantitative Biology. 51, 263-273;
- Важнейшие методы молекулярной биологии и генной инженерии;
- RK Saiki, DH Gelfand, S Stoffel, SJ Scharf, R Higuchi, et. al.. (1988). Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science. 239, 487-491;
- T. Notomi. (2000). Loop-mediated isothermal amplification of DNA. Nucleic Acids Research. 28, 63e-63;
- K. Nagamine, T. Hase, T. Notomi. (2002). Accelerated reaction by loop-mediated isothermal amplification using loop primers. Molecular and Cellular Probes. 16, 223-229;
- Kazuya Shirato, Takuya Yano, Syouhei Senba, Shigehiro Akachi, Takashi Kobayashi, et. al.. (2014). Detection of Middle East respiratory syndrome coronavirus using reverse transcription loop-mediated isothermal amplification (RT-LAMP). Virol J. 11;
- Nathan A. Tanner, Thomas C. Evans. (2014). Loop‐Mediated Isothermal Amplification for Detection of Nucleic Acids. Current Protocols in Molecular Biology. 105;
- Pooria Gill, Amir Ghaemi. (2008). Nucleic Acid Isothermal Amplification Technologies—A Review. Nucleosides, Nucleotides and Nucleic Acids. 27, 224-243;
- Kuldeep Dhama, K. Karthik, Sandip Chakrabort, Ruchi Tiwari, Sanjay Kapoor, et. al.. (2014). Loop-mediated Isothermal Amplification of DNA (LAMP): A New Diagnostic Tool Lights the World of Diagnosis of Animal and Human Pathogens: A Review. Pakistan J. of Biological Sciences. 17, 151-166;
- Shijun Fu, Guanggang Qu, Shijin Guo, Lin Ma, Na Zhang, et. al.. (2011). Applications of Loop-Mediated Isothermal DNA Amplification. Appl Biochem Biotechnol. 163, 845-850;
- 12 методов в картинках: полимеразная цепная реакция.







