Пространственное разделение транскрипции и трансляции в клетках бактерии Gemmata obscuriglobus
06 ноября 2014
Пространственное разделение транскрипции и трансляции в клетках бактерии Gemmata obscuriglobus
- 2307
- 0
- 3
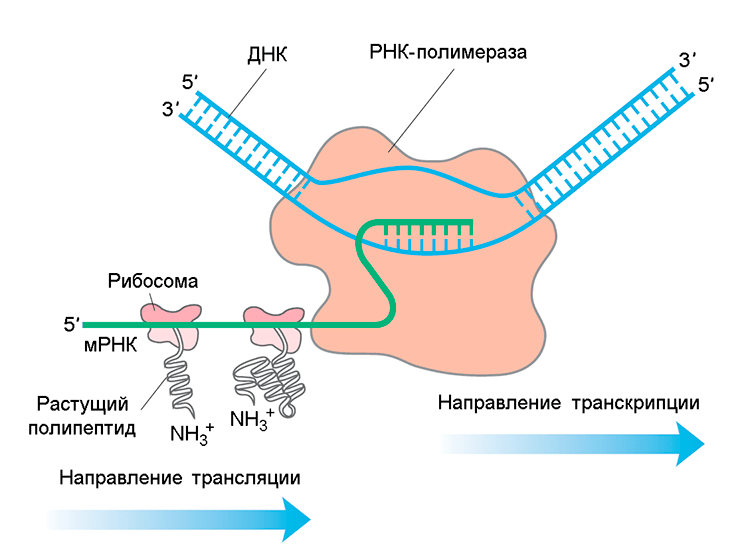
Сопряжение транскрипции и трансляции у прокариот
-
Автор
-
Редакторы
Статья на конкурс «био/мол/текст»: Клетки эукариот обладают ядерной мембраной, физически разделяющей два этапа экспрессии генов (транскрипцию и трансляцию). У прокариот (бактерий и архей) отсутствие ядерной мембраны приводит к совместной локализации транскрипции и трансляции. Однако внутри клеток бактерии Gemmata obscuriglobus обнаружена хорошо развитая сеть мембран, внешне напоминающих эукариотические, в силу чего трансляция в значительной степени не сопряжена с транскрипцией. Эти результаты расширяют наши представления о пространственной организации экспрессии бактериальных генов, базирующиеся главным образом на исследовании некоторых модельных организмов, а также весьма важны для понимания того, как в ходе эволюции могла возникнуть пространственная разобщённость этапов экспрессии генов эукариот.
Конкурс «био/мол/текст»-2014
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2014 в номинации «Лучшее новостное сообщение».
Главный спонсор конкурса — дальновидная компания «Генотек».
Конкурс поддержан ОАО «РВК».
Спонсором номинации «Биоинформатика» является Институт биоинформатики.
Спонсором приза зрительских симпатий выступила фирма Helicon.
Свой приз также вручает Фонд поддержки передовых биотехнологий.
Представление о сопряжении процессов транскрипции и трансляции у бактерий (см. заглавный рисунок) основаны на изучении относительно простых модельных микроорганизмов — Escherichia coli и Bacillus subtilis. Бактерия Gemmata obscuriglobus обладает хорошо развитой системой внутриклеточных мембран, из-за которых многие рибосомы утрачивают возможность прямого доступа к нуклеоиду. Это явление побудило авторов статьи в журнале Доклады Академии Наук США [1] проверить гипотезу о том, что у Gemmata obscuriglobus транскрипция и трансляция в значительной степени пространственно разобщены. Они впервые показали, что у Gemmata obscuriglobus эндомембраны могут являться препятствием для совместной локализации транскрипции и трансляции (о подобной роли эндомембран у прокариот до настоящего времени не было известно).
Механизмы транскрипции и трансляции высококонсервативны, но их пространственная организация у про- и эукариот отличается. Сопряжение транскрипции и трансляции у прокариот возникает из-за отсутствия физического барьера (ядерной мембраны) между нуклеоидом и цитоплазмой. Сопряжение способствует повышению стабильности мРНК и трансляционной регуляции транскрипции [3]. При исследовании Caulobacter crescentus [4] было показано, что колокализация не универсальна. Как у Bacillus subtilis [5], так и у Escherichia coli [5–7] наблюдается пространственное разделение между некоторыми рибосомами и РНК-полимеразой, ассоциированной с нуклеоидом. Таким образом, пространственная организация транскрипции и трансляции была изучена лишь у трёх модельных видов бактерий.
Бактерия Gemmata obscuriglobus (порядок Planctomycetales) обладает сложной сетью внутренних мембран [8–17]. Обнаружение эндоцитозоподобных процессов [14], [18] у этого микроорганизма указывает на то, что основной функцией мембранной сети является участие в процессах внутриклеточного транспорта. Вначале (на основании электронно-микроскопических исследований) полагали, что эти мембраны, формирующие компартменты вокруг нуклеоида [9–12], являются уникальными и отличаются от цитоплазматической мембраны. Более поздние исследования (электронная томография) [13], [15–17] как подтверждают [17], так и не подтверждают [13], [15], [16] эту точку зрения. Исследования Acehan и др. [15] и Santarella-Mellwig и др. [16] свидетельствуют о том, что эндомембраны представляют собой впячивания типичной грамотрицательной цитоплазматической мембраны, и что все цитоплазматические компартменты взаимосвязаны. До сих нет однозначного мнения об эволюционных связях G. obscuriglobus и эукариот [19–21], но сложная сеть эндомембран вне зависимости от своей эволюционной истории может представлять собой физический барьер, обусловливающий особенности пространственной организации экспрессии генов. Авторы работы [1] решили проверить гипотезу о том, что у G. obscuriglobus значительная часть трансляции может быть пространственно отделена от транскрипции.
Поскольку клеточная структура G. obscuriglobus является вариабельной и динамичной [9], [13], [15], [16], авторы сравнили при помощи просвечивающей электронной микроскопии ультраструктуру клеток G. obscuriglobus из собственных культур с другими данными, представленными в литературе ранее. Они наблюдали характерную систему внутренних мембран (одно- и двухслойные мембраны), конденсированный нуклеоид и много областей, дистальных по отношению к клеточному нуклеоиду (рис. 1). Аналогичные результаты были получены ранее в работах [9], [10], [12]. Поскольку при помощи данного метода получаются двумерные изображения, авторы не могли корректно интерпретировать число нуклеоидов (один или несколько) или степень связности различных клеточных компартментов и мембран [15], [16].
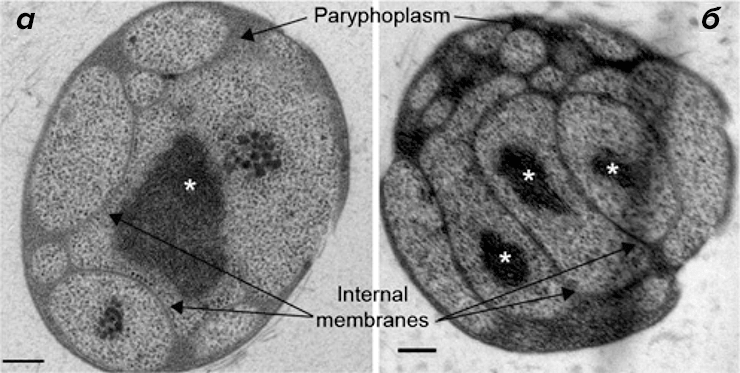
Рисунок 1. Сложная система внутренних мембран в клетках G. obscuriglobus. Изображения, полученные при помощи просвечивающей электронной микроскопии как минимум 20 клеток. Paryphoplasm — компартмент, представляющий собой свободную от рибосом область между цитоплазматической мембраной и внутренней мембраной (согласно [22]) с шириной в среднем 30–40 нм. Нуклеоид (или нуклеоиды) помечены звёздочками. а — видны расширения периферической цитоплазмы вдали от нуклеоида. Внутренние мембраны, по всей видимости, двухслойные, и на них присутствуют связанные рибосомы, как сообщалось ранее [9]. Также видно, что в некоторых клетках присутствует только один нуклеоид (а), а в других несколько нуклеоидов (б). Тем не менее сложно точно определить число нуклеоидов при отсутствии трёхмерных реконструкций. (Полоски на рисунке для масштаба 200 нм.)
Для того, чтобы определить клеточную локализацию активных транслирующих рибосом G. obscuriglobus, авторы использовали иммунофлуоресцентную микроскопию с первичными антителами к белкам S10 и EF-Tu, а также флуоресцентные красители, связывающиеся с ДНК (DAPI) и мембранами (DiOC6). Оказалось, что S10 и EF-Tu чаще встречались в участках, удалённых от нуклеоида (периферические области, где отсутствовал сигнал от DAPI; рис. 2а и 2б).
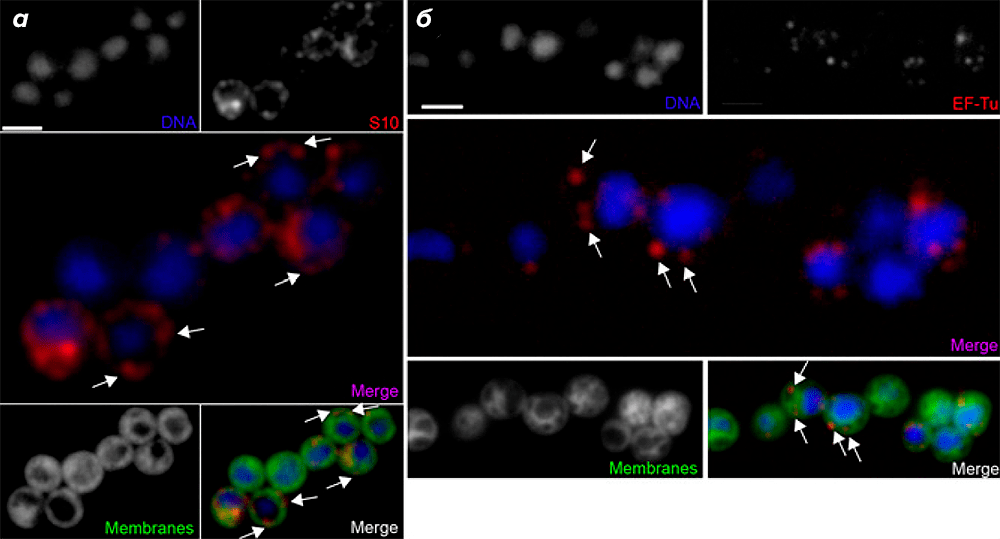
Рисунок 2. Регистрация транслирующих рибосом в клетках G. obscuriglobus при помощи иммунофлуоресцентной микроскопии. а и б — ДНК окрашена DAPI; первичные антитела к S10 (а) или EF-Tu (б) связаны с соответствующими вторичными антителами, конъюгированными с флуоресцентным красителем Texas Red; на совмещённых рисунках показаны наложенные изображения сигналов от DAPI и S10 (а) или DAPI и EF-Tu (б). Мембраны окрашены при помощи флуоресцентного красителя DiOC6. На нижних совмещённых рисунках наложены изображения от трёх сигналов: DAPI + S10 + DiOC6 (а) и DAPI + EF-Tu + DiOC6 (б). Видно, что белок S10 концентрируется в участках, удалённых от нуклеоида (а). Локализация белка EF-Tu аналогична таковой у S10. Рибосомы указаны стрелками. (Полоски на рисунке для масштаба 2 мкм.)
Для того чтобы обеспечить локализацию с высоким разрешением, авторы использовали иммуноэлектронную микроскопию. Местонахождение РНК-полимеразы в клетках G. obscuriglobus визуализировали при помощи иммуноэлектронной микроскопии с первичными антителами к β-субъединице РНК-полимеразы и вторичными антителами, конъюгированными с наночастицами золота (рис. 3а). β-субъединица РНК-полимеразы была локализована возле нуклеоида G. obscuriglobus, причём наночастицы золота группировались в 1–3 кластера. Концентрирование РНК-полимеразы в центральной области нуклеоида также было описано у B. subtilis [5].
При исследовании локализации компонентов аппарата трансляции в качестве первичных были взяты антитела к белкам S10 и EF-Tu, а вторичные антитела были конъюгированы с наночастицами золота (15 нм). Также были использованы антитела к двухцепочечной ДНК и соответствующие вторичные антитела, конъюгированные с наночастицами золота. При помощи мечения с антителами к двухцепочечной ДНК (рис. 3б) был визуализирован конденсированный нуклеоид G. obscuriglobus.
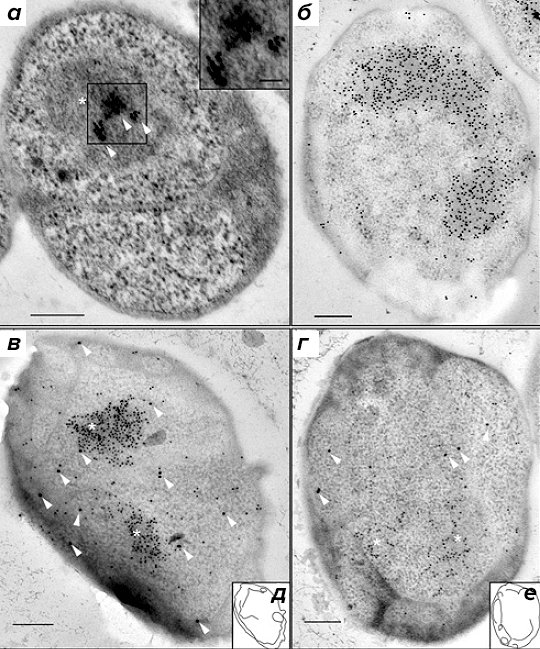
Рисунок 3. Локализация мест активной транскрипции и нуклеоида при помощи антител к β-субъединице РНК-полимеразы и к двухцепочечной ДНК, соответственно. а — Локализация вторичных антител, конъюгированных с наночастицами золота, связывающимися с первичными моноклональными антителами к β-субъединице РНК-полимеразы, показана стрелками. На вставке показан увеличенный вид наночастиц золота. Внутренняя мембрана, по-видимому, двухслойная, и с ней связаны рибосомы, как отмечалось ранее [9]. б — Мечение нуклеоида первичными антителами к двухцепочечной ДНК, связанными с вторичными антителами, конъюгированными с наночастицами золота. в и г — Нуклеоид, меченный антителами к двухцепочечной ДНК (маленькие частицы). Стрелками указаны большие частицы, представляющие собой конъюгированные с золотом вторичные антитела, связывающиеся с первичными антителами к S10 (в) или с первичными антителами к EF-Tu (г). На врезках д и е обозначены контуры клеточных мембран для в и г, соответственно. На а, в и г звёздочками обозначен нуклеоид. (Полоски на рисунке для масштаба 200 нм.)
Авторы также провели флуоресцентное мечение рибосом при помощи сульфата гентамицина, конъюгированного с сукцинимидным эфиром флуоресцентного красителя Texas Red (GTTR). Поскольку аминогликозидный антибиотик гентамицин связывается с 16S-рРНК 30S малой рибосомной субъединицы (с А-сайтом собранной рибосомы) [23], он является удобным альтернативным маркером активной трансляции. Когда клетки G. obscuriglobus были подвергнуты воздействию GTTR, сигнал от Texas Red наблюдался только в удалённых от нуклеоида областях (рис. 4а); аналогичный результат был получен для клеток дрожжей Saccharomyces cerevisiae (рис. 4б).
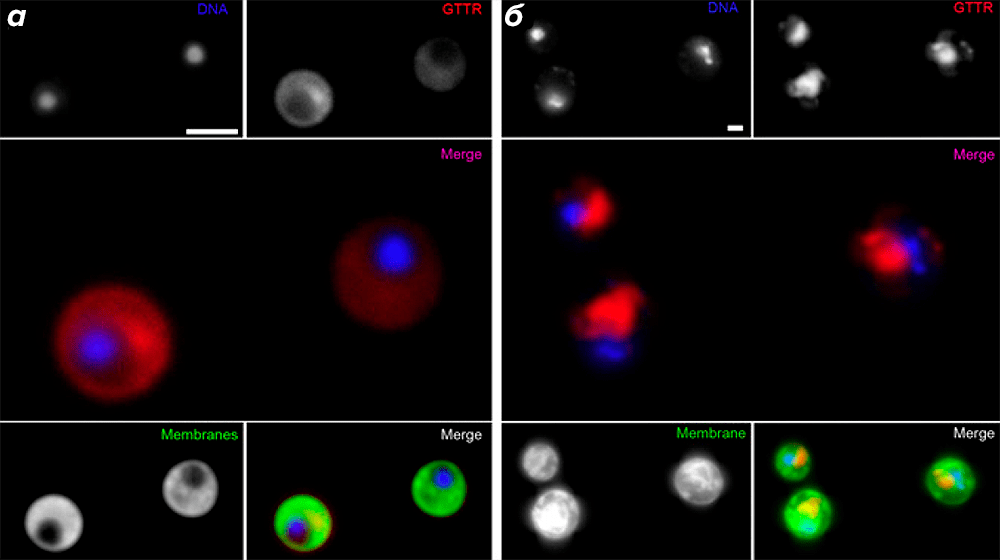
Рисунок 4. Распределение GTTR в клетках G. obscuriglobus соответствует пространственному обособлению трансляции от нуклеоида. ДНК окрашена флуоресцентным красителем DAPI. Мечение транслирующих рибосом осуществлялось с помощью GTTR; наложенное изображение получено при совмещении сигналов от DAPI и GTTR. Мембраны окрашены флуоресцентным красителем DiOC6. На нижних совмещённых рисунках наложены изображения от трёх сигналов: DAPI + GTTR + DiOC6. (Полоски на рисунке для масштаба 2 мкм.) Сигнал от GTTR, наблюдаемый в удалённых от нуклеоида областях клеток G. obscuriglobus (а), соответствует таковому в клетках дрожжей S. cerevisiae, где ядерная мембрана обусловливает пространственное разделение транскрипции и трансляции (б).
Таким образом, с использованием подхода, независимого от антител (мечение гентамицином, конъюгированным с сукцинимидным эфиром красителя Texas Red), авторами было показано то, что участки, удалённые от нуклеоида, обогащены транслирующими рибосомами, что, в свою очередь, подтверждает выводы, сделанные на основании данных иммунофлуоресценции.
Авторы представили убедительные доказательства того, что у микроорганизма G. obscuriglobus, содержащего эндомембраны, значительная часть активной трансляции, по-видимому, осуществляется в участках, удалённых от нуклеоида(ов). Это скорее всего происходит за счёт того, что мембраны отделяют нуклеоид от периферических рибосом и, следовательно, могут быть препятствием для совместной локализации процессов транскрипции и трансляции, что ранее не сообщалось для прокариотических организмов. Исследование может являться полезной основой для рассмотрения эволюции организации клеток эукариот и возникновения в её ходе пространственного разобщения процессов экспрессии генов.
Литература
- E. Y. Gottshall, C. Seebart, J. C. Gatlin, N. L. Ward. (2014). Spatially segregated transcription and translation in cells of the endomembrane-containing bacterium Gemmata obscuriglobus. Proceedings of the National Academy of Sciences. 111, 11067-11072;
- Nelson D.L. and Cox M.M. Lehninger principles of biochemistry (5 Edition). New York: W. H. Freeman and company, 2008;
- J. Gowrishankar, R. Harinarayanan. (2004). Why is transcription coupled to translation in bacteria?. Molecular Microbiology. 54, 598-603;
- Paula Montero Llopis, Audrey F. Jackson, Oleksii Sliusarenko, Ivan Surovtsev, Jennifer Heinritz, et. al.. (2010). Spatial organization of the flow of genetic information in bacteria. Nature. 466, 77-81;
- Peter J. Lewis, Shail D. Thaker, Jeffrey Errington. (2000). Compartmentalization of transcription and translation in Bacillus subtilis. EMBO J. 19, 710-718;
- Judita Mascarenhas, Michael H W Weber, Peter L Graumann. (2001). Specific polar localization of ribosomes inBacillus subtilisdepends on active transcription. EMBO Rep. 2, 685-689;
- Talukder Ali Azam, Sota Hiraga, Akira Ishihama. (2000). Two types of localization of the DNA-binding proteins within the Escherichia coli nucleoid. Genes Cells. 5, 613-626;
- P. D. Franzmann, V. B. D. Skerman. (1984). Gemmata obscuriglobus, a new genus and species of the budding bacteria. Antonie van Leeuwenhoek. 50, 261-268;
- J. A. Fuerst, R. I. Webb. (1991). Membrane-bounded nucleoid in the eubacterium Gemmatata obscuriglobus.. Proceedings of the National Academy of Sciences. 88, 8184-8188;
- J. A. Fuerst. (1995). The planctomycetes: emerging models for microbial ecology, evolution and cell biology. Microbiology. 141, 1493-1506;
- John A. Fuerst. (2005). INTRACELLULAR COMPARTMENTATION IN PLANCTOMYCETES. Annu. Rev. Microbiol.. 59, 299-328;
- Margaret Lindsay, Richard Webb, Marc Strous, Mike Jetten, Margaret Butler, et. al.. (2001). Cell compartmentalisation in planctomycetes: novel types of structural organisation for the bacterial cell. Archives of Microbiology. 175, 413-429;
- Arnon Lieber, Andrew Leis, Ariel Kushmaro, Abraham Minsky, Ohad Medalia. (2009). Chromatin Organization and Radio Resistance in the Bacterium Gemmata obscuriglobus. JB. 191, 1439-1445;
- John A. Fuerst, Evgeny Sagulenko. (2010). Protein uptake by bacteria. Communicative & Integrative Biology. 3, 572-575;
- D. Acehan, R. Santarella-Mellwig, D. P. Devos. (2014). A bacterial tubulovesicular network. Journal of Cell Science. 127, 277-280;
- Rachel Santarella-Mellwig, Sabine Pruggnaller, Norbert Roos, Iain W. Mattaj, Damien P. Devos. (2013). Three-Dimensional Reconstruction of Bacteria with a Complex Endomembrane System. PLoS Biol. 11, e1001565;
- Evgeny Sagulenko, Garry P. Morgan, Richard I. Webb, Benjamin Yee, Kuo-Chang Lee, John A. Fuerst. (2014). Structural Studies of Planctomycete Gemmata obscuriglobus Support Cell Compartmentalisation in a Bacterium. PLoS ONE. 9, e91344;
- T. G. A. Lonhienne, E. Sagulenko, R. I. Webb, K.-C. Lee, J. Franke, et. al.. (2010). Endocytosis-like protein uptake in the bacterium Gemmata obscuriglobus. Proceedings of the National Academy of Sciences. 107, 12883-12888;
- John Fuerst. (2012). Keys to eukaryality: Planctomycetes and ancestral evolution of cellular complexity. Front. Microbio.. 3;
- James O. McInerney, William F. Martin, Eugene V. Koonin, John F. Allen, Michael Y. Galperin, et. al.. (2011). Planctomycetes and eukaryotes: A case of analogy not homology. Bioessays. 33, 810-817;
- Damien P. Devos. (2012). Regarding the presence of membrane coat proteins in bacteria: Confusion? What confusion?. Bioessays. 34, 38-39;
- John A. Fuerst, Evgeny Sagulenko. (2011). Beyond the bacterium: planctomycetes challenge our concepts of microbial structure and function. Nat Rev Microbiol. 9, 403-413;
- S. Yoshizawa. (1998). Structural origins of gentamicin antibiotic action. The EMBO Journal. 17, 6437-6448.