Мобильные генетические элементы прокариот: стратификация «общества» бродяжек и домоседов
08 ноября 2014
Мобильные генетические элементы прокариот: стратификация «общества» бродяжек и домоседов
- 16452
- 4
- 24
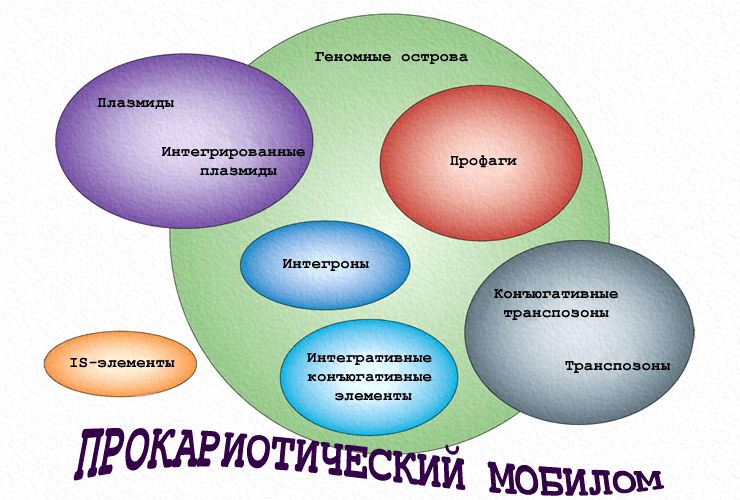
Многообразие мобильных генетических элементов
-
Автор
-
Редакторы
Статья на конкурс «био/мол/текст»: Мобильные элементы генома прокариот обсуждаются в отечественных научных и научно-популярных изданиях незаслуженно редко. Однако именно эти агенты распространяют в популяциях микроорганизмов устойчивость к физическим и химическим факторам, в том числе ультрафиолету и антибиотикам. Или дарят приютившим их клеткам селективные преимущества, кодируя метаболические пути и факторы патогенности. Эти «путешественники» преодолевают не только межклеточные и межвидовые границы, но даже междоменные, перетаскивая с собой внушительный багаж ценной генетической информации. Прокариотический мобилом поразительно мозаичен, сложно стратифицирован и напоминает своеобразный «парк юрского периода», предоставляя возможность исследовать ранние этапы эволюции механизмов наследственности и изменчивости.
Конкурс «био/мол/текст»-2014
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2014 в номинации «Биоинформатика и молекулярная эволюция».
Главный спонсор конкурса — дальновидная компания Генотек.
Конкурс поддержан ОАО «РВК».
Спонсором номинации «Биоинформатика» является Институт биоинформатики.
Спонсором приза зрительских симпатий выступила фирма Helicon.
Свой приз также вручает Фонд поддержки передовых биотехнологий.
Организмы и геномы можно таким образом расценивать как секции биосферы, по которым гены глобально циркулируют с различной интенсивностью, и в которые персональные гены и опероны могут включаться, если они предоставляют достаточные преимущества...
K. Jeon и J. Danielli [1]
Характерная, но не уникальная, особенность прокариотических организмов (бактерий и архей) — способность передавать наследственную информацию не только «вертикально» (от предков к потомкам), но и активно обмениваться ею «горизонтально» (между «соседями») — путем конъюгации, трансформации и трансдукции. Горизонтальный генетический перенос (ГГП) может происходить между прокариотическими, эукариотическими и даже между прокариотическими и эукариотическими клетками, а также в пределах одной клетки. В настоящее время ГГП признан одним из основных механизмов адаптивной эволюции [2].
ГГП осуществляют мобильные генетические элементы (МГЭ) — чрезвычайно пёстрая группа «мигрантов» с различной склонностью к оседлости, в которую входят вирусы, плазмиды, транспозоны, интегроны, геномные острова и др. Революционные темпы развития молекулярно-биологического инструментария и пополнения баз «прочитанных» нуклеотидных последовательностей способствовали открытию удивительного мира МГЭ, или мобилома. Оказалось, что в геномах бактерий эти «бродяги» не только не являются маргиналами, но и выстроили сложную иерархию. И не так их легко различить. Основной виновник этого — всё тот же ГГП, предоставляющий неисчерпаемые возможности для генетической изменчивости. В результате наследственный материал организован модульно (рис. 1) и подобно матрёшке: МГЭ меньшего размера входят в состав более крупных и т.д. И не все они легки на подъём — иногда им нужны приличные стрессы или помощь товарища. Они часто путешествуют в другие клетки «зайцами», эксплуатируя элементы более высокого ранга. А могут и предательски обездвиживать друг друга. Итак, рассмотрим основные группы прокариотического «мобильного социума».
Несколько более простых для восприятия и более коротких историй о мобильных элементах «биомолекула» уже публиковала, так что неподготовленным читателям, возможно, стóит обратиться к ним: «Геном человека: полезная книга, или глянцевый журнал?», «Alu: история одной последовательности», «Тайны „молекулярных паразитов“, или Как путешествовать по геному», «Разнообразия много не бывает: чем занимаются мобильные элементы генома в мозге». — Ред.
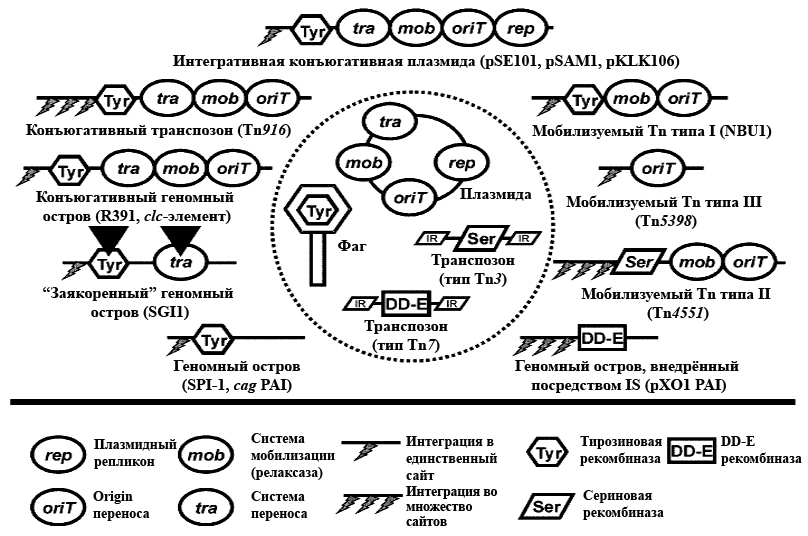
Рисунок 1. Мозаичная структура мобилизуемых и конъюгативных генетических элементов. В пунктирном круге приведены МГЭ (плазмиды, фаги, транспозоны), предоставляющие функциональные модули (указаны под чертой). Чёрные треугольники в функциональных модулях «заякоренного» геномного острова обозначают существование мутантных, нефункциональных форм этих модулей.
Вирусы: суперпаразиты и помощники
Вирусы (в т.ч. бактериофаги) и вироиды — субклеточные инфекционные агенты, которые могут воспроизводиться только внутри живых клеток («переключают» на собственное воспроизведение системы экспрессии наследственной информации клеток-хозяев), а вне клеток ведут себя как органические полимеры. Здесь я немного отклонюсь от прокариотических перипетий — уж очень интересны недавние открытия в мобиломе эукариот и даже вирусов.
Вироиды представляют собой кольцевые одноцепочечные РНК всего из нескольких сотен нуклеотидов, реплицируемые клеточной РНК-полимеразой. Они не содержат открытой рамки считывания (ОРС). Предполагается, что вироиды — это «сбежавшие интроны» (вырезанные при сплайсинге незначащие участки мРНК, приобретшие способность к репликации) или же представители добиотического «мира РНК» , эволюционные реликты. Вирусы, в отличие от вироидов, — молекулы РНК или ДНК, покрытые как минимум одной оболочкой — белковым капсидом.
«Мир РНК» — это гипотетический сценарий возникновения жизни на Земле, в основе которого лежит предположение о том, что исходно жизнь была основана только на молекулах РНК, а не ДНК или белках: «РНК у истоков жизни?». — Ред.
Вирусы паразитируют в клетках всех известных доменов — Eukarya, Archaea и Bacteria. Недавно был обнаружен ДНК-содержащий вирус «Спутник», паразитирующий на гигантском (различим с помощью светового микроскопа) мимивирусе акантамёбы (APMV) и названный по аналогии с бактериофагами вирофагом [4]. Вирофаг способен размножаться только при коинфицировании клетки амёбы мимивирусом, используя его «фабрику воспроизводства» и вызывая образование абортивных форм или патологически измененных капсидов APMV (рис. 2). ДНК «Спутника» — химера, совмещающая гены, заимствованные как у APMV, так и вирусов архей и неохарактеризованных морских вирусов.
Захватывающая история открытия вирусов вирусов рассказана в статье «...А на блохе — блошиночка поменьше». — Ред.
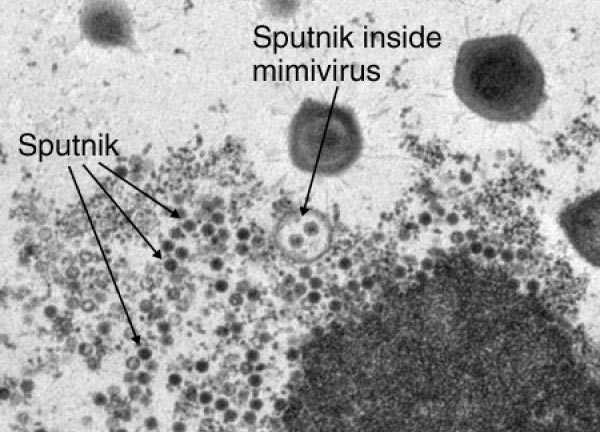
Рисунок 2. Частицы вирофага «Спутник» в мимивирусе.
В 2011 году были описаны еще 2 вирофага — OLV (Organic Lake Virophage) и Mavirus (Maverick Virus), причем множественные генетические параллели между Мавирусом и МП-транспозонами (гигантские транспозоны эукариот типа Maverick/Polinton) позволили предположить, что они эволюционировали от общего предка [6]. Отбор способствовал широкому распространению вирофага в клетках, поскольку паразитизм по отношению к гигантским вирусам повышал выживаемость инфицированных ими клеток, а приобретенная ретровирусная интеграза повышала шансы «закрепиться» в эукариотической клетке и трансформироваться в МП-транспозон.
В 2012 году охарактеризовали интегрированную в ДНК мимивирусов форму вирофага и даже новый класс МГЭ — трансповироны, способные встраиваться в ДНК и мимимирусов, и вирофагов [7]. Подобно бактериофагам, переносящим гены от одних бактерий к другим, вирофаги могут играть важную роль в ГГП между разными группами вирусов и их хозяевами.
Бактериофаги за счёт собственных тирозиновых рекомбиназ (интеграз) способны существовать в интегрированном (чаще в хромосому) состоянии — в форме профага. Однако при вырезании профага могут захватываться и упаковываться в вирусные «головки» фрагменты ДНК хозяина, которые в дальнейшем трансдуцируются в новые клетки и вместе с фаговой ДНК часто становятся «плацдармом» для рекомбинационных событий. Профаговая ДНК в некоторых случаях может составлять до 16% бактериального генома, причём постоянный обмен функциональными модулями между профагами, инфицировавшими одну клетку, приводит к формированию химерных фагов (P22 из Salmonella), а часто — к утрате способности к вырезанию и перманентному участию в эволюции бактериального генома.
Многие профаги кодируют факторы вирулентности, трансформируя нетоксигенные бактерии в агентов-убийц: профаг CTXphi из Vibrio cholerae кодирует холерный токсин, а упомянутый выше P22 — ферменты конверсии О-антигена сальмонеллы, позволяя ей уходить от иммунного надзора.
В составе фагов обнаруживают другие МГЭ (например, транспозоны, несущие гены антибиотикорезистентности) и их модули (системы репликации и переноса конъюгативных плазмид). Самый известный пример слияния модулей фагового и плазмидного происхождения — фаг Р1, способный реплицироваться и длительно поддерживаться вне хромосомы, ничем не отличаясь от плазмиды. Профаги разных бактерий детально рассмотрены в обзоре [8].
Плазмиды — маленькие гиганты больших процессов
Плазмиды — внехромосомные двуцепочечные молекулы ДНК, способные к длительному автономному существованию в клетках прокариот и некоторых эукариот. Чаще всего плазмидные ДНК суперскручены и ковалентно замкнуты в кольцо, однако у актиномицетов и спирохет встречаются и линейные формы, что обычно сочетается с линейной организацией хромосом. Размер плазмид обычно варьирует от 0,85 т.п.н. (pRKU1 из Thermotoga petrophila) до 600 т.п.н., но у бруцелл и ризобий описаны мегаплазмиды размером более 1 млн п.н., что иногда делает вопрос их дифференцировки от дополнительных хромосом риторическим [9]. Элиминация мегаплазмид, в отличие от хромосом, обычно не вызывает фатальных для бактериальной клетки последствий, однако вместе с плазмидами могут утратиться такие важные функции, как способность к фиксации азота и формированию симбиотических клубеньков (у ризобий).
На долю плазмидной ДНК может приходиться 1–15% наследственной информации бактериальной клетки, однако известны случаи, когда плазмидами контролируется до 25% (у некоторых Archaea) и даже до 40% информации (2 мегаплазмиды размером около 1,4 и 1,6 млн п.н. у Sinorhizobium meliloti).
Не являясь обязательными элементами генома, плазмиды обеспечивают селективное преимущество содержащим их микроорганизмам при освоении различных экологических ниш. При стабильном поддержании в клетке они «даруют» хозяину устойчивость к антибиотикам (R-плазмиды), солям тяжёлых металлов, ультрафиолетовому облучению, кодируют факторы патогенности, пути биосинтеза антибиотиков и бактериоцинов, системы рестрикции-модификации, симбиотические факторы, пути деградации органических и неорганических соединений (D-плазмиды), фиксации атмосферного азота [2]. Недавно показано, что некоторые плазмиды, кодируя специфические регуляторы транскрипции (AraC-, H-NS-типов и др.), могут изменять экспрессию и хозяйских генов.
Участие плазмид в ГГП обусловлено свойствами плазмидного «остова», в состав которого входят области, обеспечивающие инициацию репликации (rep-oriV, иногда — cop), стабильное наследование (у многих плазмид за это отвечают локусы par и mrs, системы постсегрегационной гибели бесплазмидных клеток), а также кластеры, обеспечивающие миграцию плазмид — системы мобилизации и конъюгации (mob, tra). Плазмиды классифицируют по размеру, копийности, кодируемым фенотипическим признакам и т.д., однако филогенетически обоснована только классификация по группам несовместимости на основе гомологии области rep-oriV.
Плазмидами часто мобилизуются сосуществующие с ними в одной клетке неконъюгативные МГЭ (но обладающие как минимум сайтом начала переноса oriT, а чаще и генами mob) и даже хромосомы. Мобилизация генов хромосомы возможна в случае интеграции в неё конъюгативной плазмиды. Это возможно, когда в обеих молекулах присутствуют одинаковые инсерционные последовательности (IS-элементы), обеспечивающие гомологичную рекомбинацию (типичный пример — F-фактор E.coli). Однако интеграция может быть и RecA-независимой, если плазмида кодирует тирозиновую интегразу (плазмиды стрептомицетов pSE101, pSAM2 и псевдомонад pKLK106). В этом случае интеграция чаще происходит в гены тРНК, но в некоторых хозяевах эта закономерность нарушается [3].
Плазмиды служат донорами функциональных модулей для других МГЭ (рис. 1), в результате чего формируются сложные мобильные мозаичные конструкции, осваивающие новые геномы. Плазмидные репликоны — «каркасы» многочисленных векторных систем и прекрасные модели для изучения механизмов экспрессии наследственной информации.
Транспозоны — универсальные генетические челноки
Транспозоны — МГЭ, перемещающиеся как в пределах одной молекулы ДНК, так и между разными репликонами одного генома (конъюгативные транспозоны — и между геномами). Фланкированы инвертированными повторами, а в центральной части содержат гены, ответственные за перемещение. Транспозоны прокариот подразделяют на IS-элементы, Tn-элементы и Mu-подобные фаги [10].
- IS-элементы — инсерционные последовательности длиной 700–1800 п.н., содержащие ОРС одного-двух белков, необходимых для перемещения (в частности, ген рекомбиназы/транспозазы), а также промотор транскрипции. IS-элементы ограничены короткими инвертированными концевыми повторами (10–40 п.н.) и обычно фланкированы прямыми повторами (2–12 п.н.), возникающими из-за удвоения ДНК-мишени в области интеграции элемента. Несмотря на то, что IS-элементы не кодируют каких-либо фенотипических признаков, их внедрение часто вызывает инсерционные мутации или же изменяет экспрессию генов в непосредственной близости от места внедрения. Внедрение двух копий IS-элемента на небольшом расстоянии друг от друга может вызывать делеции заключённого между ними наследственного материала, а присутствие копии в составе другого репликона может вести к образованию коинтегратов этих репликонов (гомологичной рекомбинации). IS-элементы очень широко представлены в хромосомах и МГЭ.
- Tn-элементы (собственно транспозоны) помимо генов, ответственных за перемещение, несут детерминанты устойчивости к тяжёлым металлам, антибиотикам, УФ, гены факторов патогенности, ферментов утилизации ксенобиотиков и т.п. «Начинка» фланкирована инвертированными повторами из 35–48 п.н. (у Tn3-семейства) или двумя идентичными IS-элементами в прямой или обратной ориентации (составные транспозоны, например, Tn5 и Tn10); как и в случае IS-элементов, характерно удвоение ДНК-мишени (небольшие прямые повторы). Транспозоны, как правило, внедряются в плазмидную и хромосомную ДНК, используя репликативный (копия — и в доноре, и в реципиенте) или консервативный (вырезание из донора — встраивание в реципиент) механизмы транспозиции, опосредованные собственной транспозазой (TnpA), а в случае Tn3-семейства — и резолвазой (TnpR), разрешающей формирующиеся при транспозиции коинтеграты. В составных транспозонах за перемещение отвечает транспозаза, кодируемая лишь одним IS-элементом, второй элемент обычно дефектен. Внедрение транспозонов, как и IS-элементов, оказывает мутагенное и рекомбиногенное действие на ДНК-мишень [10].
Многие Tn-элементы могут автономно перемещаться не только в пределах одного генома, встраиваясь в случайные места, но и между бактериями. Конъюгативные транспозоны обладают собственным (видимо, плазмидного происхождения) tra-опероном. В отличие от классических транспозонов, использующих при интеграции сериновую рекомбиназу (резолвазу/инвертазу) или DD-E-рекомбиназу (транспозазу), конъюгативные транспозоны (Tn916 из Enterococcus faecalis, Tn5253 из Streptococcus pneumoniae, CTnDOT из Bacteroides) «приручили» подобную λ-фаговой тирозиновую рекомбиназу (специфичность интеграции зависит от хозяина). Описаны также мобилизуемые транспозоны, содержащие ген рекомбиназы, oriT и mob-систему (Non-replicating Bacteroides Units — NBU1 и NBU2, Tn4555, Tn4451, Tn4453) или только oriT (Tn5398) и использующие аппарат переноса конъюгативных плазмид или других транспозонов [3]. - Mu-подобные фаги — умеренные бактериофаги γ-протеобактерий, сложнейшие транспозоны, которые помимо генов транспозиции/интеграции несут детерминанты сборки фаговых частиц и имеют внеклеточную форму существования. При первичной интеграции в геном инфицированной клетки используют механизм консервативной транспозиции, а при переходе из лизогенной стадии (профага) к литической — репликативную транспозицию.
Интеграция фага Mu происходит в случайные сайты, провоцируя инсерционный мутагенез, с дупликацией 5 п.н. ДНК-мишени. При развитии литического сценария множественные копии фага вызывают фрагментацию хромосомы, что способствует упаковке в фаговые капсиды и последующей трансдукции не только вирусной ДНК, но и ближайших фрагментов ДНК-мишени. Иногда фрагменты хромосомы при упаковке в капсид полностью заменяют фаговую ДНК [11].
Интегроны — природные системы клонирования и экспрессии
Интегроны представляют собой «заякоренные» в транспозонах, плазмидах и хромосомах «платформы», включающие ген intI тирозиновой рекомбиназы (интегразы), промотор и сайт интеграции attI для «улавливаемых» генных кассет — ДНК-сегментов, содержащих только беспромоторную ОРС с уникальным рекомбинационным сайтом attC (рис. 3, а).
В процессе перемещения от одного интегрона к другому или от одного сайта в интегроне к другому сайту, генная кассета существует как автономная и неспособная к репликации двунитевая кольцевая молекула ДНК. Кассеты, захватываемые интегронами и суперинтегронами, могут содержать гены факторов патогенности, метаболических путей, детерминанты антибиотико- и дезинфектантоустойчивости или гены рестрикционных ферментов. Интеграза IntI катализирует сайт-специфическую рекомбинацию между сайтами attI и attC, в результате чего происходит интеграция или вырезание кассеты. Множество событий интеграции ведет к образованию мультикассетных рядов, в которых все кассеты фланкированы attC-сайтами. Известны хромосомные суперинтегроны, включающие до 179 генных кассет (у Vibrio cholerae), однако среди клинически значимых бактерий большинство интегронов содержит до 5–8 генных кассет. Наиболее эффективно экспрессируются кассеты, расположенные ближе к промотору, но изменение селективного давления может способствовать перестройкам в составе интегрона.
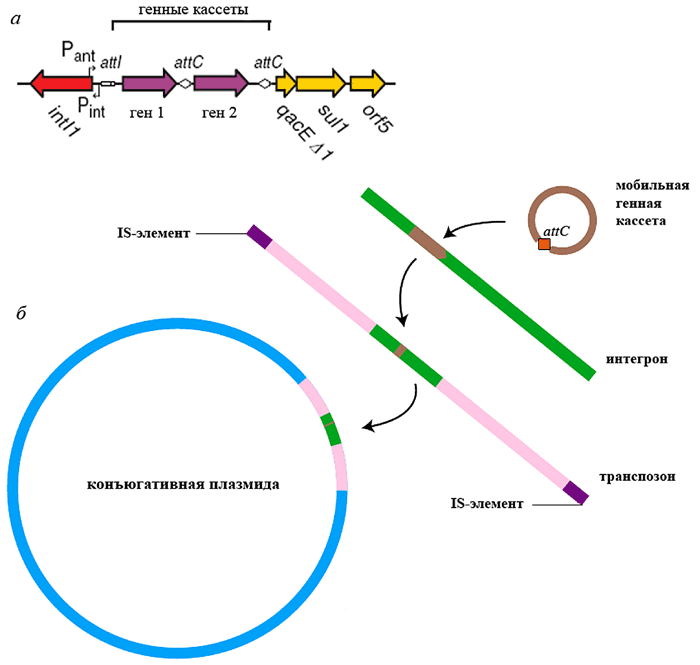
Рисунок 3. Интегрон — ДНК, улавливающая генные кассеты и распространяемая в составе более высокоорганизованных МГЭ. а — Структура интегрона класса 1. Pint — промотор интегразы, Pant — промотор кассет антибиотикорезистентности. Остальные элементы объяснены в тексте; б — Иерархическая организация МГЭ.
Все интегроны, несущие кассеты антибиотикорезистентности, разделяют на 5 классов на основании гомологии последовательностей кодируемых ими интеграз. Большинство интегронов антибиотикорезистентности относится к классу 1 (часто ассоциированы с Tn21-семейством). Они включают два концевых невариабельных региона, называемых константными последовательностями (constant sequences, CS), и высоковариабельный центральный участок. В одном конце интегрона (5’-CS), обычно находятся intI, attI и промотор, от которого экспрессируются гены кассеты. В другом конце (3’-CS), находится часть гена qacEΔ1, который, будучи интактным, несёт устойчивость к четвертичным аммониевым соединениям. За ним расположен ген sul, определяющий резистентность микроорганизма к сульфаниламидам, и 1–2 гена с неустановленной функцией — orf5 и иногда orf6 (рис. 3, а). Интегроны класса 2 ассоциированы с Tn7-семейством, классов 3 и 5 — с плазмидами, класса 4 — с конъюгативным геномным островом SXT Vibrio cholerae.
Не являясь «самораспространяемыми» элементами, интегроны активно переносятся в бактериальных популяциях за счёт МГЭ, в которых они локализованы (рис. 3, б), подвергаются рекомбинации (известны химерные интегроны из модулей разных классов), деградации, «улавливают» новые кассеты, причем функции огромного числа кассет до сих пор не известны [13].
Геномные острова — «пластилин» для эволюционного процесса
Геномные острова (genomic islands, ГО) — сегменты ДНК, присутствующие в геноме одних штаммов и отсутствующие у других, даже близкородственных штаммов одного вида. Строение «усредненного» ГО показано на рис. 4. Для геномных островов характерно:
- Размеры 10–500 т.п.н. (менее 10 т.п.н. — геномные островки (genomic islets));
- Отличные от ДНК-мишени нуклеотидные характеристики: G+C состав, частоты тетрануклеотидов и использование кодонов;
- Несут ген тирозиновой рекомбиназы (интегразы), обеспечивающей встраивание ГО в специфические районы хромосомы — гены тРНК, ген prfC у E. coli (для ГО SXT и R391). Именно избирательное встраивание ГО в 1–2 сайта хромосомы иногда помогает отличить его от «непривередливых» к месту интеграции конъюгативных транспозонов [3]. Иногда ГО не содержат или утратили ген интегразы, в результате чего оказались «заякоренными» в хромосоме хозяина. Пример такого острова — SGI1, «подаривший» штамму Salmonella enterica sv. Typhimurium DT104 множественную устойчивость к антибиотикам. Похоже, до утраты рекомбиназы этот ГО был интегративной конъюгативной плазмидой;
- Часто фланкированы 16—20-п.н. повторами ДНК-мишени, которые могут использоваться для вырезания острова. Для вырезания иногда требуется помощь собственного фермента эксцизионазы или помощь инфицировавшего клетку бактериофага. Для острова SaPI1 из Staphylococcus aureus показана мобилизация фагами 80α и φ13, причём вырезанная ДНК ГО встраивалась в новые места генома или, реплицируясь, конкурировала с фаговой ДНК за включение в фаговый капсид и переносилась путем трансдукции в другие клетки;
- Часто содержат IS-элементы и транспозоны, участвующие в приобретении или устранении генетической информации в пределах острова, причем рекомбиназы этих МГЭ могут использоваться и для интеграции/вырезания целого острова, например, ГО, обнаруженных на плазмидах pXO1 Bacillus anthracis и p33071 Rhodococcus equi [3];
- Играют важную роль в эволюции и адаптации бактерий к изменяющимся условиям среды обитания, кодируя факторы патогенности, симбиотического «стиля жизни», резистентности к тяжёлым металлам и антибиотикам, ферменты деградации ксенобиотиков и т.п. Считается, что ГО обеспечивают «эволюцию квантового скачка», драматически меняя фенотип хозяина, например, с непатогенного на патогенный или с несимбиотического на симбиотический. Предполагается, что развитие паразитического способа существования у изначально сапротрофных свободноживущих микобактерий происходило с участием ГО. Именно на ГО Rv0986 локализованы гены, кодирующие способность M. tuberculosis прикрепляться к эукариотическим клеткам [14].
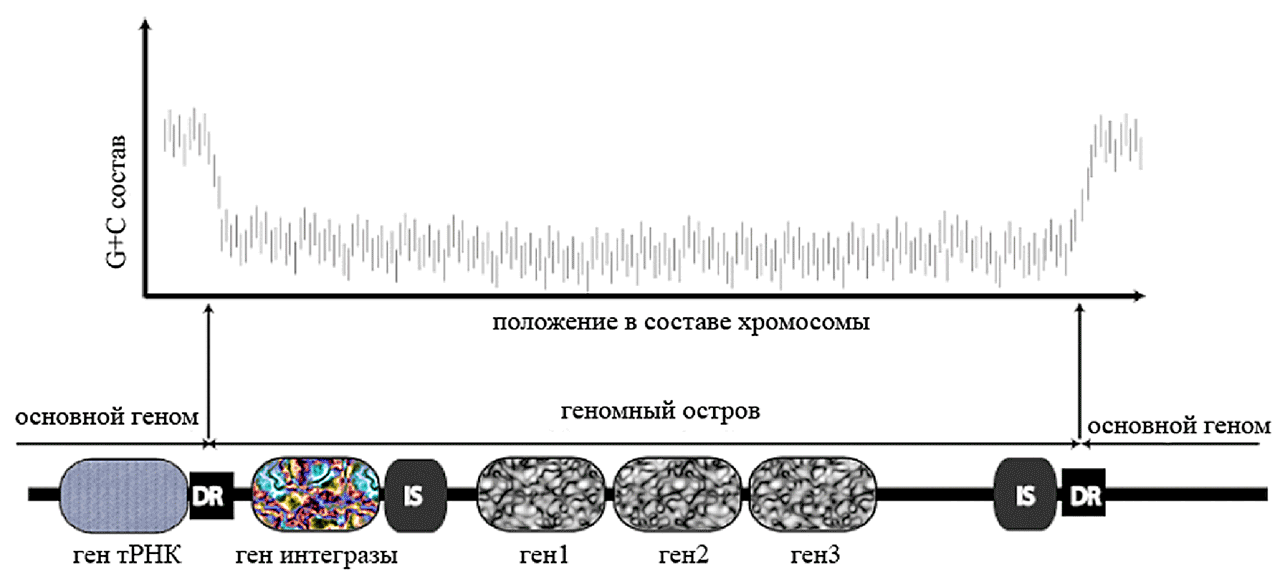
Рисунок 4. Схематическое изображение структуры геномного острова в составе бактериальной хромосомы. DR — прямые повторы ДНК хромосомы, фланкирующие ГО; IS — инсерционные элементы.
Недавно у штаммов клона С Pseudomonas aeruginosa был обнаружен ГО — pKLC102 (103,5 т.п.н.) — плазмидно-фаговый гибрид, предположительно несущий детерминанты патогенности, способный существовать как в интегрированном в хромосому состоянии, так и в форме автономной плазмиды [15]. Различные ОРС pKLC102 гомологичны типичным генам бактерий, ассоциированных с ризосферой растений, что может указывать на эволюционирование этого ГО в соответствующих микробных сообществах. У некоторых штаммов клона С, выделенных из лёгких больных муковисцидозом, этот ГО был необратимо «заякорен» в хромосоме из-за внедрения в него транспозона TNCP23 (химера из IS6100, интегрона класса 1 и плазмиды), что вело и к крупным хромосомным инверсиям. Внедрению транспозона, несущего интегрон с кассетой устойчивости к аминогликозидам, видимо, способствует лечение больных муковисцидозом соответствующими антибиотиками.
Геномные острова гонококков (GGI) кодируют систему секреции типа IV (T4SS), родственную плазмидным системам конъюгационного переноса. Посредством T4SS распространяется не только сам элемент и его продукты, но секретируется в окружающую среду и хромосома Neisseria gonorrhoeae, которая затем может трансформировать другие бактерии и участвовать в рекомбинационных событиях.
Помимо островов патогенности огромный интерес представляют «экологические» острова β- и γ-протеобактерий, многие из которых конъюгативны и кодируют ферменты биодеградации хлорированных и нитроароматических соединений. В частности, clc-элемент из Pseudomonas sp. B13 несёт amn-кластер биодеградации аминофенола.
Эксцизия, а, следовательно, и распространение ГО, строго регулируется. Часто это происходит под quorum-sensing-контролем («чувством кворума») в стационарной фазе бактериального роста. Мобилизация многих ГО происходит в результате SOS-ответа на повреждения ДНК, вызванные антибиотиками (SXT-элемент V. cholerae, способный мобилизовать и «приютившую» его ДНК) или другими факторами среды [14].
Главные нарушители границ и их мекка
Крупнейшие из самых подвижных «платформ» для сборки всевозможных мобильных элементов и модулей — плазмиды и бактериофаги, но последние обычно имеют очень ограниченный круг хозяев. Плазмиды — более широко представленный и разнообразный МГЭ прокариотического мира, поскольку этот элемент способен преодолевать не только межвидовые барьеры, но и междоменные (энциклопедический пример — Ti-плазмиды Agrobacterium tumefaciens, инфицирующие эукариотические клетки). Плазмидные модули входят в состав конъюгативных транспозонов и геномных островов (рис. 1), а в самих плазмидах находят приют IS-элементы, транспозоны, интегроны, фаговые модули и даже геномные острова. Оказалось, что влияния ГГП не избежали и внутриклеточные паразиты, которые, считалось, должны быть изолированы от потенциальных доноров ДНК: конъюгативные плазмиды, причастные к вирулентности, обнаружены у Rickettsia felis, как найдены плазмиды и у внутриклеточной бактерии Buchnera — эндосимбионта тли.
Можно выделить несколько «горячих точек» ГГП: ризосфера растений, биоплёнки, разлагающиеся материалы, клетки простейших (жизнеспособные Chlamydiae и Rickettsia обнаружены в амёбах) [16]. Всё перечисленное можно встретить в отстойниках (станциях очистки воды), где смешиваются сточные воды различного происхождения, в т.ч. больничные и фармпроизводств, обеспечивающие селективное давление для обмена МГЭ в условиях высокой бактериальной плотности и метаболической активности. Процесс трансформации клеток молекулами ДНК происходит в среде постоянно. Свободная ДНК может сохраняться в окружающей среде тысячелетиями, а природной компетентностью обладает как минимум 90 видов почвенных и водных прокариот. Однако ведущую роль в распространении между бактериями детерминант резистентности и биодеградации играют конъюгативные и мобилизуемые плазмиды.
Из обитателей отстойников, а что самое опасное — из бактерий вод, уже прошедших очистку, — в большом количестве выделяют плазмиды IncP-1-группы несовместимости (в системе классификации плазмид псевдомонад) [12]. Эти относительно небольшие молекулы дарят хозяевам массу селективных преимуществ (от множественной резистентности до биодеградации хлорорганики) и способны распространяться не только среди псевдомонад, но и практически всех грамотрицательных и некоторых грамположительных бактерий. Кроме того, они способны мобилизовать неконъюгативные R-плазмиды (например, IncP-4) к переносу в ещё более широкий спектр бактерий, а также в дрожжи и клеточные линии млекопитающих [17].
Селективное давление, оказываемое присутствием в сточных водах как лекарств, так и нефтепродуктов, тяжёлых металлов и сурфактантов, стабилизирует соответствующую плазмидную ДНК в бактериальных популяциях отстойников. Интересно, что возможна неспецифическая, «побочная», селекция R- и D-плазмид присутствием тяжёлых металлов, устойчивость к которым часто кодируется плазмидами этих двух типов. Возможна и селекция R-плазмид присутствием в среде детергентов, содержащих четвертичные аммонии: детерминанты антибиотикорезистентности и ген qacEΔ1 часто «соседствуют» в плазмидных интегронах. Кроме того, кодируемые R-плазмидами эффлюкс-помпы бактерии используют не только для выброса поступающих в клетку антибиотиков, но и для транспортировки других соединений, поэтому даже при отсутствии в среде антибиотиков соответствующие плазмиды могут оказаться полезными и стабильно поддерживаться в бактериальных популяциях. Отстойники, по сути, служат котлами по переплавке геномов, где под множественным селективным давлением происходит обмен наследственной информацией между патогенными и сапротрофными микроорганизмами, отбор оптимальных комбинаций генов, которым ничто не мешает распространяться за пределами очистных сооружений. И именно плазмиды играют важнейшую роль в поддержании бактериального «коммунального генетического пула» [9].
Биология плазмид в России и за рубежом. Что же можно возвести на крепком фундаменте?
Несмотря на колоссальный вклад плазмид в распространение бактериальной резистентности, в биодеградативный потенциал среды, в эволюцию геномов и биотехнологию, заниматься изучением этих молекул в России теперь не модно. Показательно, что в огромной стране работает всего одна (!) лаборатория, специализирующаяся на биологии плазмид (ИБФМ РАН, г. Пущино). Особо неблагодарное дело — исследовать не антибиотикорезистентность, практическая значимость которой так очевидна, а фундаментальные процессы — репликацию, сегрегацию, конъюгацию. Но именно эти процессы позволяют исследователю заглянуть в «эволюционные ясли» механизмов наследственности, лежат в основе классификации плазмид и определяют возможность поддержания клинически/биотехнологически важных признаков в конкретной бактериальной популяции.
Как же относятся к плазмидам зарубежные биологи? Статьи коллективов из Японии, Германии и Испании, посвящённые структуре и функционированию внехромосомных ДНК бактерий, регулярно появляются на страницах таких авторитетных изданий, как Nature, Journal of Bacteriology, EMBO и др. В последние годы признанных экспертов в области биологии плазмид «догнали» учёные из США, Китая, Польши, Белоруссии.
Как и в любой другой науке, практически и мировоззренчески ценные открытия в биологии плазмид опираются на возводимый десятилетиями теоретический фундамент. В этом отношении показательна «биография» плазмиды pPS10 фитопатогена Pseudomonas savastanoi. Испанские биологи копили знания о механизмах её поддержания 30 лет! В итоге на базе белка-инициатора репликации этой плазмиды (RepA) была разработана модель для изучения амилоидных протеинопатий, наблюдаемых при нейродегенеративных заболеваниях человека [18]. Кроме того, обнаружение структурного сходства белка RepA с компонентами эукариотического origin recognition complex заставило учёных выдвинуть предположение о слиянии на каком-то эволюционном этапе гена инициатора репликации хромосомы общего предка эукариот и архей и гена repA бактериальных плазмид типа pPS10 [19]. Таким образом, некоторые молекулярные модули эукариотических организмов, включая человека, могут быть унаследованы напрямую от плазмид прокариот.
Внушителен и «послужной список» плазмиды биодеградации карбазола pCAR1: японские микробиологи по ней написали не менее пяти работ, причём именно гены биодеградации их интересовали в последнюю очередь, а в первую — механизмы поддержания и распространения, взаимодействие с бактериальной хромосомой. Авторы «CARологии» показали, например, что глобальный регулятор транскрипции H-NS-семейства, кодируемый pCAR1, модулирует экспрессию хромосомных генов, в том числе, усиливает транскрипцию mexEF-oprN-оперона (гены эффлюкс-системы), повышая устойчивость бактерии-хозяина к антибиотикам [20].
С годами становится ясно, что самое интересное лежит не на поверхности. Связь плазмид с распространением антибиотикорезистентности очевидна, и соответстующие гены изучаются давно, однако борьба с этим опасным явлением пока успехом не увенчалась. И вот тут-то на помощь могут прийти всё те же, такие «неинтересные», области rep-ori-par, ответственные за поддержание плазмидной ДНК. Именно их рассматривают в качестве мишеней антиплазмидной терапии, которой в перспективе можно было бы дополнить лечение некоторых бактериальных инфекций.
Потенциал мобильных элементов прокариот как объектов исследования почти неиссякаем. На какие же аспекты функционирования «мобильного социума» будут направлены взгляды учёных в будущем? Разумеется, микробиологи продолжат «вербовать» эти элементы для удовлетворения биотехнологических нужд общества (векторы, препараты для биоремедиации) и искать способы борьбы с негативными для человека последствиями горизонтального генетического переноса (бактериальная резистентность и вирулентность). В то же время, внимание научного сообщества будет направлено и на дальнейшее вскрытие вклада МГЭ в эволюцию организмов. В ближайшие годы также прогнозируется массовое обращение микробиологов к мобилому кишечной микрофлоры: публикации о циркуляции плазмид в этих загадочных бактериальных коллективах уже появляются в респектабельных изданиях. Поиски будут направлены, в том числе, на выяснение роли МГЭ наших симбионтов в формировании метаболических путей и развитии заболеваний.
Литература
- Jeon K.W. and Danielli J.F. (1971). Micrurgical Studies with Large Free-Living Amebas. Int. Rev. Cytol. 30, 49–89;
- Top E.M., Moenne-Loccoz Y., Pembroke T., Thomas C.M. Phenotypic traits conferred by plasmids. In: The horizontal gene pool: bacterial plasmids and gene spread / ed. by Thomas C.M. Amsterdam: Harwood Academic Publishers, 2000. P. 246–285;
- A Mark Osborn, Dietmar Böltner. (2002). When phage, plasmids, and transposons collide: genomic islands, and conjugative- and mobilizable-transposons as a mosaic continuum. Plasmid. 48, 202-212;
- Bernard La Scola, Christelle Desnues, Isabelle Pagnier, Catherine Robert, Lina Barrassi, et. al.. (2008). The virophage as a unique parasite of the giant mimivirus. Nature. 455, 100-104;
- Pennisi E. (2008). Virus gets a taste of its own medicine. Science News;
- M. G. Fischer, C. A. Suttle. (2011). A Virophage at the Origin of Large DNA Transposons. Science. 332, 231-234;
- C. Desnues, B. La Scola, N. Yutin, G. Fournous, C. Robert, et. al.. (2012). Provirophages and transpovirons as the diverse mobilome of giant viruses. Proceedings of the National Academy of Sciences. 109, 18078-18083;
- C. Canchaya, C. Proux, G. Fournous, A. Bruttin, H. Brussow. (2003). Prophage Genomics. Microbiology and Molecular Biology Reviews. 67, 238-276;
- A. Norman, L. H. Hansen, S. J. Sorensen. (2009). Conjugative plasmids: vessels of the communal gene pool. Philosophical Transactions of the Royal Society B: Biological Sciences. 364, 2275-2289;
- Рыбчин В.Н. Основы генетической инженерии. 2-е изд., перераб. и доп.: Учебник для вузов. СПб: Изд-во СПбГТУ, 2002. — 522 с.;
- N D F Grindley, R R Reed. (1985). Transpositional Recombination in Prokaryotes. Annu. Rev. Biochem.. 54, 863-896;
- Andreas Schlüter, Rafael Szczepanowski, Alfred Pühler, Eva M. Top. (2007). Genomics of IncP-1 antibiotic resistance plasmids isolated from wastewater treatment plants provides evidence for a widely accessible drug resistance gene pool. FEMS Microbiol Rev. 31, 449-477;
- A.C. Fluit, F.-J. Schmitz. (2004). Resistance integrons and super-integrons. Clinical Microbiology and Infection. 10, 272-288;
- Mario Juhas, Jan Roelof van der Meer, Muriel Gaillard, Rosalind M. Harding, Derek W. Hood, Derrick W. Crook. (2009). Genomic islands: tools of bacterial horizontal gene transfer and evolution. FEMS Microbiol Rev. 33, 376-393;
- J. Klockgether, O. Reva, K. Larbig, B. Tummler. (2004). Sequence Analysis of the Mobile Genome Island pKLC102 of Pseudomonas aeruginosa C. Journal of Bacteriology. 186, 518-534;
- M. A. Ragan, R. G. Beiko. (2009). Lateral genetic transfer: open issues. Philosophical Transactions of the Royal Society B: Biological Sciences. 364, 2241-2251;
- Virginia L. Waters. (2001). Conjugation between bacterial and mammalian cells. Nat Genet. 29, 375-376;
- Rafael Giraldo, María Elena Fernández-Tresguerres. (2010). Voyage of RepA protein from plasmid DNA replication through amyloid aggregation towards synthetic biology. Journal of Applied Biomedicine. 8, 151-158;
- Rafael Giraldo. (2003). Common domains in the initiators of DNA replication inBacteria, ArchaeaandEukarya: combined structural, functional and phylogenetic perspectives. FEMS Microbiol Rev. 26, 533-554;
- C.-S. Yun, C. Suzuki, K. Naito, T. Takeda, Y. Takahashi, et. al.. (2010). Pmr, a Histone-Like Protein H1 (H-NS) Family Protein Encoded by the IncP-7 Plasmid pCAR1, Is a Key Global Regulator That Alters Host Function. Journal of Bacteriology. 192, 4720-4731.