Это чума
29 октября 2011
Это чума
- 3593
- 2
- 4
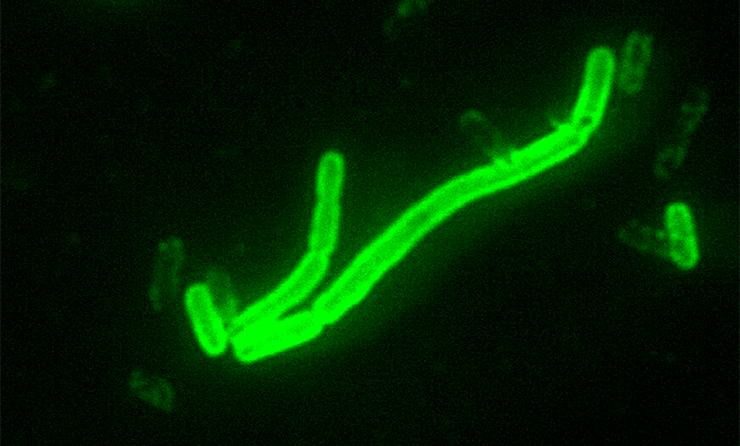
Бактерия Yersinia pestis при увеличении 200×. Переносящаяся блохами, она нанесла человечеству в свое время многомиллионный урон.
-
Автор
-
Редакторы
Статья на конкурс «био/мол/текст»: Под именем Yersinia pestis скрывается патоген, в свое время уничтоживший треть населения Европы, а также еще массу народа в Африке и Азии. Он ответственен, в частности, за то, что называют «Черной смертью» — одну из трех крупнейших пандемий чумы, зародившуюся в Монголии и уничтожавшую европейцев в период с 1346 по 1351 год. Спустя почти 7 веков человечество решило если не отомстить бактерии, то хотя бы разобраться, как ей удалось достичь тогда таких чудовищных результатов. Молекулы ДНК черной смерти подняли из могил и секвенировали.
Конкурс «био/мол/текст»-2011
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2011 в номинации «Лучшее новостное сообщение».
Изучать древних патогенов важно для того, чтобы понимать, какими извилистыми путями шагает их эволюция, и каких сюрпризов стоит ожидать от наших микроскопических врагов в будущем. Современные технологии секвенирования и совершенствование методов манипуляции с ДНК позволили коллективу ученых из Канады, США и Германии реконструировать геном возбудителя европейской эпидемии бубонной чумы XIV века. Генетический материал удалось получить из останков жертв эпидемии, захороненных в Восточном Смитфилде — районе современного Лондона. Статью о первом секвенированном геноме древней патогенной бактерии опубликовал в октябре этого года журнал Nature [1].
«Биомолекула» уже многократно рассказывала про успехи и методы секвенирования: «Важнейшие методы молекулярной биологии и генной инженерии» [7], «Огурцы-убийцы, или Как встретились Джим Уотсон и Гордон Мур» [8], «Код жизни: прочесть не значит понять» [9] и «454-секвенирование (высокопроизводительное пиросеквенирование ДНК)» [10]. — Ред.
Исследовать древнюю ДНК непросто. Во-первых, она довольно плохого качества, поскольку деградирует от смены температур, влажности и времени. Приходится иметь дело с фрагментированными и поперечно-сшитыми молекулами. Во-вторых, не всегда легко доказать множеству скептиков, что выделил именно то, что нужно для каких бы то ни было громких выводов. В случае с Yersinia pestis споров было много. Некоторые считали, что дошедшие до нас сведения об эпидемии указывают на то, что это была не чума, а скорее какой-то вариант геморрагической лихорадки типа той, что вызывает вирус Эбола [2]. В 2000 году коллектив под руководством Дидье Рауля (Didier Raoult) опубликовал результаты работы, в ходе которой была выделена ДНК из зубов людей, погребенных в одном из мест массового захоронения жертв эпидемии XIV века [3]. Ученые амплифицировали фрагменты ДНК при помощи ПЦР и показали, что в образцах есть последовательности генома Yersinia pestis, и, следовательно, в те годы в Европе бушевала именно чума. Однако повторение эксперимента другими авторами под руководством Томаса Гилберта с использованием более крупной выборки результатов не дало [4]. Команда пришла к обратному выводу, объяснив результат коллег тем, что они амплифицировали ДНК какой-то другой бактерии или засорили образцы современной чумной палочкой.
Самая свежая работа по теме была проведена под руководством Хендрика Пойнара (Hendrik Poinar) и Йоханнеса Краузе (Johannes Krause). Их сотрудники использовали принципиально другой подход с применением секвенаторов нового поколения. Такой подход уже применялся ранее при изучении генома неандертальца [5], однако авторы работы про древнюю чумную палочку утверждают, что их исследование было куда более сложным, поскольку ДНК патогенных бактерий в костях и зубах давно умерших людей чрезвычайно мáло.
Для того чтобы достать из древних образцов нужную ДНК, использовались микрочипы с ДНК ныне живущих штаммов чумной палочки. Это позволило исключить из работы почвенных бактерий и прочий генетический мусор. Полученная фрагментированная ДНК (фрагменты генома в среднем были длиной по 55 нуклеотидов) секвенировалась с использованием секвенаторов фирмы Illumina. При этом по оценкам авторов каждая из «букв» генома древней бактерии была прочитана порядка 30 раз, что делает полученную последовательность весьма достоверной.
Геном, который получился в итоге, сравнили с ДНК 17 современных штаммов Yersinia pestis и обнаружили, что он находится примерно в корне эволюционного древа этих последовательностей (рис. 1), что свидетельствует о том, что штамм «Черная смерть» имеет много близких родственников среди ныне циркулирующих чумных палочек.
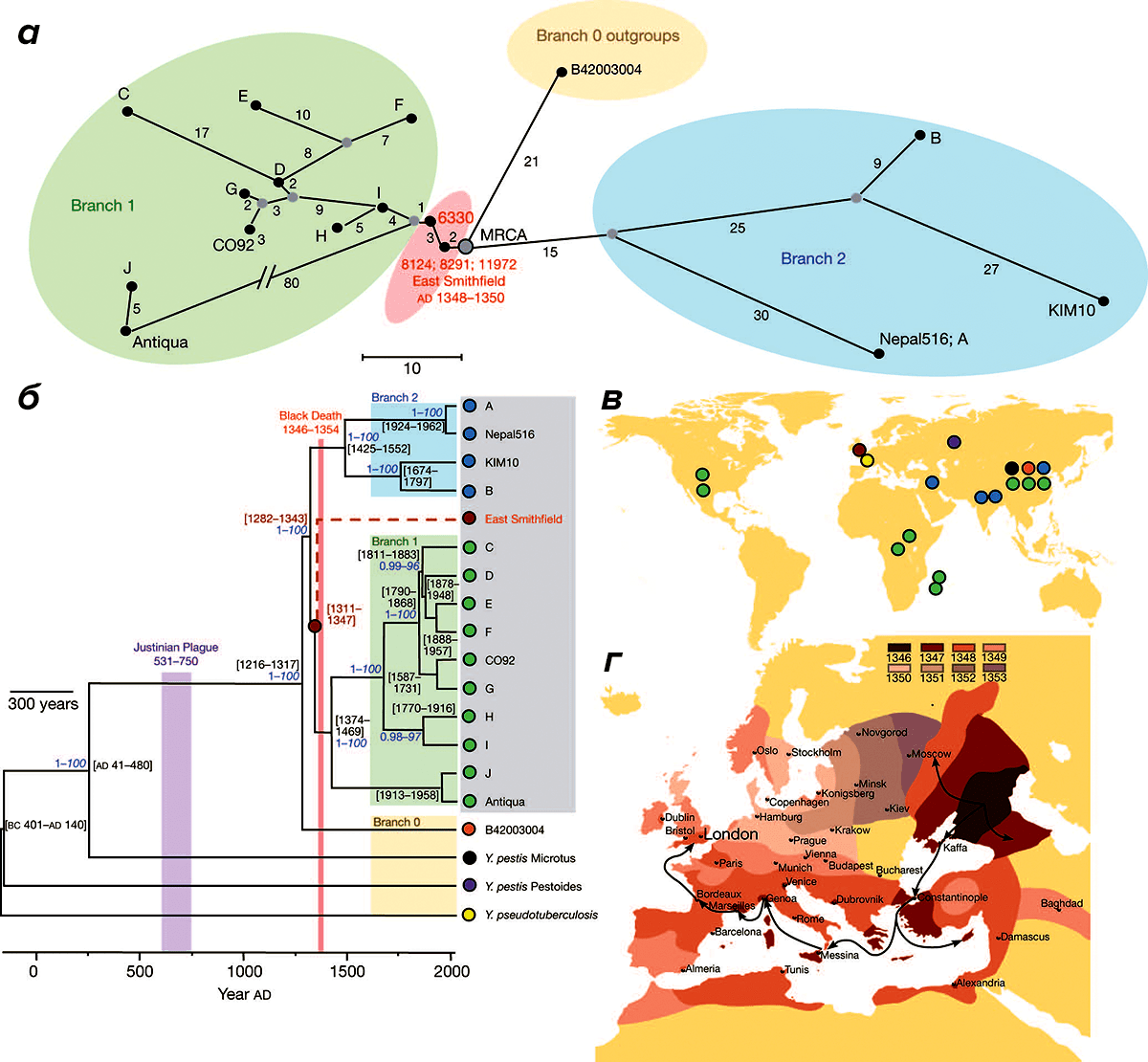
Рисунок 1. Филогенетическое положение и история штамма из Восточного Смитфилда. а и б — Разные графические представления филогенетических деревьев, построенных на основе последовательностей генома древнего и современных штаммов чумной палочки. Цветами показаны разные ветви ныне живущих штаммов. в — Регионы, из которых брались образцы ДНК чумных палочек для исследования. г — Схема распространения Черной смерти по Европе и прилегающим территориям.
Авторы исследования считают, что общий предок секвенированного ими штамма и ныне живущих возбудителей чумы циркулировал буквально за столетие до того, как появилась Черная смерть, что якобы говорит о том, что линии чумной палочки, ответственные за более ранние эпидемии (например, Юстинианова чума 551–580 гг.), бесследно вымерли. Однако с этим не согласны многие исследователи, указывающие на то, что команда Пойнара и Краузе не рассмотрела многие штаммы чумы из восточной Азии, которые, как считается, имеют более древних предков.
Анализ генома средневековой Yersinia pestis не выявил радикальных отличий от геномов современных штаммов. Авторы склоняются к тому, что то невероятное количество жертв, которое унесла Черная смерть, связано не столько с особенностями штамма, но в значительной мере с факторами окружающей среды, динамикой распространения через организмов-носителей (некоторые ученые предполагают, что бубонную чуму XIV века могли переносить не блохи, а кто-то другой) и восприимчивостью к патогену тогдашнего населения Европы.
Написано по материалам Nature News [6].
Литература
- Kirsten I. Bos, Verena J. Schuenemann, G. Brian Golding, Hernán A. Burbano, Nicholas Waglechner, et. al.. (2011). A draft genome of Yersinia pestis from victims of the Black Death. Nature. 478, 506-510;
- Scott S. and Duncan C.J. Biology of plagues: evidence from historical populations. Cambridge: Cambridge University Press, 2001;
- D. Raoult, G. Aboudharam, E. Crubezy, G. Larrouy, B. Ludes, M. Drancourt. (2000). Molecular identification by "suicide PCR" of Yersinia pestis as the agent of Medieval Black Death. Proceedings of the National Academy of Sciences. 97, 12800-12803;
- M. T. P. Gilbert. (2004). Absence of Yersinia pestis-specific DNA in human teeth from five European excavations of putative plague victims. Microbiology. 150, 341-354;
- R. E. Green, J. Krause, A. W. Briggs, T. Maricic, U. Stenzel, et. al.. (2010). A Draft Sequence of the Neandertal Genome. Science. 328, 710-722;
- Ewen Callaway. (2011). Plague genome: The Black Death decoded. Nature. 478, 444-446;
- Важнейшие методы молекулярной биологии и генной инженерии;
- Огурцы-убийцы, или Как встретились Джим Уотсон и Гордон Мур;
- Код жизни: прочесть не значит понять;
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК).