Процессы и эпигеном
11 февраля 2022
Процессы и эпигеном
- 1395
- 0
- 12
Транспозаза встраивает ДНК-транспозон, взаимодействуя с белками транскрипции на хроматине
иллюстрация Михаила Гурьева
-
Автор
-
Редакторы
-
Рецензенты
-
Иллюстратор
Уже разобравшись в рамках спецпроекта «Эпигенетика», что такое хроматин и почему он организуется в домены, сегодня мы остановимся на эпигенетической стороне такой «классики» молекулярной биологии, как транскрипция, репликация и репарация ДНК; обсудим транспозицию; и отдельное внимание уделим взаимодействию этих процессов. Также мы поговорим об эпигенетике в контексте общебиологических процессов: дифференцировки клеток, развития, видообразования и иммунного ответа.
Эпигенетика
Эпигенетика — с одной стороны, молодая и бурно развивающаяся наука. С другой стороны, в этой науке уже сложился фундамент основных понятий и концепций. Цель спецпроекта — рассказать об основных концепциях эпигенетики просто, но в то же время со всех сторон. Читатель узнает, какие молекулы и структуры стоят за передачей наследственной информацией и какими механизмами осуществляется эта передача. Часть контента позаимствована из блога автора Friends of Chromatin.

Генеральный партнер спецпроекта — компания «Диаэм».
Это крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
В предыдущих статьях спецпроекта по эпигенетике мы рассказали, из чего состоит хроматин и почему в ядре он организован по доменному принципу [1], [2]; а также рассмотрели наследование эпигенетической информации в контексте репликации, митоза и целого организма [3].
Сегодня мы рассмотрим, как клетка изменяется в течение всего клеточного цикла, и какие эпигенетические процессы сопровождают эти изменения. Мы уделим внимание как делению клетки, так периоду между делениями. В частности, мы рассмотрим такую «классику» молекулярной биологии, как транскрипция, удвоение, починка поломок в ДНК и транспозиция, а также взаимодействие между этими процессами — и всё это в эпигенетическом свете. В заключение мы поговорим о роли эпигенетических явлений в контексте общебиологических процессов: например, в развитии и нейрогенезе.
Клеточный цикл, митоз и мейоз — удивительное деление клетки
Жизненный путь клетки
В качестве введения мы рассмотрим клеточный цикл и место в нем классических процессов молекулярной биологии. Тут будет совсем немного эпигенетики, но зато читатель получит базу для понимания того, какую роль она играет на всем протяжении жизненного пути клетки. Цикл соматической клетки — это интервал между «рождением» клетки путем деления родительской до того момента, как она уже сама поделится, давая жизнь своему клеточному потомству. Традиционно этот цикл делят на четыре периода: фазы G1 и G2 (от англ. gap — «промежуток»), S (от англ. synthesis — «синтез» [ДНК]) и M (от англ. mitosis — «митоз» ), когда клетка делится (рис. 1).
Кроме митоза есть и другой тип клеточного деления — мейоз: таким путем получаются уже не диплоидные соматические, а гаплоидные половые клетки, — однако подробно на этом мы останавливаться не будем.
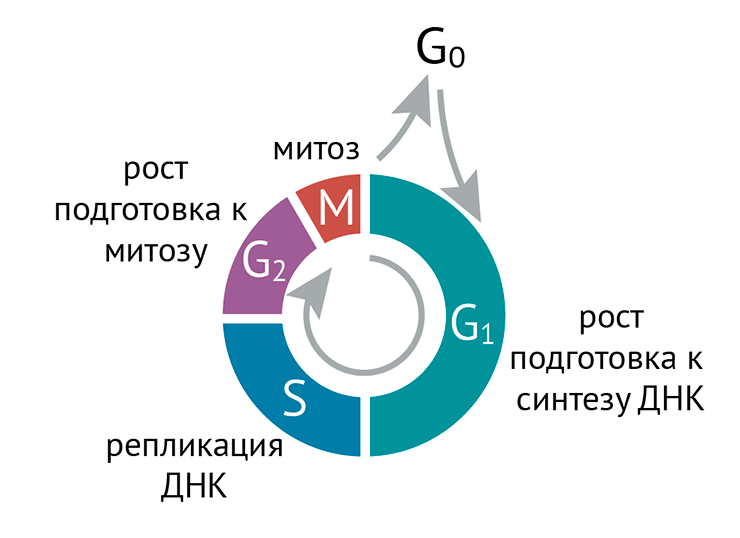
Рисунок 1. Схема клеточного цикла. Фаза G1 следует сразу за митозом, вслед за ней — S-фаза, во время которой удваивается ДНК; потом идет G2, а за ней уже вновь настает очередь митоза. Относительная длительность фаз у разных клеток разная.
иллюстрация Михаила Гурьева по материалам Thermofisher
Переход между фазами регулируют специальные белки — циклины и циклин-зависимые киназы (Cyclin-Dependent Kinases, CDKs), выборочно взаимодействующие друг с другом на разных этапах клеточного цикла. При формировании таких комплексов CDKs активируются и могут фосфорилировать другие белки — например, транскрипционные факторы, запускающие чтение генов клеточного цикла. Митоз тоже делят на определенные периоды — фазы (рис. 2).
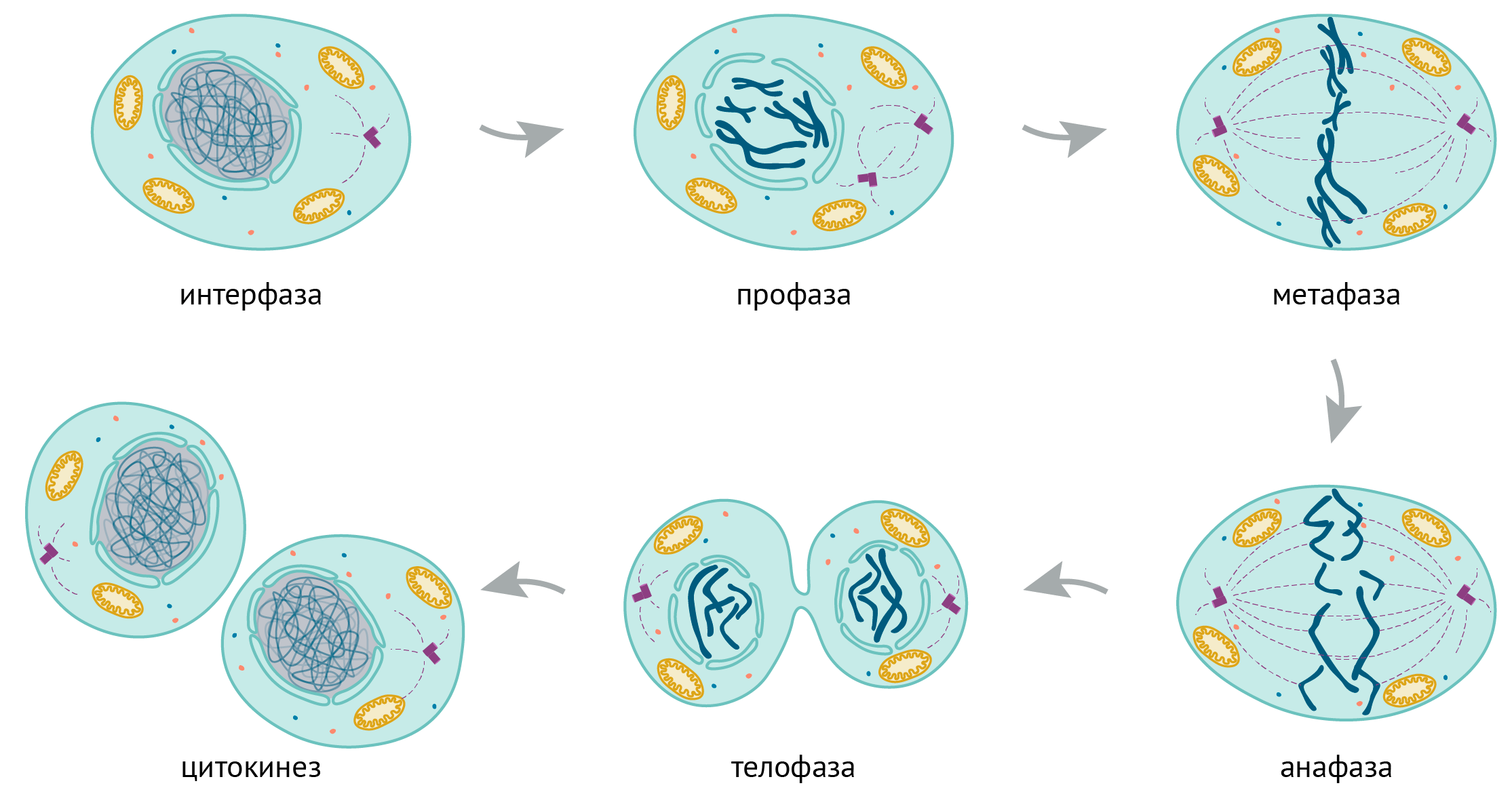
Рисунок 2. Фазы митоза. Во время профазы хромосомы конденсируются — особый белковый комплекс конденсин укладывает хроматин в петли, исходящие из остова, сформированного конденсином и топоизомеразой II [4]. Оболочка ядра в профазе разбирается. Во время прометафазы формируется веретено деления — симметричные пучки микротрубочек, исходящие из двух центров — центросом. Такие микротрубочки крепятся к центромерам хромосом через сложные белковые структуры — кинетохоры — и подталкивают хромосомы к экватору клетки. Во время метафазы сестринские хроматиды выстраиваются парами вдоль экватора друг напротив друга, сцепляясь когезином. Позже когезин расщепляется белком сепаразой, и это дает начало анафазе: микротрубочки укорачиваются и утаскивают хроматиды от экватора к полюсам клетки — происходит расхождение хромосом. Вслед за этим веретено деления разбирается, хромосомы становятся менее компактными. Другими словами, хромосомы деконденсируются — то есть исчезают конденсиновые петли и постепенно устанавливается интерфазная организация генома (уже когезином). Вновь строится оболочка ядра. Эта стадия называется телофазой. Потом клетка делится на две — происходит цитокинез. Что регулирует компактизацию хромосом? Очень вероятно, что это модификации гистонов. Это было показано для дрожжей [5], а также в экспериментах на куриных клетках: если очистить гистоны из хроматина интерфазных клеток, то они будут образовывать менее компактные структуры, чем очищенные гистоны митотических клеток [100].
иллюстрация Михаила Гурьева по материалам CK-12
Конденсин может участвовать в формировании петель и трехмерных доменов и во время интерфазы. Это характерно для покоящихся клеток дрожжей — не делящихся, но потенциально способных к этому [6].
Не все процессы молекулярной биологии протекают в течение клеточного цикла равномерно: например, транскрипция в митозе по большей части выключается (за исключением центромер), а репарация не останавливается и в митотической фазе. Гомологичная рекомбинация, для которой нужны две гомологичные хромосомы, не происходит в фазе G1, пока ДНК не реплицировалась. А сама репликация происходит по большей части в S-фазе, хотя некоторые сайты реплицируются и в митозе [7].
Как изучают деление клетки?
Казалось бы, всё описанное выше — хорошо известные вещи, давно нашедшие пристанище в учебниках (в том числе школьных) и на страницах «Википедии». Однако детали всех этих процессов до сих пор довольно туманны, так что исследования продолжаются. Какие же технологии можно применить, чтобы разглядеть нюансы клеточного деления? Самое первое, что тут придумали — это микроскопия [8], в том числе флуоресцентная [9]. Но, конечно, этого мало. В микроскоп можно смотреть только на отдельные клетки, да и то с небольшим разрешением.
Чтобы изучить отдельные стадии клеточного цикла, было бы неплохо «застопорить» клетки в митозе и уже потом «разобрать» их на части (белки, РНК и т.д.), а еще их можно проанализировать такими мощными современными методиками, как Hi-C [10]. Добиться такой «заморозки» можно с помощью химической деполимеризации клеточного цитоскелета (например, колхицином или нокодазолом [11]), что приведет к «накоплению» клеток в стадии митоза — однако входить в него они будут не синхронно, а потому тонкие эффекты отдельных митотических стадий ускользнут от взора исследователей.
Есть и другой способ: если заблокировать циклин-зависимую киназу 1 (Cdk1), клетки вообще не войдут в митоз, а будут накапливаться в фазe G2 (рис. 1). Так вот, есть искусственный вариант этого белка, который можно обратимо заблокировать в G2 перед митозом, застопорить клетки, а затем синхронно выпустить их в митоз. Таким образом можно изучать, как меняется композиция митотических хромосом по мере деления клетки на разных стадиях [12], [13].
Ранее мы рассказывали об определении структуры хроматина для одиночных клеток [14], [15]. Оказывается, эти методы тоже позволяют детализировано исследовать процесс клеточного цикла. Так, в результате Hi-C одиночных клеток можно получить контактные карты хромосом клеток на всевозможных стадиях цикла. С помощью биоинформатического анализа их можно разделить на M-, S- и G-фазы и исследовать сразу тысячи структур хромосом разных стадий [16].
В молекулярной биологии очень много исследований, когда «выключают» тот или иной белок [17] и смотрят, «чего будет». Так же и с митозом, но только здесь мы имеем дело с довольно быстрым процессом, а потому распространенная методика «выключения» белка РНК-интерференцией не очень-то подходит: на такое воздействие клетка реагирует несколько дней , а в этом случае хочется ответа за час-другой (чтобы взять и «выключить» интересующий белок прямо перед митозом).
Соответственно, нужно с осторожностью интерпретировать результаты работ, где уровень белка снижается РНК-интерференцией, поскольку клетки часто успевают адаптироваться, пока белок из них медленно исчезает. Было показано, например, что если «плавно выключить» конденсин в клетках РНК-интерференцией и «вырубить» его прямо перед митозом, то это ведет к абсолютно разным фенотипам [18], [19].
Однако какая технология позволяет «вырубить» белок за считанные часы? Это направленная деградация : изучаемый белок сшивают с дегроном AID (auxin inducible degron) — белком, который быстро распадается после взаимодействия с (внезапно) растительным гормоном ауксином [20]. В клетках синтезируют дополнительный белок TIR1 (transport inhibitor response), который и направляет AID на деградацию вместе со слитым с ним митотическим белком в ответ на взаимодействие с ауксином. Эта технология пригодна для любой стадии клеточного цикла, но ее часто применяют для изучения именно митоза.
Принцип направленной деградации используют и в фармакологии, когда похожим дегроном метят неугодную мишень для убиквитиновой деградации [21].
Ошибки в делении клетки — что может пойти не так с хромосомами
Как и в любом процессе, в делении клетки возможны ошибки. Например, гомологичные хромосомы могут сливаться своими концами — теломерами, — формируя ковалентные мостики. Это приводит не только к неравным наборам хромосом при делении, но и к патологическим перестройкам в хромосомах на протяжении последующих делений клетки [22]. Некоторые хромосомы могут запаздывать с расхождением, формировать отдельные микроядра в дочерних клетках. Всё это вызывает нестабильность генома и может вести к различным заболеваниям, в том числе к раку.
Репликация ДНК: удвоить и пересобрать
Вспомнив в общих чертах клеточный цикл, давайте теперь обратимся к конкретным процессам молекулярной биологии, имеющим эпигенетическую регулировку. И здесь в деле все игроки эпигенетики, которые нам уже встречались ранее — это и гистоновые модификации, и ремоделеры, и транскрипционные факторы. Что касается репликации: в ее регуляции специальная роль отведена гистоновым модификациям и белкам, которые ими манипулируют, — «читателям» и «писателям». Они особо отмечают новосинтезированный хроматин, а также маркируют места ранней и поздней репликации. Однако сначала напомним азы процесса копирования ДНК — ну а те, кто хорошо это знают, могут врезку смело пропустить.
Какие гистоновые модификации регулируют репликацию? Оказывается, отсутствие модификации тоже может служить сигналом: с меткой H4K20me0 (неметилированным остатком лизина K20 на гистоне H4) связываются специальные белки TONSL и BARD1, причем последний привлекает белок репарации ДНК BRCA1 (прославившийся своей ролью в возникновении рака груди [24], [25]) . Похоже, это взаимодействие инициирует починку двухцепочечных (дц) разрывов ДНК — гомологичную рекомбинацию (см. далее [26], [27]). Таким образом чинится новореплицированная ДНК. В мутантах BARD1, которые не могут связывать H4K20me0, гомологичная рекомбинация не работает.
Да, получается, что это белки-читатели немодифицированного гистона. Открыли их, инкубировав такие немодифицированные нуклеосомы с клеточным экстрактом и посмотрев, какие белки с ними связались.
Более того, не все домены реплицируются одновременно, и гистоновые модификации коррелируют со временем репликации. Точек начала репликации — так называемых ориджинов — очень много. И репликация начинается на них не одновременно: сначала реплицируются «ранние» фрагменты хромосомы, потом «поздние», и всё это сшивается воедино лигазой.
Домены, которые реплицируются раньше, обогащены активными гистоновыми метками (например, H3K4me3 и H3K36me3). Более поздние несут на себе метки неактивного хроматина (например, H3K9me3) [28]. В раковых клетках, помимо потери метилирования ДНК на поздних участках репликации, H3K9me3 по большей части заменяется на H3K27me3. Многие «поздние» участки в этих клетках переходят на темную сторону и начинают реплицироваться «рано», меняя рисунок генетической экспрессии.
Гистоновые метки не просто маркируют, но и управляют временем репликации: белки, навешивающие эти метки, регулируют и старт удвоения ДНК [29]. Например, в дрожжах, мутантных по гистондеацетилазе, гистоны ацетилированы (это активные метки), что приводит к ранней активации поздних участков репликации. Однако нужно понимать, что эффект может быть непрямым — гистоновые модификации могут, например, повлиять на транскрипцию или укладку хроматина в пространстве, что уже аукнется времени старта репликации.
Как изучают репликацию?
Значительная часть методов изучения репликации основана на синхронизации (накоплении) клеток в S-фазе и инкубации их с биотинилированными дезоксинуклеотидами, которые вставляются в ДНК во время репликации. Соответственно, новосинтезированные фрагменты ДНК можно изолировать и изучить — какие белки связываются с новосинтезированным хроматином [30]; или сконцентрироваться на одном белке или одной гистоновой модификации и сделать аналог ChIP-секвенирования на новосинтезированном хроматине [31]; или сделать ATAC-секвенирование, которое позволяет посмотреть, насколько «открыт» хроматин в отдельных местах [32].
Эти методы выявили много новых репликационных белков, показали аккуратное перераспределение гистонов с родительского дуплекса на дочерние цепи (см. статью «Наследование и эпигеном» [3]) и обнаружили, что рестарт транскрипции (когда она запускается уже на новосинтезированной ДНК) нужен не просто для того, чтобы шла экспрессия генов, но и чтобы установить открытое состояние хроматина (когда он менее компактный и более рыхлый) после репликации — это занимает до 30 минут после репликации.
Если же ингибировать транскрипцию, то паттерн ATAC-секвенирования (а следовательно, и паттерн доступности) новореплицированного хроматина изменится, показывая, что рестарт транскрипции не выполнил этой своей служебной функции.
ДТП на магистралях хроматина: репликационная вилка въезжает в РНК-полимеразу
Как и в течение всего клеточного цикла, по мере работы репликационной вилки в ДНК возникают дц-разрывы, починка которых основана на гомологичной рекомбинации. Когда эти разрывы возникают под репликационной вилкой, активируются сигнальные каскады, запускающие починку. Это может сопровождаться резекцией синтезирующихся цепей на репликационной вилке (образованием протяженных одноцепочечных участков), обращением вилки вспять (когда только что синтезированные одноцепочечные цепи ДНК откидываются назад и отжигаются друг на друге, рис. 3), а также разрезанием репликационной вилки [33].
В месте репликационной вилки хроматин отличается по своим свойствам — ведь в нем есть длинные одноцепочечные участки ДНК, затравки из РНК, а еще весь арсенал репликационных белков.
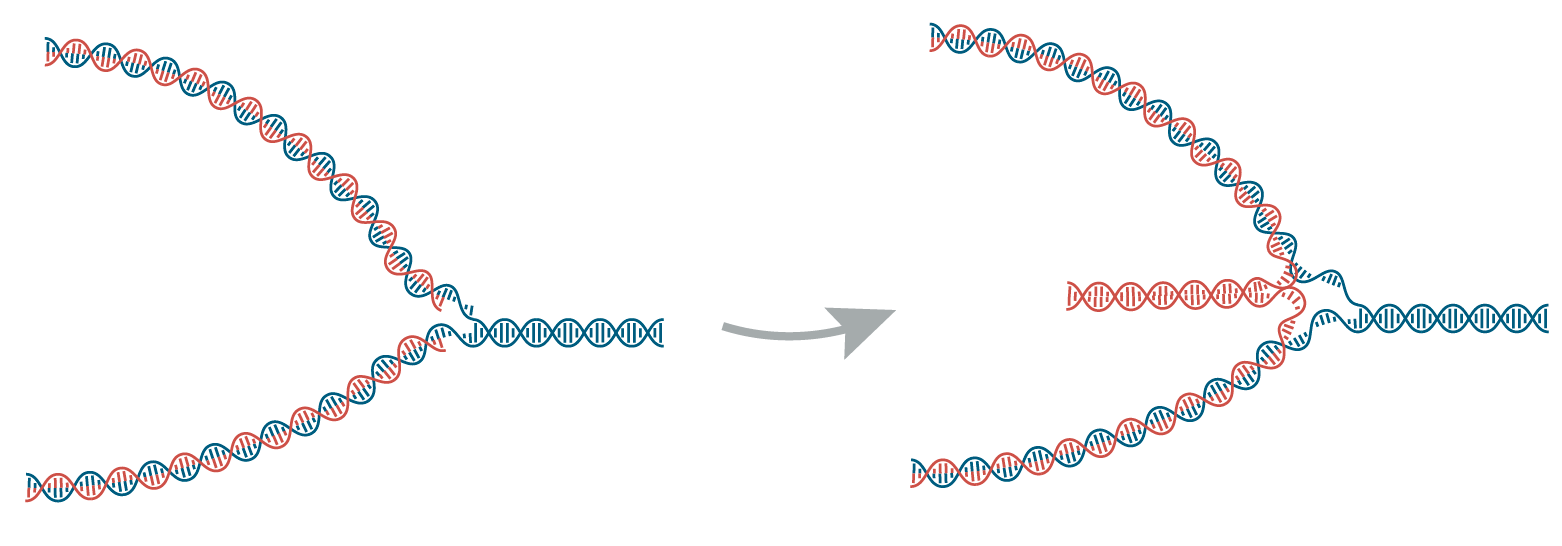
Рисунок 3. Обращение репликационной вилки вспять. Родительские цепи ДНК показаны синим, дочерние — красным. Синтезирующиеся участки дочерних цепей (справа) откидываются назад (налево) и отжигаются друг на друге.
иллюстрация Михаила Гурьева
Последние два процесса могут происходить и в том случае, если репликационная вилка встречает РНК—ДНК-гибриды, образующиеся по мере транскрипции, — то есть когда репликация «врезается» в транскрипцию, которая не прекращается и в S-фазе (ведь нужно же синтезировать гистоны, например). При этом репликационная вилка может скинуть РНК-полимеразу с ДНК [34], но иногда этого недостаточно — уже синтезированный РНК-транскрипт отжигается на ДНК и тоже мешает репликации. Когда в клетке слишком много таких РНК—ДНК-гибридов, активируется тот же самый сигнальный каскад, который запускает репарацию ДНК; центральную роль тут играет киназа ATR .
Активация киназы ATR требует двух вещей: обращения репликационной вилки вспять (рис. 3) и ее разрезания эндонуклеазой MUS81, которая, отработав, «утихомиривается» от излишней активности той же ATR.
Получается, репликационная вилка намеренно разрезается, чтобы просигналить клетке о поломках ДНК [35] — это как дернуть стоп-кран на полном ходу, чтобы дать клетке возможность ликвидировать бардак в форме излишков РНК—ДНК-гибридов. Прежде чем клетка сможет продолжить расти и делиться дальше, должна пройти репарация, в ходе которой РНКаза H вырежет РНК из гибридов, иными словами, устранит «ДТП» с участием репликационных белков и аппарата транскрипции [36]. У читателя может возникнуть законный вопрос: а можно ли было на время репликации вообще остановить транскрипцию? Наверное, можно, но, как это принято говорить, «так пошла эволюция», а ее путь — не всегда самый прямой и короткий.
Транскрипция, или как читается книга генома
Транскрипция — это «переписывание» части генетической информации с «бобины» ДНК на «временный носитель» — РНК (рис. 4); при этом дуплекс ДНК расплетается, а РНК считывается с кодирующей цепи при помощи РНК-полимеразы (это комплекс из множества субъединиц, различающихся для разных изоформ фермента). Транскрипция реализует экспрессию генов — когда информация, лежащая на ДНК «мертвым грузом», идет в работу, определяя дальнейший путь клетки и организма.
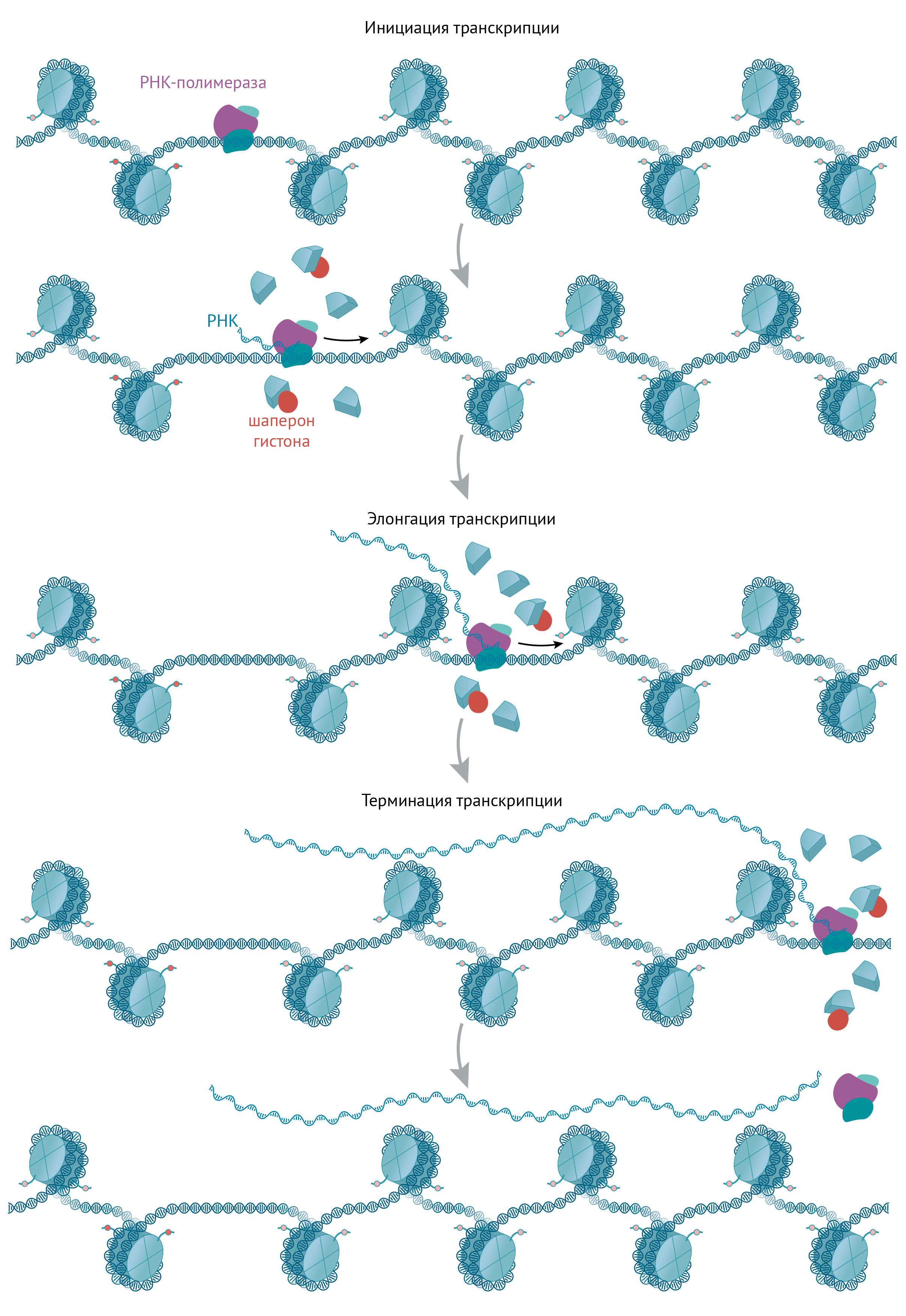
Рисунок 4. Стадии транскрипции. Начало процесса называют инициацией транскрипции, продолжение — элонгацией, а окончание — терминацией [37]. При этом в зависимости от стадии транскрипции некоторые субъединицы РНК-полимеразы могут присоединяться и отсоединяться (есть факторы инициации, элонгации и терминации транскрипции), а часть одного из белков комплекса, называемая «хвостом» РНК-полимеразы, может по-разному фосфорилироваться [38]. Факторами транскрипции могут быть не только белки, но и молекулы РНК, например, 7SK РНК в комплексе с белками регулирует элонгацию транскрипции [39].
иллюстрация Михаила Гурьева
После того как РНК-полимераза II транскрибирует 20–120 нуклеотидных остатков, она делает паузу. Для того чтобы она читала дальше, требуются факторы элонгации: это дополнительный способ регуляции транскрипции [40]. Элонгация — процесс непостоянный, который может прерываться не только из-за остановки РНК-полимеразы, но и при столкновении с нуклеосомами и другими полимеразами на хроматине (в том числе репликационными), а также в связи с поломками в самой ДНК [37].
Как прочитать ДНК и кто помогает в этом полимеразе?
Как и в репликации, в транскрипции играют роль эпигенетические регуляторы. В предыдущих статьях спецпроекта мы уже разбирали, как транскрипция регулируется гистоновыми модификациями и ремоделерами, которые позиционируют нуклеосомы вокруг сайта начала транскрипции; а теперь давайте посмотрим, как на нее влияют шапероны гистонов и транскрипционные факторы.
Как уже упоминалось в статье «Ядро и эпигеном» [2], во время транскрипции нуклеосомы, которые попадаются по пути РНК-полимеразе, разбираются шаперонами гистонов, а потом собираются обратно. Пожалуй, самый изученный такой шаперон — FACT: он умеет как собирать нуклеосомы, так и разбирать их, «вынимая» оттуда один из димеров H2A-H2B и немного разворачивая ДНК [41], [42]. Недавно лаборатория Каролин Лугер определила строение FACT с полной и неполной нуклеосомой (рис. 5) [41] — то есть прямо за работой! FACT не одинок в своей работе по регуляции транскрипции: тут задействованы и другие связанные друг с другом шапероны. Если нарушить взаимодействие между шаперонами Spt6 и Spn1 в дрожжах, то количество FACT на хроматине сильно повышается, и в транскрипции возникают дефекты. Это можно преодолеть, заставив FACT меньше взаимодействовать с хроматином [43].
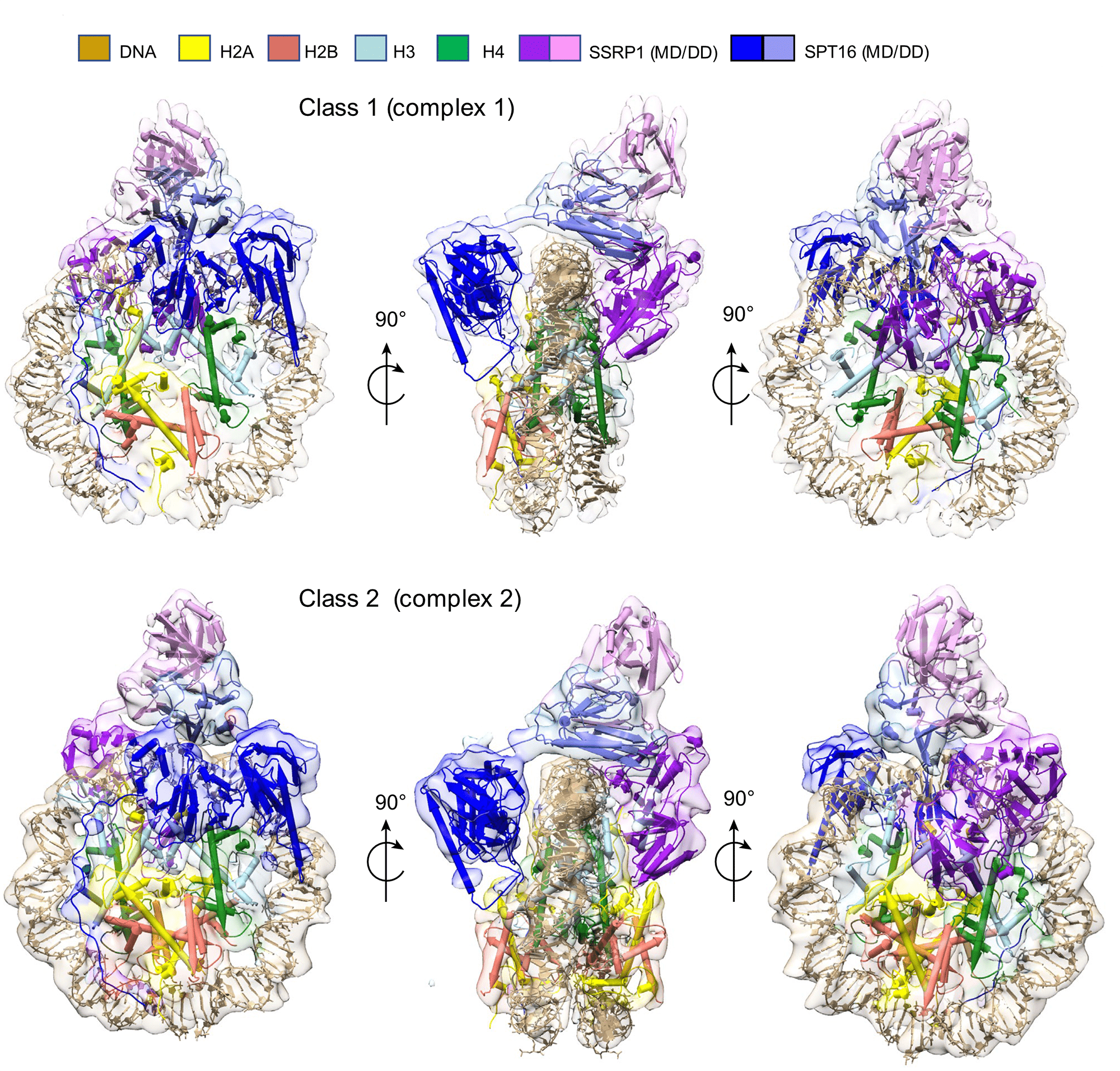
Рисунок 5. FACT за работой. Это изображение суммирует структуру FACT (субъединицы SSRP1 и SPT16) в комплексе с полной и неполной нуклеосомой. Тут FACT напоминает седло, которое располагается на колесе (тетрамере H3-H4). К колесу присоединяются одна или две «педальки» — димеры H2A-H2B.
Для транскрипции важны не только шапероны, но и ремоделеры, которые четко позиционируют нуклеосомы на промоторе относительно сайта начала транскрипции: это нужно для корректного связывания транскрипционных факторов (ТФ), которые уже помогают полимеразе сесть на промотор. Если ремоделер подвинет нуклеосому, которая в результате закроет сайт посадки ТФ, транскрипция может пойти хуже или вовсе остановиться. Более того, некоторые ремоделеры могут вообще «столкнуть» ТФ с хроматина [44]!
Транскрипция встречается с выпетливанием хроматина — столкновение гигантов
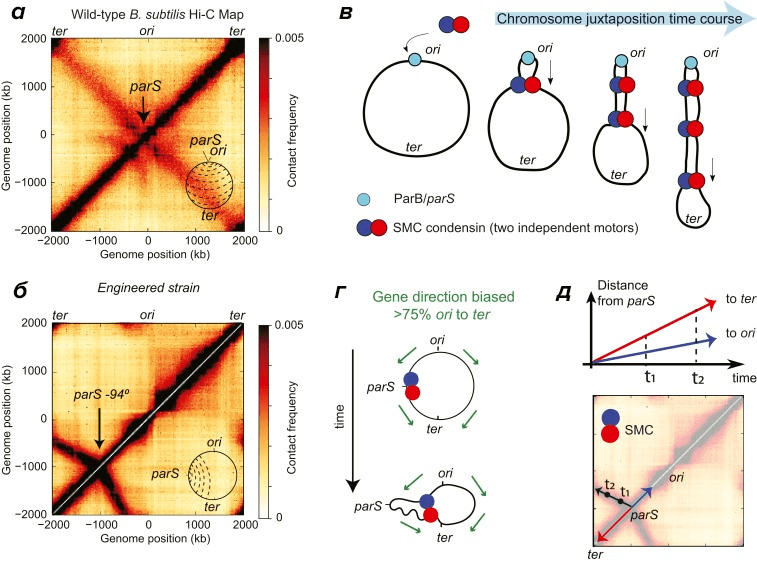
Выпетливание хроматина, о котором уже шла речь раньше [2], — важный этап пространственной организации генома: белки конденсины или когезины окольцовывают обе цепи ДНК и формируют петлю, активно по ним перемещаясь. Конечно, на образование петель влияют и прочие хроматиновые процессы, тем самым изменяя и трехмерную структуру генома. Самый очевидный такой процесс — конечно, транскрипция. То, как она затрагивает выпетливание, лучше всего изучено в бактериях. Хромосомы у них кольцевые, и конденсин к ним крепится белками ParB, распознающими в ДНК особые последовательности ParS. Расположены они рядом с ориджинами репликации (ori) — местами начала удвоения ДНК (рис. 6).
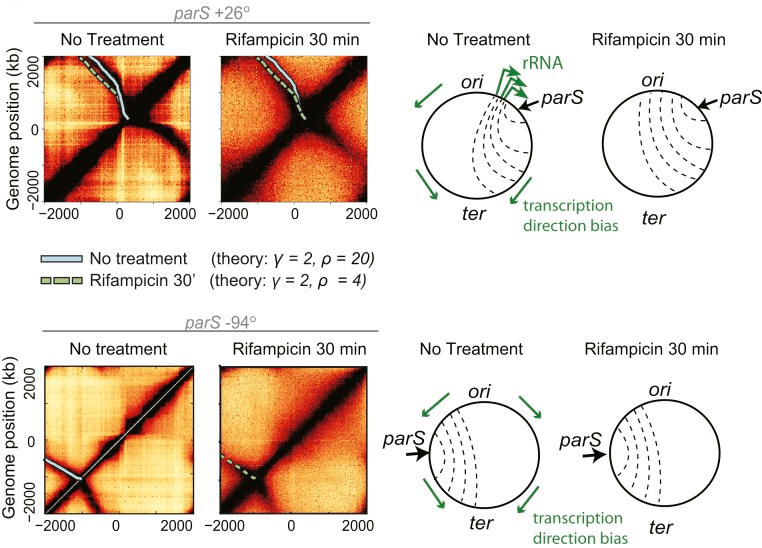
Похоже, когда конденсин перемещается по ДНК в бактериях, полимеразы ему не мешают. Только очень интенсивная транскрипция мешает перемещению конденсина по ДНК, тормозя его встречными РНК-полимеразами и соответственно влияя на организацию генома в пространстве. Похожая ситуация наблюдается у млекопитающих: на активных генах когезин накапливается возле сайтов начала транскрипции и на 3′-концах генов (как и РНК-полимераза II). Ученые думают, что РНК-полимераза — передвижной барьер на пути выпетливания хроматина. В согласии с этой моделью, в клетках, где транскрипция ингибирована, структура генома вокруг активных генов ослабевает (и хроматин делается более рыхлым) [46].
Репарация ДНК
На хроматине могут возникать поломки: разрывы одной или сразу двух цепочек ДНК; отсутствие части одной цепи ДНК; замена нуклеотидных остатков в ДНК (так что нарушается комплементарность); сшивки оснований, вызванные ультрафиолетом; а также сшивки ДНК с хроматиновыми белками (рис. 7). Кое-что из этого возникает в норме (сопутствуя нормальным процессам на хроматине), но чаще всего это признак поломки. А поломки необходимо чинить — и применительно к ДНК это называется репарацией. Мы рассмотрим те из поломок и способы их репарации, где изучена связь с эпигенетикой — а именно, починку дц-разрывов ДНК и вставку урацила в ДНК.
Ну а если их не чинить, или когда клетка или организм не справляются с большим числом поломок, возникают заболевания. Например, рак, атаксия-телеангиэктазия или пигментная ксеродерма — когда кожа обладает повышенной чувствительностью к ультрафиолету. Именно поэтому так важно, чтобы все белки, задействованные в починке ДНК, функционировали как надо.
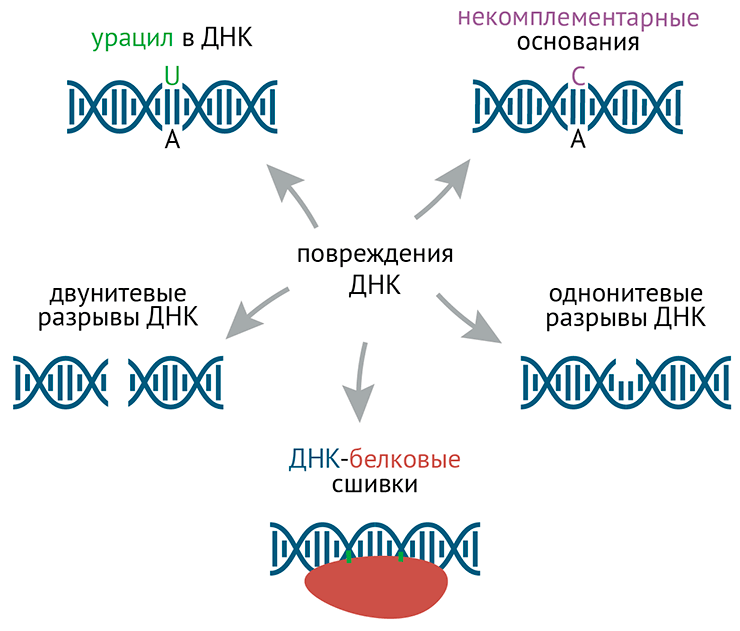
Рисунок 7. Типы поломок ДНК на хроматине
иллюстрация Михаила Гурьева
Вставка урацила в ДНК
Частный случай поломок в ДНК — вставка урацила (основания РНК) вместо тимина (см. рис. 7); он попадает туда во время синтеза ДНК или получается дезаминированием цитозина. Урацил в ДНК обычно распознается как ошибка и замещается на «правильный» тимин белком UNG (урацил-N-гликозилазой). Места замены T → U можно картировать, если использовать мутантный UNG, распознающий урацил, но не вырезающий его из ДНК: эти места можно пометить антителами к ферменту, а потом выделить и секвенировать.
Однако бывает и так, что урацил появляется в ДНК «законно» в нормальных процессах — например, при рекомбинантной сборке генов антител.
Парадоксально, что эта, казалось бы, случайная мутация T → U может быть управляемой: те точки, куда должен вставиться урацил, помечает эпигенетика!
Оказалось, что урацил «в норме» вставляется преимущественно в AT-богатые участки гетерохроматина, а если ингибировать UNG, то и в GC-богатые, и в хроматиновые домены Polycomb [47]. Таким образом, не все гены равновероятно получат урациловую мутацию. Поскольку урацил вставляется в ДНК в том числе во время репликации, изменение клеточного цикла повлечет за собой и изменение урацилового паттерна. Ингибирование UNG задерживает клеточный цикл, и урацил начинает встраиваться в места ранней репликации.
Таким образом, определенные гены имеют больший шанс «заразиться» урацилом. Но если ингибировать UNG, то клеточный цикл смещается, и под угрозой оказываются уже другие гены в совсем других местах генома.
Починка двухцепочечных разрывов в ДНК
Дц-разрывы в ДНК опасны для генома, поскольку неправильная починка может привести к рекомбинации и повлечь заболевания, в том числе — рак. Существует два основных пути починки таких поломок — негомологичное соединение концов и гомологичная рекомбинация.
Места дц-разрыва помечаются фосфорилированием H2AX (варианта гистона H2A) при помощи киназ ATM (ataxia telangiectasia mutated, мутированной в атаксии-телеангиэктазии), ATR (ataxia telangiectasia and Rad3-related protein) и других. Эти киназы, активируемые комплексом Ku70/80, инициируют починку ДНК и останавливают клеточный цикл, если разрывов очень много. Есть и другие гистоновые модификации, отмечающие место разрыва [33], — все они привлекают белки, зашивающие разрыв.
По одной из гипотез, фосфорилированный H2AX повышает доступность хроматина, а также помогает тасовать нуклеосомы в местах разрыва, «расчищая» место работы репарационных белков [49].
H2AX фосфорилируется при разрывах на протяжении всего клеточного цикла — как в интерфазе, так и в митозе. Эти модификации удается зарегистрировать по иммунофлуоресценции в форме небольших пятнышек, число которых соответствует скоплениям разрывов ДНК. Поскольку репарацию ДНК часто ингибируют при лечении рака (чтобы дестабилизировать геном раковых клеток и убить их), количество пятнышек H2AX используют как маркер починки ДНК в опухолях пациентов [49].
Что определяет выбор пути починки дц-разрывов ДНК? Во-первых, это фаза клеточного цикла — гомологичная рекомбинация возможна только в S- и G2-фазах. Во-вторых, присутствие длинных одноцепочечных участков ДНК на концах в месте разрыва — если они есть, то выбор останавливается на гомологичной рекомбинации. В-третьих, это масштаб повреждений — если нужно быстро сшить много разрывов (возникших, например, при ультрафиолетовом облучении), в ход идет негомологичное соединение концов. На выбор пути починки влияет и контекст хроматина в месте разрыва [33]: например, в фазе G2 гетерохроматин по большей части застраивается гомологичной рекомбинацией.
ChIP-секвенирование некоторых факторов починки показало, что они кучкуются на активных генах: вероятно, потому что изрядная часть поломок происходит в местах столкновения транскрипции и репликации (о чем мы уже писали выше). Но нельзя исключить, что есть какие-то дополнительные (кроме H2AX) эпигенетические метки или факторы транскрипции, привлекающие белки репарации [33].
Таким образом, хроматиновый контекст отчасти определяет паттерн урациловых мутаций, а также привлекает белки, зашивающие дц-разрывы ДНК, и помогает выбрать путь починки таких разрывов.
Транспозиция и как ей помешать
Транспозоны (одна из разновидностей мобильных генетических элементов) — это последовательности ДНК, которые могут перемещаться по геному. Выделяют ДНК-транспозоны, которые вырезаются из дуплекса и встраиваются в другое место генома; и ретротранспозоны, в жизненном цикле которых присутствует стадия РНК [50–55]. Они транскрибируются, а перед тем, как встроиться в другое место хроматина, осуществляют обратную транскрипцию: РНК → ДНК. Вставкой ДНК транспозона в геном занимается специальный белок — интеграза. Места встраивания определяются взаимодействиями интегразы с хроматиновыми белками (рис. 8) — часто это участники транскрипции или репликации ДНК.
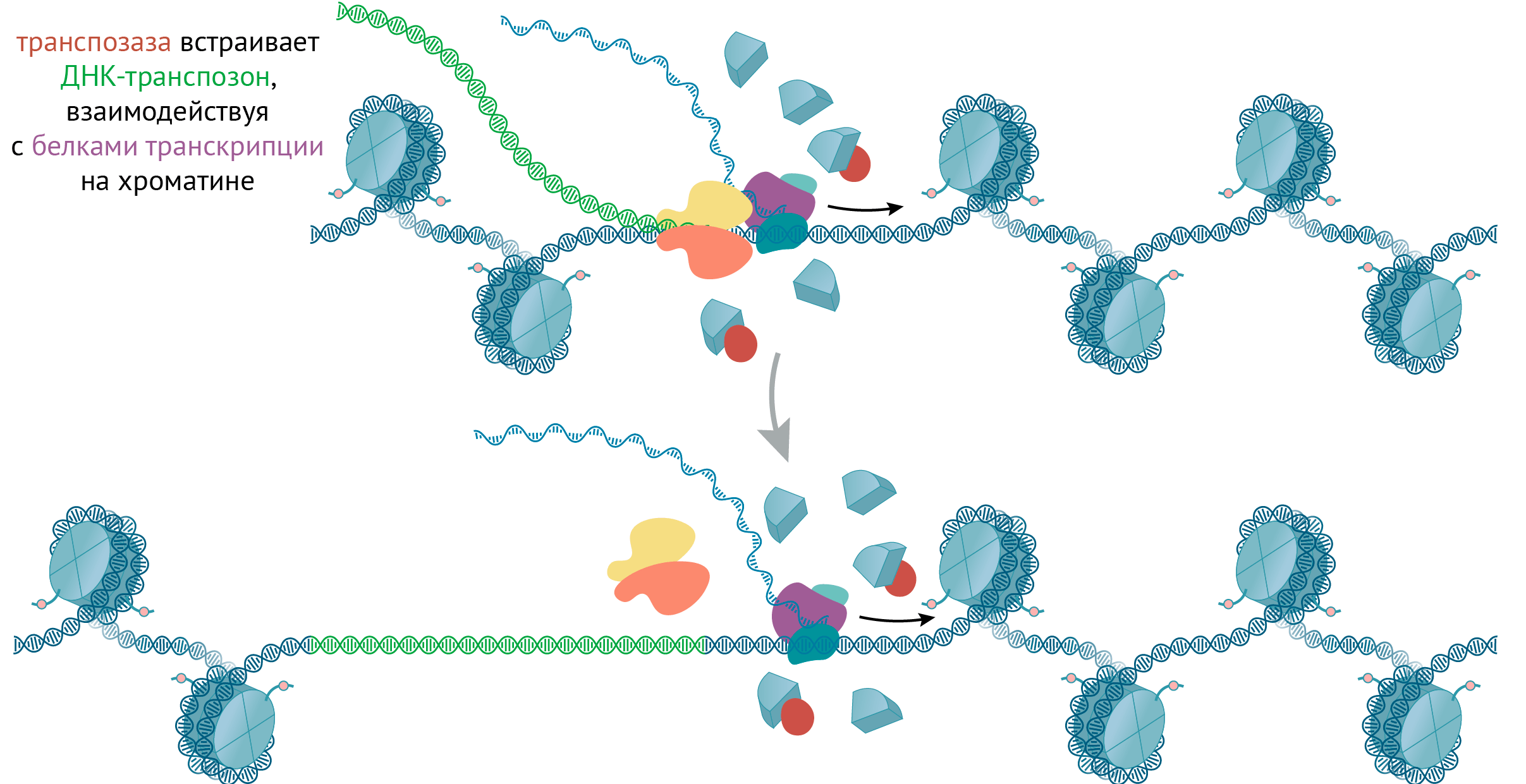
Рисунок 8. Транспозаза встраивает ДНК-транспозон, взаимодействуя с белками транскрипции на хроматине. Часто транспозазы взаимодействуют с белками транскрипции или репликации и встраивают транспозоны туда, где идут эти процессы.
иллюстрация Михаила Гурьева
Перемещение транспозонов может посеять хаос в клетке, поскольку эти повторяющиеся последовательности могут рекомбинировать и приводить к перестройкам генома. Более того, дц-разрывы ДНК, которые образуются при транспозиции (рис. 8), сами по себе способны привести к рекомбинации. Во избежание этих рисков, клетки направляют некоторые из своих эпигенетических механизмов на замалчивание транспозонов. Так, многие транспозоны метилированы [3] и помечены главным маркером гетерохроматина — H3K9me3 [56].
Почему эпигенетические метки оказываются именно на транспозонах? В половых и плюрипотентных стволовых клетках это происходит посредством пиРНК (об этом мы уже рассказывали в статье «Наследование и эпигеном» [3]), а также транскрипционных факторов KRAB (Krüppel-associated box), узнающих своими «цинковыми пальцами» специфические «транспозоновые» последовательности ДНК и содействующих их «выключению» . Считается, что в эволюции KRAB’ы произошли от белка, который обладает как цинковыми пальцами, так и умением метить гистоны — вроде PRDM9 (того самого, который опосредует сшивку разрывов в мейозе [57]).
KRAB’ы привлекают такие белки как Su(var)3-9 и ДНК-метилтрансферазы, помогая тем самым поставить транспозоны «на mute».
KRAB’ы и транспозоны коэволюционируют. Из анализа 16 геномов млекопитающих выяснилось, что чем больше там транспозонов типа LTR (с длинными повторами на концах), тем больше и KRAB’ов [50], [58]. Но такая гонка не может быть единственным фактором эволюции. Во-первых, некоторым транспозонам удается продолжать встраиваться даже в присутствии вроде бы блокирующих их KRAB’ов. Во-вторых, некоторые KRAB’ы ошибочно нацелены даже на транспозоны, уже потерявшие способность к встраиванию. Почему — до конца не понятно, но ученые полагают, что они регулируют транскрипцию генов, близких к транспозонам, а также подавляют рекомбинацию по повторяющимся последовательностям [57].
Таким же подавлением транспозонов занимается комплекс HUSH, один из белков которого связывает РНК, наводя ученых на мысль, что «замалчивать» транспозоны помогает эта молекула-посредник [59]. Действует он (в отличие от KRAB) не только в плюрипотентных стволовых клетках, но и в дифференцированных. Похоже, что HUSH заточен на поиск ретроэлементов, только что встроившихся в новое место генома: заставляет «замолчать» он только последовательности без интронов [60].
Гонка транспозиции и починки ДНК во время репликации
Навесить эпигенетические метки на транспозоны, чтобы те затихли, — не единственная стратегия противодействия. Можно повлиять и на саму транспозицию, зашивая дц-разрывы, сопровождающие встраивание транспозона.
Используя хитрую методику (см. врезку выше), ученые выяснили, что перемещение LINE-1 по геному замедляется белком BRCA1 — тем самым, мутации которого вызывают рак груди [24]. Он участвует в резекции, умеет вешать «черную метку» (убиквитин [61]) на другие белки и задействован в репарации дц-разрывов ДНК. Когда же ученые посмотрели, где расположены белки, кодируемые самим транспозоном LINE-1, то оказалось, что один из них — ORF1 — облепляет места удвоения ДНК. Получается, что белки, чинящие ДНК во время репликации, одновременно регулируют и перемещение ретротранспозонов.
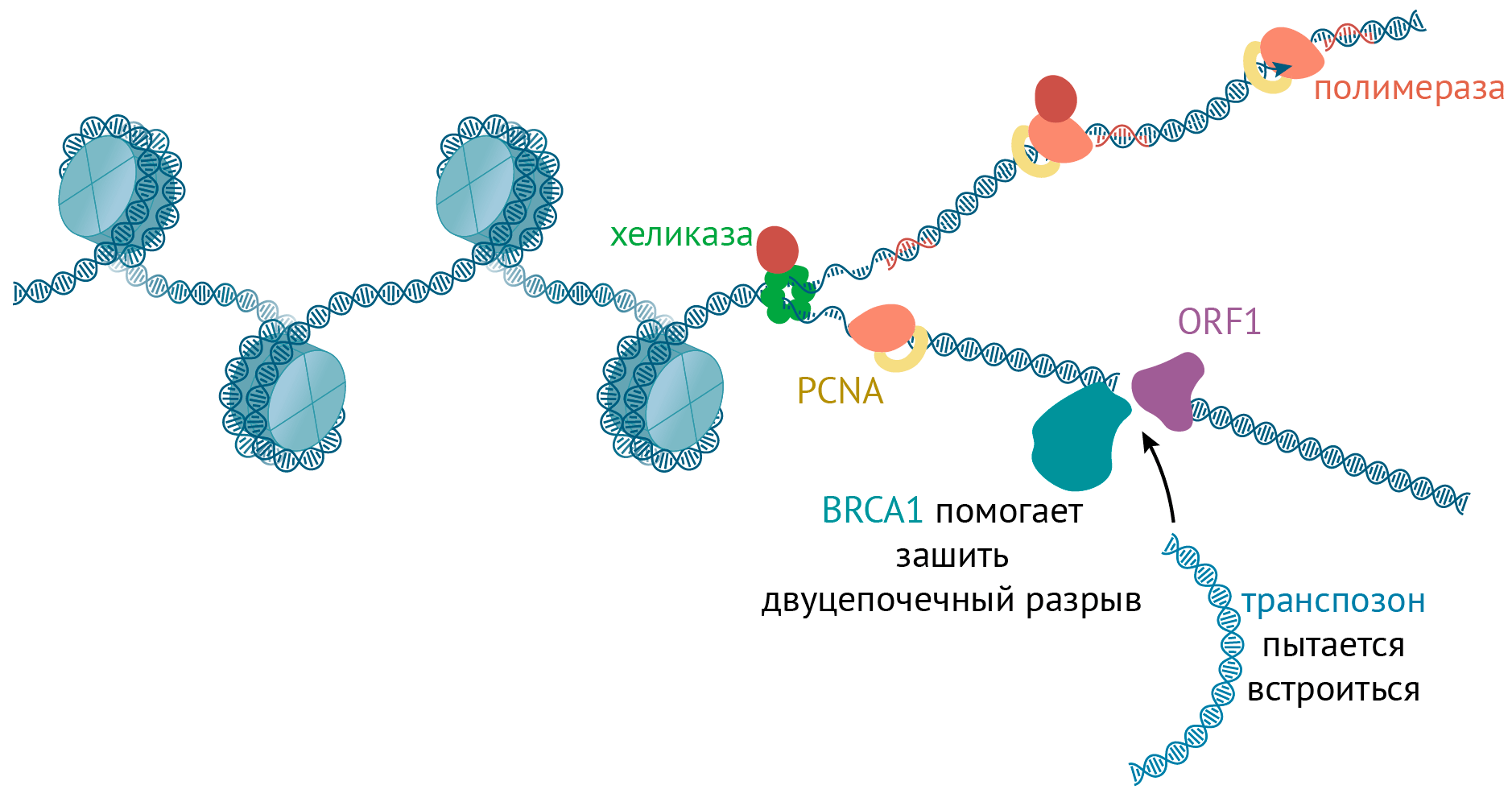
Рисунок 9. Соревнование транспозиции и починки ДНК во время репликации. Транспозон пытается встроиться в место удвоения ДНК, а клетка пытается зашить вносимые транспозоном дц-разрывы и помешать этому.
иллюстрация Михаила Гурьева
Таким образом, репликационные вилки — настоящие «горячие точки» столкновений между встраиванием LINE-1, вносящим разрывы в ДНК, и репарацией, стремящейся это безобразие предотвратить (рис. 9). Оба процесса пытаются обогнать друг друга, как спринтеры на стадионе 🤷 [62].
Эпигенетика в контексте биологии
Мы рассмотрели транскрипцию, репликацию, репарацию ДНК и транспозицию. Но эпигенетика работает не только на уровне отдельных молекул и хроматина, но отражается и на уровне клеток, организмов и даже популяций. Давайте поговорим об эпигенетических эффектах в «программировании» стволовых клеток, развитии, врожденном иммунитете и образовании видов.
Эпигенетика, стволовые клетки и нейрогенез
Стволовые клетки (СК) [63–65] в организме, подобно ученикам, могут превращаться в прочие типы клеток (взрослых специалистов) [64]. Плюрипотентные СК могут давать начало всем клеткам организма, тотипотентные — всем клеткам организма и внеэмбриональным тканям (например, плаценте). Постепенная дифференцировка клеток сопровождается (и, наверное, правильно будет даже сказать — управляется) эпигенетическими перестройками.
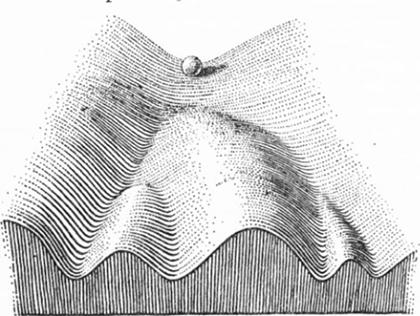
Рисунок 10. Ландшафт Уоддингтона метафорически изображает, как клетка по мере развития из стадии стволовой выбирает всё более специализированный путь, далее уже не пересекающийся с дорогами других клеток.
Постепенное превращение стволовой клетки в другие типы клеток наглядно иллюстрирует так называемый ландшафт Уоддингтона (рис. 10) [66], [67]. Представим себе гору, с которой скатывается мячик. Скатываясь в ту или иную долину, он уже не может взобраться обратно или перепрыгнуть в соседнюю. Каждая долина представляет собой определенный путь дифференцировки, а конечная точка, докуда допрыгает мячик — конечную стадию дифференцировки [68].
Дифференцировка находится под эпигенетическим контролем, который фактически определяет картину экспрессии генов и, в конечном счете, чем клетка является и во что она может превратиться. Поэтому ключевые моменты «биографии» развивающихся клеток — транскрипция тех или иных генов и «переключение» транскрипционных паттернов. Включив определенные транскрипционные факторы (ТФ) [69] и запустив соответствующие гены, можно инициировать и обратный процесс — превращение дифференцированной клетки в плюрипотентную стволовую; причем этого можно достичь синтезом всего четырех ТФ. Один из наборов, включающий в себя Oct4, Sox2, c-Myc и Klf4, был открыт Синьей Яманакой, другой набор — открыт Джеймсом Томпсоном и состоит из Oct4, Sox2, Nanog и Lin28 [70], [71].
Как и любая дифференцировка, нейрогенез [72] — образование нейронов из клеток-предшественников — определяется эпигенетически. Процесс направляют ТФ, действующие в комбинации с другими молекулами (например, ремоделерами хроматина): они могут запускать определенные пути развития и подавлять альтернативные [73]. Один из хорошо изученных примеров — комплекс ТФ Pax6 и ремоделера хроматина BAF, нужный для дифференцировки в промежуточные предшественники нейронов кортекса [73], [74]. Получается, транскрипционные факторы в коллаборации с ремоделерами управляют развитием мозга и определяют судьбы его разных клеток.
Одни и те же ТФ могут участвовать в дифференцировке клеток разных тканей. Например, тот же Pax6 нужен для развития глаза, причем у довольно широкого спектра организмов — от плодовой мушки дрозофилы до человека [75].
Эпигенетика и развитие
По мере роста организма эпигенетические черты изменяются [76]. Пожалуй, самые изученные драйверы развития — ТФ: они действуют в разных частях организма и запускают программы развития. Выбранный путь влияет не только на судьбу клетки, но и на хроматин и его пространственную организацию. Рассмотрим, как именно.
ТФ дирижируют развитием печени мыши: точная программа
Как ТФ действуют во время развития, хорошо изучено на примере печени мыши. ТФ, действующие во время развития, подразделяются на пионерные и непионерные. Первые связываются с неактивными участками ДНК в раннем развитии и «открывают» хроматин (делают его рыхлым и доступным для остальных молекул), позволяя генам читаться. Вторые связываются попозже уже с «открытым» хроматином и запускают собственно чтение генов.
Однако раннее связывание ТФ — необходимое, но недостаточное условие активации генов. Часто на область перед одним геном садится сразу несколько молекул разных (и это важно!) ТФ. Более того, это связывание может быть динамическим: одни ТФ могут приходить, а другие — покидать область промотора. Часто бывает так, активация гена происходит сильно позже связывания на нем ТФ: фактически, ТФ «размечают» гены, которые должны заработать в будущем [77].
Трехмерная организация генома на примере раннего развития
Наша ДНК в ядре уложена по определенным закономерностям. Благодаря методу Hi-C, который позволяет посмотреть, с какой частотой участки ДНК контактируют в пространстве, всё большую поддержку получает модель фрактальной глобулы, согласно которой маленькие глобулы из скомканной ДНК с навешенными хроматиновыми «аксессуарами» собираются в глобулы побольше, те — в глобулы еще большего размера и так далее [78]. Глобулы размером от 100 тысяч до 5 миллионов нуклеотидных остатков получили название топологически ассоциированных доменов (ТАДов) [79–82]. По данным Hi-C можно выделить два типа взаимодействий между участками ДНК, условно названных компартментами А и B. Участки ДНК в А взаимодействуют пореже, чем в B.
Следует сказать две вещи про эти компартменты. Во-первых, они есть не всегда: например, в мышиных женских пронуклеусах их нет [83], зато они постепенно появляются после оплодотворения [84], [85]. Во-вторых, B-компартмент преимущественно находится возле оболочки ядра — ядерной ламины, а А-компартмент — внутри ядра [86]. Причем ядерная ламина важна для того, чтобы А- и В-компартменты укладывались правильным образом [87].
Пространственную укладку генома по мере развития неплохо изучили флуоресцентной гибридизацией на примере хромосомы V червячка C. elegans (рис. 11) [88]. Фиксированные клетки с нарушенной мембраной инкубировали с разноцветными последовательностями ДНК, комплементарными изучаемой. Две комплементарные последовательности ДНК спариваются, и по разноцветным меткам на флуоресцентном микроскопе можно отследить нахождение изучаемой последовательности.
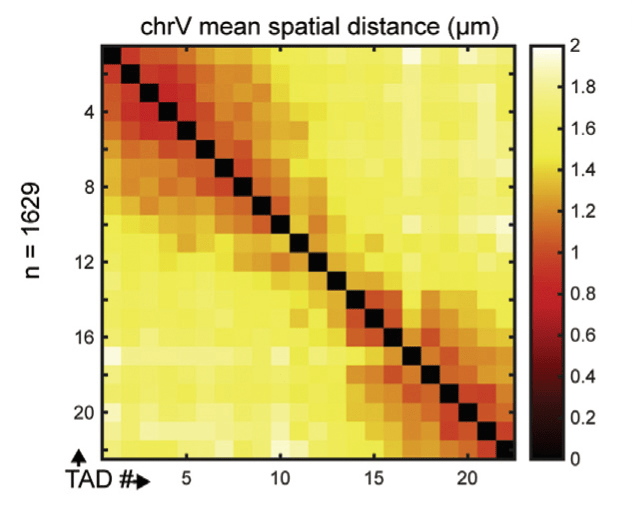
Рисунок 11. Карта гибридизации V хромосомы C. elegans. Исследователи сделали комплементарные пробы к огромному участку хромосомы из 22-х ТАДов и на основе данных микроскопии построили двумерную карту расстояний между ТАДами.
Эксперименты показали:
- компартменты А и B окончательно формировались только к поздним стадиям развития червячка;
- на ранних стадиях эмбриогенеза они были уложены необычно. Хромосома имела форму гантельки, с А-компартментом в середине и В-компартментами по бокам. Причем на более поздних стадиях развития гантелька схлопывалась, и B-компартменты одной гантельки взаимодействовали. Гантельку можно было схлопнуть и на ранних стадиях эмбриогенеза, удалив белок, который заякоривает неактивный хроматин на ядерной ламине.
Получается, ламина заякоривает и растягивает хромосомы на ранних этапах развития червячка [88].
Врожденный иммунный ответ на патогенную ДНК
Иммунитет тоже может регулироваться эпигенетически. В частности, врожденный иммунитет включает систему распознавания двуцепочечной ДНК проникших в клетку вирусов — но, конечно, не своей собственной. Давайте попытаемся разобраться, как это происходит.
Эта система основана на ферменте cGAS — сенсоре дцДНК, синтезирующем в ответ на связывание с ней циклический ГМФ-АМФ (цГМФ-АМФ). Эта молекула, в свою очередь, через каскад с участием белка STING активирует ТФ, ну а они уже запускают транскрипцию генов врожденного иммунитета [89]. cGAS находится в основном в цитоплазме, но та небольшая его часть, что расположена в ядре, связана с хроматином в неактивном состоянии и не узнает ядерную ДНК. Что же не дает cGAS’у среагировать на свою собственную ДНК, которой вокруг в избытке? Оказывается, это белок BAF: если выключить его, то гены врожденного иммунитета будут прекрасно активироваться в ответ на свою собственную (!) ДНК. Являясь одним из ключевых ДНК-связывающих белков, BAF, видимо, не дает cGAS реагировать на собственную ДНК клетки, уберегая нас от ужасных аутоиммунных реакций [90].
Хроматин может управлять процессом образования видов
Читатели «Биомолекулы» не сомневаются, что в ходе эволюции образуется множество новых видов — причем происходило это не только в каком-то седом кембрии, но происходит и прямо сейчас. И оказывается, что эпигенетика и тут играет свою на первый взгляд незаметную, но крайне важную роль.
Виды образуются в результате репродуктивной изоляции, с какого-то момента делающей невозможным скрещивание особей двух разных популяций друг с другом (а значит, одновременно прекращается и обмен генами). Часто это происходит из-за того, что хромосомы начинают расходиться неправильно — и тут-то на сцене и появляется эпигенетика. Репродуктивная изоляция может возникать до или после формирования зиготы. Примером случая «до» является географическое разделение двух популяций (например, океаном или горами). Изоляция «после» возникает вследствие стерильности или нефертильности гибридов между близкородственными видами или подвидами. Этот вид репродуктивной изоляции регулируется специальными генами, фактически стимулируя видообразование. Еще удивительнее, что если эти гены нарушить, разделившиеся было популяции вновь обретут способность плодотворно скрещиваться.
Один из таких генов у мышей — метилтрансфераза PRDM9. Обычно, если скрещиваются два подвида мыши — Mus musculus domesticus и Mus musculus musculus, — их потомство будет стерильным. Однако если нарушить у domesticus’ов ген Prdm9 [91] или искусственно вставить туда область цинковых пальцев из человеческого гена [92], потомство окажется плодовитым! Prdm9 дикого типа у стерильных гибридов приводит к неправильному выстраиванию хромосом при мейозе вследствие неспособности зашить разрывы при рекомбинации, а потому половые клетки у таких животных получаются дефектными, и потомства они уже не имеют.
Еще один пример репродуктивной изоляции — неспособность скрещиваться двух близкородственных видов плодовой мушки дрозофилы — Drosophila melanogaster и Drosophila simulans. Этот случай репродуктивной изоляции хорошо соответствует модели Добжанского—Мюллера. По этой модели, два разных функциональных участка ДНК в близкородственных видах эволюционируют и изменяются до такой степени, что разные участки ДНК из разных видов не могут сосуществовать. В данном случае эти участки — гены Hmr (hybrid male rescue, спасение гибридных самцов) и Lhr (lethal hybrid rescue, спасение летальных гибридов). Если скрестить самок Drosophila melanogaster и самцов Drosophila simulans, то женское потомство стерильно, а мужское умирает. Но если нарушить ген Hmr у Drosophila melanogaster или ген Lhr у Drosophila simulans, то самцы выживают [93]. Так чем занимаются эти белки у плодовых мушек? Если посмотреть на мозг Drosophila melanogaster, в которых отсутствуют эти белки, то можно увидеть многочисленные проблемы при делении клетки, подразумевающие проблемы со сцеплением хромосом когезином [94]. Похоже, Hmr и Lhr нужны именно для правильного сцепления хромосом во время метафазы.
Оба этих белка располагаются на цетромерах, стабилизируя друг друга — если убрать один из них, второй тоже деградирует. Белка HMR больше у Drosophila melanogaster, LHR — у Drosophila simulans. В гибридах много обоих белков. Комплекса HMR/LHR становится так много, что он начинает распространяться с центромер по другим местам генома [95]. У гибридов наблюдаются проблемы с конденсацией хромосом [96], а если включить экспрессию HMR/LHR на полную мощность, то конденсин не так эффективно связывается с хроматином [97]. Вероятно, в гибридах этот комплекс, присутствуя в излишних количествах, убирает часть конденсина с хроматина, и хромосомы не могут так эффективно конденсироваться. В результате потомство мух разных видов оказывается, увы, стерильным.
Напоследок о процессах
Итак, мы рассмотрели, как эпигенетика дирижирует процессами молекулярной биологии на хроматине — репликацией, транскрипцией, репарацией и даже транспозицией. Именно эпигенетика определяет развитие, комбинируя и направляя разные транскрипционные факторы на целевые гены и таким образом взращивая организм. Важна она и для иммунитета — в частности, благодаря ей специальные сигнальные белки узнают только чужую, но не свою ДНК, а на свою ДНК врожденный иммунный ответ не запускается. Наконец, в процессе эволюции эпигенетика управляет видообразованием, создав особые механизмы нарушения (!) клеточного цикла, чтобы сделать потомство близкородственных, но уже различающихся, видов стерильным. Грубо, но зато на пользу эволюции!
Литература
- Молекулы и эпигеном ;
- Ядро и эпигеном;
- Наследование и эпигеном;
- Paul Batty, Daniel W. Gerlich. (2019). Mitotic Chromosome Mechanics: How Cells Segregate Their Genome. Trends in Cell Biology. 29, 717-726;
- Bryan J. Wilkins, Nils A. Rall, Yogesh Ostwal, Tom Kruitwagen, Kyoko Hiragami-Hamada, et. al.. (2014). A Cascade of Histone Modifications Induces Chromatin Condensation in Mitosis. Science. 343, 77-80;
- Sarah G. Swygert, Seungsoo Kim, Xiaoying Wu, Tianhong Fu, Tsung-Han Hsieh, et. al.. (2019). Condensin-Dependent Chromatin Compaction Represses Transcription Globally during Quiescence. Molecular Cell. 73, 533-546.e4;
- Morgane Macheret, Rahul Bhowmick, Katarzyna Sobkowiak, Laura Padayachy, Jonathan Mailler, et. al.. (2020). High-resolution mapping of mitotic DNA synthesis regions and common fragile sites in the human genome through direct sequencing. Cell Res. 30, 997-1008;
- 12 методов в картинках: микроскопия;
- Флуоресцентные репортеры и их молекулярные репортажи;
- Johan H. Gibcus, Kumiko Samejima, Anton Goloborodko, Itaru Samejima, Natalia Naumova, et. al.. (2018). A pathway for mitotic chromosome formation. Science. 359, eaao6135;
- Kristin Abramo, Anne-Laure Valton, Sergey V. Venev, Hakan Ozadam, A. Nicole Fox, Job Dekker. (2019). A chromosome folding intermediate at the condensin-to-cohesin transition during telophase. Nat Cell Biol. 21, 1393-1402;
- Helfrid Hochegger, Donniphat Dejsuphong, Eiichiro Sonoda, Alihossein Saberi, Eeson Rajendra, et. al.. (2007). An essential role for Cdk1 in S phase control is revealed via chemical genetics in vertebrate cells. Journal of Cell Biology. 178, 257-268;
- Feroz R. Papa, Chao Zhang, Kevan Shokat, Peter Walter. (2003). Bypassing a Kinase Activity with an ATP-Competitive Drug. Science. 302, 1533-1537;
- Неуловимая архитектура хроматина мухи;
- Половая жизнь хроматина;
- Takashi Nagano, Yaniv Lubling, Csilla Várnai, Carmel Dudley, Wing Leung, et. al.. (2017). Cell-cycle dynamics of chromosomal organization at single-cell resolution. Nature. 547, 61-67;
- Обо всех РНК на свете, больших и малых;
- Kumiko Samejima, Daniel G. Booth, Hiromi Ogawa, James R. Paulson, Linfeng Xie, et. al.. (2018). Rapid degradation of condensins and 3D-EM reveal chromatin volume is uncoupled from chromosome architecture in mitosis. Journal of Cell Science;
- Paola Vagnarelli, Damien F. Hudson, Susana A. Ribeiro, Laura Trinkle-Mulcahy, Jennifer M. Spence, et. al.. (2006). Condensin and Repo-Man–PP1 co-operate in the regulation of chromosome architecture during mitosis. Nat Cell Biol. 8, 1133-1142;
- Ауксин — великий мотиватор;
- Голодные химеры: направленный протеолиз в качестве лекарства;
- Neil T. Umbreit, Cheng-Zhong Zhang, Luke D. Lynch, Logan J. Blaine, Anna M. Cheng, et. al.. (2020). Mechanisms generating cancer genome complexity from a single cell division error. Science. 368;
- Kathleen R. Stewart-Morgan, Nataliya Petryk, Anja Groth. (2020). Chromatin replication and epigenetic cell memory. Nat Cell Biol. 22, 361-371;
- Рак молочной железы с семейной историей;
- Дело о BRCA-мутациях: выводим преступников на чистую воду;
- Kyosuke Nakamura, Giulia Saredi, Jordan R. Becker, Benjamin M. Foster, Nhuong V. Nguyen, et. al.. (2019). H4K20me0 recognition by BRCA1–BARD1 directs homologous recombination to sister chromatids. Nat Cell Biol. 21, 311-318;
- Giulia Saredi, Hongda Huang, Colin M. Hammond, Constance Alabert, Simon Bekker-Jensen, et. al.. (2016). H4K20me0 marks post-replicative chromatin and recruits the TONSL–MMS22L DNA repair complex. Nature. 534, 714-718;
- Qian Du, Saul A. Bert, Nicola J. Armstrong, C. Elizabeth Caldon, Jenny Z. Song, et. al.. (2019). Replication timing and epigenome remodelling are associated with the nature of chromosomal rearrangements in cancer. Nat Commun. 10;
- Jennifer G. Aparicio, Christopher J. Viggiani, Daniel G. Gibson, Oscar M. Aparicio. (2004). The Rpd3-Sin3 Histone Deacetylase Regulates Replication Timing and Enables Intra-S Origin Control in Saccharomyces cerevisiae. Mol Cell Biol. 24, 4769-4780;
- Constance Alabert, Carolin Loos, Moritz Voelker-Albert, Simona Graziano, Ignasi Forné, et. al.. (2020). Domain Model Explains Propagation Dynamics and Stability of Histone H3K27 and H3K36 Methylation Landscapes. Cell Reports. 30, 1223-1234.e8;
- Nazaret Reverón-Gómez, Cristina González-Aguilera, Kathleen R. Stewart-Morgan, Nataliya Petryk, Valentin Flury, et. al.. (2018). Accurate Recycling of Parental Histones Reproduces the Histone Modification Landscape during DNA Replication. Molecular Cell. 72, 239-249.e5;
- Kathleen R. Stewart-Morgan, Nazaret Reverón-Gómez, Anja Groth. (2019). Transcription Restart Establishes Chromatin Accessibility after DNA Replication. Molecular Cell. 75, 284-297.e6;
- Ralph Scully, Arvind Panday, Rajula Elango, Nicholas A. Willis. (2019). DNA double-strand break repair-pathway choice in somatic mammalian cells. Nat Rev Mol Cell Biol. 20, 698-714;
- Jan-Gert Brüning, Kenneth J Marians. (2021). Bypass of complex co-directional replication-transcription collisions by replisome skipping. Nucleic Acids Research. 49, 9870-9885;
- Dominick A. Matos, Jia-Min Zhang, Jian Ouyang, Hai Dang Nguyen, Marie-Michelle Genois, Lee Zou. (2020). ATR Protects the Genome against R Loops through a MUS81-Triggered Feedback Loop. Molecular Cell. 77, 514-527.e4;
- Pradeep S. Pallan, Martin Egli. (2008). Insights into RNA/DNA hybrid recognition and processing by RNase H from the crystal structure of a non-specific enzyme-dsDNA complex. Cell Cycle. 7, 2562-2569;
- Melvin Noe Gonzalez, Daniel Blears, Jesper Q. Svejstrup. (2021). Causes and consequences of RNA polymerase II stalling during transcript elongation. Nat Rev Mol Cell Biol. 22, 3-21;
- Justyna Zaborowska, Sylvain Egloff, Shona Murphy. (2016). The pol II CTD: new twists in the tail. Nat Struct Mol Biol. 23, 771-777;
- Cécilia Studniarek, Sylvain Egloff, Shona Murphy. (2021). Noncoding RNAs Set the Stage for RNA Polymerase II Transcription. Trends in Genetics. 37, 279-291;
- Karen Adelman, John T. Lis. (2012). Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nat Rev Genet. 13, 720-731;
- Yang Liu, Keda Zhou, Naifu Zhang, Hui Wei, Yong Zi Tan, et. al.. (2020). FACT caught in the act of manipulating the nucleosome. Nature. 577, 426-431;
- Tao Wang, Yang Liu, Garrett Edwards, Daniel Krzizike, Hataichanok Scherman, Karolin Luger. (2018). The histone chaperone FACT modulates nucleosome structure by tethering its components. Life Sci. Alliance. 1, e201800107;
- Olga Viktorovskaya, James Chuang, Dhawal Jain, Natalia I. Reim, Francheska López-Rivera, et. al.. (2021). Essential histone chaperones collaborate to regulate transcription and chromatin integrity. Genes Dev.. 35, 698-712;
- Ming Li, Arjan Hada, Payel Sen, Lola Olufemi, Michael A Hall, et. al.. (2015). Dynamic regulation of transcription factors by nucleosome remodeling. eLife. 4;
- Hugo B. Brandão, Payel Paul, Aafke A. van den Berg, David Z. Rudner, Xindan Wang, Leonid A. Mirny. (2019). RNA polymerases as moving barriers to condensin loop extrusion. Proc Natl Acad Sci USA. 116, 20489-20499;
- Banigan E.J., Tang W., van den Berg A.A., Stocsits R.R., Wutz G., Brandão H.B. et al. (2022). Transcription shapes 3D chromatin organization by interacting with loop-extruding cohesin complexes. bioRxiv;
- Hajnalka L Pálinkás, Angéla Békési, Gergely Róna, Lőrinc Pongor, Gábor Papp, et. al.. (2020). Genome-wide alterations of uracil distribution patterns in human DNA upon chemotherapeutic treatments. eLife. 9;
- T Mimori, M Akizuki, H Yamagata, S Inada, S Yoshida, M Homma. (1981). Characterization of a high molecular weight acidic nuclear protein recognized by autoantibodies in sera from patients with polymyositis-scleroderma overlap.. J. Clin. Invest.. 68, 611-620;
- L-J Mah, A El-Osta, T C Karagiannis. (2010). γH2AX: a sensitive molecular marker of DNA damage and repair. Leukemia. 24, 679-686;
- Мода на ретро. Где встречается обратная транскрипция, и как она эволюционировала;
- Ген древних ретровирусов на службе у мозга млекопитающих;
- Мобильные генетические элементы прокариот: стратификация «общества» бродяжек и домоседов;
- Alu: история одной последовательности;
- Разнообразия много не бывает: чем занимаются мобильные элементы генома в мозге;
- Тайны «молекулярных паразитов», или Как путешествовать по геному;
- R. Keith Slotkin, Robert Martienssen. (2007). Transposable elements and the epigenetic regulation of the genome. Nat Rev Genet. 8, 272-285;
- Peng Yang, Yixuan Wang, Todd S. Macfarlan. (2017). The Role of KRAB-ZFPs in Transposable Element Repression and Mammalian Evolution. Trends in Genetics. 33, 871-881;
- James H. Thomas, Sean Schneider. (2011). Coevolution of retroelements and tandem zinc finger genes. Genome Res.. 21, 1800-1812;
- Christopher H. Douse, Iva A. Tchasovnikarova, Richard T. Timms, Anna V. Protasio, Marta Seczynska, et. al.. (2020). TASOR is a pseudo-PARP that directs HUSH complex assembly and epigenetic transposon control. Nat Commun. 11;
- Marta Seczynska, Stuart Bloor, Sergio Martinez Cuesta, Paul J. Lehner. (2022). Genome surveillance by HUSH-mediated silencing of intronless mobile elements. Nature. 601, 440-445;
- Вездесущий убиквитин;
- Paolo Mita, Xiaoji Sun, David Fenyö, David J. Kahler, Donghui Li, et. al.. (2020). BRCA1 and S phase DNA repair pathways restrict LINE-1 retrotransposition in human cells. Nat Struct Mol Biol. 27, 179-191;
- Была клетка простая, стала стволовая;
- Ствол и ветки: стволовые клетки;
- Такие разные стволовые клетки;
- Эпигенетика: невидимый командир генома;
- Melinda Bonnie Fagan. (2012). Waddington redux: models and explanation in stem cell and systems biology. Biol Philos. 27, 179-213;
- Jonathan M. W. Slack. (2002). Conrad Hal Waddington: the last Renaissance biologist?. Nat Rev Genet. 3, 889-895;
- Кем быть? Как гемопоэтическая стволовая клетка выбирает профессию;
- Нобелевская премия по физиологии и медицине (2012): индуцированные стволовые клетки;
- Guilai Shi, Ying Jin. (2010). Role of Oct4 in maintaining and regaining stem cell pluripotency. Stem Cell Research & Therapy. 1, 39;
- Как сделать нейрон из фибробласта?;
- Jovica Ninkovic, Magdalena Götz. (2015). How to make neurons—thoughts on the molecular logic of neurogenesis in the central nervous system. Cell Tissue Res. 359, 5-16;
- Jovica Ninkovic, Andrea Steiner-Mezzadri, Melanie Jawerka, Umut Akinci, Giacomo Masserdotti, et. al.. (2013). The BAF Complex Interacts with Pax6 in Adult Neural Progenitors to Establish a Neurogenic Cross-Regulatory Transcriptional Network. Cell Stem Cell. 13, 403-418;
- Walter J Gehring, Kazuho Ikeo. (1999). Pax 6: mastering eye morphogenesis and eye evolution. Trends in Genetics. 15, 371-377;
- Эпигенетические часы: сколько лет вашему метилому?;
- Panagiota Karagianni, Panagiotis Moulos, Dominic Schmidt, Duncan T. Odom, Iannis Talianidis. (2020). Bookmarking by Non-pioneer Transcription Factors during Liver Development Establishes Competence for Future Gene Activation. Cell Reports. 30, 1319-1328.e6;
- E. Lieberman-Aiden, N. L. van Berkum, L. Williams, M. Imakaev, T. Ragoczy, et. al.. (2009). Comprehensive Mapping of Long-Range Interactions Reveals Folding Principles of the Human Genome. Science. 326, 289-293;
- Новый взгляд на геном: не просто цепочка генов, а трехмерная сеть, интегрирующая функциональные домены ядра;
- Неуловимая архитектура хроматина мухи;
- Пространственный контакт;
- Jesse R. Dixon, Siddarth Selvaraj, Feng Yue, Audrey Kim, Yan Li, et. al.. (2012). Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 485, 376-380;
- Ilya M. Flyamer, Johanna Gassler, Maxim Imakaev, Hugo B. Brandão, Sergey V. Ulianov, et. al.. (2017). Single-nucleus Hi-C reveals unique chromatin reorganization at oocyte-to-zygote transition. Nature. 544, 110-114;
- Yuwen Ke, Yanan Xu, Xuepeng Chen, Songjie Feng, Zhenbo Liu, et. al.. (2017). 3D Chromatin Structures of Mature Gametes and Structural Reprogramming during Mammalian Embryogenesis. Cell. 170, 367-381.e20;
- Zhenhai Du, Hui Zheng, Bo Huang, Rui Ma, Jingyi Wu, et. al.. (2017). Allelic reprogramming of 3D chromatin architecture during early mammalian development. Nature. 547, 232-235;
- Bas van Steensel, Andrew S. Belmont. (2017). Lamina-Associated Domains: Links with Chromosome Architecture, Heterochromatin, and Gene Repression. Cell. 169, 780-791;
- Sergey V. Ulianov, Semen A. Doronin, Ekaterina E. Khrameeva, Pavel I. Kos, Artem V. Luzhin, et. al.. (2019). Nuclear lamina integrity is required for proper spatial organization of chromatin in Drosophila. Nat Commun. 10;
- Ahilya N. Sawh, Maxwell E.R. Shafer, Jun-Han Su, Xiaowei Zhuang, Siyuan Wang, Susan E. Mango. (2020). Lamina-Dependent Stretching and Unconventional Chromosome Compartments in Early C. elegans Embryos. Molecular Cell. 78, 96-111.e6;
- Jonny Hertzog, Jan Rehwinkel. (2020). Regulation and inhibition of the DNA sensor cGAS. EMBO Reports. 21;
- Baptiste Guey, Marilena Wischnewski, Alexiane Decout, Kristina Makasheva, Murat Kaynak, et. al.. (2020). BAF restricts cGAS on nuclear DNA to prevent innate immune activation. Science. 369, 823-828;
- Ondrej Mihola, Zdenek Trachtulec, Cestmir Vlcek, John C. Schimenti, Jiri Forejt. (2009). A Mouse Speciation Gene Encodes a Meiotic Histone H3 Methyltransferase. Science. 323, 373-375;
- Benjamin Davies, Edouard Hatton, Nicolas Altemose, Julie G. Hussin, Florencia Pratto, et. al.. (2016). Re-engineering the zinc fingers of PRDM9 reverses hybrid sterility in mice. Nature. 530, 171-176;
- Nicholas J. Brideau, Heather A. Flores, Jun Wang, Shamoni Maheshwari, Xu Wang, Daniel A. Barbash. (2006). Two Dobzhansky-Muller Genes Interact to Cause Hybrid Lethality in Drosophila. Science. 314, 1292-1295;
- Jacob A Blum, Silvia Bonaccorsi, Marta Marzullo, Valeria Palumbo, Yukiko M Yamashita, et. al.. (2017). The Hybrid Incompatibility Genes Lhr and Hmr Are Required for Sister Chromatid Detachment During Anaphase but Not for Centromere Function. Genetics. 207, 1457-1472;
- Andreas W. Thomae, Georg O.M. Schade, Jan Padeken, Marc Borath, Irene Vetter, et. al.. (2013). A Pair of Centromeric Proteins Mediates Reproductive Isolation in Drosophila Species. Developmental Cell. 27, 412-424;
- Bonnie J Bolkan, Ronald Booker, Michael L Goldberg, Daniel A Barbash. (2007). Developmental and Cell Cycle Progression Defects in Drosophila Hybrid Males. Genetics. 177, 2233-2241;
- Natalia Y Kochanova, Tamas Schauer, Grusha Primal Mathias, Andrea Lukacs, Andreas Schmidt, et. al.. (2020). A multi-layered structure of the interphase chromocenter revealed by proximity-based biotinylation. Nucleic Acids Research. 48, 4161-4178;
- Robert G. Roeder. (2019). 50+ years of eukaryotic transcription: an expanding universe of factors and mechanisms. Nat Struct Mol Biol. 26, 783-791;
- Marco Saponaro. (2022). Transcription–Replication Coordination. Life. 12, 108;
- Alisa Zhiteneva, Juan Jose Bonfiglio, Alexandr Makarov, Thomas Colby, Paola Vagnarelli, et. al.. (2017). Mitotic post-translational modifications of histones promote chromatin compaction in vitro. Open Biol.. 7, 170076.