
Таинственный код нашего генома
21 января 2014
Таинственный код нашего генома
- 5049
- 1
- 7
Расшифровка генетического код стала важным научным событием двадцатого века. Сейчас перед учеными появляются новые загадки о функционировании нашего генома.
-
Автор
-
Редакторы
Последовательность ДНК определяет строение белка с помощью триплетного генетического кода, в котором каждой аминокислоте соответствует три нуклеотида. Случайные мутации приводят к изменению последовательности нуклеотидов, в результате чего появляются новые варианты белков. Именно так до недавнего времени представляли себе ученые эволюцию белков. Но благодаря исследованиям последних лет оказалось, что помимо генетического кода есть и другие «коды», которые диктуют эволюции белков свои правила.
Одним из важных свойств генетического кода является его избыточность — каждая аминокислота, как правило, кодируется не одним, а 2–6 кодонами. Интересно, что при этом частота использования разных кодонов, отвечающих за одну и ту же аминокислоту, различается как в прокариотических, так и в эукариотических геномах [1]. У организмов с коротким жизненным циклом предпочтения одних кодонов другим связывают с необходимостью в увеличении эффективности транскрипции и стабильности мРНК [2], [3]. Однако в случае геномов млекопитающих такое объяснение подходит лишь для небольшого количества случаев, поэтому в последние годы ученые активно занимаются изучением особенностей геномов млекопитающих и причин предпочтительного использования тех или иных кодонов.
Важное значение в частоте использования кодонов играют транскрипционные факторы — к такому выводу пришла группа ученых из Университета Вашингтона под руководством Джона Стаматояннопоулоса (John A. Stamatoyannopoulos). В опубликованной в журнале Science статье обсуждается, как транскрипционные факторы могут управлять эволюцией белков посредством влияния на частоту использования кодонов [4].
Транскрипционные факторы (ТФ) — это белки, регулирующие транскрипцию генов при связывании с ДНК. ТФ могут повышать транскрипцию или снижать ее, влияя, таким образом, на количество мРНК и белка, соответствующих определенному гену. Долгое время считалось, что ТФ связываются только в некодирующей (не содержащей генов) части ДНК. В своем новом исследовании группа Стаматояннопоулоса выяснила, что на самом деле во многих генах человека ТФ связываются с кодирующими последовательностями ДНК (т.е. с теми, которые являются частью генов). Так как эффективность связывания ТФ с ДНК зависит от того, какие именно нуклеотиды находятся в сайте связывания, ТФ могут снижать возможное разнообразие кодонов в местах своей посадки (рис. 1). При этом даже нейтральные с точки зрения белка мутации (те, при которых последовательность аминокислот не меняется благодаря избыточности генетического кода) могут изменять эффективность связывания ТФ с ДНК и становиться материалом для естественного отбора. Получается, что эволюция белков определяется не только хорошо изученным генетическим кодом, но и другим особенным кодом — «кодом связывания ТФ». Ранее были описаны и некоторые другие «регуляторные» коды, которые контролируют организацию хроматина [5], пространственную структуру и сплайсинг мРНК [5], [6], эффективность трансляции [7], ко-трансляционный фолдинг белков [8] (рис. 2). Все они могут влиять на предпочтительное использование тех или иных кодонов.
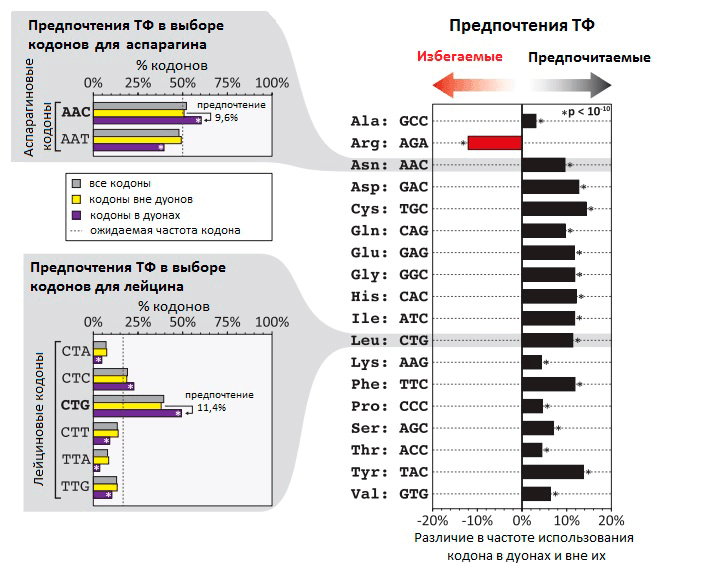
Рисунок 1. Неслучайная частота использования кодов в дуонах в местах связывания ТФ с ДНК. На гистограмме видно, что частота использования некоторых кодонов на 5–15% выше в дуонах, чем вне дуонов. В случае аргинина кодон AGA, напротив, гораздо реже встречается в дуонах, чем в других участках генома. В левой части рисунка — распределение частоты использования разных кодонов на примере кодонов для аспарагина и лейцина.
Насколько в геноме распространено применение дополнительных «регуляторных» кодов, которые перекрывают генетический код, и какое влияние они оказывают на эволюцию белков? Сотрудники лаборатории Стаматояннопоулоса попытались ответить на этот вопрос при исследовании «кода связывания ТФ». Чтобы выявить участки ДНК, связывающиеся с ТФ, они применили метод картирования с помощью дезоксирибонуклеазы I. Этот фермент разрушает одноцепочечные участки ДНК — если только они в этот момент не связаны с ТФ (в таком случае они сохранятся). Ученые исследовали 81 тип человеческих клеток, определив точные нуклеотидные последовательности связанных с ТФ участков генов. Оказалось, что приблизительно 14% кодонов в 86,9% генов человека связаны с различными транскрипционными факторами. В своей статье исследователи предлагают называть эти участки генов «дуонами», т.к. они кодируют два типа информации — информацию о белковой последовательности в виде генетического кода и информацию об экспрессии гена с помощью связывания ТФ. Для нормальной экспрессии гена необходимо связывание ДНК с ТФ, поэтому существуют определенные ограничения на использование различных кодонов, обусловленные строением ДНК-связывающего участка ТФ.
В геноме человека широко распространены однонуклеотидные полиморфизмы (single nucleotide polymorphisms, SNP) — различия последовательности гомологичных генов разных людей на один нуклеотид. Могут ли такие однонуклеотидные различия повлиять на эффективность связывания ТФ с ДНК? Чтобы узнать это, ученые из лаборатории Стаматояннопоулоса нашли на полученной ими карте дуонов почти 600 тыс. известных сайтов SNP, связанных с развитием какого-либо заболевания или проявлением определенного фенотипического признака. Оказалось, что 17,4% сайтов полиморфизма изменяют результаты картирования с помощью дезоксирибонуклеазы I, т.е. они, вероятно, снижают эффективность связывания ТФ с ДНК. Это изменение не зависит от того, является ли данный полиморфизм синонимичным или несинонимичным (т.е. влияет ли замена нуклеотида на замену аминокислоты в белке). Интересно, что значительная часть несинонимичных замен, хотя и приводит к изменению последовательности белка, не приводит к нарушению его функций. В этих случаях изменения нуклеотидной последовательности приводят только к нарушению связывания ТФ с ДНК. Эта находка поддерживает гипотезу о том, что SNP в кодирующей ДНК могут приводить к развитию заболеваний без влияния на функцию белка [9], [10]. Поэтому при изучении роли SNP в различных заболеваниях и при исследовании экзома необходимо учитывать весь спектр «регуляторных кодов», взаимодействующих с последовательностью гена.
«Регуляторные коды» далеко не всегда мирно и гармонично сосуществуют. В генах плодовой мушки Drosophila melanogaster ближе к концу экзонов наблюдается резкое снижение частоты использования оптимальных для трансляции кодонов и повышение частоты использования кодонов, которые облегчают сплайсинг мРНК [11]. Это показывает, что в ходе эволюции потребность в точном сплайсинге была выше, чем потребность в более эффективной трансляции. Также при исследовании дуонов и других ТФ-связывающих участков ДНК оказалось, что среди этих последовательностей нет стоп-кодонов.
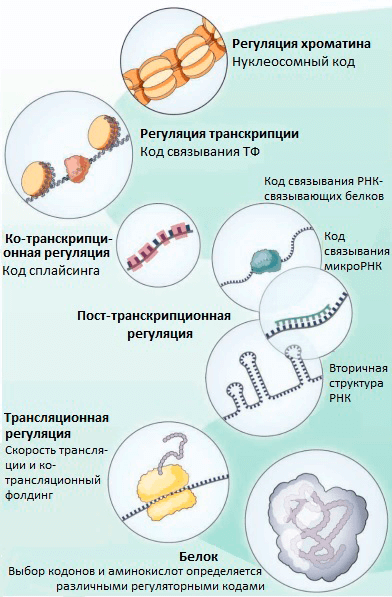
Рисунок 2. «Тайные коды» нашего генома, которые определяют частоту использования кодонов и выбор аминокислот в эволюции белков, независимо от выполнения белком его функций
Что же может обеспечить взаимовыгодное соседство «регуляторных» и генетического кодов? Одним из ключевых ограничений для белок-кодирующих генов является то, что последовательность гена должна обеспечивать нормальный фолдинг кодируемого белка. Мутации, нарушающие правильную укладку, с большой вероятностью будут отсеяны как вредные. Можно предположить, что когда необходимость правильного фолдинга отсутствует (например, в неструктурированных белках [12]), белок-кодирующая последовательность может содержать большее количество регуляторных элементов для различных «регуляторных кодов». Действительно ли это так, помогут узнать дальнейшие исследования.
Несмотря на то, что в работе Стаматояннопоулоса и его коллег было сделано много интересных наблюдений о функционировании «кода связывания ТФ», некоторые вопросы остаются открытыми. Например, авторы статьи отмечают, что ТФ гораздо реже связываются с генами с высокой экспрессией, но не ясно, как ТФ при связывании с белок-кодирующими участками ДНК могут воздействовать на транскрипцию этих генов. Возможно, что связывание ТФ в данном случае вызывает активацию альтернативного промотора или соседнего гена, снижая таким образом экспрессию гена с ТФ-связывающей последовательностью. С другой стороны, этот эффект может быть связан с перестройкой хроматина, которая приводит к снижению экспрессии ряда генов.
Новые исследования помогут ученым лучше понять, как различные «регуляторные коды» взаимодействуют друг с другом и с генетическим кодом. Интересно узнать, всегда ли природа могла найти оптимальное решение при сочетании разных кодов, или иногда возникали противоречия, приводящие к неоптимальным или вредным последствиям. Например, может оказаться, что белок-кодирующие последовательности ДНК, которым «трудно справиться» с обилием и разнообразием регуляторных элементов, активно используются патогенами при инфицировании хозяина. Обнаружение перекрывающихся «регуляторных кодов» в нашем геноме открывает новые перспективы для интерпретации различий и особенностей в последовательностях ДНК и указывает на то, что исследование генетического кода еще не подошло к концу.
Перевод редакционной колонки журнала Science [13].
Литература
- R. Grantham, C. Gautier, M. Gouy, M. Jacobzone, R. Mercier. (1981). Codon catalog usage is a genome strategy modulated for gene expressivity. Nucl Acids Res. 9, 213-213;
- Carlini D.B. and Stephan W. (2003). In vivo introduction of unpreferred synonymous codons into the drosophila Adh gene results in reduced levels of ADH protein. Genetics. 163, 239–243;
- M. d. Reis. (2004). Solving the riddle of codon usage preferences: a test for translational selection. Nucleic Acids Research. 32, 5036-5044;
- A. B. Stergachis, E. Haugen, A. Shafer, W. Fu, B. Vernot, et. al.. (2013). Exonic Transcription Factor Binding Directs Codon Choice and Affects Protein Evolution. Science. 342, 1367-1372;
- Tobias Warnecke, Claudia C. Weber, Laurence D. Hurst. (2009). Why there is more to protein evolution than protein function: splicing, nucleosomes and dual-coding sequence. Biochemical Society Transactions. 37, 756-761;
- S. A. Shabalina. (2006). A periodic pattern of mRNA secondary structure created by the genetic code. Nucleic Acids Research. 34, 2428-2437;
- Kurt Fredrick, Michael Ibba. (2010). How the Sequence of a Gene Can Tune Its Translation. Cell. 141, 227-229;
- Sebastian Pechmann, Judith Frydman. (2013). Evolutionary conservation of codon optimality reveals hidden signatures of cotranslational folding. Nat Struct Mol Biol. 20, 237-243;
- Zuben E. Sauna, Chava Kimchi-Sarfaty. (2011). Understanding the contribution of synonymous mutations to human disease. Nat Rev Genet. 12, 683-691;
- Joshua B. Plotkin, Grzegorz Kudla. (2011). Synonymous but not the same: the causes and consequences of codon bias. Nat Rev Genet. 12, 32-42;
- Tobias Warnecke, Laurence D. Hurst. (2007). Evidence for a Trade-Off between Translational Efficiency and Splicing Regulation in Determining Synonymous Codon Usage in Drosophila melanogaster. Molecular Biology and Evolution. 24, 2755-2762;
- За пределами порядка;
- R. J. Weatheritt, M. M. Babu. (2013). The Hidden Codes That Shape Protein Evolution. Science. 342, 1325-1326.





