«Пластик, сэр!», или Бактериальная диета в стиле хай-тек
09 августа 2016
«Пластик, сэр!», или Бактериальная диета в стиле хай-тек
- 2230
- 0
- 8
Ideonella sakaiensis — первая всесторонне описанная бактерия, которая полностью деградирует и усваивает пластик.
сайт onlinesource.co
-
Автор
-
Редакторы
ПЭТ (полиэтилентерефталат) — вещество, трудно разлагаемое в природе, но давно и широко применяемое для изготовления пластиковых емкостей, пленок и других изделий. Многократно увеличившееся с годами количество ПЭТ-мусора сейчас напрямую влияет на стабильность естественных экосистем. Не дожидаясь помощи от людей, природа решила бороться с таким загрязнением сама — с помощью микроорганизмов. Недавно биологам удалось познакомиться с этими «спецназовцами» — бактериями, способными не только разрушать ПЭТ, но и неплохо его усваивать.
Ежегодно в мире производится 311 млн тонн пластмасс. Более 50 млн тонн из них — это продукция из полиэтилентерефталата (ПЭТ): упаковка для продуктов питания, строительная пленка, волокна для изготовления одежды и т.д. Но лишь около 14% пластиковых изделий собирают для вторичной переработки. Основная масса остается в окружающей среде и представляет большую опасность для экосистем, особенно океанических . ПЭТ, получаемый из нефтепродуктов и содержащий высокий процент ароматических компонентов, — химически инертное, а потому чрезвычайно устойчивое в природных условиях соединение: у микробов либо нет ферментов, которые могли бы его разрушить , либо их активность низка [1].
Подробно о загрязнении мирового океана пластиковым мусором «биомолекула» писала ранее — «А ты такой холодный, как... пластик в океане» [2].
Чем именно ограничена биодоступность нефтепродуктов в природных системах, рассказано в статье «Пределы биодоступности углеводородов в грунтах» [3].
До недавнего времени не было известно ни одного микроорганизма, который мог бы деградировать ПЭТ или использовать его для поддержания своего роста. Ситуация изменилась в 2007 году: немецкие ученые опубликовали статью о нитчатых грибах Fusarium oxysporum и F. solani, которые могли расти на минеральной среде, содержащей нити полиэтилентерефталата [4]. В 2011 году в Саргассовом море, где циркулирует более 1100 тонн пластикового мусора, обнаружили консорциум бактерий, питающихся пластиком, но отдельных форм так и не выделили [5]. В феврале 2016 года та же группа немецких биологов сообщила о гидролазе актиномицета Thermobifida fusca, способной расщеплять эфирные связи в ПЭТ [6]. На этом всё и закончилось. А вот если бы удалось изолировать отдельный бактериальный штамм, полностью разлагающий полиэтилентерефталат, изучить его катаболические ферменты и, что не менее важно, гены этих ферментов, то такой организм вполне можно было бы использовать для биологической переработки ПЭТ-мусора.
О впечатляющем спектре особенностей и способностей актиномицетов читайте в конкурсной работе 2015 года «„Зеленые“ революционеры» [7].
Ideonella sakaiensis, микроб-гурманофутурист
Не секрет, что население Японского архипелага давно обитает в мире высоких технологий. Так почему же тамошние микробы должны отставать? Сосуке Ёсида и его коллеги в марте 2016 года сообщили об обнаружении бактерии, которая полностью деградирует и усваивает полиэтилентерефталат в качестве единственного источника углерода [15]. Авторы работы взяли на заводе по переработке пластиковых бутылок 250 проб почвы, сточных вод и активного ила, загрязненных ПЭТ, и провели отбор микроорганизмов, растущих на ПЭТ-пленке. Лишь в одной пробе оказался микробный консорциум (рис. 2а), который индуцировал морфологические изменения пленки (рис. 2б). Сообщество бактерий, дрожжеподобных клеток и простейших успешно деградировало поверхность пленки со скоростью 0,13 мг × см−2 × сут−1 при 30 °C (рис. 2в).

Рисунок 2. Микробный рост на ПЭТ. На рисунках а-в показана деградация пленки ПЭТ (60 мг, 20 × 15 × 0,2 мм) при 30 °С микробным консорциумом, на ж-з — одним бактериальным видом. а — Рост на пленке после 20 дней эксперимента. б — Деградированная ПЭТ-пленка через 70 дней (изображение со сканирующего электронного микроскопа, СЭМ). На врезке — нетронутая пленка. Шкала — 1 мм. в — Ход деградации пленки микробным консорциумом. На рисунках г-е представлены СЭМ-изображения клеток Ideonella sakaiensis, выращенных на ПЭТ-пленке в течение 60 часов. Шкала — 1 мм. Стрелки на левой части рисунка г указывают на точки контакта бактериальных отростков с другими клетками и пленкой (справа — увеличения), на рисунке е — с пленкой.
Последующие эксперименты показали, что единственным микроорганизмом, ответственным за биодеградацию ПЭТ, является Ideonella sakaiensis — грамотрицательная аэробная бета-протеобактерия (рис. 2г) [16]. Клетки идеонеллы оказались связанными друг с другом длинными отростками (рис. 2д), а с ПЭТ-пленкой — короткими (рис. 2г и 2е). Возможно, эти отростки задействованы в доставке секретируемых бактерией ферментов к пленке. При росте I. sakaiensis полиэтилентерефталатная пленка сильно повреждалась (рис. 2ж) и через шесть недель при 30 °С практически исчезала (рис. 2з).
Шесть недель — это, конечно, очень большой срок при таких размерах ПЭТ-пленки, однако эксперимент дает толчок для развития этого направления и, возможно, получения более эффективного штамма, который уже можно будет использовать для вторичной переработки ПЭТ-отходов.
«Нож» и «вилка» для нового блюда
Ёсида с сотрудниками полностью секвенировали геном I. sakaiensis и идентифицировали два ключевых фермента, участвующих в деградации ПЭТ [16]:
- ПЭТазу, секретируемую из клетки и «нарезающую» полиэтилентерефталат на гетеродимеры, представляющие собой моно-(2-гидроксиэтил)-терефталевую кислоту (МГЭТ);
- МГЭТазу — внутриклеточный фермент, который гидролизует поглощенную клеткой МГЭТ до двух мономеров: этиленгликоля и терефталата (рис. 3).
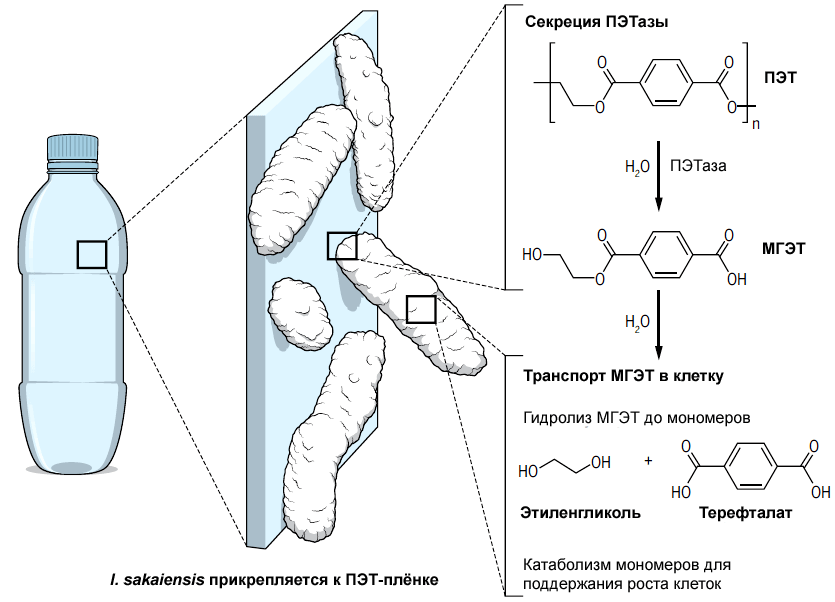
Рисунок 3. Вкусный пластик. Бактерии I. sakaiensis прикрепляются к поверхности ПЭТ-изделия и производят в числе прочих два гидролитических фермента (ПЭТазу и МГЭТазу), которые катализируют деградацию волокон ПЭТ до мономеров двух типов. Мономеры затем утилизируются бактерией в качестве источников углерода и энергии.
[16], рисунок адаптирован
ПЭТаза показала лишь 51% гомологии аминокислотной последовательности с гидролазой T. fusca и предпочитала иной спектр субстратов. МГЭТазу отнесли к семейству ферментов танназ, однако она не проявляла активности в отношении ароматических эфиров, обычно преобразуемых танназами. Кроме того, оказалось, что экспрессия генов ПЭТазы и МГЭТазы позитивно регулируется самим полиэтилентерефталатом. На основании этого Ёсида сделал вывод, что оба фермента уникальны и необходимы именно для деградации ПЭТ [16]. К сожалению, авторы пока не установили, где конкретно находятся гены ферментов — на хромосоме или на внехромосомных элементах, — то есть могут ли они передаваться горизонтально между бактериями, что дало бы возможность без лишних усилий подобрать для экспрессии этих генов более эффективный штамм.
Нет пока ответа и на другой важный вопрос: что идеонеллы используют в качестве вилки — как получают доступ к волокнам ПЭТ на гладкой поверхности пластика? Например, при деградации целлюлозы решающее значение имеют специфические белки с углевод-связывающими модулями (УСМ), которые поднимают с поверхности отдельные волокна целлюлозы, что делает их доступными для гликозидаз и других гидролитических ферментов [17]. Японским ученым придется проверить, производят ли идеонеллы подобные УСМ-содержащие ферменты или же приноровились подбираться к волокнам полиэтилентерефталата иначе.
Заключение: а приборчики-то откуда?
Человечество производит изделия из полиэтилентерефталата и загрязняет ими окружающую среду почти 70 лет. Могли ли описанные японцами гидролитические ферменты появиться за столь короткий период, позволить бактериям питаться новым для них соединением и обеспечить преимущество в борьбе за существование?
Как ни странно, существует пример такой быстрой естественной эволюции — атразинхлоргидролаза. Гербицид атразин широко используется всего лишь с 1950-х годов и содержит в своей молекуле атом хлора. Атразинхлоргидролаза, разрушающая связь C-Cl, развилась из меламиндезаминазы, которая «неравнодушна» к связи C-N меламина. В результате мутаций всего девяти аминокислот меламиндезаминаза потеряла интерес к C-N-связи и стала атразинхлоргидролазой [18].
О быстрой эволюции бактериальных белков-регуляторов «биомолекула» рассказывала в статье «Бактерии способны заменить утраченный белок всего за 96 часов» [19].
Возможно, ПЭТаза посредством нескольких мутаций образовалась из кутиназы, деградирующей алифатический полимер кутин, но проявляющей небольшую активность и в отношении ПЭТ [20].
В этом контексте очень интересен вопрос: как происходит эволюция ферментов, как развиваются их новые активности? Есть предположение, что новые функции фермента (гена) возникают, когда селекция идет по его минорной побочной активности (как в случае с кутиназой). В подходящих условиях эта активность повышается чаще всего путем дупликации гена. Поскольку в результате копирования не страдает исходная функция, а предпочтительная в новых условиях активность возрастает, возникает платформа для отбора различных вариантов путем рекомбинации и точечных мутаций. Так происходит дивергенция с родительским геном, и модифицированные копии постепенно распространяются по популяции [21]. Группе Ёсиды, чтобы выяснить родословную ферментов идеонеллы, придется изучить не только полиморфизм генов ПЭТазы и МГЭТазы, но и их трехмерные структуры в сравнении с филогенетически близкими ферментами.
Литература
- Müller R.J., Kleeberg I., Deckwer W.D. (2001). Biodegradation of polyesters containing aromatic constituents. J. Biotechnol. 86, 87–95;
- А ты такой холодный, как... пластик в океане;
- Пределы биодоступности углеводородов в грунтах;
- Nimchua T., Punnapayak H., Zimmermann W. (2007). Comparison of the hydrolysis of polyethylene terephthalate fibers by a hydrolase from Fusarium oxysporum LCH I and Fusarium solani f. sp. pisi. Biotechnol. J. 2, 361–364;
- Пластик на завтрак;
- Wei R., Oeser T., Schmidt J., Meier R., Barth M., Then J., Zimmermann W. (2016). Engineered bacterial polyester hydrolases efficiently degrade polyethylene terephthalate due to relieved product inhibition. Biotechnol. Bioeng. 113, 1658–1665;
- «Зеленые» революционеры;
- Top E.M., Springael D., Boon N. (2002). Catabolic mobile genetic elements and their potential use in bioaugmentation of polluted soils and waters. FEMS Microbiol. Ecol. 42, 199–208;
- Мобильные генетические элементы прокариот: стратификация «общества» бродяжек и домоседов;
- Lu X.Y., Zhang T., Fang H.H. (2011). Bacteria-mediated PAH degradation in soil and sediment. Appl. Microbiol. Biotechnol. 89, 1357–1371;
- Chakraborty J. and Das S. (2016). Molecular perspectives and recent advances in microbial remediation of persistent organic pollutants. Environ. Sci. Pollut. Res. Int. 23, 16883–16903;
- Биодеградация белого фосфора: как яд стал удобрением;
- Есикова Т.З., Волкова О.В., Таран С.А., Боронин А.М. (2015). Ключевая роль dca-генов в катаболизме epsilon-капролактама у бактерий рода Pseudomonas. Микробиология. 84, 616–619;
- Zhang S., Xia X., Xia N., Wu S., Gao F., Zhou W. (2013). Identification and biodegradation efficiency of a newly isolated 2, 2’, 4, 4’-tetrabromodiphenyl ether (BDE-47) aerobic degrading bacterial strain. Int. Biodeter. Biodegr. 76, 24–31;
- Yoshida S., Hiraga K., Takehana T., Taniguchi I., Yamaji H., Maeda Y. et al. (2016). A bacterium that degrades and assimilates poly(ethylene terephthalate). Science. 351, 1196–1199;
- Bornscheuer U.T. (2016). Feeding on plastic. Science. 351, 1154–1155;
- Bornscheuer U., Buchholz K., Seibel J. (2014). Enzymatic degradation of (ligno)cellulose. Angew. Chem. Int. Ed. Engl. 53, 10876–10893;
- Seffernick J.L., de Souza M.L., Sadowsky M.J., Wackett L.P. (2001). Melamine deaminase and atrazine chlorohydrolase: 98 percent identical but functionally different. J. Bacteriol. 183, 2405–2410;
- Бактерии способны заменить утраченный белок всего за 96 часов;
- Espino-Rammer L., Ribitsch D., Przylucka A., Marold A., Greimel K.J., Herrero Acero E. et al. (2013). Two novel class II hydrophobins from Trichoderma spp. stimulate enzymatic hydrolysis of poly(ethylene terephthalate) when expressed as fusion proteins. Appl. Environ. Microbiol. 79, 4230–4238;
- Bergthorsson U., Andersson D.I., Roth J.R. (2007). Ohno’s dilemma: evolution of new genes under continuous selection. Proc. Natl. Acad. Sci. USA. 104, 17004–17009;
- Тейлор Д., Грин Н., Стаут У. Биология. Том 1 (3-е изд.). М.: «МИР», 2004. — 454 с.