Танец знамений: убиквитин и протеасома в судьбе внутриклеточных белков
19 марта 2024
Танец знамений: убиквитин и протеасома в судьбе внутриклеточных белков
- 1786
- 1
- 12
Запутанные сети убиквитинового сигналинга, покруче хорошего детектива.
рисунок авторов
-
Авторы
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: Каждая клетка старается содержать внутреннее пространство в чистоте и избавляться от утративших свои функции компонентов. Поддержание внутриклеточного белкового гомеостаза в эукариотических клетках преимущественно осуществляется за счет убиквитин-протеасомной системы, обеспечивающей селективную деградацию субстрата, модифицированного убиквитином — маленьким, но распространенным сигнальным белком. В настоящее время известно, что различные модификации субстрата убиквитином опосредуют и другие клеточные процессы. Как же устроен убиквитиновый код? Что лежит в основе разнообразия сигналов которые формируются на его основе? Как осуществляется убиквитин-зависисмая деградация белка в протеасомах? С ответами на эти вопросы, а также с различными типами протеасом и их главными помощниками мы предлагаем вам познакомиться в этой статье.
Конкурс «Био/Мол/Текст»-2023/2024
Эта работа опубликована в номинации «Свободная тема» конкурса «Био/Мол/Текст»-2023/2024.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

Партнер номинации — компания SkyGen: передовой дистрибьютор продукции для life science на российском рынке.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Введение
В процессе развития человеческих обществ росла потребность в точной передаче разнообразных данных как между отдельными людьми, так и между городами, государствами и поколениями. Так, за свою историю человеком были разработаны различные способы хранения и передачи информации. Любопытным способом пользовались древние инки. Для кодирования информации они использовали последовательности узелков (а также их цвет и длину) в связке шерстяных или хлопковых нитей. Такая система получила название кипу. С ее помощью инки передавали сообщения, вели календарь и проводили арифметические вычисления. Интересно, что в живых клетках, из которых мы состоим, существует сигнальная система, в своей основе похожая на кипу. Роль узелков в ней играет молекула небольшого белка — убиквитина (от ubiquitous — вездесущий) [1], получившего свое название за счет широкого распространения в клетках и тканях эукариотических организмов.
Данный белок под действием ряда ферментов присоединяется к субстрату (обычно — другому белку) и формирует различные виды цепочек. В их длине, а также в типе связи молекул убиквитина между собой кодируется сигнал, который определяет дальнейшую «судьбу» субстрата [2].
Сигнальная система на основе убиквитина участвует в регуляции основных клеточных процессов, среди которых клеточный цикл, апоптоз, репарация, транскрипция, внутриклеточная передача сигнала, иммунный ответ и другие [3]. Особая роль данной системы заключается в поддержании белкового гомеостаза в клетке которое осуществляется за счет направленной деградации дефектных или короткоживущих внутриклеточных белков, помеченных убиквитином, в специальной молекулярной машине — протеасоме [4]. Система, основанная на слаженной работе убиквитина и протеасомы получила название «убиквитин-протеасомная система».
Из первой части статьи вы узнаете о структуре и свойствах убиквитина, о ферментах, участвующих в убиквитиновом сигналинге, а также о том, как устроен убиквитиновый код. Также в первой части содержится дополнительная информация, которая касается неканонических мишеней убиквитина и происхождения убиквитинового сигналинга эукариот. Вторая часть статьи посвящена убиквитин-зависимой деградации белка, строению и разнообразию протеасом и взаимодействующих с ними регуляторных компонентов.. Ключевая цель данной работы — продемонстрировать, насколько велико молекулярное разнообразие компонентов и функций убиквитин-протеасомной системы. Уверяем, что это не менее удивительно, чем наблюдаемое разнообразие живых организмов, населяющих различные уголки нашей планеты. Давайте же вместе прогуляемся по внутреннему миру эукариот и познакомимся с одной из множества взаимосвязанных и интересно устроенных молекулярных систем.
Убиквитин и убиквитиновый сигналинг
Вершитель судеб — убиквитин
Для начала мы познакомимся с главным героем нашего рассказа, 76-аминокислотным высококонсервативным белком эукариот — убиквитином. Он обладает высокой стабильностью и компактностью за счет своей характерной структуры, получившей название β-grasp fold — пять β-цепей, частично обернутых вокруг центральной α-спирали [5].
На С-конце убиквитина находится единственный разупорядоченный и подвижный участок молекулы, заканчивающийся глицином (точнее, остатком глицина Gly-76). Он участвует в образовании ковалентной связи с субстратом (подразумевается, что субстрат — белок, если не указано другое, см. врезку 1) (рис. 1). Также важную роль во взаимодействии убиквитина с субстратом и в формировании сигнальных полиубиквитиновых цепей играет аминокислота лизин (обозначается как Lys или K). Она примечательна тем, что содержит дополнительную аминогруппу (NH2) в боковой цепи. Данная аминогруппа тоже может образовывать пептидную связь (пептидная связь между аминокислотами, образованная с участием боковой цепи по крайней мере одной из аминокислот, называется изопептидной).
Таким типом связи обусловлено каноническое взаимодействие убиквитина с субстратом [2]. В нем участвует боковая аминогруппа лизина входящего в состав субстратного белка и карбоксильная группа С-концевого глицина убиквитина (рис 1б). Наращивание убиквитиновой цепи происходит по такому же принципу — С-концевой глицин вышележащего убиквитина взаимодействует с одним из лизинов нижележащего (по направлению от субстрата).
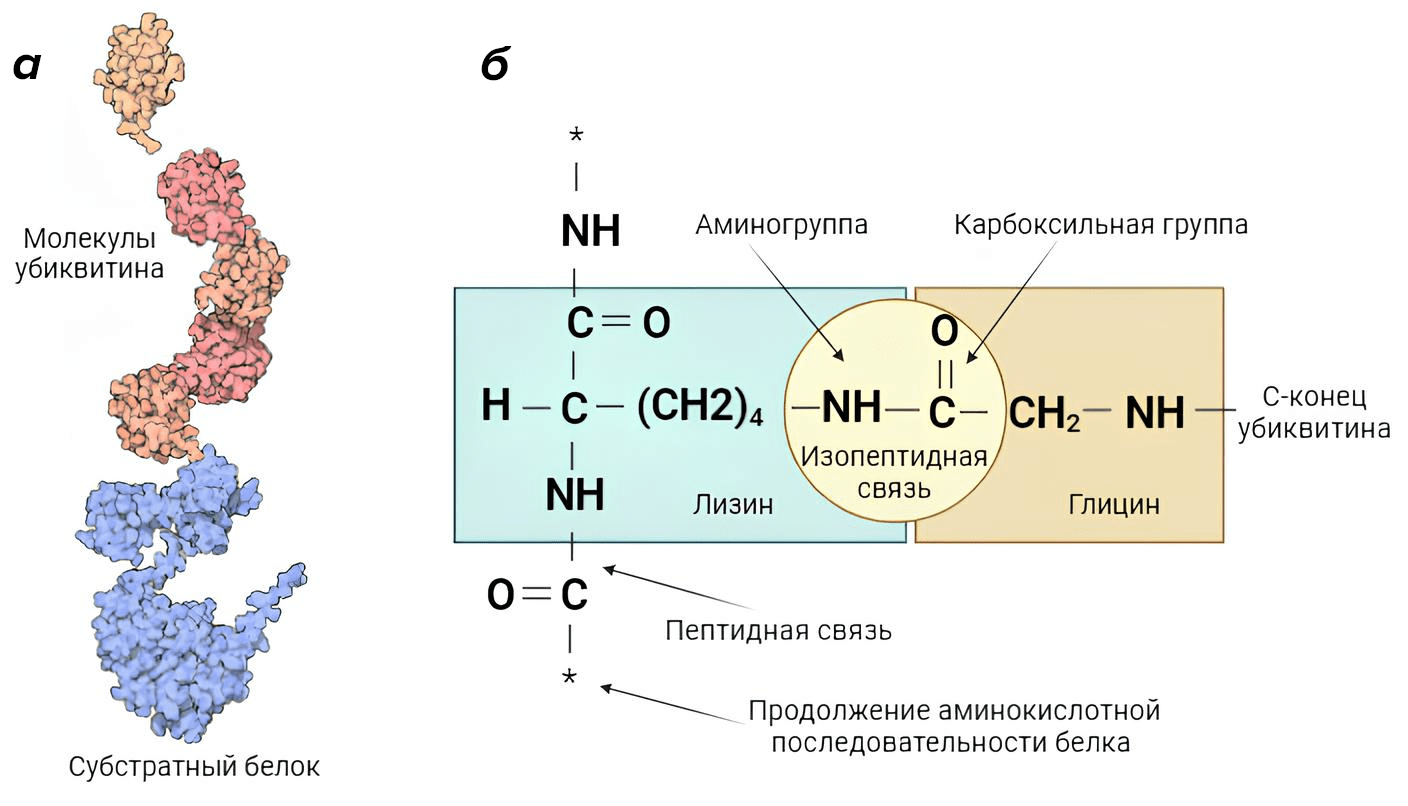
Рисунок 1. (а) Полиубиквитиновая цепь связанная с субстратным белком. (б) Изопептидная связь между лизином субстратного белка и глицином убиквитина. Также взаимодействуют и молекулы убиквитина между собой в составе полиубиквитиновой цепи.
[6], рисунок авторов
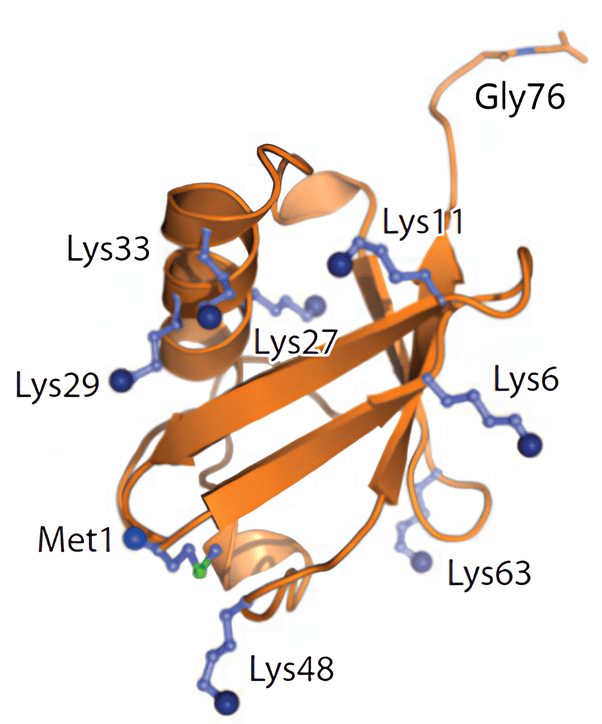
Рисунок 2. Трехмерная структура убиквитина, выделены семь остатков лизина (Lys), N-концевой метионин (Met-1) и С-концевой глицин (Gly-76).
Всего в состав молекулы убиквитина входит семь остатков лизина, которые могут участвовать в формировании полиубиквитиновой цепочки (рис. 2) [1]. Этим в частности обусловлено основное разнообразие сигнальных последовательностей на основе убиквитина. Стоит отметить, что помимо полиубиквитиновых цепей, в которых взаимодействие осуществляется через лизин, известны так называемые линейные цепи, построенные через N-концевой метионин (Met1, M1) и также регулирующие клеточные процессы посредством направленной деградации белков [7].
Формирование, декодирование и удаление убиквитинового сигнала
Прежде чем двигаться дальше и перейти к рассмотрению разнообразия кодируемых убиквитином сигналов, следует обратить внимание на ключевые ферменты, которые участвуют в самом процессе убиквитинирования, удалении убиквитиновой метки и распознавании сигнала.
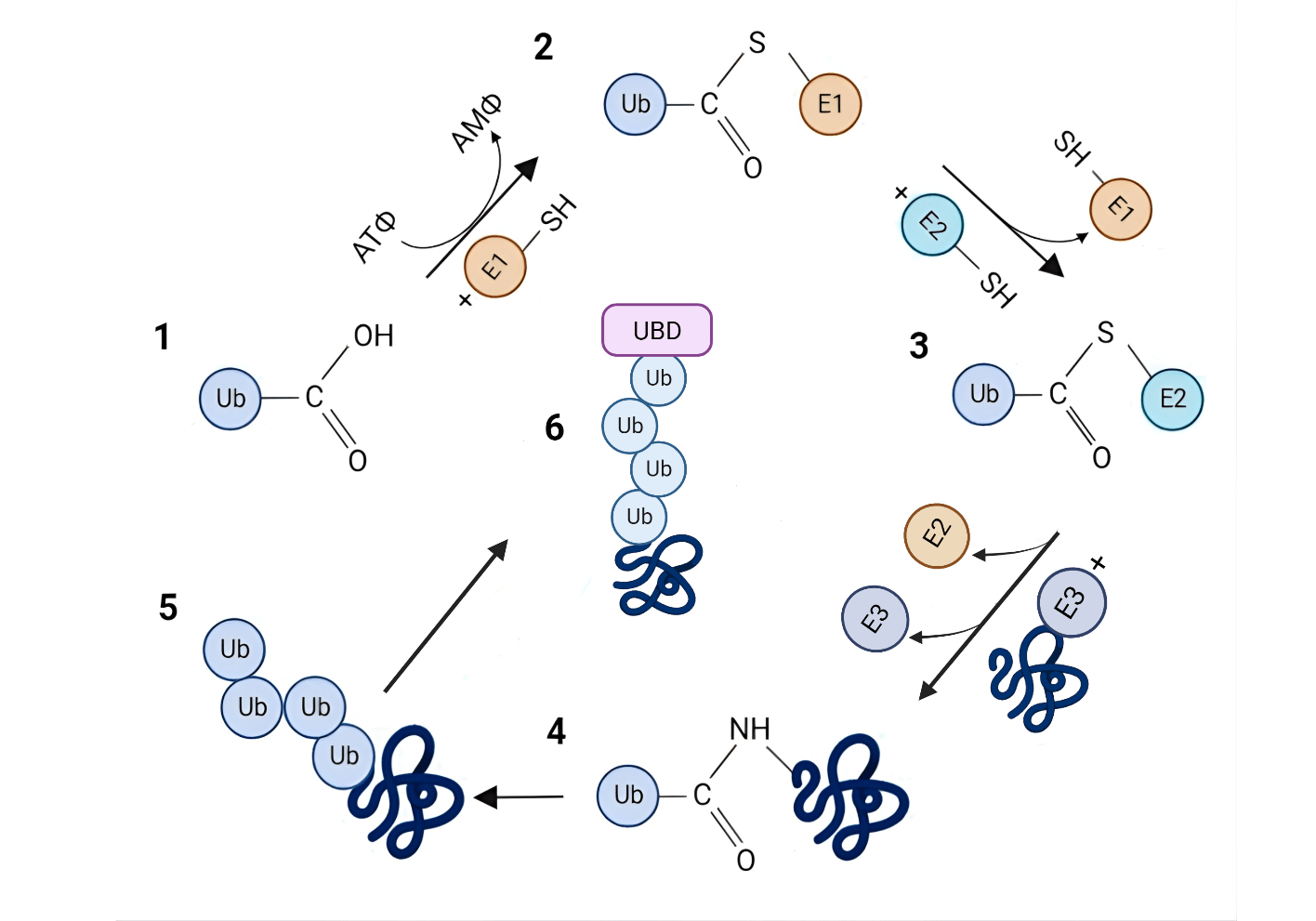
Процесс присоединения убиквитина к субстрату включает три основные стадии: активация-конъюгация-лигирование, катализируемые соответствующими ферментами E1, E2 и E3. На первой стадии под действием убиквитин-активирующего фермента E1 происходит АТФ-зависимое аденилирование C-концевой карбоксильной группы убиквитина, которая далее участвует в образовании тиоэфирной связи с каталитическим остатком цистеина Е1. После этого осуществляется перенос убиквитина с E1 на каталитический цистеин фермента Е2. На следующем этапе убиквитин-лигаза E3 взаимодействует с белком мишенью и ферментом E2, сближая их на расстояние, достаточное для переноса убиквитина на субстрат [10], [11] (рис 3). Именно ферменты E3 обладают субстратной специфичностью и, следовательно, высоким разнообразием. В клетках эукариот обнаружено 2 фермента типа E1, около 40 ферментов типа E2 и более 600 ферментов типа E3 [11]. Ферменты E3 на основе структурного сходства, механизма переноса убиквитина и функциональных классов субстратов, которые они распознают, группируются в четыре основных семейства: RING, HECT, RBR и U-box [12].
Распознавание убиквитиновых сигналов, в свою очередь обусловлено белками, содержащими один или несколько убиквитин-связывающих доменов (UBD, от ubiquitin-binding domains) и получивших обобщенное название «ридеры или декодеры» [13]. Белки, содержащие UBD, расшифровывают убиквитиновый код (тип связи и длину цепи) и опосредуют осуществление определенных клеточных процессов, например, доставляют убиквитинированные субстраты к протеасоме (RAD23 и UBQLN) или к аутофагосомным вакуолям (p62), другие участвуют в несвязанных с протеолизом процессах например, в повреждении ДНК и воспалении [14]. В геноме человека кодируется не менее 20 типов UBD, содержащихся в сотнях белков [15].
Наличие убиквитиновой метки на молекуле субстрата еще не гарантирует выполнение функции, обусловленной сигнальной убиквитиновой последовательностью. Убиквитиновый сигнал может быть удален с помощью деубиквитиназ (DUB) путем расщепления изопептидной связи убиквитина на конце цепи (экзопептидазная активность) или внутри полимера (эндопептидазная активность). Деубиквитиназ в геноме человека насчитывается около 100 [16].
Таким образом, ферменты участвующие в убиквитиновом сигналинге можно разделить на три ключевые группы:
- Writers — писатели. Ферменты, формирующие сигнал (E3 — убиквитин-лигазы).
- Erasers — ластики. Ферменты, стирающие убиквитиновый сигнал — деубиквитиназы (DUB).
- Decoders — декодеры. Белки, декодирующие сигнал. Содержат домен UBD.
Разнообразие модификаций субстрата убиквитином и кодируемые ими сигналы
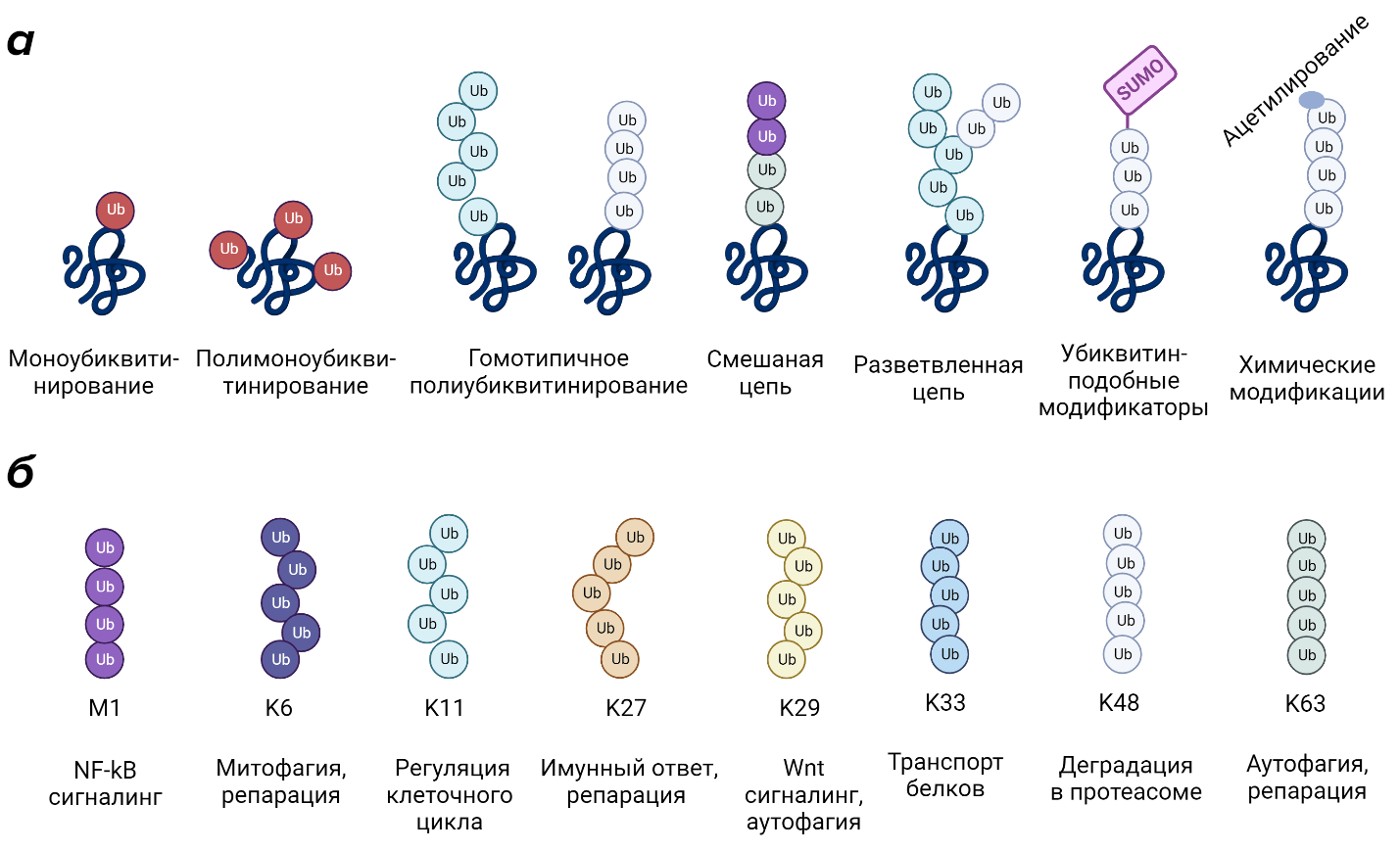
Рассмотрим разнообразие сигнальных последовательностей на основе убиквитина, а заодно и обратим внимание на многообразие опосредуемых ими клеточных процессов. В порядке увеличения сложности сигнала,которая выражается в конфигурации цепи, принято выделять следующие типы последовательностей (рис. 4а):
- Моноубиквитинирование и полимоноубиквитирование — присоединение одной или нескольких одиночных молекул убиквитина к разным участкам субстрата. Данные виды модификаций могут служить сигналом для эндоцитоза трансмембранных рецепторов, регулировать активность, субклеточную локализацию и белок-белковые взаимодействия субстрата. Моноубиквитинирование гистонов задействовано в регуляции транскрипции и репарации ДНК за счет изменения структуры хроматина и привлечения белков репарации соответственно [17], [18].
- Гомотипичное полиубиквитинирование — в данном случае цепь состоит из нескольких молекул убиквитина. В образовании такой цепи задействован лишь один из остатков лизина, входящих в состав убиквитина, например, цепи, образованные через K63, K48, K11 и др., либо цепь, в которой молекулы убиквитина связаны между собой через N-концевой метионин (Met1). Всего возможно образование 8 типов цепей данной конфигурации (K6, K11, K27, K29, K33, K48, K63, Met1). Они кодируют разнообразные сигналы, среди которых деградация субстрата в протеасоме (K48), регуляция клеточного цикла (K11), митофагия (K6), иммунный ответ (К27), аутофагия (К29, К63), репарация ДНК (К6, К27, К63), транспорт белков (К33), NF-kB сигналинг (M1) и другие [3] (рис. 4б).
- Гетеротипичные смешанные цепи — это полиубиквитиновые цепи, в образовании которых участвуют несколько аминокислотных остатков убиквитина, например, K63/Met1. Данный вид цепей собирается комплексом LUBAC (от linear ubiquitin chain assembly complex) и участвует в изменении активности транскрипционного фактора NF-κB, тем самым влияя на регуляцию процессов апоптоза, клеточного цикла и иммунного ответа [7], [19], [20].
- Разветвленные цепи — полиубиквитиновые цепи, которые по мере роста разделяются на несколько ветвей. В качестве примера можно привести модификацию регуляторов клеточного цикла и неправильно свернутых белков разветвленной цепью K11/K48, которая приводит к их направленной деградации [21], [22].
- Убиквитин в составе цепи сам может быть модифицирован за счет фосфорилирования, ацетилирования и других посттрансляционных модификаций. Например, ацетилирование убиквитина по сайтам лизина блокирует их дальнейшее участие в формировании полиубиквитиновых последовательностей [9], [23].
- Убиквитин-подобные белки (UBL от ubiquitin-like protein). Помимо самого убиквитина, в клетках присутствуют различные белки, осуществляющие схожие функции, например SUMO (от small ubiquitin-like modifier) который вовлечен в регуляцию таких клеточных процессов, как ядерный транспорт, транскрипция, репарация и репликация ДНК, апоптоз и стабилизация белков [3], [5], [24].
Убиквитин-опосредованная деградация белка в протеасоме
Разнообразие убиквитиновых сигналов велико, а сам убиквитиновый код неоднозначный и запутанный. В рамках данной статьи мы подробно рассмотрим только убиквитин-зависимую деградацию белка в протеасоме, за раскрытие механизма которой, Аврам Гершко, Аарон Чехановер и Ирвин Роуз были удостоены Нобелевской премии по химии в 2004 году (рис. 5) [36].

Рисунок 5. Нобелевские лауреаты по химии 2004 года. Слева направо: Аврам Гершко, Аарон Чехановер, Ирвин Роуз.
Поддержание белкового гомеостаза в клетке в основном осуществляется за счет убиквитин-протеасомной (УПС) и аутофаго-лизосомальной (АЛС) систем [23]. АЛС участвует в утилизации крупных белковых агрегатов, органелл и патогенов. Протеасомной деградации в клетке, в свою очередь, подвергаются белки, поврежденные в ходе стресса, разупорядоченные или неправильно свернутые, а также короткоживущие, например, циклины. УПС и АЛС ответственны примерно за 80–90% и 10–20% клеточного протеолиза соответственно. Соотношение может варьировать в зависимости от физиологического состояния и типа клеток [37]. Кроме того, работа данных систем имеет перекрывающиеся механизмы, ведь деградация белка как в УПС, так и в АЛС может быть инициирована убиквитином [38], [39].
Классическим сигналом для деградации субстрата в протеасоме является полиубиквитиновая цепь из четырех молекул, связанных по К48 [40]. Тем не менее, не все так однозначно, и механизм убиквитин-зависимой деградации белка в протеасоме намного сложнее устроен и может быть опосредован не только канонической последовательностью из четырех убиквитинов. Например, деградация белка в протеасоме может быть опосредована смешанной цепью убиквитина K11/K48, разветвленной цепью К48/К63 или моноубиквитинированием по нескольким сайтам [41], [42]. Кроме того, возможна и убиквитин-независимая деградация белка в протеасоме [43], например, такому типу утилизации подвергается известный опухолевый супрессор p53 [44].
Строение и разнообразие протеасом
На этом этапе мы с вами детально познакомимся со вторым, но не менее интересным и важным героем запутанных клеточных сетей — протеасомой. Протеасома — это молекулярная машина, предназначенная для разрушения дефектных или короткоживущих белков в клетке. Она, имеет бочкообразную форму и представляет из себя крупномолекулярную мультикаталитическую протеазу, которая способна гидролизовать белки до более мелких пептидов как по убиквитинзависимому, так и по убиквитиннезависимому механизму [43]. Она состоит из каталитически активной коровой частицы 20S массой 750 кДа и набора различных регуляторных компонентов, способных при взаимодействии с коровой частицей менять субстратную специфичность протеасомы [45]. Регуляторы, взаимодействующие с протеасомой, мы рассмотрим немного позже. А сейчас давайте разберемся в строении частицы 20S.
Коровая частица протеасомы 20S
Базовая архитектура протеасомы представлена коровой частицей 20S которая имеет форму полого цилиндра состоящего из четырех сложенных в стопку кольцевых структур — двух идентичных внешних α-колец и двух идентичных внутренних β-колец. Данные кольца состоят из родственных субъединиц, которые являются продуктами двух групп гомологичных генов (α и β) возникших в результате дупликации еще на заре эволюции. . В состав каждого кольца входит по 7 обособленных субъединиц (рис. 6а). У эукариот, в отличие, от бактерий и архей все α и β субъединицы различны [46].
Функция внешних α-колец заключается в контроле доступа субстрата к внутренней протеолитической полости протеасомы. Также они служат для взаимодействия с различными регуляторными компонентами. Во внутренних β-кольцах локализованы каталитические субъединицы протеасомы — β1, β2 и β5. Они обладают каспазной, трипсиновой и химотрипсиновой активностью и могут гидролизовать белки по кислым, основным и гидрофобным аминокислотным остаткам соответственно (рис. 6а). Длина эукариотической 20S коровой частицы протеасомы составляет около 150 Å, а диаметр — около 115 Å [4].
Внутри протеасомы находятся три взаимосвязанные полости: две из них образованы соседними α- и β-кольцами (протеасомные ворота), а центральная каталитическая полость ограничена двумя внутренними β-кольцами (рис. 6б). Активные центры каталитических субъединиц протеасомы изолированы внутри центральной полости, а входные отверстия закрыты. Таким образом, окружающие протеасому ядерные или цитоплазматические белки защищены от неконтролируемой деградации [4].
Доступ субстрата в протеолитическую полость осуществляется за счет конформационных перестроек в N-концевых участках α-субъединиц. Они могут быть индуцированы регуляторными частицами после связывания с убиквитинированным субстратом или опосредованы взаимодействием с неструктурированным белком, имеющим специфичную последовательность [47].
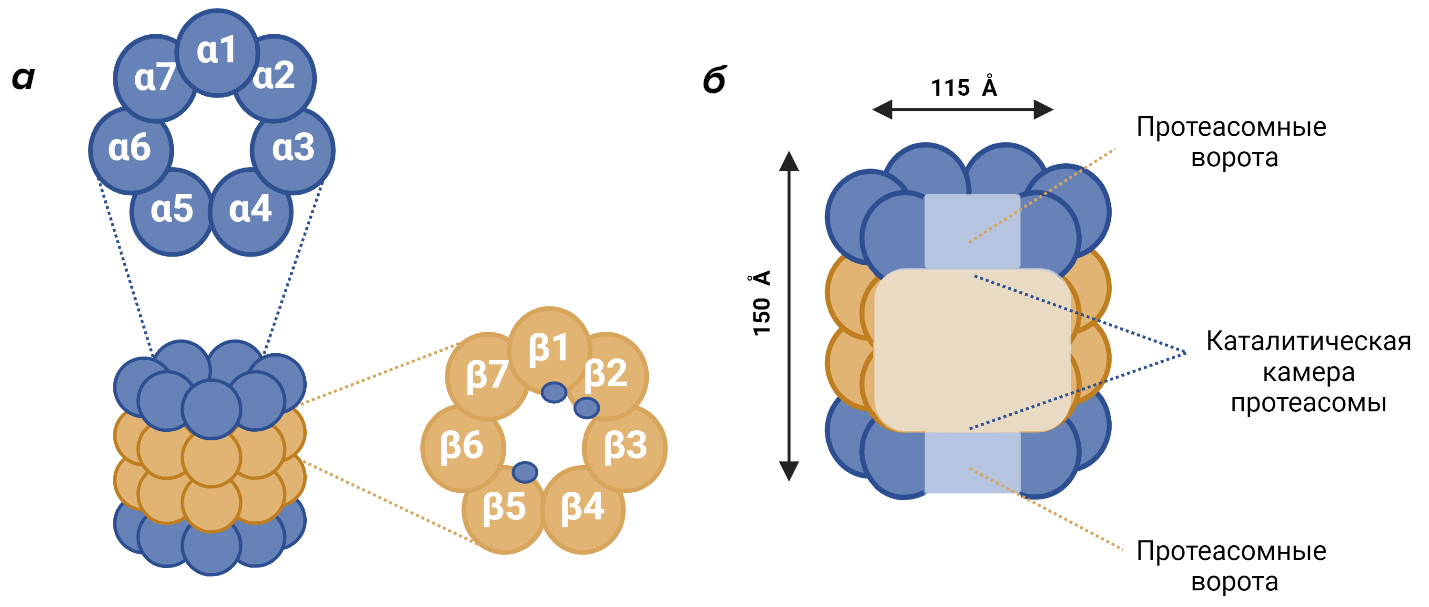
Рисунок 6. Строение коровой 20S частицы протеасомы эукариот. Представлены α и β-кольца (а) и основные внутренние полости протеасомы (б).
оригинальная иллюстрация на основе литературных данных [4]
Протеасомы с одинаковой архитектурой обнаружены у всех царств живых организмов: эукариот, архей и некоторых бактерий. Протеасома — очень древняя молекулярная машина, формирование которой, вероятно, началось еще у LUCA (от last universal common ancestor — последний универсальный общий предок). В отличие от ферментов каскада убиквитинирования, гены, кодирующие субъединицы протеасом, гомологичны у архей, бактерий и эукариот, что указывает на то, что они появились еще до разделения бактерий и архей, но у ряда бактерий были утрачены в течение эволюции [48].
Типы 20S протеасом
В 20S частице при определенных условиях или тканеспецифично могут быть заменены α или β субъединицы [49]. В настоящее время известно четыре типа 20S протеасом — конститутивная, иммунопротеасома, тимопротеасома и сперматопротеасома (рис. 7), а также несколько промежуточных типов [4].
Иммунопротеасома продуцируется и играет особенно важную роль в лимфоидных тканях. Кроме того, ее образование может быть индуцировано в других тканях организма во время иммунного ответа, направленного на борьбу с инфекциями, вызванными внутриклеточными патогенами [50], или при окислительном стрессе. Так, при взаимодействии клетки с медиаторами воспаления (интерферон γ, фактор некроза опухоли) происходит замена конститутивных каталитических субъединиц β1, β2, β5 индуцибельными субъединицами β1i, β2i, b5i, формируя таким образом иммунопротеасому. Известно, что иммунопротеасома эффективнее генерирует пептиды, выступающие в качестве антигенов для презентации главным комплексом гистосовместимости I класса (MHC-I). Это обусловлено более выраженной химотрипсиновой активностью иммунопротеасомы (табл. 1), которая обеспечивает гидролиз субстрата после гидрофобных аминокислот [4]. Также известна роль иммунопротеасом в деградации окисленных белков [51].
При замене в иммунопротеасоме β5i на β5t образуется тимопротеасома, которая является компонентом кортикальных эпителиальных клеток тимуса и участвует в селекции T-лимфоцитов [52]. Также известен особый вид протеасомы, обнаруженный в сперматоцитах — сперматопротеасома. Она отличается от стандартной протеасомы заменой α4 субъединицы на α4s и участвует в деградации ацетилированных гистонов и поддержании структуры хромосом [4], [53].
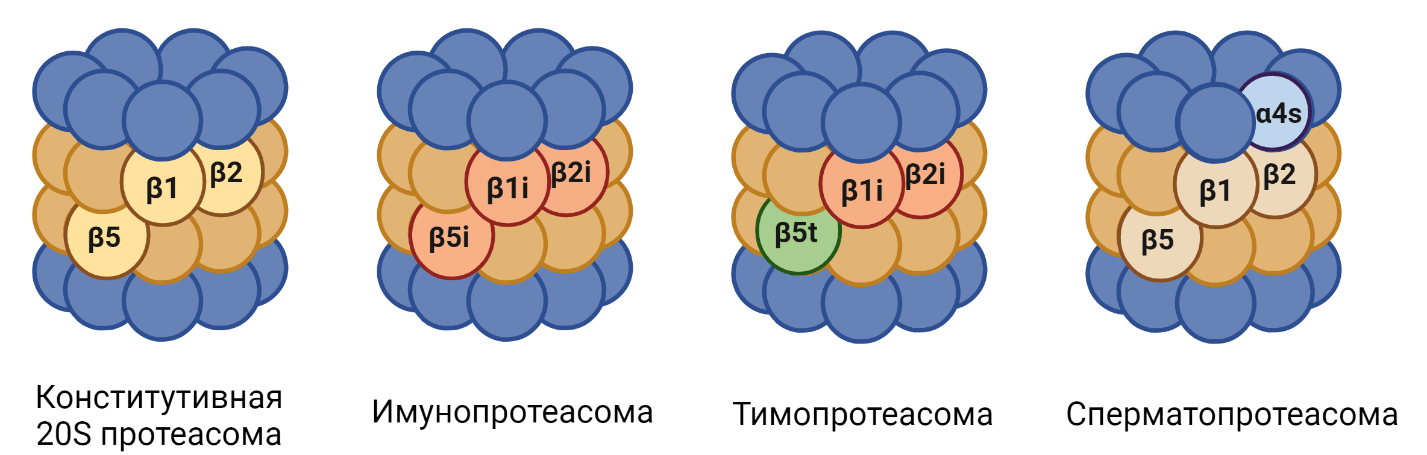
Рисунок 7. Индуцибельные субъединицы различных типов 20S протеасом.
рисунок авторов
| Субъединица | Пептидазная активность |
|---|---|
| β1 | Каспазная |
| β2 | Трипсиновая |
| β5 | Химотрипсиновая |
| β1i | Химотрипсиновая |
| β2i | Трипсиновая |
| β5i | Химотрипсиновая |
| β5t | Сниженная химотрипсиновая |
| α4s | - |
Регуляторные частицы протеасомы
Вот мы наконец-то добрались и до регуляторных компонентов протеасомы, о которых упоминали ранее. Наиболее распространенной регуляторной частицей (RP от regulatory particle) является 19S RP массой 700 кДа. Она может быть связана с внешними α-кольцами коровой 20S частицы с одной или с двух сторон, формируя, таким образом, 26S или 30S протеасому соответственно. Регулятор 19S RP можно разделить на два субкомплекса — «основание» и «крышка». Основание состоит из кольца, включающего 6 гомологичных субъединиц с АТФ-азной активностью (Rpt1-6), и 4-ех субъединиц, не обладающих АТФ-азной активностью (Rpn1, Rpn2, Rpn10, Rpn13) (рис. 8а). Кольцо из АТФаз взаимодействуют с внешним α–кольцом 20S частицы и осуществляет открытие доступа к протеолитической полости, АТФ-зависимое разворачивание и транслокацию субстрата. Субъединицы Rpn1, Rpn10 и Rpn13 являются рецепторами убиквитина и необходимы соответственно для распознавания убиквитинированного субстрата [54].
В «крышке» 19S локализованы неАТФ-азные субъединицы (Rpn3, Rpn5-Rpn9, Rpn11, Rpn12, Rpn15). Rpn11 является деубиквитиназой, которая отщепляет молекулы убиквитина от субстрата, предназначенного для деградации, а остальные субъединицы играют структурную функцию, в том числе обеспечивающую расположение деубиквитиназы Rpn11 над АТФазным кольцом (рис. 8а).
Таким образом, ключевые функции регулятора 19S — распознавание убиквитинированных белков, открытие α-кольца, АТФ-зависимое разворачивание и транслокация субстрата в протеолитическую полость, а также предшествующее деградации деубиквитинирование субстрата [54], [55]. За счет наличия трех рецепторов убиквитина в 19S RP возможно узнавание различных полиубиквитиновых цепей (рис. 8б) [42].
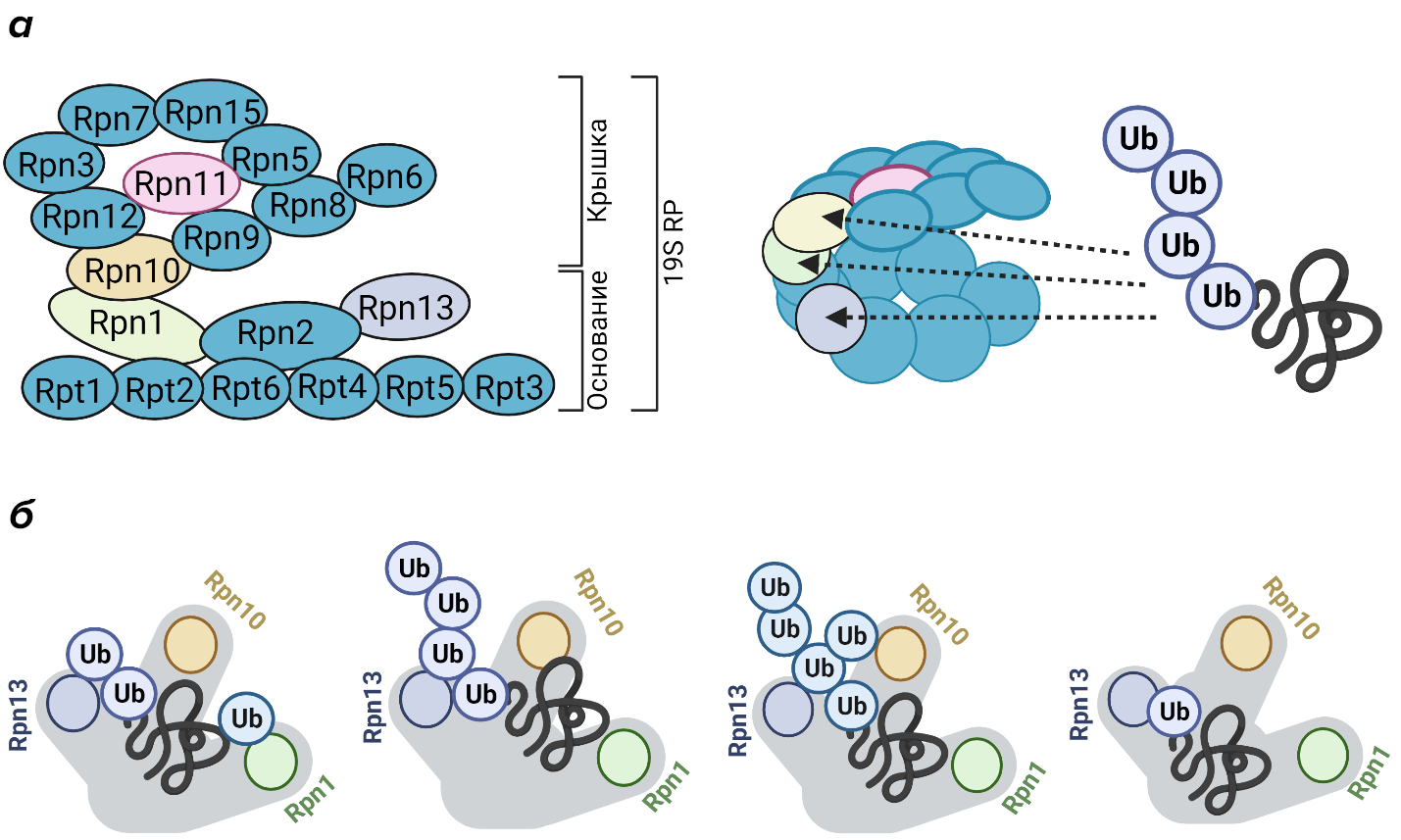
Рисунок 8. Строение регуляторной частицы протеасомы 19S (а) и ее взаимодействие с убиквитинированными белками (б).
оригинальная иллюстрация на основе литературных данных [41]
Разнообразие регуляторных компонентов протеасомы
Конечно, одним регулятором не обойтись и эукариотическая клетка запаслась еще несколькими. Среди них регулятор PA28 (он же REG). PA28 представляет из себя кольцеобразную гепта или гексамерную структуру, субъединичный состав которой может быть представлен разным сочетанием двух гомологичных α (REGα) и β (REGβ) субъединиц (PA28αβ) либо одной γ субъединицей (PA28γ) [51].
Экспрессия PA28α и PA28β наблюдается совместно с иммуносубъединицами протеасомы β1i, β2i и β5i, что приводит к образованию иммунопротеасом PA28αβ-20S. Регулятор PA28αβ, также как иммунопротеасома, принимает участие в поддержании белкового гомеостаза во время окислительного стресса, а также в генерации пептидов для презентации антигена [50], [51].
Функции PA28γ (REGγ) активно изучаются, например, недавно была показана его роль в поддержании плюрипотентности эмбриональных стволовых клеток мыши за счет регуляции образования P-телец [56].
Известны и другие типы регуляторов протеасом, например, белок PA200/Blm10, высокий уровень продукции которого наблюдается в семенниках [57], а также белок PI31, способный ингибировать 20S протеасому [58], и Ecm29, который опосредует взаимодействие протеасомы с различными клеточными компартментами [59]. Кроме того, гексамер VCP/P97 участвует, помимо прочего, в деградации белков, ассоциированных с эндоплазматическим ретикулумом [60].
Гетерогенность популяции протеасом в клетках эукариот также обусловлена формированием гибридных протеасом, которые образуются за счет взаимодействия 20S коровой частицы с двумя типами различных регуляторов, например, 19S-20S-PA200 [49]. Теоретически возможное разнообразие таких структур достаточно велико, и существование не всех из них еще доказано экспериментально (рис 9).
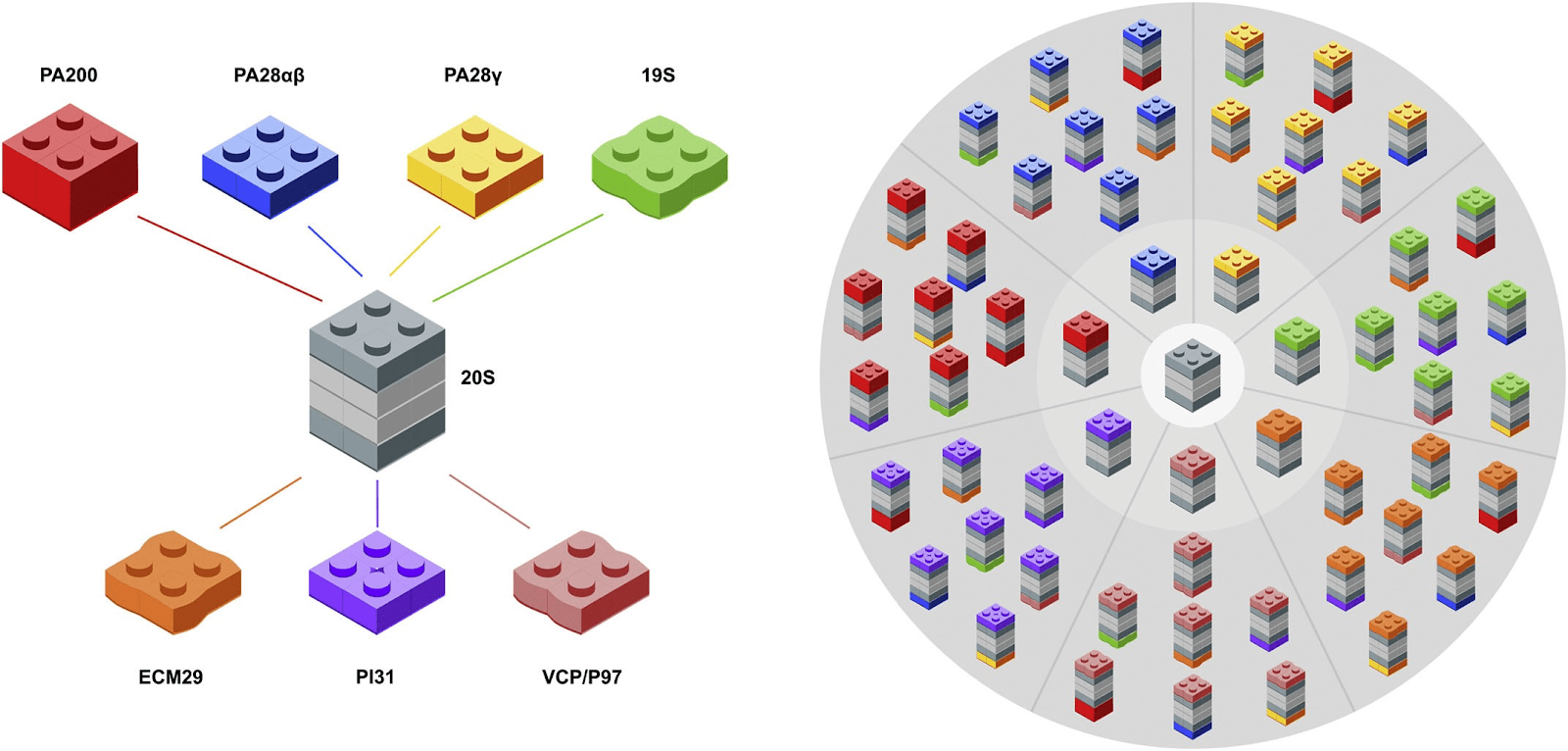
Рисунок 9. Регуляторные компоненты протеасомы и теоретически возможное разнообразие типов протеасом на основе 20S коровой частицы и различных регуляторов.
Убиквитин-протеасомная система в патогенезе и медицине
Как уже было сказано, за счет способности к избирательной деградации белков убиквитин-протеасомная система участвует в регуляции основных клеточных процессов. Ее нормальная работа необходима для поддержания гомеостаза клеток и, следовательно, всего организма как при нормальных условиях, так и в условиях стресса. Неудивительно, что данная система также задействована и в патогенезе различных заболеваний. Нарушения в УПС могут приводить к дезактивации или аберрантной активации сигнальных путей, дефектам при иммунном ответе, протеотоксическому стрессу ввиду накопления поврежденных или неправильно свернутых белков [62], [63]. Данные эффекты, в свою очередь, ассоциированы с онкологическими [64], аутоиммунными [65] и нейродегенеративными заболеваниями соответственно [66]. В настоящее время разрабатываются различные медицинские препараты, мишенями для которых выступают компоненты УПС.
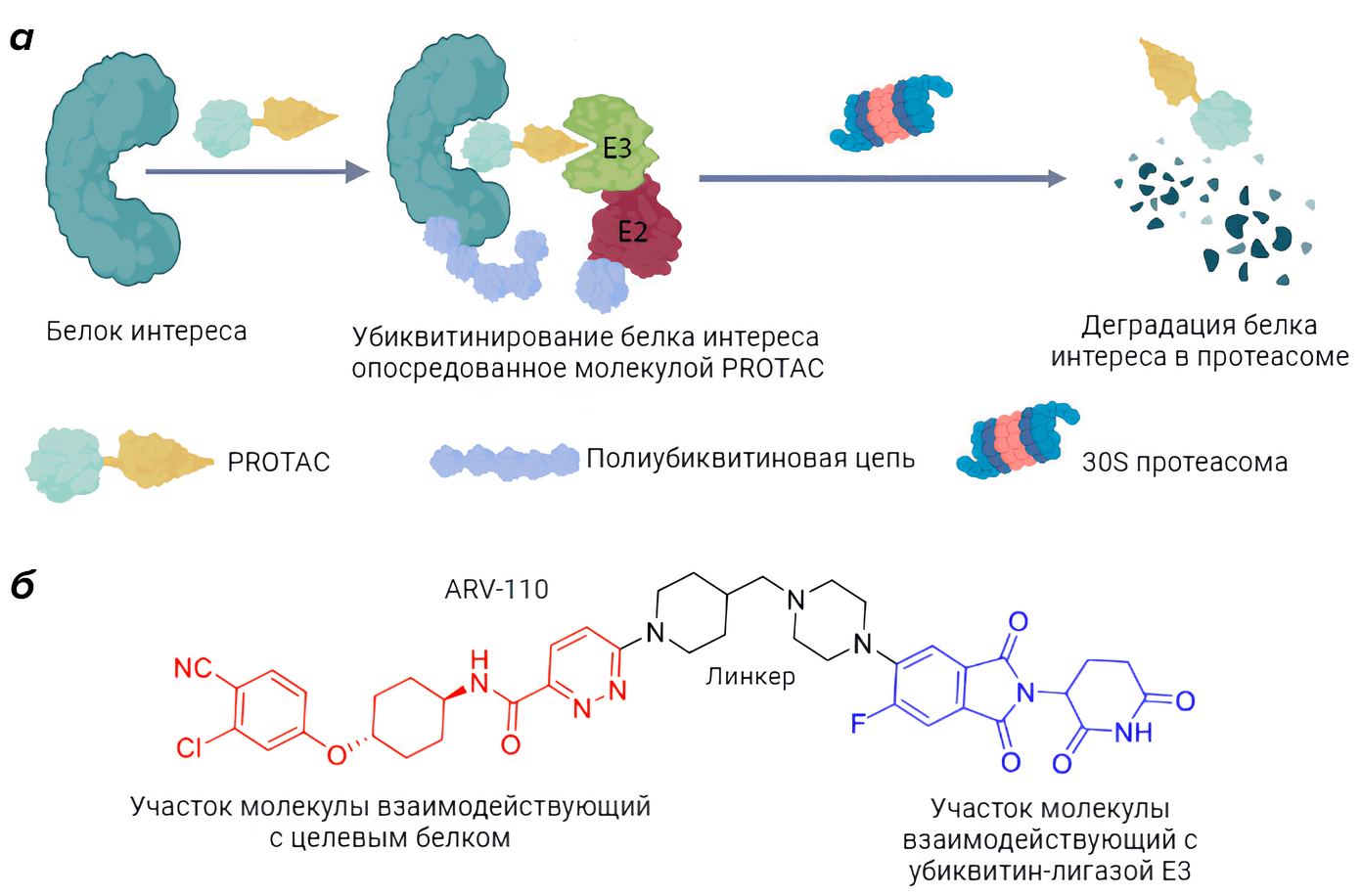
Кроме того, значительный интерес как для прикладных, так и для фундаментальных исследований представляет возможность управлять убиквитинированием белков в целях регуляции клеточных процессов. Наиболее привлекательная сегодня технология заключается в разработке молекул PROTACs (PROteolysis TArgeting Chimeras). PROTAC представляет из себя бифункциональную молекулу, способную взаимодействовать с убиквитинлигазой E3 и белком интереса. В результате такого взаимодействия белок интереса подвергается убиквитинированию и последующей деградации в протеасоме (рис. 10А) [67], [68]. На основе данной технологии разрабатываются лекарственные препараты, например ARV-110 (рис. 10Б) который предназначен для лечения тяжелой формы рака предстательной железы и уже успешно прошедший 1 и 2 фазу клинических испытаний [69].
Заключение
В заключение хотелось бы вернуться к поражающему разнообразию компонентов убиквитин-протеасомной системы. Учитывая ферменты принимающие участие в убиквитиновом сигналинге, различные конфигурации цепей на основе убиквитина, типы протеасом и их регуляторные частицы, число компонентов превышает тысячу. Таким образом убиквитин-протеасомная система «следит» практически за всем, что происходит в клетке и регулирует протекающие в ней процессы. Конечно, осталось еще много интересных вопросов, которые мы не успели разобрать, но авторы надеются, что этот запутанный молекулярный мир, в котором убиквитин и протеасомы вершат «судьбы» белков, стал для читателей чуточку понятнее.
Литература
- David Komander, Michael Rape. (2012). The Ubiquitin Code. Annu. Rev. Biochem.. 81, 203-229;
- Eugene Oh, David Akopian, Michael Rape. (2018). Principles of Ubiquitin-Dependent Signaling. Annu. Rev. Cell Dev. Biol.. 34, 137-162;
- Kirby N Swatek, David Komander. (2016). Ubiquitin modifications. Cell Res. 26, 399-422;
- Joanna Abi Habib, Julie Lesenfants, Nathalie Vigneron, Benoit J. Van den Eynde. (2022). Functional Differences between Proteasome Subtypes. Cells. 11, 421;
- Laurent Cappadocia, Christopher D. Lima. (2018). Ubiquitin-like Protein Conjugation: Structures, Chemistry, and Mechanism. Chem. Rev.. 118, 889-918;
- Goodsell D.S. Bionanotechnology: Lessons from Nature (Life Sciences). NJ: Wiley-Blackwell, 2004. — 347 p.;
- Daisuke Oikawa, Yusuke Sato, Hidefumi Ito, Fuminori Tokunaga. (2020). Linear Ubiquitin Code: Its Writer, Erasers, Decoders, Inhibitors, and Implications in Disorders. IJMS. 21, 3381;
- Elsje G. Otten, Emma Werner, Ana Crespillo-Casado, Keith B. Boyle, Vimisha Dharamdasani, et. al.. (2021). Ubiquitylation of lipopolysaccharide by RNF213 during bacterial infection. Nature. 594, 111-116;
- Ivan Dikic, Brenda A. Schulman. (2023). An expanded lexicon for the ubiquitin code. Nat Rev Mol Cell Biol. 24, 273-287;
- Gary Kleiger, Thibault Mayor. (2014). Perilous journey: a tour of the ubiquitin–proteasome system. Trends in Cell Biology. 24, 352-359;
- Kirandeep K. Deol, Sonja Lorenz, Eric R. Strieter. (2019). Enzymatic Logic of Ubiquitin Chain Assembly. Front. Physiol.. 10;
- Quan Yang, Jinyao Zhao, Dan Chen, Yang Wang. (2021). E3 ubiquitin ligases: styles, structures and functions. Mol Biomed. 2;
- Daniel Scott, Neil J. Oldham, Jo Strachan, Mark S. Searle, Robert Layfield. (2015). Ubiquitin‐binding domains: Mechanisms of ubiquitin recognition and use as tools to investigate ubiquitin‐modified proteomes. Proteomics. 15, 844-861;
- Ivan Dikic, Soichi Wakatsuki, Kylie J. Walters. (2009). Ubiquitin-binding domains — from structures to functions. Nat Rev Mol Cell Biol. 10, 659-671;
- Claire Heride, Sylvie Urbé, Michael J. Clague. (2014). Ubiquitin code assembly and disassembly. Current Biology. 24, R215-R220;
- Yi-Zheng Fang, Li Jiang, Qiaojun He, Ji Cao, Bo Yang. (2023). Deubiquitination complex platform: A plausible mechanism for regulating the substrate specificity of deubiquitinating enzymes. Acta Pharmaceutica Sinica B. 13, 2955-2962;
- Yujie Chen, Dandan Zhou, Yinan Yao, Yutong Sun, Fan Yao, Li Ma. (2022). Monoubiquitination in Homeostasis and Cancer. IJMS. 23, 5925;
- Liat Oss-Ronen, Tzlil Sarusi, Idan Cohen. (2022). Histone Mono-Ubiquitination in Transcriptional Regulation and Its Mark on Life: Emerging Roles in Tissue Development and Disease. Cells. 11, 2404;
- Fuminori Tokunaga, Kazuhiro Iwai. (2012). LUBAC, a novel ubiquitin ligase for linear ubiquitination, is crucial for inflammation and immune responses. Microbes and Infection. 14, 563-572;
- Kazuhiro Iwai, Hiroaki Fujita, Yoshiteru Sasaki. (2014). Linear ubiquitin chains: NF-κB signalling, cell death and beyond. Nat Rev Mol Cell Biol. 15, 503-508;
- Richard G. Yau, Kerstin Doerner, Erick R. Castellanos, Diane L. Haakonsen, Achim Werner, et. al.. (2017). Assembly and Function of Heterotypic Ubiquitin Chains in Cell-Cycle and Protein Quality Control. Cell. 171, 918-933.e20;
- Fumiaki Ohtake. (2022). Branched ubiquitin code: from basic biology to targeted protein degradation. The Journal of Biochemistry. 171, 361-366;
- Yong Tae Kwon, Aaron Ciechanover. (2017). The Ubiquitin Code in the Ubiquitin-Proteasome System and Autophagy. Trends in Biochemical Sciences. 42, 873-886;
- David A. Pérez Berrocal, Katharina F. Witting, Huib Ovaa, Monique P. C. Mulder. (2020). Hybrid Chains: A Collaboration of Ubiquitin and Ubiquitin-Like Modifiers Introducing Cross-Functionality to the Ubiquitin Code. Front. Chem.. 7;
- Sjoerd JL van Wijk, Simone Fulda, Ivan Dikic, Mike Heilemann. (2019). Visualizing ubiquitination in mammalian cells. EMBO Reports. 20;
- Mingwei Sun, Xiaofei Zhang. (2022). Current methodologies in protein ubiquitination characterization: from ubiquitinated protein to ubiquitin chain architecture. Cell Biosci. 12;
- E. Ozkaynak, D. Finley, M. J. Solomon, A. Varshavsky. (1987). The yeast ubiquitin genes: a family of natural gene fusions.. The EMBO Journal. 6, 1429-1439;
- Michael J. Pearce, Julian Mintseris, Jessica Ferreyra, Steven P. Gygi, K. Heran Darwin. (2008). Ubiquitin-Like Protein Involved in the Proteasome Pathway of Mycobacterium tuberculosis. Science. 322, 1104-1107;
- Mikhail Kavalchuk, Ahmad Jomaa, Andreas U. Müller, Eilika Weber-Ban. (2022). Structural basis of prokaryotic ubiquitin-like protein engagement and translocation by the mycobacterial Mpa-proteasome complex. Nat Commun. 13;
- Gilad Lehmann, Ronald G. Udasin, Ido Livneh, Aaron Ciechanover. (2017). Identification of UBact, a ubiquitin-like protein, along with other homologous components of a conjugation system and the proteasome in different gram-negative bacteria. Biochemical and Biophysical Research Communications. 483, 946-950;
- Takuro Nunoura, Yoshihiro Takaki, Jungo Kakuta, Shinro Nishi, Junichi Sugahara, et. al.. (2011). Insights into the evolution of Archaea and eukaryotic protein modifier systems revealed by the genome of a novel archaeal group. Nucleic Acids Research. 39, 3204-3223;
- Rory Hennell James, Eva F. Caceres, Alex Escasinas, Haya Alhasan, Julie A. Howard, et. al.. (2017). Functional reconstruction of a eukaryotic-like E1/E2/(RING) E3 ubiquitylation cascade from an uncultured archaeon. Nat Commun. 8;
- Adrian C. D. Fuchs, Lorena Maldoner, Matthias Wojtynek, Marcus D. Hartmann, Jörg Martin. (2018). Rpn11-mediated ubiquitin processing in an ancestral archaeal ubiquitination system. Nat Commun. 9;
- Katarzyna Zaremba-Niedzwiedzka, Eva F. Caceres, Jimmy H. Saw, Disa Bäckström, Lina Juzokaite, et. al.. (2017). Asgard archaea illuminate the origin of eukaryotic cellular complexity. Nature. 541, 353-358;
- Tomoyuki Hatano, Saravanan Palani, Dimitra Papatziamou, Ralf Salzer, Diorge P. Souza, et. al.. (2022). Asgard archaea shed light on the evolutionary origins of the eukaryotic ubiquitin-ESCRT machinery. Nat Commun. 13;
- Aaron Ciechanover. (2005). Intracellular Protein Degradation: From a Vague Idea, through the Lysosome and the Ubiquitin–Proteasome System, and onto Human Diseases and Drug Targeting (Nobel Lecture). Angew Chem Int Ed. 44, 5944-5967;
- Catherine Meyer-Schwesinger. (2019). The ubiquitin–proteasome system in kidney physiology and disease. Nat Rev Nephrol. 15, 393-411;
- Martina P. Liebl, Thorsten Hoppe. (2016). It's all about talking: two-way communication between proteasomal and lysosomal degradation pathways via ubiquitin. American Journal of Physiology-Cell Physiology. 311, C166-C178;
- Ivan Dikic. (2017). Proteasomal and Autophagic Degradation Systems. Annu. Rev. Biochem.. 86, 193-224;
- J. S. Thrower. (2000). Recognition of the polyubiquitin proteolytic signal. The EMBO Journal. 19, 94-102;
- Guinevere L. Grice, James A. Nathan. (2016). The recognition of ubiquitinated proteins by the proteasome. Cell. Mol. Life Sci.. 73, 3497-3506;
- Takuya Tomita. (2022). Structural and biochemical elements of efficiently degradable proteasome substrates. The Journal of Biochemistry. 171, 261-268;
- Indrajit Sahu, Sachitanand M. Mali, Prasad Sulkshane, Cong Xu, Andrey Rozenberg, et. al.. (2021). The 20S as a stand-alone proteasome in cells can degrade the ubiquitin tag. Nat Commun. 12;
- P Tsvetkov, N Reuven, Y Shaul. (2010). Ubiquitin-independent p53 proteasomal degradation. Cell Death Differ. 17, 103-108;
- Keiji Tanaka. (2009). The proteasome: Overview of structure and functions. Proceedings of the Japan Academy. Ser. B: Physical and Biological Sciences. 85, 12-36;
- Adrian C. D. Fuchs, Marcus D. Hartmann. (2019). On the Origins of Symmetry and Modularity in the Proteasome Family. BioEssays. 41;
- Indrajit Sahu, Michael H. Glickman. (2021). Structural Insights into Substrate Recognition and Processing by the 20S Proteasome. Biomolecules. 11, 148;
- Adrian C D Fuchs, Vikram Alva, Andrei N Lupas. (2021). An astonishing wealth of new proteasome homologs. Bioinformatics. 37, 4694-4703;
- Alexey V. Morozov, Vadim L. Karpov. (2018). Biological consequences of structural and functional proteasome diversity. Heliyon. 4, e00894;
- Shigeo Murata, Yousuke Takahama, Masanori Kasahara, Keiji Tanaka. (2018). The immunoproteasome and thymoproteasome: functions, evolution and human disease. Nat Immunol. 19, 923-931;
- Bart L. van den Eshof, Lobna Medfai, Emanuele Nolfi, Magdalena Wawrzyniuk, Alice J. A. M. Sijts. (2021). The Function of Immunoproteasomes—An Immunologists’ Perspective. Cells. 10, 3360;
- Yousuke Takahama, Kensuke Takada, Shigeo Murata, Keiji Tanaka. (2012). β5t-containing thymoproteasome: specific expression in thymic cortical epithelial cells and role in positive selection of CD8+ T cells. Current Opinion in Immunology. 24, 92-98;
- Zi-Hui Zhang, Tian-Xia Jiang, Lian-Bin Chen, Wenhui Zhou, Yixun Liu, et. al.. (2021). Proteasome subunit α4s is essential for formation of spermatoproteasomes and histone degradation during meiotic DNA repair in spermatocytes. Journal of Biological Chemistry. 296, 100130;
- Jared A.M. Bard, Ellen A. Goodall, Eric R. Greene, Erik Jonsson, Ken C. Dong, Andreas Martin. (2018). Structure and Function of the 26S Proteasome. Annu. Rev. Biochem.. 87, 697-724;
- Yasushi Saeki. (2017). Ubiquitin recognition by the proteasome. J Biochem. mvw091;
- Federico Pecori, Nanako Kondo, Chika Ogura, Taichi Miura, Masahiko Kume, et. al.. (2021). Site-specific O-GlcNAcylation of Psme3 maintains mouse stem cell pluripotency by impairing P-body homeostasis. Cell Reports. 36, 109361;
- Laura Gómez-H, Natalia Felipe-Medina, Yazmine B. Condezo, Rodrigo Garcia-Valiente, Isabel Ramos, et. al.. (2019). The PSMA8 subunit of the spermatoproteasome is essential for proper meiotic exit and mouse fertility. PLoS Genet. 15, e1008316;
- Xiaohua Li, David Thompson, Brajesh Kumar, George N. DeMartino. (2014). Molecular and Cellular Roles of PI31 (PSMF1) Protein in Regulation of Proteasome Function. Journal of Biological Chemistry. 289, 17392-17405;
- Andrea Lehmann, Agathe Niewienda, Katharina Jechow, Katharina Janek, Cordula Enenkel. (2010). Ecm29 Fulfils Quality Control Functions in Proteasome Assembly. Molecular Cell. 38, 879-888;
- Johannes van den Boom, Hemmo Meyer. (2018). VCP/p97-Mediated Unfolding as a Principle in Protein Homeostasis and Signaling. Molecular Cell. 69, 182-194;
- Xinyuan Wang, Thomas Meul, Silke Meiners. (2020). Exploring the proteasome system: A novel concept of proteasome inhibition and regulation. Pharmacology & Therapeutics. 211, 107526;
- Michael Rape. (2018). Ubiquitylation at the crossroads of development and disease. Nat Rev Mol Cell Biol. 19, 59-70;
- Aaron Ciechanover. (2005). Intracellular Protein Degradation: From a Vague Idea, through the Lysosome and the Ubiquitin–Proteasome System, and onto Human Diseases and Drug Targeting (Nobel Lecture). Angew Chem Int Ed. 44, 5944-5967;
- Gabriel LaPlante, Wei Zhang. (2021). Targeting the Ubiquitin-Proteasome System for Cancer Therapeutics by Small-Molecule Inhibitors. Cancers. 13, 3079;
- Dhananjay Yadav, Ji Yeon Lee, Nidhi Puranik, Pallavi S. Chauhan, Vishal Chavda, et. al.. (2022). Modulating the Ubiquitin–Proteasome System: A Therapeutic Strategy for Autoimmune Diseases. Cells. 11, 1093;
- Saeideh Momtaz, Zahra Memariani, Fardous F. El-Senduny, Nima Sanadgol, Fereshteh Golab, et. al.. (2020). Targeting Ubiquitin-Proteasome Pathway by Natural Products: Novel Therapeutic Strategy for Treatment of Neurodegenerative Diseases. Front. Physiol.. 11;
- Xiuyun Sun, Hongying Gao, Yiqing Yang, Ming He, Yue Wu, et. al.. (2019). PROTACs: great opportunities for academia and industry. Sig Transduct Target Ther. 4;
- Zi Liu, Mingxing Hu, Yu Yang, Chenghao Du, Haoxuan Zhou, et. al.. (2022). An overview of PROTACs: a promising drug discovery paradigm. Mol Biomed. 3;
- Xiaojuan Jia, Xin Han. (2023). Targeting androgen receptor degradation with PROTACs from bench to bedside. Biomedicine & Pharmacotherapy. 158, 114112.