Фальшивые мотивы в ДНК: как геномные варианты меняют поведение транскрипционных факторов
09 августа 2021
Фальшивые мотивы в ДНК: как геномные варианты меняют поведение транскрипционных факторов
- 1245
- 1
- 3
-
Автор
-
Редакторы
Работа клетки подобна работе оркестра, только вместо музыки она производит белки и РНК. Для правильного функционирования всей системы каждый ген должен «вступать» в нужный момент, скоординировано с другими генами, и давать столько продукта, сколько потребуется. Это значит, что транскрипция каждого гена должна происходить в определенное время и с определенной интенсивностью. Дирижерами процесса выступают специальные белки — факторы транскрипции. Партитура при этом записана в самой ДНК: партию определяют регуляторные последовательности, с которыми транскрипционный фактор связывается и в результате усиливает или ослабляет транскрипцию соответствующих генов. Замены в таких последовательностях могут приводить к изменению силы связывания и, как следствие, фальши в транскрипции: неверной или не вовремя сыгранной партии конкретного гена. Современные биологи активно пытаются решить вопрос о том, как устроены эти последовательности для каждого транскрипционного фактора и какие мутации в них будут влиять на связывание с белком. Одним из подходов к расшифровке клеточной партитуры является изучение аллель-специфичного связывания: когда варианты регуляторной последовательности, унаследованные от матери и от отца, различаются, можно изучать, с каким из них транскрипционный фактор связывается лучше. Несмотря на прозрачную постановку задачи, на пути к ее решению возникает ряд проблем. Мы придумали, как их преодолеть, и обнаружили сотни тысяч событий аллель-специфичного связывания, попутно показав их вклад в предрасположенность ко многим заболеваниям. Работа недавно опубликована в журнале Nature Communications.
Факторы транскрипции регулируют работу одного или нескольких генов, связываясь со специфическими последовательностями (сайтами связывания) в некодирующей части генома. О том, в каких именно местах транскрипционные факторы связываются с ДНК, мы узнаём, в основном, из данных экспериментов ChIP-Seq. Эксперимент ChIP-Seq дает нам прочтения (прочтенные, то есть отсеквенированные, нуклеотидные последовательности), попадающие в районы связывания интересующего нас белка. Имея такие прочтения и последовательность всего генома, мы можем узнать, где именно расположен сайт связывания. При этом чем больше покрытие некоторого участка генома в таком эксперименте (то есть число прочтений, приходящихся на этот участок), тем более вероятно, что с ним специфично связывается белок, который мы изучаем. Мы использовали данные более 7,5 тысяч ChIP-Seq-экспериментов для более чем 500 различных типов клеток, чтобы проанализировать влияние однонуклеотидных замен в сайтах связывания на, собственно, связывание транскрипционных факторов [1].
Каждый участок генома в человеческой клетке, как правило, представлен двумя аллелями: одна из аллелей наследуется от матери, вторая — от отца. Мы сосредоточили свое внимание на гетерозиготных участках, содержащих известные однонуклеотидные полиморфизмы (мутации, встречающиеся в человеческой популяции) (рис. 1).
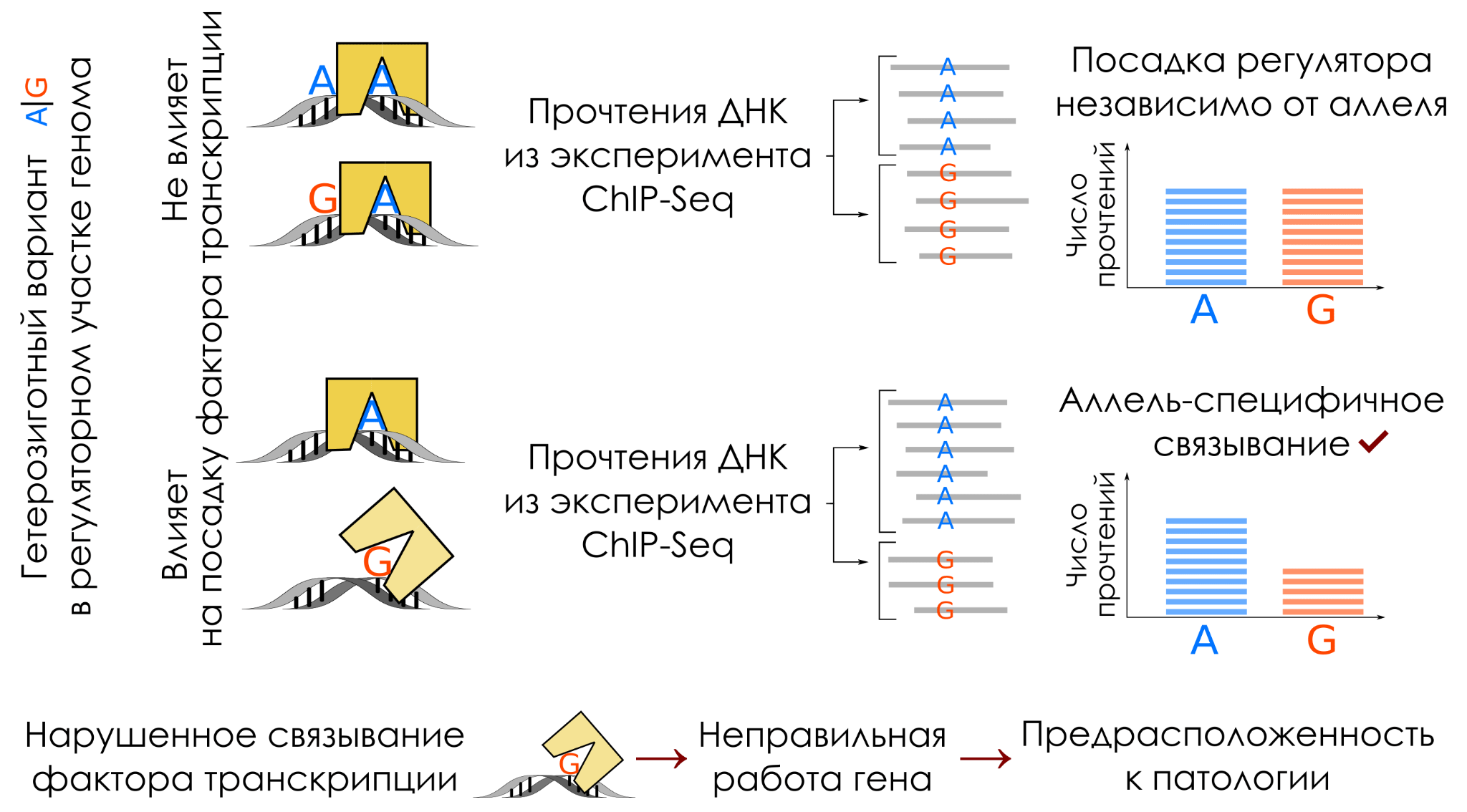
Рисунок 1. Сценарии, при которых гетерозиготный вариант в регуляторном участке генома либо не влияет (верхняя часть рисунка, слева направо), либо влияет (в центре рисунка, слева направо) на посадку фактора транскрипции. Внизу схематично показано, к каким последствиям может привести нарушение связывания транскрипционного фактора.
рисунок автора статьи и соавторов научной публикации
Казалось бы, исходя из соотношения ChIP-Seq-покрытий двух аллелей легко понять, с каким из вариантов белок связывается лучше. Например, если мы видим, что прочтений на обеих аллелях примерно поровну, то, значит, никакого аллель-специфичного связывания здесь нет. А что если ChIP-Seq-покрытие одной из аллелей в два раза больше, чем другой? Кажется, разница здесь значительная и предпочтение налицо. Однако не всё так однозначно. Основная проблема кроется в том, что на деле не все клетки диплоидны — число копий ДНК от мамы и/или папы в клетке не равно 1. Обрабатываемые данные из экспериментов ChIP-Seq были проведены на клеточных линиях, многие из которых относятся к опухолевым, а для них характерна анеуплоидия — недостаток или избыток копий целых хромосом, а также многочисленное копирование каких-то отдельных хромосомных участков. Таким образом, видимое предпочтение одной из аллелей транскрипционным фактором может объясняться тем, что данная аллель просто представлена большим числом копий (рис. 2).
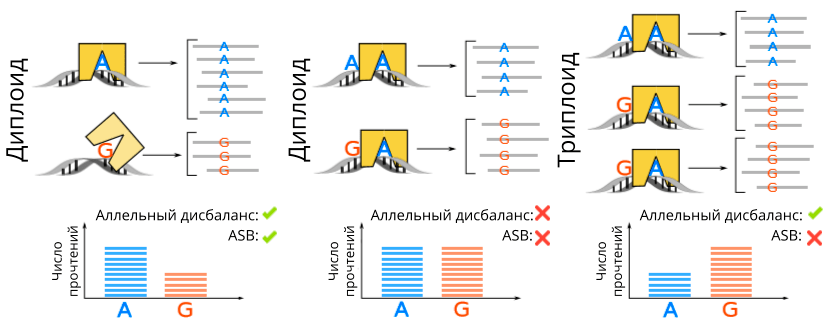
Рисунок 2. Аллельный дисбаланс, то есть неодинаковое число прочтений на разных аллелях, не всегда говорит об аллель-специфичном связывании (ASB, allele-specific binding). На правой картинке соотношение прочтений на разных аллелях 2:1, но это объясняется не предпочтениями транскрипционного фактора, а тем, что аллель G представлена в геноме в два раза большим числом копий, чем аллель A.
рисунок автора статьи и соавторов научной публикации
Но откуда узнать «фоновое» соотношение аллелей, чтобы сделать на него поправку? Мы придумали, как оценить его из тех же данных, не прибегая к каким-либо дополнительным источникам информации. Давайте разберем этот подход поподробнее: так как аллель-специфичное связывание — событие редкое, то для большинства гетерозиготных сайтов, находящихся по соседству от интересующего, соотношение покрытий ChIP-Seq будет просто отражать относительную представленность в геноме этих сайтов. Иными словами, неспецифичное связывание (в соседних сайтах) задаст некое фоновое распределение числа прочтений, а аллель-специфичное будет выглядеть как выброс. Мы построили алгоритм, разбивающий весь геном на сегменты с примерно постоянным соотношением копий двух аллелей и оценивающий это фоновое соотношение для каждого сегмента.
Вооружившись локальными оценками копийности и сделав на них поправки, получилось собрать самую большую на сегодняшний день базу данных событий аллель-специфичного связывания ADASTRA (Allelic Dosage-corrected Allele-Specific human Transcription factor binding sites). О чем могут рассказать полученные данные? Во-первых, они помогут в построении и проверке моделей для описания взаимодействия транскрипционного фактора и ДНК. Например, накопление новых данных позволяет уточнять мотивы транскрипционных факторов (этим музыкальным словом называются характерные паттерны в последовательности ДНК, которые транскрипционный фактор распознает). Кроме того, помимо самой последовательности, для связывания важен еще один фактор: открытость хроматина. ДНК в эукариотической клетке связана с гистонами и упакована сложным образом. Для транскрипции ДНК необходимо распаковать, то есть перевести хроматин из закрытой конформации в открытую. Определенные транскрипционные факторы способны инициировать открытие хроматина, после чего другие регуляторные белки приходят уже на подготовленную площадку и вступают в работу. Таким образом, если связывание первых, открывающих, факторов нарушится, то и последующие не придут, даже если последовательность для них будет «подходящая». Действительно, опубликованные ранее данные по аллель-специфичной доступности хроматина [2] хорошо согласуются с представленными в ADASTRA, и это отличный пример того, какие биологические закономерности можно наблюдать, комбинируя одни большие данные с другими. Подробнее об использовании данных по доступности хроматина в задаче предсказания связывания транскрипционных факторов можно прочитать в статье «Биомолекулы» «Мечту вызывали?» [3].
Данные по аллель-специфичному связыванию имеют также и практическое значение. Для изучения генетических детерминант различных полигенных признаков, в том числе заболеваний, сейчас активно используется метод полногеномного поиска ассоциаций (GWAS). Однако для того, чтобы среди ассоциированных с заболеванием вариантов выделить действительно приводящие к нему («причинные»), нужно постараться. Дело в том, что соседние геномные варианты «путешествуют» в популяции не независимо: как и гены, варианты наследуются сцепленно, если расположены достаточно близко друг к другу на хромосоме. Из-за этого, проводя полногеномный поиск ассоциаций, мы зачастую находим ассоциации с целыми островками в геноме, внутри которых один или несколько вариантов являются причинными, а все остальные — просто их случайные попутчики. На помощь приходит функциональная аннотация: при помощи дополнительных данных мы пытаемся понять, на что влияет конкретный вариант. Конечно, наиболее подозрительны варианты, приводящие к несинонимичным заменам в белках.
А что если в нашем списке подозреваемых только варианты из некодирующей области? Мы предположили, что изменение связывания фактора транскрипции может быть хорошим критерием функциональности варианта. Как выяснилось, такие замены действительно чаще ассоциированы с различными заболеваниями. При этом они часто находятся в локусах количественных признаков, то есть действительно связаны с изменением активности определенных генов. Для 30% регулируемых генов показана их клиническая значимость в ClinVar. Таким образом, влияние на посадку фактора транскрипции может стать весомым аргументом в пользу «причинности» варианта для развития патологии. Немаловажным является и тот факт, что помимо самого варианта нам известен еще и регуляторный белок, чье связывание из-за него нарушается, что позволяет строить предположения о молекулярном механизме возникновения заболевания. Кстати, в статье «Беспечный фактор транскрипции: что он скрывает от системы репарации» [4], опубликованной ранее на «Биомолекуле», можно прочесть о том, как изменение связывания транскрипционного фактора может мешать работе системы репарации.
Интересно, что наиболее сильные ассоциации с фенотипами показывают «переключающие» однонуклеотидные замены: их аллели предпочтительно связываются разными транскрипционными факторами. Мы предполагаем, что такие регуляторные варианты способны кардинально менять характер транскрипции соответствующих генов: в зависимости от варианта в геноме, руководство над их транскрипцией будет вверено разным белкам, каждый из которых может изменять свою активность в ответ на специфические сигналы.
Итак, ADASTRA содержит ценную биологическую информацию, которая может пригодиться как медицинским генетикам для определения функциональной значимости отдельных вариантов, так и вычислительным биологам для построения моделей связывания транскрипционных факторов с ДНК. История создания ADASTRA подтверждает: не нужно бояться шумных экспериментальных данных. Если данных много, то при помощи аккуратных алгоритмов все равно можно различить среди шума подлинные мотивы.
Автор выражает благодарность Ивану Кулаковскому, Марине Фридман, Дмитрию Пензару, Сергею Абрамову, Александру Бойцову, Арсению Зинкевичу и Надежде Потаповой за помощь в работе над текстом.
Литература
- Sergey Abramov, Alexandr Boytsov, Daria Bykova, Dmitry D. Penzar, Ivan Yevshin, et. al.. (2021). Landscape of allele-specific transcription factor binding in the human genome. Nat Commun. 12;
- Matthew T Maurano, Eric Haugen, Richard Sandstrom, Jeff Vierstra, Anthony Shafer, et. al.. (2015). Large-scale identification of sequence variants influencing human transcription factor occupancy in vivo. Nat Genet. 47, 1393-1401;
- Мечту вызывали?;
- Беспечный фактор транскрипции: что он скрывает от системы репарации.