
Сиквенс лишним не бывает или Зачем мы повторно секвенируем вирусы?
27 марта 2024
Сиквенс лишним не бывает или Зачем мы повторно секвенируем вирусы?
- 562
- 0
- 3
Барт Симпсон записывает результаты ресеквенирования геномов.
Рисунок в полном размере.
иллюстрация сделана с помощью генератора ranzey.com
-
Автор
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: ОРВИ срывает планы, мешает жить и иногда может протекать долго и тяжело. Возбудителями сезонных простудных заболеваний является целый ряд вирусов, но особый интерес у исследователей вызывают наиболее опасные вирусы, которые могут приводить к смерти: вирусы гриппа, SARS-СoV-2 и РСВ. Но почему всего для нескольких вирусов, вызывающих ОРВИ, в базах данных можно найти 17 миллионов последовательностей геномов? Зачем исследователям повторно определять последовательности геномов вирусов? О том, что повторное секвенирование не такое уж повторное и зачем нам столько данных о генетике вирусов, читайте в этой статье.
Конкурс «Био/Мол/Текст»-2023/2024
Эта работа опубликована в номинации «Своя работа» конкурса «Био/Мол/Текст»-2023/2024.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.
Партнер номинации — компания «Хеликон».

«Книжный» спонсор конкурса — «Альпина нон-фикшн».
С приходом дождливых дней осени мы начинаем больше болеть и обычно не сильно интересуемся природой инфекции, объясняя болезнь сквозняками и промокшей обувью. Да и прикладного смысла для больного в этой информации нет: эффективных лекарств от респираторных вирусных заболеваний пока не разработали [1]. Тем не менее, каждая «простуда» вызвана каким-то вирусом [2], и иногда эти вирусы могут представлять опасность для жизни. Поэтому важно знать, какие вирусы вообще циркулируют в городе и в стране и исследовать их генетическое разнообразие. В том числе этим и занимается НИИ гриппа имени А.А. Смородинцева в Санкт-Петербурге.
До наступления эпохи ковида повторно секвенировали (то есть определяли) последовательности геномов вирусов гриппа, респираторно-синцитиального вируса (РСВ) и некоторых других — в базах данных не набралось бы и полумиллиона геномов на все вирусы вместе взятые. Но с появлением новой коронавирусной инфекции ресеквенирование вышло на совершенно другой уровень. Ресеквенирование — это секвенирование геномов или генов, последовательность которых уже приблизительно известна. А геном — это совокупность наследственного материала, из которого по большей части и состоят вирусы (рис. 1). Сейчас в международных базах генетической информации вирусов более 16 миллионов последовательностей геномов вируса SARS-CoV-2, а новый коронавирус SARS-CoV-2 — самый генетически охарактеризованный организм на планете.
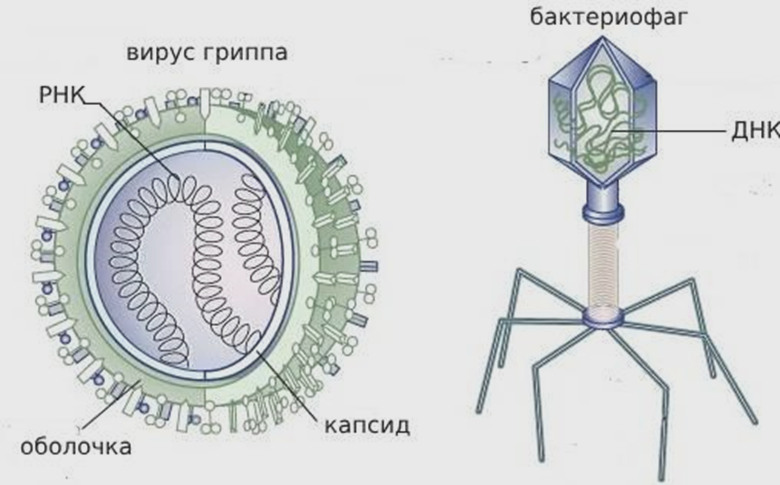
Рисунок 1. Схематичное изображение вируса гриппа и вируса бактерий — бактериофага.
Секвенировать первый геном неизвестного ранее вируса — самое важное, что можно сделать при появлении новой инфекции. Это дает возможность не только поставить диагноз пациенту с инфекционным заболеванием неясной природы, но и создать диагностические системы для детекции такого вируса, начать разработку вакцин и изучение нового патогена. Принципиально, что последовательность генома нового вируса будет быстро загружена в одну из международных баз, доступных ученым по всему миру. Это позволит озаботиться диагностическими методами и профилактикой новой инфекции или нового варианта ранее известного вируса в лабораториях по всему миру еще до того, как вирус окажется в этих странах.
Так и произошло с новой коронавирусной инфекцией — всего через пару дней после того, как стало ясно, что врачи столкнулись с неизвестной ранее инфекцией (все тесты на известные патогены были отрицательными), геном нового вируса был секвенирован. С публикацией последовательности SARS-CoV-2 было некоторое промедление, но через пару недель после первого заболевшего последовательность вируса стала общедоступной.
А ведь еще в конце прошлого века на схожую задачу по идентификации вируса гепатита С ушло более 10 лет [3]! В 1980-е гг. для открытия нового вируса ученым пришлось проводить сложную работу по поиску РНК вируса среди всей найденной в крови больных РНК. Как это было устроено: на базе всех РНК крови получали белки, их проверяли на взаимодействие с сыворотками пациентов, пока не обнаружили ту самую сыворотку, в которой произошло связывание антител с одним таким белком.
Сейчас такая задача может быть решена при помощи большого количества данных секвенирования и их биоинформатической обработки [4] . Благодаря базам данных можно быстро определить, к геномам каких организмов относятся прочтения (полученные в ходе секвенирования последовательности), оценить, сколько прочтений приходится на какой организм и обнаружить геномы новых видов, если какую-то часть данных секвенирования не удается соотнести с существующими геномами (рис. 2).
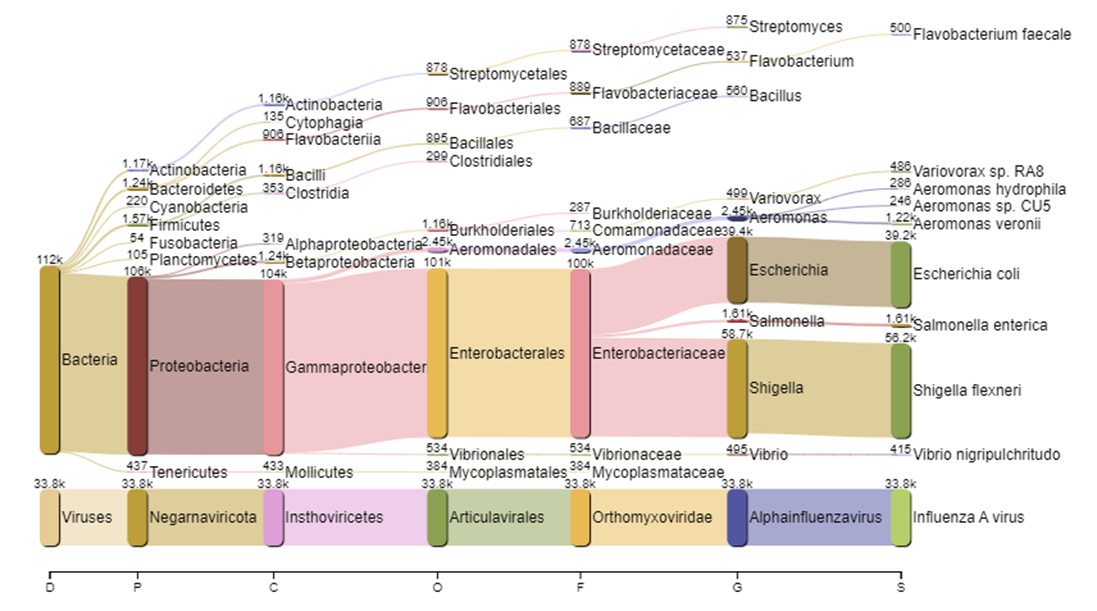
Рисунок 2. Пример обработки данных секвенирования одного образца с соотнесением полученных прочтений с геномами разных видов микроорганизмов. Классификация последовательностей — Kraken, визуализация результатов классификации — Pavian.
Но зачем же секвенировать такое огромное количество геномов одного вируса и все загружать в базы данных?
Вирусы постоянно и быстро мутируют [5], поэтому результаты секвенирования одного и того же вируса от разных пациентов будут немного отличаться. И даже в одном длительно болеющем пациенте вирус будет эволюционировать, меняя последовательность своей РНК [6]. Так что повторное секвенирование вирусов не такое уж и повторное.
Эволюционные изменения вирусов сказываются на эффективности работы диагностических тестов, созданных для их обнаружения. Когда перестает работать диагностический тест, можно посмотреть на свежие последовательности геномов вируса в базах данных и понять, в чем дело. А еще лучше — предположить потенциальную проблему с детекцией заранее, отслеживая появление новых вариантов (новых разновидностей вируса) и проверяя тесты in silico.
Важно, что ресеквенирование технологически значительно проще и дешевле, чем первое прочтение генома, поскольку уже есть образец последовательности, на который будут похожи результаты следующего секвенирования: новые варианты вирусов обычно отличаются лишь небольшим набором мутаций (кроме случаев рекомбинаций и реассортаций — ситуаций когда вирусы меняются целыми кусками геномов). В таком случае сборка генома de novo, как в первый раз, не требуется, процесс сборки проще, а данных секвенирования (прочтенных последовательностей) требуется меньше.
Имея много последовательностей вирусных геномов, можно выделить наименее изменчивые, консервативные участки генома и создать диагностический тест, детектирующий все существующие варианты вируса, а также его будущие потенциальные варианты (рис. 3). А если нам известны наименее изменчивые участки генома, значит, известны и наименее изменчивые участки белков, закодированных в этих генах. Поэтому имея много последовательностей вируса, полученных из образцов в разных странах мира на протяжении нескольких лет, можно разработать максимально эффективную вакцину: так же как и с диагностическим тестом, в вакцину лучше включать актуальные и наименее изменчивые участки белков или нуклеиновых кислот вируса.

Рисунок 3. Фрагмент «выравнивания» нуклеотидных последовательностей разных геномов вируса, иллюстрирующий сходства и различия геномов разных вариантов этого вируса. Визуализация множественного выравнивания в программе UGENE.
Кроме того, ресеквенирование позволяет находить новые варианты вирусов и исследовать их. В современной лаборатории можно получить (изолировать) вирус из образца и исследовать in vitro его особенности: вирулентность (насколько он вредит человеку), его тропность (сродство) к разным тканям, контагиозность (насколько он заразен) и прочее. А также, если от вируса существует вакцина, можно in vitro оценить, насколько хорошо вакцина работает против нового варианта. Конечно, чтобы обнаружить такого мутанта, придется повторно секвенировать много уже известных вариантов.
Сравнение пациентов, инфицированных разными вариантами вируса, дает возможность определять связь тяжести течения с вариантом вируса, что может позволить врачам лучше прогнозировать течение заболевания. Часто, если такая взаимосвязь наблюдается, (например, как с высокопатогенным гриппом [7]) для детекции дополнительно разрабатывают и применяют более дешевый и быстрый метод — ПЦР (полимеразную цепную реакцию). Но и метод ресеквенирования вирусов используется для диагностики, как, например, в случае c ВИЧ и устойчивостью к терапии некоторых вариантов вируса, которая обусловлена определенным набором мутаций [8].
Помимо этого, ресеквенирование вирусов из большого количества образцов позволяет проводить анализ их родства для расследования вспышек инфекции на всех уровнях: от коллектива до межконтинентального распространения во время пандемии (рис. 4). По результатам таких исследований становится ясно, как быстро и каким образом распространяется инфекция среди населения — такая информация полезна для определения источников инфекции и выбора мер контроля ее распространения.
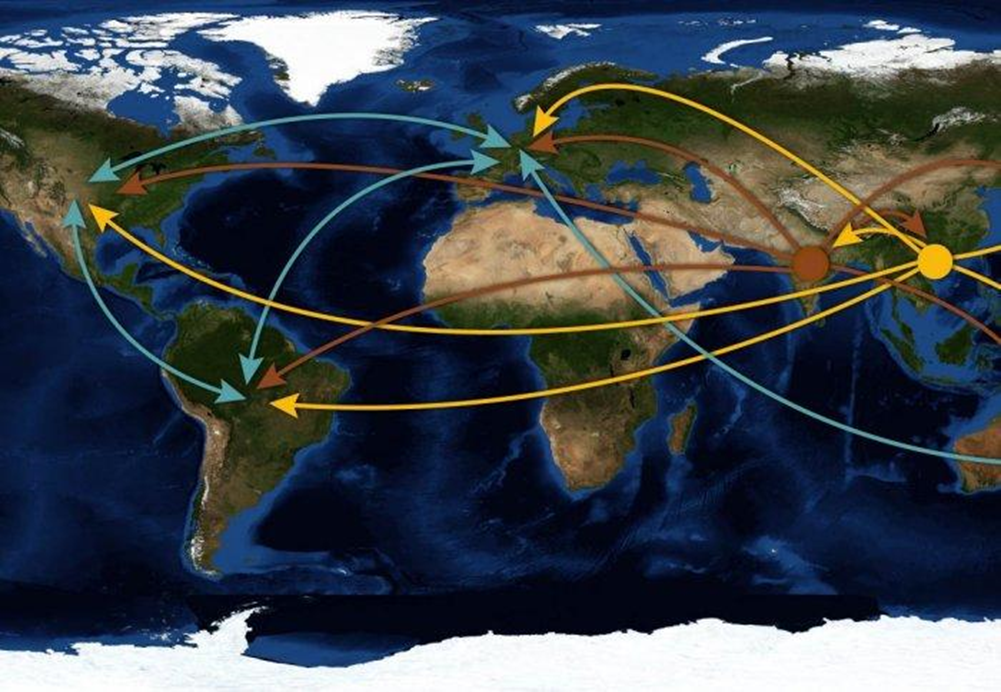
Рисунок 4. Схема глобального движения сезонных вирусов гриппа.
Colin Russell / NASA Visible Earth
Количество нередко переходит в качество — при накоплении большого количества последовательностей вируса исследователи неизбежно обращают внимание на закономерности, которые могут и не иметь отношения к конкретной инфекции, но в дальнейшем оказываются в основе новых технологий.
Например, благодаря большому количеству последовательностей геномов бактерий в них была обнаружена закономерность в виде повторов фрагментов ДНК. В ходе изучения природы этих повторов был открыт механизм противовирусного иммунитета у бактерий, а на базе белков, задействованных в этом иммунитете, получены новые генно-инженерные и диагностические инструменты CRISPR-Cas (рис. 5). Впрочем, все инструменты для генной инженерии ученые позаимствовали у вирусов или бактерий, секвенируя и аннотируя их геномы — определяя не только саму последовательность, но и закодированные в ней гены и регуляторные элементы.
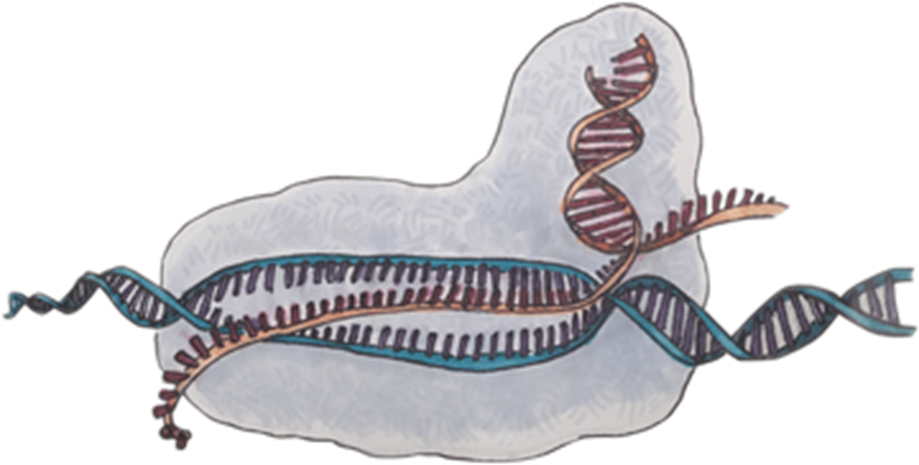
Рисунок 5. Схематическое изображение комплекса CRISPR-Cas9.
«Биомолекула»: CRISPR
На самом деле, огромное количество проведенных ресеквенирований SARS-CoV-2 связано не только с тем, что это полезно и интересно, но и с некоторыми другими факторами. Во-первых, секвенирование за последнее десятилетие стало дешевле и доступнее: на рынке представлено несколько платформ для секвенирования (Illumina, Oxford Nanopore, Ion Torrent и др.), и некоторые даже не требуют дорогостоящего оборудования. Например, в пандемию гриппа 2009 года секвенаторов на планете было меньше, секвенирование стоило дороже и секвенированием гриппа занимались значительно меньше. Во-вторых, геном SARS-CoV-2 относительно невелик — меньше человеческого в 100 000 раз. Ну а в-третьих, повлиял и масштаб пандемии: связанные с появлением новой инфекции ограничения проникли во все области жизни и коснулись всех жителей планеты, поэтому государства готовы были финансировать такие исследования.
Но важно не только секвенировать, но и делиться полученными данными. Смысл ресеквенирования геномов будет в значительной мере утерян, если полученные последовательности не будут депонированы для всеобщего пользования. Одним из крупнейших международных хранилищ данных последовательностей SARS-CoV-2, гриппа и других вирусов является GISAID. Причем работать с этими данными могут не только вирусологи и биоинформатики по всему миру, но и любой желающий — используя инструменты ресурсов NextStrain и NextClade, которые позволяют фильтровать и визуализировать данные GISAID (рис. 6). Еще один важный репозиторий генетической информации о вирусах — различные базы данных NCBI.
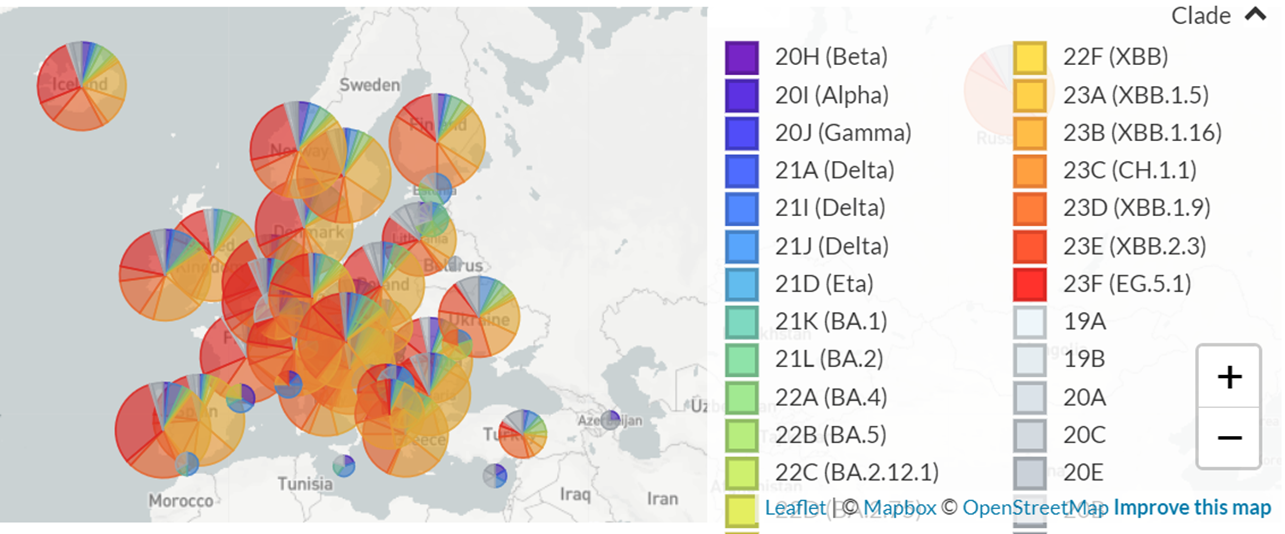
Рисунок 6. Пример визуализации данных GISAID с помощью NextStrain — инфографика позволяет оценить распространение различных вариантов вирусов по странам.
Мы в лаборатории молекулярной вирусологии НИИ гриппа секвенируем и ресеквенируем вирусы как в рамках федеральных программ, так и для международных проектов. Мы являемся Национальным центром ВОЗ по гриппу и референс-центром по SARS-CoV-2 и изучаем эти и многие другие вирусы и генетическое разнообразие каждого из них. С учетом наших данных эксперты ВОЗ принимают решения по составу противогриппозных вакцин на предстоящий сезон . А совместно с коллегами из Сколково и Института проблем передачи информации (команда Егора Базыкина) мы проводим филогенетический анализ вирусов [9], [10], в России и мире и исследуем эволюцию вирусов SARS-CoV-2 у пациентов c хроническим ковидом [6]. Эта же группа ученых реализовала визуализацию частот вариантов SARS-CoV-2, циркулирующих в РФ, на основе, в том числе, наших данных.
Также мы разрабатываем тест-системы для детекции вирусов (вызывающих ОРВИ и не только), которые используются как в исследованиях, так и коммерческими производителями. А проводить клинические исследования таких тестов мы можем благодаря внушительному биобанку клинических материалов, содержащих генетически охарактеризованные вирусы.
Если подытожить, то вирусы, вызывающие грипп, ковид и другие респираторные инфекции — это преимущественно нуклеиновые кислоты, последовательности которых постоянно меняются. А определение этих последовательностей — основа для борьбы с ними. Что бы мы ни задумали сделать — тест, вакцину или лекарство — нам в первую очередь надо знать, какие бывают генетические варианты того или иного вируса. Хочется верить, что с каждой новой прочитанной последовательностью мы становимся на шаг ближе к миру без вирусов. Ну или хотя бы к тому, чтобы реже болеть.
Литература
- Diane E. Pappas. (2018). The Common Cold. Principles and Practice of Pediatric Infectious Diseases. 199-202.e1;
- J. Owen Hendley. (1998). Epidemiology, pathogenesis, and treatment of the common cold. Seminars in Pediatric Infectious Diseases. 9, 50-55;
- Michael Houghton. (2009). The long and winding road leading to the identification of the hepatitis C virus. Journal of Hepatology. 51, 939-948;
- Simon H. Ye, Katherine J. Siddle, Daniel J. Park, Pardis C. Sabeti. (2019). Benchmarking Metagenomics Tools for Taxonomic Classification. Cell. 178, 779-794;
- Rafael Sanjuán, Miguel R. Nebot, Nicola Chirico, Louis M. Mansky, Robert Belshaw. (2010). Viral Mutation Rates. JVI. 84, 9733-9748;
- Oksana V. Stanevich, Evgeniia I. Alekseeva, Maria Sergeeva, Artem V. Fadeev, Kseniya S. Komissarova, et. al.. (2023). SARS-CoV-2 escape from cytotoxic T cells during long-term COVID-19. Nat Commun. 14;
- Timothy M. Uyeki. (2009). Human Infection with Highly Pathogenic Avian Influenza A (H5N1) Virus: Review of Clinical Issues. CLIN INFECT DIS. 49, 279-290;
- Pleuni S. Pennings. (2013). HIV drug resistance: problems and perspectives. Infectious Disease Reports. 5, e5;
- Galya V. Klink, Ksenia R. Safina, Sofya K. Garushyants, Mikhail Moldovan, Elena Nabieva, et. al.. (2022). Spread of endemic SARS-CoV-2 lineages in Russia before April 2021. PLoS ONE. 17, e0270717;
- Galya V. Klink, Daria Danilenko, Andrey B. Komissarov, Nikita Yolshin, Olga Shneider, et. al.. (2023). An Early SARS-CoV-2 Omicron Outbreak in a Dormitory in Saint Petersburg, Russia. Viruses. 15, 1415.






