
Альтернативная «сварка» мРНК: как из одного гена получается несколько белков
12 февраля 2022
Альтернативная «сварка» мРНК: как из одного гена получается несколько белков
- 3571
- 0
- 12
«Сварка» мРНК
Рисунок в полном размере.
коллаж автора с использованием изображений из открытого доступа
-
Авторы
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: Любому, кто не прогуливал уроки биологии в школе, знакома центральная догма молекулярной биологии, сформулированная еще на этапе открытия структуры ДНК Фрэнсисом Криком. Это обобщающее правило гласит: из ДНК посредством транскрипции получается РНК, а из РНК — белок благодаря процессу трансляции.
На самом деле, на практике все не так просто: к примеру, всего 5% генома человека составляют последовательности, кодирующие белки. Более того, они расположены не друг за другом, а разделяются некодирующими фрагментами. Что еще более удивительно: в клетках эукариот из одного гена может образовываться несколько различных по свойствам белков. Как же так получается? Обо всем по порядку.
За возможность получения нескольких различных белков (изоформ) отвечает сложный и до конца не изученный процесс — альтернативный сплайсинг, напоминающий работу конструкторского бюро. Этому процессу подвержены около 94% всех генов человека, и любая ошибка может очень сильно повлиять на весь организм. К примеру, была доказана связь альтернативного сплайсинга генов, экспрессирующихся в нервной системе, с болезнью Альцгеймера и расстройствами аутистического спектра. В этой статье мы отметим основные достижения молекулярной биологии в исследовании его механизмов и влияния на живые организмы (прежде всего, человека).
Конкурс «Био/Мол/Текст»-2021/2022
Эта работа опубликована в номинации «Свободная тема» конкурса «Био/Мол/Текст»-2021/2022.

Партнер номинации — компания SkyGen: передовой дистрибьютор продукции для life science на российском рынке.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

Генеральный партнер конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Конструкторское бюро РНК
Однако прежде чем говорить об альтернативном сплайсинге, стоит вспомнить (или узнать), что он из себя, собственно, представляет.Сразу после окончания транскрипции молекула РНК еще не готова выполнять свою основную функцию — быть матрицей для белкового синтеза, т.е. трансляции. Она должна претерпеть превращения, называемые постранскрипционным процессингом. Частью этого процесса является вышеупомянутый сплайсинг, который представляет собой механизм точного вырезания некодирующих участков (интронов) и сшивания кодирующих участков (экзонов) первичного транскрипта или пре-мРНК, который проводится главным «сварщиком» — комплексом РНК (точнее, малых ядерных рибонуклеопротеидов — snPRP, small nuclear PNP) и белковых молекул, который называется сплайсосомой [1].
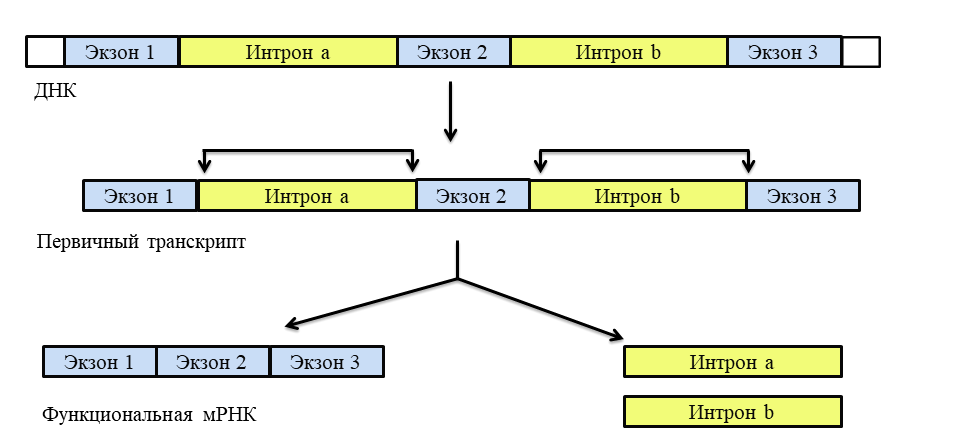
Рисунок 1. Сплайсинг первичного транскрипта у эукариот. Угловыми стрелками указаны места соединения экзонов 1, 2 и 3 после удаления интронов a и b.
[1], с модификациями
Сплайсинг характерен для нуклеиновых кислот и белков; нас в большей степени интересует первый процесс. ДНК эукариот, в отличие от прокариот, не представляет собой непрерывный сценарий белкового синтеза. Она больше напоминает журнал или газету, содержащую «полезные статьи» — экзоны — кодирующие участки — и так называемые «рекламные вставки» — интроны, участки, которые ничего не кодируют.
Казалось бы, для чего они тогда нужны в клетке? Неужели клетки прокариот «чище» и эффективнее? Ответ — нет! Благодаря наличию интронов, которые вырезаются из транскрипта, образуются фрагменты, которые могут сшиваться в разном порядке, образуя разные мРНК. Более того, интроны могут оставаться в транскрипте, а экзоны — вырезаться [2].
Помимо всего прочего, интроны на своих краях содержат последовательности длиной в два нуклеотида, которые сигнализируют о начале и об окончании сплайсинга, точнее, о вырезании конкретного интрона. Первые два нуклеотида интрона, чаще всего это гуанин и урацил — GU, — это так называемый донорный или 5’-cайт. Донорным его назвали, потому что на нем происходит сборка сплайсосомы: с 5’-сайтом связываются snRNP и белковые факторы сплайсинга. Акцепторный, или 3’-сайт, — это два нуклеотида, аденин и гуанин (AG), маркирующие конец интрона. Экзоны, находящиеся до донорного и после акцепторного сайтов, сшиваются, а интрон высвобождается и разрушается [1].
Мы можем представить пре-мРНК как набор деталей, из которых составляются разные конструкции. Какие-то детали мы задействуем, какие-то — нет, и, «сваривая» нужные фрагменты, мы получим некоторое количество различных структур — вариаций мРНК — и, как следствие, белков всего лишь из одного гена. В этом и есть суть альтернативного сплайсинга.
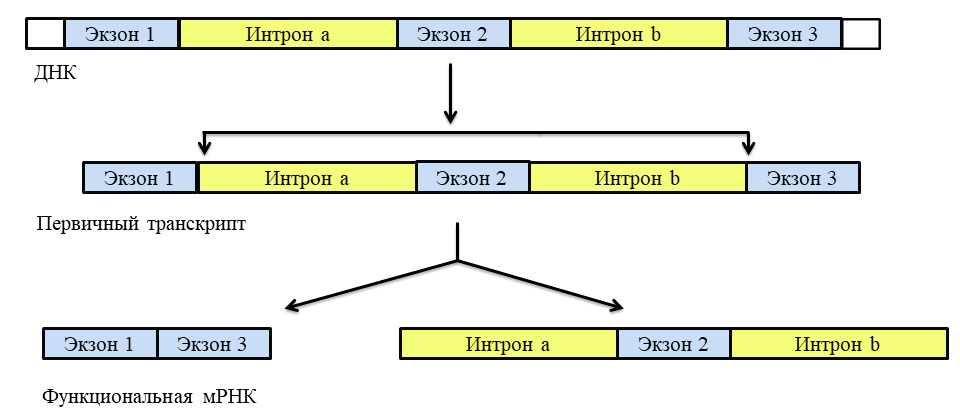
Рисунок 2. Альтернативный сплайсинг первичного транскрипта у эукариот. Стрелками указаны места соединения экзонов после удаления интронов. Экзон 2, фланкируемый интронами 1 и 2, вырезается из первичного транскрипта, а экзоны 1 и 3 соединяются с образованием функциональной мРНК.
[1], с модификациями
Как люди обнаружили «сварку» мРНК?
Этот интереснейший механизм обработки генетической информации был обнаружен, можно сказать, случайно: никто не предполагал о его существовании. При изучении экспрессии, т.е. считывания генов, эукариот в 1970-е годы выяснилось, что гены не просто «компилируются» подряд, как строчки программного кода. Внутри клеток происходят сложные превращения на уровне синтеза мРНК, от которых зависит, какой ген и каким именно образом будет переведен на язык всего остального организма — язык белков.
Такое представление сформировалось на основе полученных данных о нахождении мРНК, кодирующих особые белки, только в тех клетках, которыми эти белки производятся. К примеру, остатки мРНК, кодирующей глобины (белки-предшественники гемоглобина, миоглобина и т.д.) были найдены только в ретикулоцитах (клетки-предшественники эритроцитов, составляют примерно 1% эритроцитов в крови) [3].
Было выдвинуто две гипотезы о механизмах регуляции экспрессии генов: согласно первой, различные участки генома клетки «переписываются» в мРНК в случаях, когда клетка нуждается в белках, кодируемых ими. Эти мРНК — либо прямые продукты транскрипции, либо получены из неких схожих по размерам предшественников. Это предположение основано на экспериментах с клетками прокариот, но, как мы знаем, геномы большинства прокариот считываются непрерывно и не могут помочь изучении геномов эукариот.
Вторая гипотеза гласит, что мРНК получаются из очень длинных молекул-предшественников, которые проходят через множество сложных реакций, включающих их расщепление. Это предположение гораздо сильнее похоже на современные представления о сплайсинге пре-мРНК.
В 1977 году группа ученых из университета Рокфеллера представила на симпозиуме в лаборатории Колд Спринг Харбор результаты своей работы, суть которой заключалась в исследовании молекул мРНК культивированных клеток, зараженных аденовирусом 2. На поздних стадиях заражения было замечено, что их цитоплазма содержит множество новых РНК, которые, словно детали одного пазла, при сборке в одно целое, отражают практически всю информацию о геноме вируса, а в ядре была найдена очень длинная цепочка пре-мРНК вируса, состоящая из примерно 20 тысяч пар нуклеотидов.
С помощью меченых нуклеотидов [4] ученые смогли выяснить, что «большой» первичный транскрипт разделяется на части и «сшивается» несколькими способами, образуя цепочки мРНК, кодирующие различные вирусные белки.
Этот доклад вошел в выпуск журнала Nature № 268 под заголовком Adenovirus amazes at Cold Spring Harbor («Аденовирус поражает в Колд Спринг Харбор»). Действительно, все присутствовавшие были поражены результатами, ведь выяснилось то, о чем никто не предполагал: мРНК, синтезированные на основе ДНК аденовируса, представляют собой молекулы в технике patch-work : они состоят из фрагментов, комплементарных нескольким несмежным фрагментам вирусного генома [5].
Техника шитья из лоскутков ткани.
Несколькими годами позже, в 1981 году, был описан первый пример альтернативного сплайсинга гена самого организма, а не вирусной ДНК [6]. Этим геном оказался ген кальцитонина крысы. Как нетрудно догадаться из названия, он несет информацию о кальцитонине — гормоне щитовидной железы, регулирующем кальций-фосфорный обмен.
Организм может экспрессировать его несколькими способами: он кодирует не только кальцитонин, но и белок, называемый CGRP (calcitonin gene-related peptide), образующийся в клетках нервной системы [7]. Что интересно, с кальций-фосфорным обменом его функции не связаны: был замечен его вклад в развитие мигрени, поэтому уже производятся препараты на основе моноклональных антител к этому белку [8], [9].
Но мы говорим о другом: как можно видеть на рисунке 3, у мРНК кальцитонина и CGRP общий первичный транскрипт. Однако далее жизненный путь этого транскрипта идет по двум разным сюжетным линиям: клетки щитовидной железы выбирают «ветку» с кальцитонин-кодирующим экзоном, а в клетках мозга выбор падает на CGRP-кодирующий.
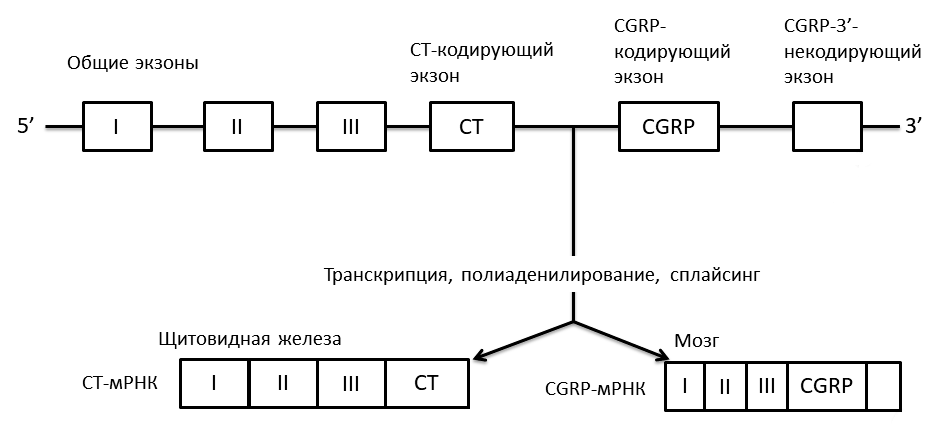
Рисунок 3. Тканеспецифический альтернативный сплайсинг в экспрессии гена кальцитонина крысы.
[7], с модификациями
С тех пор было доказано, что альтернативный сплайсинг распространен среди всех эукариот, благодаря чему вся совокупность белков в наших организмах столь обширна и разнообразна!
Есть ли у сплайсосомы инструкция по сборке?
Безусловно, было бы любопытно разобрать «по косточкам» весь процесс альтернативного сплайсинга. Но, увы, полную картину происходящих с мРНК превращений после транскрипции мы пока не имеем. Если инструкция и есть, то она крайне изменчива и зависит от условий жизни организма. Однако даже общий механизм альтернативного сплайсинга малоизучен: до сих пор остается неясным до конца, как контролируется этот процесс и от чего зависит порядок соединения фрагментов. На данный момент известно 5 основных механизмов альтернативного сплайсинга [10]:
- Пропуск экзона. Кодирующая последовательность вырезается из транскрипта и «погибает».
- Взаимоисключающие экзоны. Только один из двух экзонов останется в каком-либо конечном варианте мРНК (как в вышеописанном случае сплайсинга гена кальцитонина). Необязательно «сильнейший»: как мы уже убедились, останется тот экзон, который является «деталькой в пазле» мРНК белка, необходимого органу или клетке.
- Альтернативный донорный сайт. В этом случае интрон содержит несколько 5’-сайтов, при взаимодействии с каждым из которых меняется длина сохраняемого фрагмента.
- Альтернативный акцепторный сайт. Этот случай противоположен предыдущему: в интроне имеется несколько акцепторных сайтов; также меняется длина сохраненного участка.
- Удержание интрона. Некодирующий фрагмент сохраняется в транскрипте. Чаще всего такие мРНК нежизнеспособны: интрон, находящийся в кодирующей последовательности, может кодировать стоп-кодон или сдвинуть рамки считывания (последовательности, потенциально кодирующие аминокислоты). Это может привести к потере функциональности белка, поэтому данный механизм крайне редко встречается у млекопитающих [2].
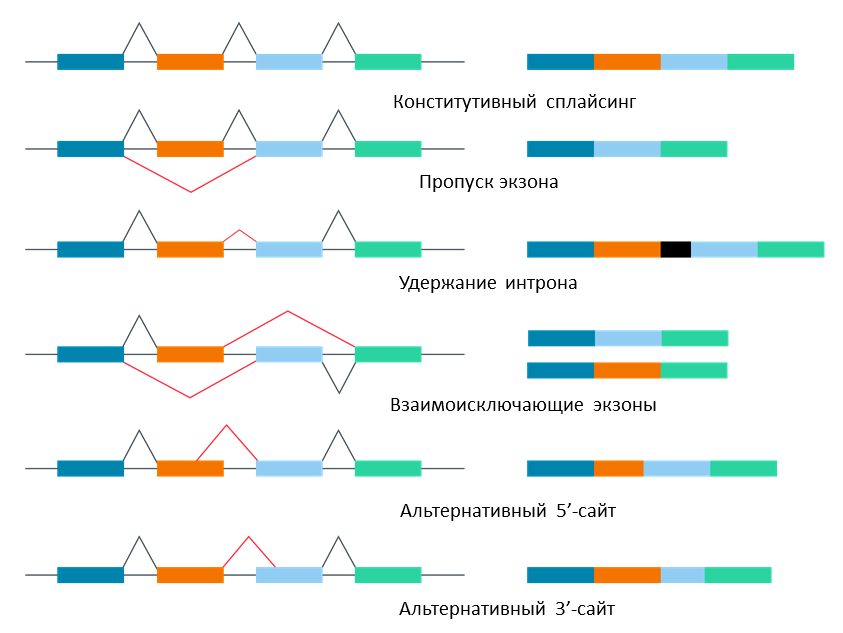
Рисунок 4. Иллюстрация различных сценариев сплайсинга.
Конститутивный сплайсинг — сплайсинг, проходящий по «классическому» (неальтернативному) механизму.
Oxford Nanopore, с модификациями
Самый распространенный механизм в геноме человека — пропуск экзона. Мы можем наблюдать его в действии при альтернативном сплайсинге мРНК, кодирующей белок-антиген CD45.
CD45 (трансмембранная тирозиновая фосфатаза) — сигнальная молекула, участвующая в регуляции митотического цикла, онкогенеза, дифференцировании клеток — и не только. Однако в первую очередь — это важный маркер, по изоформам которого на поверхности Т-лимфоцитов можно судить о течении инфекционного заболевания [11].
У этого белка имеется 8 изоформ, образующихся посредством, опять-таки, разнообразных результатов считывания гена Pprc (Protein Tyrosine-Phosphatase, Receptor Type) [11]. Как можно видеть на рисунке 5, экзоны 4, 5 и 6 «выключаются» в некоторых изоформах.
Экспрессия этого гена напрямую зависит от различных внешних воздействий, главным образом, от попадания в организм какого-либо антигена, например, вируса. При введении в организм антигенов, экзоны 4 и 6 в рабочую мРНК практически не включаются.
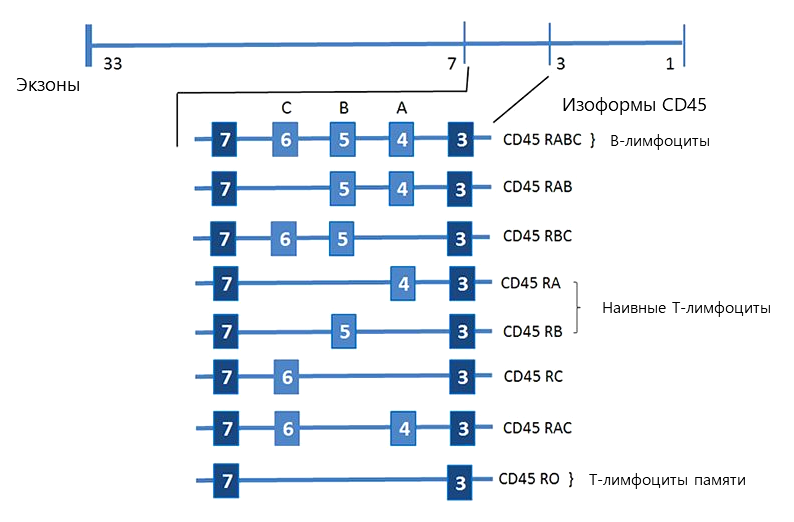
Рисунок 5. Экзон-интрон структура гена CD45.
Bio-Rad-Antibodies, с модификациями
Другие механизмы встречаются несколько реже. Например, использование разных донорных сайтов характерно для альтернативного сплайсинга пре-мРНК обезьяньего вируса SV40, которым в 1960-е годы заразились миллионы людей из-за его присутствия в вакцине против полиомиелита.
Известно, что при использовании разных 5’-сайтов могут образовываться две мРНК, кодирующих два вирусных антигена: большой Т и малый t. Оба эти белка вызывают изменения в зараженных клетках и способствуют онкогенезу, но в разной степени и с помощью разных механизмов. К примеру, большой Т-антиген способен связываться с ДНК и с некоторыми факторами транскрипции, нарушая нормальное считывание генов зараженного организма. В свою очередь, малый t-антиген способен связываться с ферментом фосфатазой PP2A, катализирующим отщепление фосфорной кислоты, что приводит к нарушению фосфорного обмена и, как следствие, к нарушению метаболизма и расхода/накопления энергии на биохимические реакции [12].
Доподлинно неизвестно, от чего зависит путь сплайсинга, который выберет клетка, и можно ли предугадать, какой будет рабочая молекула мРНК. Однако известны имена некоторых помощников сплайсосомы, работающих не покладая атомов на благо корректной «сшивке» главного носителя информации о белках нашего организма.
Есть ли у сплайсосомы помощники?
Сплайсосома выполняет очень сложную работу по сшиванию необходимых кусочков мРНК. Но она бы не справилась с такой работой в одиночестве: ей помогают не только другие молекулы-факторы сплайсинга, но и ДНК. Таким образом, на ход сплайсинга могут влиять некоторые последовательности ДНК, так называемые цис-действующие элементы: они помогают синтезу РНК еще на этапе транскрипции. Среди цис-элементов выделяют промоторы — стартовые площадки транскрипции, энхансеры — последовательности-стимуляторы — и сайленсеры — глушители, которые ускоряют или, соответственно, замедляют синтез пре-мРНК в соответствующих участках ДНК.
Другие, чаще всего белковые молекулы, участвующие в сплайсинге, называются транс-действующими факторами. Среди них выделяют множество семейств, некоторые из которых мы рассмотрим далее.
Исследования показали, что контроль альтернативного сплайсинга осуществляется с помощью регуляторных белков (РНК-связывающих белков, или RBP, RNA-binding proteins), связывающихся с экзонами, которые либо вырезаются, либо остаются в конечной мРНК. Эти белки принято разделять на два семейства: способствующие пропуску экзонов (рибонуклеопротеины, или hnRNP, heterogeneous nuclear ribonucleoproteins) и их включению (серин-аргинин-богатые белки, или SR). Предполагается, что RS-домены (участки, богатые чередующимися остатками аргинина и серина) SR-белков работают как детекторы: при связывании с экзонами в пре-мРНК, их RS-домены взаимодействуют с RS-доменами специфичного белка сплайсосомы для содействия определению экзона [13].
Помимо этих белков, были выделены дополнительные белки-помощники: например, полипиримидин-связывающие белки (PTB) и специфические РНК-связывающие факторы Nova. Эти белки регулируют сплайсинг в зависимости от нахождения экзона: например, они подавляют включение некоторого экзона при связывании с ним, но при связывании с интроном ниже от этого экзона, они, наоборот, активируют его включение [14].
Рассмотрим совместное действие SR-белков и белков Nova:
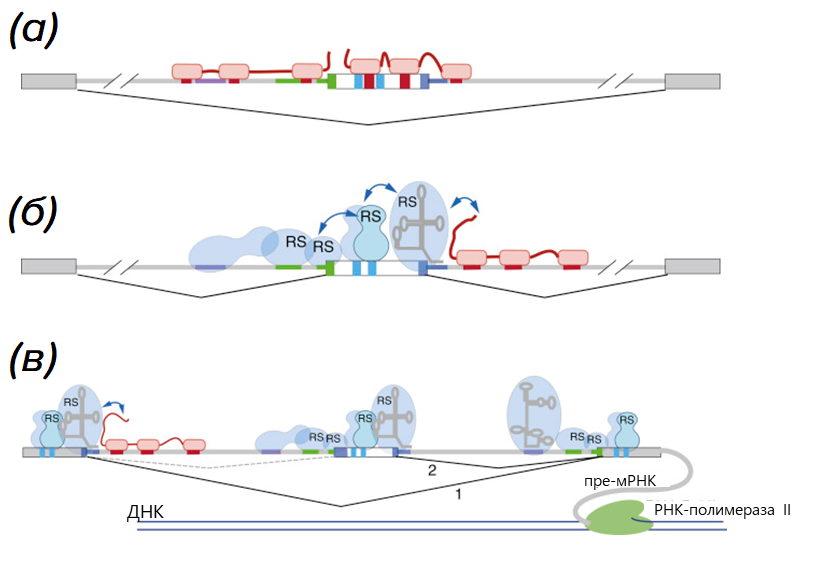
Рисунок 6. Механизмы регулирования альтернативного сплайсинга.
(а) — белки Nova (отмечены красными линиями), «вклинившиеся» между сайтами сплайсинга и усиливающими белками, привлекают RBP-репрессоры, которые могут конкурировать за связывание с пре-мРНК с другими факторами сплайсинга.
(б) — если белки Nova расположены ниже экзона, то многие RBP могут связываться, скорее всего, со сплайсосомой для поддержки определения экзона.
(в) — белки Nova могут косвенно регулировать альтернативный сплайсинг путем усиления включения некоторого экзона, что способствует пропуску его соседа спереди (путь 1). Также существует другой путь включения экзона, путь 2: первым делом удаляется интрон, находящийся перед тем самым соседом, удаляющимся в первом пути. В таком случае белки Nova, вероятно, способствуют пропуску экзона, влияя на конкуренцию между путями 1 и 2.
[14], с модификациями
Далее обсудим некоторые детали работы сплайсосомы и то, что происходит при нарушении ее работы.
Как «сварщик» понимает, где нужно начинать работу?
У многих может возникнуть вопрос: как же из множества схожих последовательностей клетка может понять, куда именно «пристроить» сплайсосому для правильной работы? Ведь донорные сайты состоят всего лишь из двух нуклеотидов, по всей пре-мРНК может быть разбросано очень много участков «гуанин—урацил».
Все дело в расположении сайтов сплайсинга: так называемые «скрытые» (либо «альтернативные») сайты можно отличить от канонических отчасти потому, что их расположение несовместимо с действиями, нужными для определения сайта: чаще всего они расположены внутри экзонов и интронов. Более того, они часто окружены интронными или экзонными сайленсерами в пре-мРНК, подавляющими их распознавание сплайсосомой [15–17]. Альтернативные сайты используются только в том случае, если канонические сайты сплайсинга повреждены, и это приводит к укорочению последовательности, в которой был «скрыт» этот сайт.
Что делать, если не получается с первого раза?
Иногда случается так, что сплайсинг не проходит «идеально» с первого раза, и приходится его повторять. Это явление называется рекурсивный сплайсинг (ReS) — процесс, посредством которого интроны удаляются в несколько этапов [18]. Он чаще всего присутствует в длинных интронах генов человека и способствует повышенной точности сплайсинга [19–21]. Этот механизм необходим для пропуска скрытых экзонов, обнаруженных в сайтах рекурсивного сплайсинга (далее ReS-экзоны) [21], [22]. На самом деле, ReS-экзоны — это не экзоны, а сохранившиеся при первом этапе сплайсинга участки интронов.
Схема механизма, лежащего в основе рекурсивного сплайсинга, и его регулирование различными белковыми факторами представлена на рисунке 6.
Сначала вырезается интрон, в котором «спрятался» ReS-экзон (Интрон 1). После этого восстанавливается 5’-сайт ReS-экзона, после чего с экзоном, идущим перед ReS-экзоном, связываются белковые факторы сплайсинга. Это приводит к тому, что сплайсинг может пойти по двум взаимоисключающим путям. Если сплайсосома собирается на восстановленном 5’-сайте, то это может привести к удалению ReS-экзона (как показано стрелкой 1). Однако случается так, что белковые комплексы подавляют распознавание 5’-сайта ReS-экзона, способствуя его включению (как показано стрелкой 2).
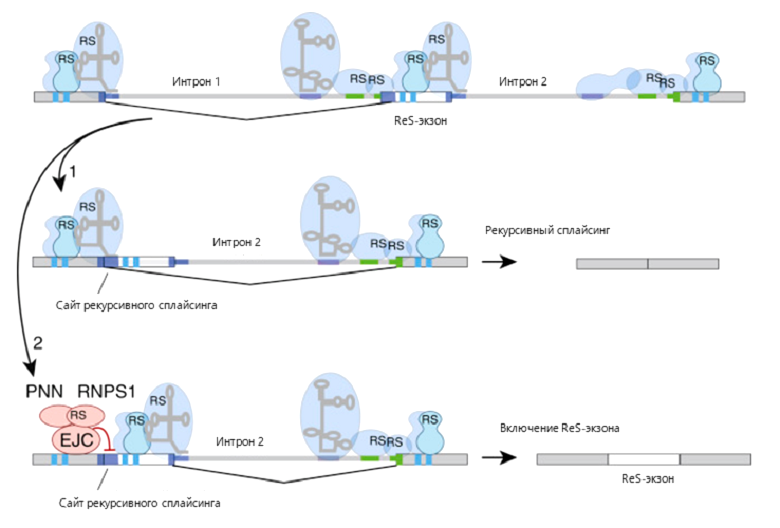
Рисунок 7. Рекурсивный сплайсинг и его регулирование.
ReS-экзоны содержат стоп-кодоны, и их включение вызывает нонсенс-опосредованный распад (NMD) — удаление мРНК [23]. Дело в том, что ReS-экзоны почти всегда удаляются из нормальных пре-мРНК, но было обнаружено, что они сохраняются при сплайсинге дефектных транскриптов, например, когда вышестоящий экзон был «переписан» с аберрантной (т.е. отличающейся от нормы) последовательности ДНК или когда произошла ошибка при сплайсинге [24]. NMD запускается только в том случае, когда процесс шел неправильно с самого начала либо когда неисправность подкралась незаметно для бдительной сплайсосомы.
Таким образом, рекурсивный сплайсинг нужен для контроля качества транскриптов, чтобы предотвратить образование аберрантных белков [21].
Ошибки на «производстве»: к чему приводят нарушения сплайсинга
Как известно, чем сложнее система, тем проще она выводится из строя. Выше мы смогли убедиться в сложности процессов регуляции альтернативного сплайсинга, поэтому неудивительно, что «неполадки» в этом процессе могут произойти в любой момент.
Нарушения сплайсинга связаны с генетическими заболеваниями человека, включая расстройства мозга. Например, семейная дизавтономия вызывается мутацией 5’-сайта сплайсинга гена IKBKAP [25]. Эта мутация снижает экспрессию IKBKAP из-за альтернативного сплайсинга, ведущего к нонсенс-опосредованному распаду, что приводит к ухудшению развития нейронов вегетативной нервной системы. Суть этой мутации в том, что из-за нее ухудшается связывание донорных сайтов со сплайсосомой, что приводит к пропуску экзона 20, что, в свою очередь, вызывает сдвиг рамки считывания и генерацию стоп-кодона в экзоне 21 мРНК IKBKAP, что, как мы узнали в прошлом разделе, как раз и приводит к NMD [26]. Снижение экспрессии происходит из-за того, что мРНК, «переписанные» с этого гена, уничтожаются и не вступают на путь трансляции.
Благодаря исследованиям GWAS, обнаружена связь аномалий в гене фактора сплайсинга RBFOX1 с расстройствами аутистического спектра, болезнью Альцгеймера и агрессивным поведением [27–29]. Аллель С этого гена связали с расстройством поведения, которое проявляется при отсутствии материнского тепла. С болезнью Альцгеймера этот ген связывает то, что нарушения его экспрессии приводят к потере серого вещества в височной доле мозга у пожилых людей с когнитивными нарушениями [30].

Рисунок 8. Функция сплайсинга регуляторных белков в нервной системе млекопитающих.
Факторы сплайсинга участвуют в ряде различных процессов во время развития мозга, включая:
(а) — самообновляющееся деление и дифференцировку нейрональных клеток;
(б) — миграцию новорожденного нейрона во время формирования коры головного мозга;
(в) — синаптогенез.
Кроме того, мРНК, кодирующая рецептор ApoER2, также подвергается альтернативному сплайсингу. Корректная изоформа этого рецептора, содержащая определенный экзон, важна для формирования и функционирования синапсов [31]. Включение этого экзона уменьшается в мозге пациентов с болезнью Альцгеймера из-за действия фактора сплайсинга из семейства SR — SRSF1 (один из тех случаев, когда фактор семейства SR не способствует сохранению экзона).
Однако было показано, что блокирование сайтов связывания SRSF1 с помощью антисмыслового (комплементарного этому фрагменту мРНК) олигонуклеотида способно существенно помочь в лечении болезни Альцгеймера [32].
Каждый из нас — сложнейший механизм
Мы в очередной раз убедились в том, насколько удивителен и сложен наш организм. Прямо сейчас в каждой клетке происходят многостадийные процессы: ДНК режется, с нее «списываются» первичные транскрипты, которые потом разбираются и собираются множеством способов в разные мРНК, служащие конвейером для сборки белков.
Альтернативный сплайсинг идет вразрез с догмой «один ген — один белок», ведь благодаря этому механизму практически каждый ген универсален: с помощью одного набора кодирующих последовательностей-«деталей» многие клетки могут собирать матрицы для производства самых разнообразных белков, необходимых ей. При этом каждая клетка собирает свой белок всего лишь из одного гена.
Любое наше движение, любая мысль, любое желание является следствием считывания и обработки генетического «сценария» нашего организма, а сами эти процессы напоминают слаженную работу всех элементов сложнейшего механизма, запутанность которого порой приводит к ошибкам и поломкам.
Но благодаря тому, что ученые непрерывно исследуют работу этого механизма, в будущем мы сможем не только «чинить», но и предугадывать и заранее предотвращать любые поломки, вызванные ошибками во время альтернативного сплайсинга.
Литература
- Глик Б. Молекулярная биотехнология. Принципы и применение. М.: «Мир», 2002. — 589 с.;
- Aishwarya G. Jacob, Christopher W. J. Smith. (2017). Intron retention as a component of regulated gene expression programs. Hum Genet. 136, 1043-1057;
- Andrew G. STEWART, Patricia M. CLISSOLD, Henry R. V. ARNSTEIN. (1976). The Initiation of Globin Synthesis in Differentiating Rabbit-Bone-Marrow Erythroid Cells. Eur J Biochem. 65, 349-355;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- J. Sambrook. (1977). Adenovirus amazes at Cold Spring Harbor. Nature. 268, 102-104;
- Michael G. Rosenfeld, Susan G. Amara, Bernard A. Roos, Estelita S. Ong, Ronald M. Evans. (1981). Altered expression of the calcitonin gene associated with RNA polymorphism. Nature. 290, 63-65;
- Ашмарян И.П., Антипенко А.Е., Ашапкин В.В. Нейрохимия: учебник для биологических и медицинских вузов. М: Изд. Института биомедицинской химии РАМН, 1996. — 470 с.;
- Азимова Ю.Э., Ашихмин Я.И., Кукушкин М.Л. (2019). Моноклональные антитела к CGRP: новое слово в лечении мигрени. Российский журнал боли. 17, 61–66;
- Моноклональные антитела;
- YAN WANG, JING LIU, BO HUANG, YAN-MEI XU, JING LI, et. al.. (2015). Mechanism of alternative splicing and its regulation. Biomedical Reports. 3, 152-158;
- Юрова К. А., Хазиахматова О. Г., Сохоневич Н. А. (2015). Альтернативный сплайсинг молекулы CD45 в механизмах молекулярно-генетического контроля дифференцировки Т-клеток. Российский иммунологический журнал. 9, 186–193;
- Ugo Moens, Ole Morten Seternes, Bjarne Johansen, Ole Petter Rekvig, Ole Petter Rekvig. (1997). . Virus Genes. 15, 135-154;
- John Karijolich, Yi-Tao Yu. (2010). Spliceosomal snRNA modifications and their function. RNA Biology. 7, 192-204;
- Jane Y. Wu, Tom Maniatis. (1993). Specific interactions between proteins implicated in splice site selection and regulated alternative splicing. Cell. 75, 1061-1070;
- Jernej Ule, Benjamin J. Blencowe. (2019). Alternative Splicing Regulatory Networks: Functions, Mechanisms, and Evolution. Molecular Cell. 76, 329-345;
- Christopher R. Sibley, Lorea Blazquez, Jernej Ule. (2016). Lessons from non-canonical splicing. Nat Rev Genet. 17, 407-421;
- Zefeng Wang, Michael E. Rolish, Gene Yeo, Vivian Tung, Matthew Mawson, Christopher B. Burge. (2004). Systematic Identification and Analysis of Exonic Splicing Silencers. Cell. 119, 831-845;
- C. Zhang, W.-H. Li, A. R. Krainer, M. Q. Zhang. (2008). RNA landscape of evolution for optimal exon and intron discrimination. Proceedings of the National Academy of Sciences. 105, 5797-5802;
- Allyson R Hatton, Vaidyanathan Subramaniam, A.Javier Lopez. (1998). Generation of Alternative Ultrabithorax Isoforms and Stepwise Removal of a Large Intron by Resplicing at Exon–Exon Junctions. Molecular Cell. 2, 787-796;
- James M Burnette, Etsuko Miyamoto-Sato, Marc A Schaub, Jamie Conklin, A Javier Lopez. (2005). Subdivision of Large Introns in Drosophila by Recursive Splicing at Nonexonic Elements. Genetics. 170, 661-674;
- Michael O. Duff, Sara Olson, Xintao Wei, Sandra C. Garrett, Ahmad Osman, et. al.. (2015). Genome-wide identification of zero nucleotide recursive splicing in Drosophila. Nature. 521, 376-379;
- Christopher R. Sibley, Warren Emmett, Lorea Blazquez, Ana Faro, Nejc Haberman, et. al.. (2015). Recursive splicing in long vertebrate genes. Nature. 521, 371-375;
- Как избавиться от РНК за несколько минут;
- Heidi Cook-Andersen, Miles F. Wilkinson. (2015). Splicing does the two-step. Nature. 521, 301-301;
- Brian Joseph, Shu Kondo, Eric C. Lai. (2018). Short cryptic exons mediate recursive splicing in Drosophila. Nat Struct Mol Biol. 25, 365-371;
- Anthony J Hinrich, Francine M Jodelka, Jennifer L Chang, Daniella Brutman, Angela M Bruno, et. al.. (2016). Therapeutic correction of ApoER2 splicing in Alzheimer's disease mice using antisense oligonucleotides. EMBO Mol Med. 8, 328-345;
- Susan A. Slaugenhaupt, Anat Blumenfeld, Sandra P. Gill, Maire Leyne, James Mull, et. al.. (2001). Tissue-Specific Expression of a Splicing Mutation in the Gene Causes Familial Dysautonomia. The American Journal of Human Genetics. 68, 598-605;
- Mayumi Yoshida, Naoyuki Kataoka, Kenjyo Miyauchi, Kenji Ohe, Kei Iida, et. al.. (2015). Rectifier of aberrant mRNA splicing recovers tRNA modification in familial dysautonomia. Proc Natl Acad Sci USA. 112, 2764-2769;
- GWAS и психогенетика: консорциумы в поисках ассоциаций;
- Chun-Hao Su, Dhananjaya D, Woan-Yuh Tarn. (2018). Alternative Splicing in Neurogenesis and Brain Development. Front. Mol. Biosci.. 5;
- Noèlia Fernàndez-Castillo, Gabriela Gan, Marjolein M.J. van Donkelaar, Mariliis Vaht, Heike Weber, et. al.. (2020). RBFOX1, encoding a splicing regulator, is a candidate gene for aggressive behavior. European Neuropsychopharmacology. 30, 44-55;
- Uwe Beffert, Edwin J. Weeber, Andre Durudas, Shenfeng Qiu, Irene Masiulis, et. al.. (2005). Modulation of Synaptic Plasticity and Memory by Reelin Involves Differential Splicing of the Lipoprotein Receptor Apoer2. Neuron. 47, 567-579.






