Бедные родственники, или незаслуженно забытые нуклеазы транспозонов
25 октября 2021
Бедные родственники, или незаслуженно забытые нуклеазы транспозонов
- 693
- 0
- 3
Бактериальные транспозоны группы IS200/IS605 кодируют три семейства РНК-программируемых нуклеаз, два из которых являются предками белков Cas9 и Cas12.
Рисунок в полном размере.
сайт Pixels
-
Автор
-
Редакторы
Темы
Известная каждому биологу система CRISPR-Cas9, нашедшая широчайшее применение в области редактирования геномов, произошла от нуклеаз IscB, которые кодируют транспозоны группы IS200/IS605. Хотя этот факт был установлен довольно давно по меркам молекулярной биологии — в 2015 году — функции IscB так и остались неизученными. До недавнего времени было неизвестно даже, способны ли они разрезать ДНК-мишень при участии гидовой РНК подобно их потомкам — нуклеазам Cas9. Недавнее исследование американских ученых наконец позволило пролить свет не только на свойства IscB и других нуклеаз, кодируемых транспозонами, но и детально разобраться в их эволюции и происхождении Cas9.
Вряд ли найдется биолог, который бы не слышал о редактировании геномов с помощью систем CRISPR-Cas9. Ключевой элемент этой системы, нуклеаза Cas9, которая, привлекаемая гидовой РНК, разрезает ДНК-мишень, произошла от нуклеаз IscB, которые кодируют транспозоны группы IS200/IS605. Эти же транспозоны кодируют нуклеазы TnpB, от которых, скорее всего, произошли нуклеазы Cas12, которые также нашли применение в геномном редактировании. Хотя нуклеазы транспозонов, давшие начало нуклеазам Cas9 и Cas12, известны уже несколько лет, до недавнего момента про их свойства не было известно ровном счетом ничего. Недавняя работа американских ученых, опубликованная в Science, устранила эту вопиющую несправедливость. Авторы исследования не только досконально изучили биохимические свойства IscB и другие нуклеазы транспозонов, эволюционно связанные с Cas9 и Cas12, но и смогли восстановить «родственные» отношения между ними. Правда, клеточные функции этих нуклеаз так и остались невыясненными.
Подробнее о том, что такое системы CRISPR-Cas и как они работают, читайте в статьях «Биомолекулы» [1], [2].
Нуклеаза IscB устроена примерно так же, как Cas9: она содержит эндонуклеазный домен RuvC, который разбит на две части небольшой спиралью-мостиком, и эндонуклеазный домен HNH. Кроме того, у нее есть N-концевой участок с непонятной функцией, которого нет у Cas9. Этот домен авторы работы обозначили как PLMP (рис. 1). Авторы исследования показали, что IscB и Cas9 являются единственными из известных на данный момент белков, которые обладают описанной выше доменной архитектурой, включающей нуклеазные домены RuvC и HNH в составе одного белка [3]. Гены, кодирующие Cas9, ассоциированы с локусами CRISPR, со спейсеров в которых и считываются направляющие РНК. А есть ли нуклеазы IscB, ассоциированные с CRISPR? Раньше такая связь отрицалась, однако авторы обсуждаемой работы показали, что в действительности по крайней мере часть IscB связана с CRISPR. Примечательно, что связь с CRISPR приобретали неродственные друг другу IscB, что указывает на то, что связь IscB с CRISPR в ходе эволюции возникала несколько раз. Одна из таких связей в конечном итоге привела к появлению нуклеаз Cas9. Но могут ли IscB, ассоциированные с CRISPR, функционировать как направляемые РНК нуклеазы?
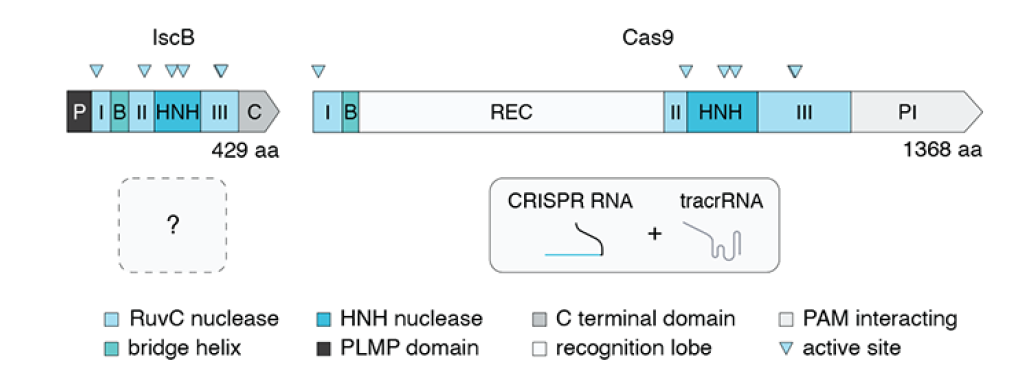
Рисунок 1. Доменная организация IscB и Cas9. Цветами показаны нуклеазные домены RuvC и HNH, мостиковая спираль, домен PLMP и C-концевой домен, домен REC, участвующий в распознавании гидовой РНК, и домен, распознающий PAM (protospacer adjacent motif). Треугольниками отмечены активные нуклеазные сайты.
Авторы показали, что нуклеазы IscB, ассоциированные с CRSIPR, действительно, могут связывать РНК. Более того, РНК, с которыми взаимодействуют IscB, считываются с прямого повтора в локусе CRISPR и части спейсера. Оказалось, что IscB и вправду вносят разрывы в ДНК, на которую «указывает» связанная с ними гидовая РНК. Однако большая часть IscB все же не связана CRISPR. Авторы показали, что перед геном, кодирующим IscB, находится консервативный межгенный участок длиной примерно 300 нуклеотидов. Его последовательность такова, что допускает образование шпилек в считываемых с него направляющих РНК. Такие направляющие РНК, содержащие несколько шпилек, авторы работы обозначили как ω-РНК. Таким образом, ген iscB по крайней мере в ряде случаев идет «в комплекте» с вышележащим межгенным участком, кодирующим направляющую РНК с несколькими шпильками (рис. 2). Ученые предположили, что ген, кодирующий направляющую РНК для IscB, является эволюционным предком локусов CRISPR.
Чтобы выяснить, идет ли транскрипция с предполагаемых участков, кодирующих направляющие РНК для IscB, авторы исследования отсеквенировали малые РНК почвенной бактерии Ktedonobacter racemifer штамма SOSP1-21, в геноме которой содержится 49 локусов IscB, и показали, что, действительно, с этих локусов считываются ω-РНК. В роли направляющей РНК, по-видимому, выступает 5’-концевой участок ω-РНК, который, к слову, обладает наименьшей консервативностью и, следовательно, может быть подстроен под разные мишени. Ближе к 3’-концу в ω-РНК располагаются шпильки, которые в распознавании ДНК-мишени не участвуют. По сути, локус IscB содержит ген, кодирующий собственно белок IscB, и область, с которой считывается одна или несколько ω-РНК.
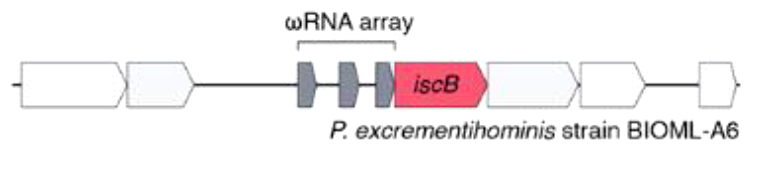
Рисунок 2. Строение локуса IscB: перед геном, кодирующим белок iscB, находится кластер, кодирующий несколько ω-РНК.
Насколько IscB можно перепрограммировать на разрезание другой мишени? Как показали эксперименты с IscB вышеупомянутой бактерии Ktedonobacter racemifer, нуклеаза IscB вносит разрывы в ДНК-мишени, соответствующие ω-РНК, причем направлять фермент можно на разные мишени, меняя последовательность ω-РНК. Таким образом, IscB — настоящая РНК-программируемая нуклеаза. Стоит отметить, что, помимо комплементарности 5’-концевой части ω-РНК, для распознавания мишени IscB рядом с ее 3’-концом должна располагаться особая последовательность, обозначаемая TAM (от англ. target-adjacent motif). Однако для дальнейших экспериментов авторы работы выбрали IscB из другой бактерии, Allochromatium warmingii, как более эффективный вариант. Оказалось, что этот фермент, как и Cas9, вносит разрыв в последовательность мишень ровно на три нуклеотида выше TAM! Сходство IscB с Cas9, как говорится, налицо.
Любопытно, что гены, кодирующие ω-РНК, встречаются и там, где нет генов, кодирующих IscB. Авторы предполагают, что такие «сильные и независимые» ω-РНК могут быть использованы IscB для перепрограммирования на другие мишени. Экспериментальная проверка этой гипотезы на IscB Ktedonobacter racemifer, у которых экспрессируются девять одиночных ω-РНК, показала, что, действительно, пять из них могут взаимодействовать с IscB и направлять ее на другие мишени. Примерно треть ω-РНК направляют IscB на последовательности в собственном прокариотическом геноме, поэтому, как полагают ученые, системы на основе IscB выполняют какие-то функции, не связанные с защитой от инородных генетических элементов — в противном случае нуклеазы IscB просто порезали бы собственный геном клетки.
Авторы работы произвели биоинформатический поиск других нуклеаз, которые были бы структурно близки к IscB и Cas9. Им удалось найти еще одно семейство нуклеаз, которые кодируют транспозоны IS200/IS605 — его обозначили как IsrB. Белки IsrB имеют PLMP-домен (тот самый странный N-концевой домен IscB) и разбитый спиралью надвое нуклеазный домен RuvC, но не имеют нуклеазного домена HNH. Чтобы изучить, как все эти белки — IscB, IsrB, Cas9 — связаны друг с другом эволюционно, ученые построили филогенетическое дерево на основе выравниваний последовательностей домена RuvC и разделяющей его надвое спиралью-мостика (рис. 3). На построенном дереве все указанные белковые семейства — IscB, IsrB, Cas9 — формируют четко обособленные клады, что свидетельствует о том, что каждое семейство нуклеаз возникло самостоятельно в ходе эволюции. Гены IscB и IsrB очень редко ассоциированы с локусами CRISPR и никогда не связаны с генами белков Cas, участвующих в адаптации — процессе внедрения в локус CRISPR новых спейсеров. Эта особенность фундаментально отличает IscB и IsrB от Cas9. Тем не менее, общность эволюционного происхождения IscB, IsrB и Cas9 не вызывает сомнений. Авторы работы полагают, что самым древним предковым семейством были нуклеазы IsrB, которые не имеют домена HNH. IsrB, вероятно, произошло от некой эндонуклеазы, имевшей домен RuvC. Способность к взаимодействию с направляющей РНК, которая направляет нуклеазу на мишень, скорее всего, появилась на ранних этапах эволюции нуклеаз IscB, IsrB и Cas9, примерно тогда, когда домен RuvC приобрел вставки, делающими возможным его взаимодействие с ω-РНК. Стоит отметить, что гены IsrB тоже ассоциированы с ω-РНК, однако ω-РНК IsrB и IscB существенно различаются друг от друга по вторичной структуре.
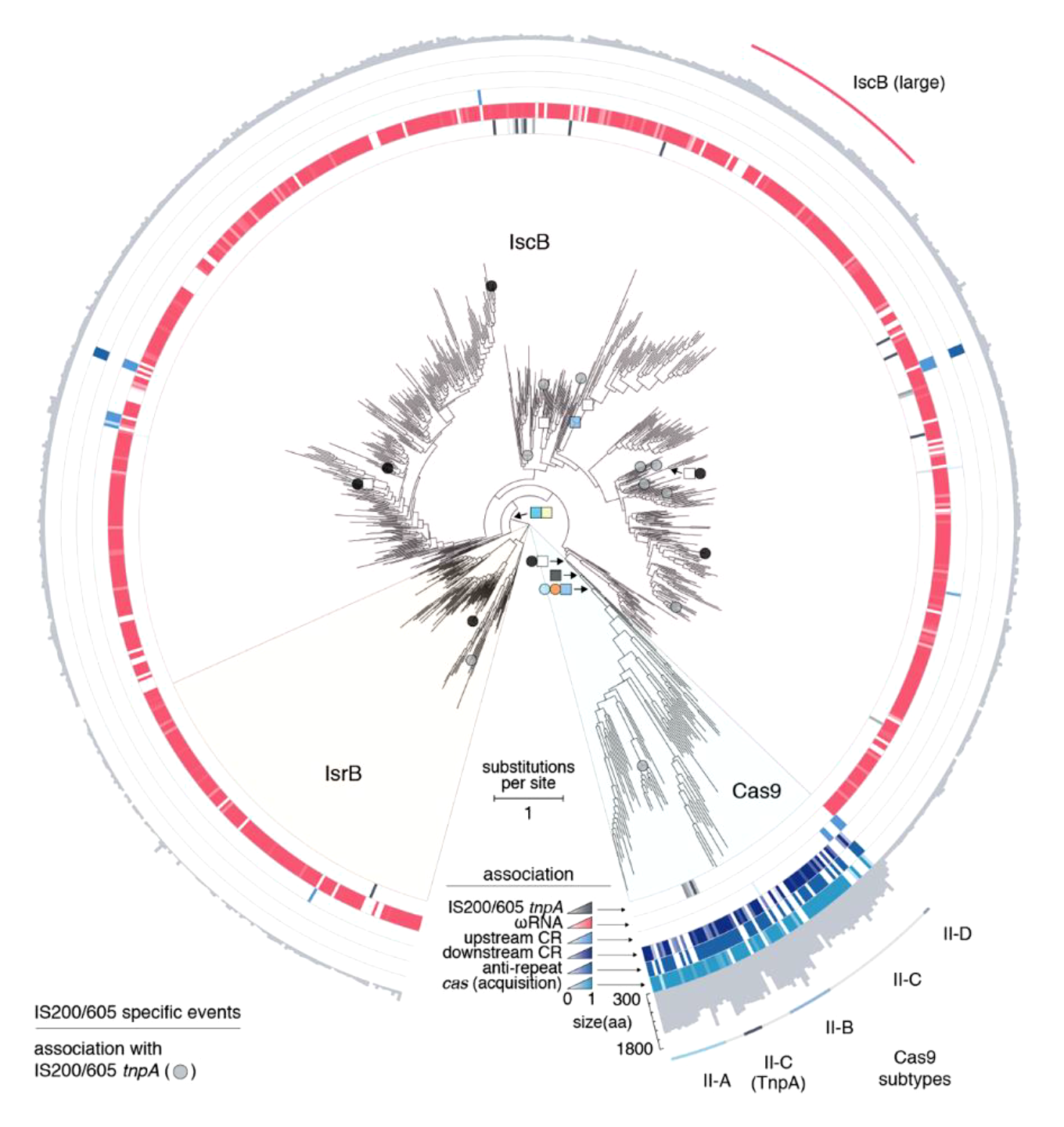
Рисунок 3. Филогенетическое дерево, построенное на основе выравниваний последовательностей домена RuvC и спирали-мостика. Видно, что IsrB, IscB и Cas9 имеют общее эволюционное происхождение, однако обособление каждого из семейств нуклеаз происходило независимо, поскольку они образуют четко оформленные клады.
Связь с CRISPR в семействе IscB в ходе эволюции появлялась несколько раз независимо. Локусы CRISPR с повторами и спейсерами могли возникнуть в результате дупликаций участков, кодирующих ω-РНК. Эволюция Cas9 из нуклеаз IscB сопровождалась появлением ряда вставок в домене RuvC и мостиковой спирали. Ассоциация с белками Cas, участвующими в процессе адаптации, привела к появлению массы вариантов Cas9 у разных бактерий, которыми они также активно обменивались с помощью горизонтального переноса генов.
Удивительно, но нуклеазы бактериальных транспозонов есть и у эукариот! Авторы исследования смогли найти множество локусов iscB в хлоропластах наземной зеленой водоросли Ignatius tetrasposus. Хотя в большинстве из них открытая рамка считывания IscB разрушена разнообразными мутациями, один локус все же кодирует функциональную нуклеазу IscB и реально транскрибируемую ω-РНК. В чем функциональная роль этих элементов, к сожалению, остается неизвестным.
Тот факт, что нуклеазы IscB и IsrB можно направлять на разные мишени, меняя последовательность ω-РНК, делает их привлекательными инструментами для внесения мутаций в геном. Авторы исследования проверили способность нескольких IscB делать вставки и делеции в клетках HEK293. Один из проверенных белков действительно оказался способен вносить мутации в клетках человека, правда, с невысокой эффективностью — порядка 4,4%. Так что белки IscB теоретически могут быть использованы для редактирования геномов, однако эффективность нуклеаз дикого типа пока оставляет желать лучшего.
Транспозоны IS200/IS605 кодируют еще одно семейство нуклеаз с доменом RuvC — TnpB. По данным предыдущих работ, белки этого семейства стали предками Cas12. Эти нуклеазы также находят все более широкое применение в геномном редактировании. Авторы работы выяснили, что непосредственно к генам TnpB прилегают консервативные участки, с которых считываются некодирующие РНК, причем их 3’-конец частично захватывает открытую рамку считывания TnpB. В условиях in vitro нуклеазы TnpB действительно были способны вносить разрезы в ДНК-мишень, соответствующую направляющей РНК. Примечательно, что семейство TnpB оказалось существенно более многочисленным, чем IscB: авторы работы выявили более миллиона предполагаемых локусов TnpB в геномах бактерий и архей, что делает их одним из самых многочисленных прокариотических семейств генов.
Итак, транспозоны кодируют несколько семейств РНК-программируемых нуклеаз: IsrB, IscB и TnpB, которые авторы исследования предлагают в совокупности обозначать как Ω (от OMEGA: Obligate Mobile Element Guided Activity). К сожалению, мы пока так и не знаем, что эти ферменты делают в клетках прокариот. Возможно, они являются частью своеобразной системы токсин–антитоксин, в которой транспозон является токсином, а нуклеаза, которая его вырезает, — антитоксином. А может быть, они, напротив, обеспечивают направляемое РНК перемещение транспозонов. Обнаружение функционально активной нуклеазы IscB в хлоропластах показывает, что РНК-направляемые нуклеазы, разрезающие ДНК, могут играть важную роль в функционировании и эукариотической клетки тоже.
Литература
- Просто о сложном: CRISPR/Cas;
- Разнообразие и эволюция систем CRISPR/Cas;
- Han Altae-Tran, Soumya Kannan, F. Esra Demircioglu, Rachel Oshiro, Suchita P. Nety, et. al.. (2021). The widespread IS200/IS605 transposon family encodes diverse programmable RNA-guided endonucleases. Science. 374, 57-65.