Разнообразие и эволюция систем CRISPR/Cas
24 мая 2019
Разнообразие и эволюция систем CRISPR/Cas
- 4338
- 1
- 13
Пространственные структуры эффекторных комплексов CRISPR/Cas-систем классов 1 и 2
-
Автор
-
Редактор
Системы CRISPR/Cas вот уже несколько лет триумфально шествуют по лабораториям мира, и неудивительно: эта простая, дешевая и быстрая технология редактирования генома нашла самое широкое применение в генетической инженерии. Однако биология систем CRISPR/Cas, а именно их функционирование в качестве противовирусных иммунных агентов бактерий и архей, не менее интересна и увлекательна: разнообразие молекулярных механизмов и в то же время их фундаментальное сходство и простота общей идеи поражают воображение. Наш обзор посвящен разнообразию естественных систем CRISPR/Cas.
Системы CRISPR/Cas защищают клетки бактерий и архей от вирусов, мобильных генетических элементов и прочей инородной ДНК (а некоторые виды систем — и РНК) . CRISPR (дословно переводится как «короткие палиндромные повторы, регулярно расположенные группами» от англ. Clustered Regularly Interspaced Short Palindromic Repeats) представляют собой особым образом устроенные локусы генома, в которых короткие палиндромные (то есть одинаково читающиеся в обоих направлениях) повторы разделяются небольшими уникальными последовательностями — спейсерами. Неподалеку от локуса CRISPR на ДНК находятся гены, кодирующие белки Cas, которые играют важнейшую роль во всех этапах работы этой защитной системы бактерий.
Подробнее об общих принципах работы систем CRISPR/Cas можно прочесть в статьях «Просто о сложном: CRISPR/ Cas» [1] и «CRISPR-системы: иммунизация прокариот» [2].
В общем случае при первом столкновении прокариотической клетки с вирусом из его ДНК белки Cas вырезают небольшой фрагмент (его еще называют протоспейсером) и обеспечивают его интеграцию в локус CRISPR в качестве нового спейсера. При следующей встрече с тем же вирусом с соответствующего спейсера считывается РНК (ее обозначают CRISPR-РНК, или, сокращенно, crРНК), комплементарная участку его генома, которая взаимодействует с вирусом и привлекает к его ДНК другие белки Cas, разрезающие вирусный геном на кусочки, тем самым инактивируя его. Эта базовая идея лежит в основе работы всех систем CRISPR/Cas, однако детали у разных типов и подтипов различаются.
В большинстве систем CRISPR/Cas за этап адаптации, то есть встраивания в CRISPR нового спейсера, отвечают белки Cas1 и Cas2. Они формируют комплекс, где Cas1 функционирует как эндонуклеаза (задействованная и в вырезании фрагмента из вирусной ДНК, и во внесении разрывов в локус CRISPR) и интеграза (которая встраивает новый спейсер в бактериальный геном), а Cas2 обычно формирует структурную опору (рис. 1). Во многих системах CRISPR/Cas в адаптации принимают участие и другие белки Cas (Cas3, Cas4 или Cas9), а если фрагмент вырезается из генома РНК-вируса, то и обратная транскриптаза, причем нередко перечисленные белки сливаются с Cas1 или Cas2 и синтезируются в виде единой молекулы. Разнообразие систем CRISPR/Cas связано преимущественно с эффекторными белками, которые обеспечивают уничтожение чужеродной ДНК [3].
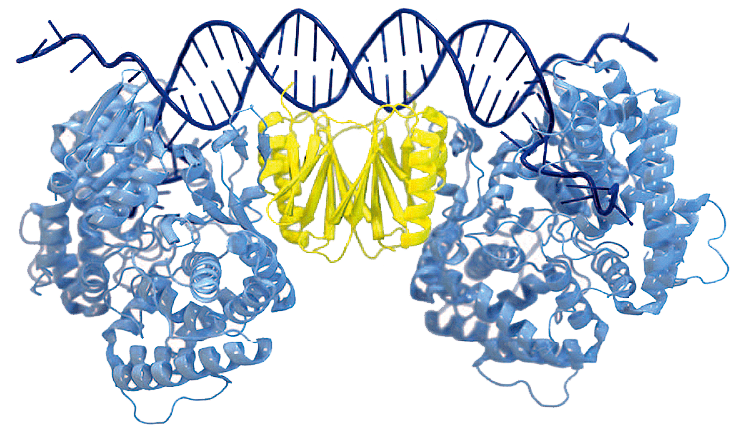
Рисунок 1. Кристаллическая структура комплекса Cas1—Cas2, связанного с ДНК. Cas1 показан голубым цветом, Cas2 — желтым.
Классификация
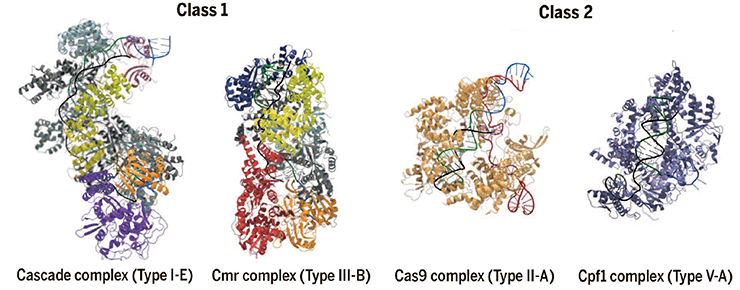
На основе устройства эффекторных белковых комплексов системы CRISPR/Cas разделяют на два класса. В системах класса 1 эффекторный комплекс образован множеством субъединиц, в том числе несколькими белками Cas, а в системах класса 2 эффектором является один большой многодоменный белок. К сожалению, белки Cas, и даже белок Cas1 — самый эволюционно консервативный из белков Cas, — в силу разнообразия аминокислотных последовательностей и независимой эволюции не могут быть использованы для воссоздания филогении систем CRISPR/Cas [3].
Классы систем CRISPR/Cas подразделяются на несколько типов. К классу 1 относятся типы I, III и IV, а к классу 2 — типы II, V и VI. Деление на типы основано на устройстве эффекторных комплексов, причем в системах одного и того же типа, как правило, в состав комплекса входит особый сигнатурный белок, уникальный для систем этого типа. Типы, в свою очередь, делятся на подтипы, различающиеся особенностями строения локуса CRISPR и, в некоторых случаях, наличием уникальных белков Cas (рис. 2) [3].
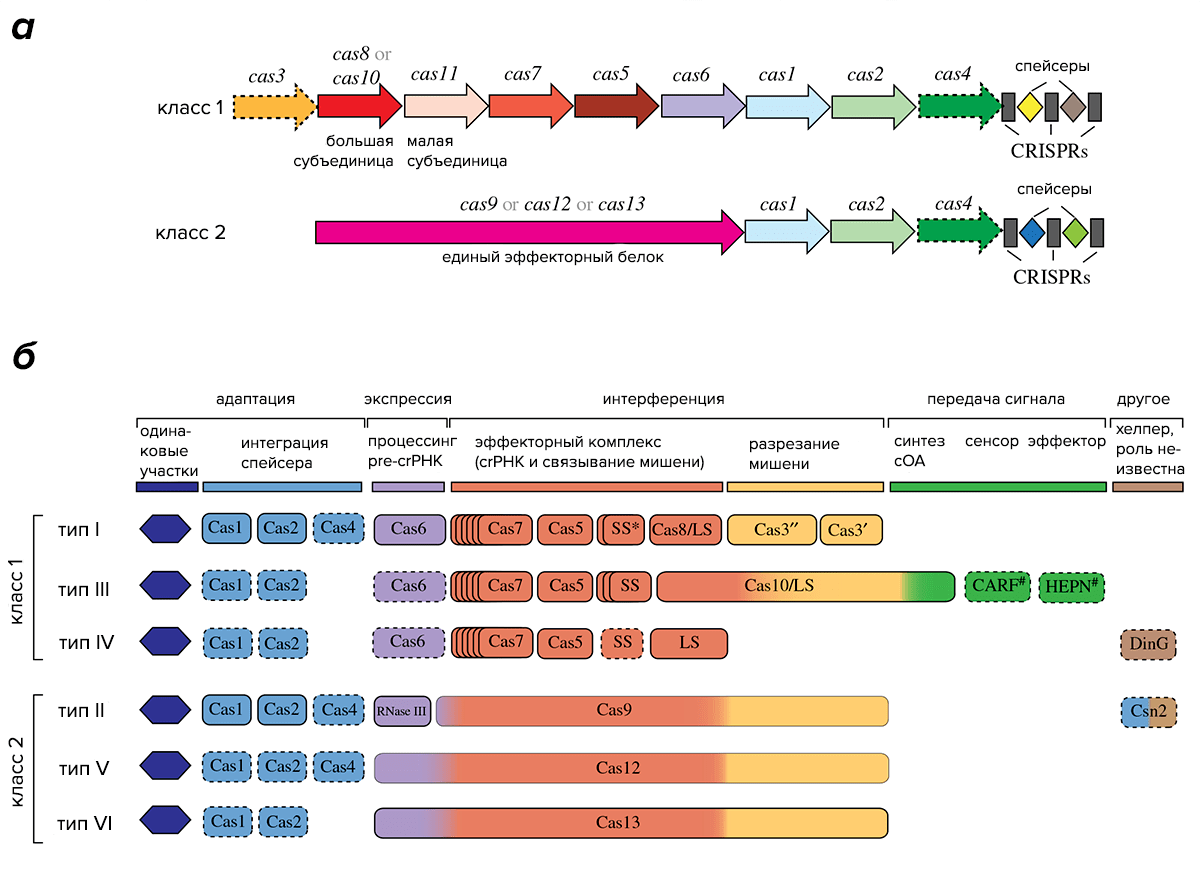
Рисунок 2. Разнообразие систем CRISPR/Cas. а — Общая схема устройства систем классов 1 и 2. б — Схема отдельных типов систем.
[3], рисунок адаптирован
В системах класса 1 созревание (процессинг) crРНК катализирует сложный комплекс из белков Cas, известный как Cascade (от англ. CRISPR-associated complex for antiviral defence). Он связывается с транскриптом-предшественником crРНК (пре-crРНК) и привлекает дополнительный белок Cas6 (реже Cas5) — нуклеазу, осуществляющую процессинг [3].
В наиболее изученных представителях класса 2 — системах типа II — процессинг катализирует фермент, не имеющий отношения к системе CRISPR/Cas, — бактериальная РНКаза III — при участии дополнительных РНК — транс-действующих CRISPR-РНК (tracrРНК от trans-acting). tracrРНК описаны также в системах типа V класса 2. Однако в системах этого типа и типа VI пока еще не охарактеризована нуклеаза, которая осуществляет процессинг предшественника crРНК. Но известно, что она также входит в состав эффекторного комплекса, разрушающего чужеродную ДНК [3].
Различия между системами классов 1 и 2 становятся особенно очевидны на этапе интерференции, когда crРНК распознает свою мишень и связывается с ней. В системах типа I, относящихся к классу 1, белковый комплекс, содержащий зрелую crРНК, распознает комплементарную ей последовательность в мишени и привлекает дополнительный белок Cas — Cas3, — который обладает хеликазной активностью, благодаря чему расплетает двойную спираль ДНК-мишени, а также имеет нуклеазный домен, который разрезает мишень. В системах типа III класса 1 нуклеаза, которая разрушает молекулу-мишень, входит в состав процессирующего комплекса, причем для деградации чужеродной ДНК ей не требуется дополнительной хеликазной активности. Кроме того, в ходе работы систем типа III специальная РНКаза, ассоциированная с локусами CRISPR, разрушает еще и транскрипты, считанные с генома-мишени. В системах же класса 2 за разрушение мишени отвечают различные домены одного и того же большого эффекторного белка [3].
Как мы уже отмечали, восстанавливать филогенетические отношения между различными типами систем CRISPR/Cas даже по довольно консервативному белку Cas1, присутствующему во всех системах, невозможно из-за того, что системы CRISPR/Cas разных типов и подтипов часто обмениваются друг с другом своими эффекторными модулями. Особенно ярко эта особенность прослеживается в тех подтипах систем III типа, которые работают с РНК-мишенями и потому включают в себя обратную транскриптазу. Они часто обмениваются эффекторными модулями с системами других подтипов III типа. Вероятно, такой обмен служит своего рода ответом на постоянно повышающееся разнообразие быстро эволюционирующих вирусов. Обмен эффекторными модулями также помогает защититься от белков анти-CRISPR (Acr) , которыми обзавелись многие бактериофаги [3].
Подробнее о системах анти-CRISPR читайте в статье «Анти-CRISPR: ответ вирусов» [4].
В случае многих типов систем CRISPR/Cas эволюция защитных функций приводила к их вовлечению в другие процессы, не связанные с адаптивным иммунитетом [3]. «Биомолекула» уже писала о нескольких любопытных примерах участия систем CRISPR/Cas в процессах, не связанных с защитой от вирусов и мобильных генетических элементов [5].
Происхождение адаптационного модуля
Детальное изучение генов cas показало, что ген белка Cas1 (как мы помним, этот белок отвечает за интеграцию спейсеров в локусы CRISPR) не всегда связан с локусами CRISPR. Выяснилось, что в ряде случаев ген cas1 оказывается внутри участков ДНК длиной от 12 до 18 тысяч пар оснований, которые фланкированы концевыми инвертированными повторами, а это одна из характерных черт транспозонов . Оказалось, что эти предполагаемые транспозоны несут не только ген cas1, но и ген ДНК-полимеразы семейства B. Кроме того, в них часто содержатся дополнительные гены, кодирующие разнообразные нуклеазы и ДНК-связывающие белки. Более того, интегразы транспозонов и Cas1, который исполняет роль интегразы при вставке новых спейсеров, функционируют по очень похожим механизмам. Новая группа транспозонов получила название «каспозоны». Каспозоны способны синтезировать сами себя за счет ДНК-полимеразы семейства B и используют механизм «скопировать—вставить» для перемещения по геному. Примечательно, что, хотя подобный тип транспозонов распространен среди эукариот, у прокариот он идентифицирован впервые [3].
О транспозонах и других мобильных генетических элементах рассказывает статья «Мобильные генетические элементы прокариот: стратификация “общества” бродяжек и домоседов» [6].
Дальнейшее изучение каспозонов показало, что они гораздо больше распространены среди архей, чем среди бактерий. Пока что перемещение каспозонов не удалось продемонстрировать экспериментально, однако геномный анализ нескольких штаммов археи Methanosarcina mazei позволил найти следы их недавних перемещений, так что по крайней мере некоторые из каспозонов действительно являются активными транспозонами [3].
Предполагают, что адаптационный модуль систем CRISPR/Cas произошел от предкового каспозона (рис. 4), который, кроме того, мог содержать дополнительные гены cas. Хотя идентифицированные на данный момент каспозоны не кодируют Cas2, некоторые из них кодируют нуклеазы, гомологичные Cas4, и ряд дополнительных нуклеаз. Возможно, в процессе формирования адаптационного модуля CRISPR/Cas произошла иммобилизация каспозона, при этом часть его генов — в том числе ген ДНК-полимеразы — были утеряны [3].
Происхождение систем класса 1
Хотя аминокислотные последовательности эффекторных белков типов I и III класса 1 мало похожи, можно не сомневаться, что эффекторные комплексы этих систем имеют общее происхождение. Их пространственные структуры поразительно похожи, и в обоих случаях структурная основа эффекторного комплекса образована несколькими копиями белка Cas7 (рис. 3). Также комплексы содержат белок Cas5, приходящийся дальним родственником белку Cas7, и так называемые малую и большую субъединицы. Малые субъединицы очень близки структурно и сходны с C-концевым доменом белка Cas10. В системах I типа роль большой субъединицы исполняет белок Cas8, аминокислотная последовательность которого высоко вариабельна даже в пределах систем различных подтипов типа I. В системах III типа большая субъединица представлена белком Cas10, который содержит два РНК-связывающих домена, причем один из них очень похож на домен-«ладонь», имеющийся у многих РНК- и ДНК-полимераз. Отметим, что Cas8 структурно лишь отдаленно напоминает белок Cas10, однако общность структурной организации и наличие опорного элемента из белков Cas5 и Cas7 почти не оставляет сомнений, что системы I и III типов класса 1 произошли от одного предка. Любопытно, что на долю этих типов приходится около 90% всех идентифицированных систем CRISPR/Cas и почти 100% этих систем у архей [3].
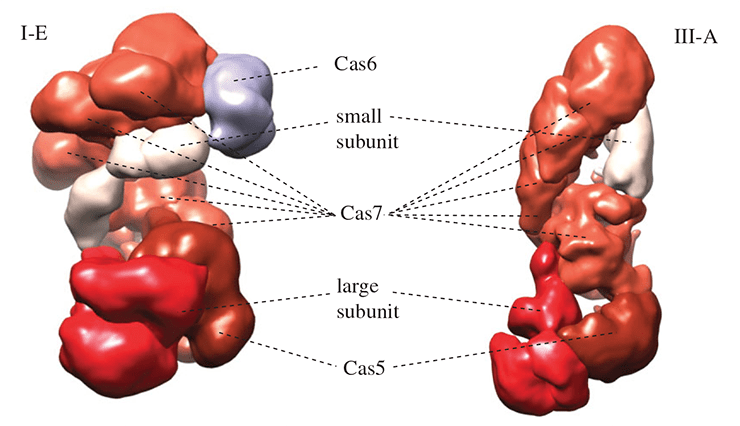
Рисунок 3. Сравнение строения эффекторных комплексов систем I и III типов класса 1
Стоит отметить, что распознавание мишени системами III типа запускает полимеразную активность белка Cas10, который начинает производить циклические олиго-(А)-нуклеотиды (cOA). Большинство систем CRISPR/Cas III типа включает ряд белков, которые не относятся к числу белков Cas, но необходимы для функционирования системы. Таковыми являются, в частности, белки Csm6 и Csx1. Csm6 связывает cOA, за счет чего активируется его РНКазный домен, который разрушает и РНК-мишень (напомним, многие системы CRISPR/Cas III типа действуют на РНК, а не на ДНК), и другие молекулы РНК, «подвернувшиеся под руку». Следствием такого массового неспецифического разрушения РНК является или переход клетки в состояние покоя, или ее программируемая гибель. Оба этих варианта предотвращают дальнейшее распространение вируса по бактериальной колонии и массовую гибель других бактериальных клеток [3].
Так как последствием активации пути Cas10—cOA может стать программируемая клеточная гибель, существует предположение, что эффекторные комплексы некоторых систем CRISPR/Cas произошли от систем ответа на стресс, которые запускали гибель клетки. Ген возможного белка, который дал эволюционное начало сигнальной системе с защитной функцией, удалось идентифицировать в геномах нескольких бактерий. Этот белок содержит единственный домен-«ладонь», гомологичный полимеразно-циклазному домену Cas10. Этот факт служит еще одним подтверждением предположения, что адаптивный иммунитет прокариот, то есть системы CRISPR/Cas, произошли от древних форм защиты бактериальных сообществ, запускавших гибель клетки или переводящих ее в состояние покоя. Системы III типа с двойной функцией — разрушение чужеродной ДНК и запуск состояния покоя или программируемой гибели клетки — считаются одними из самых сложных систем CRISPR/Cas [3].
Системы IV типа встречаются в основном в составе плазмид и профагов (рис. 4) и подробнее рассматриваются ниже. На данный момент достоверных сведений об их происхождении нет.
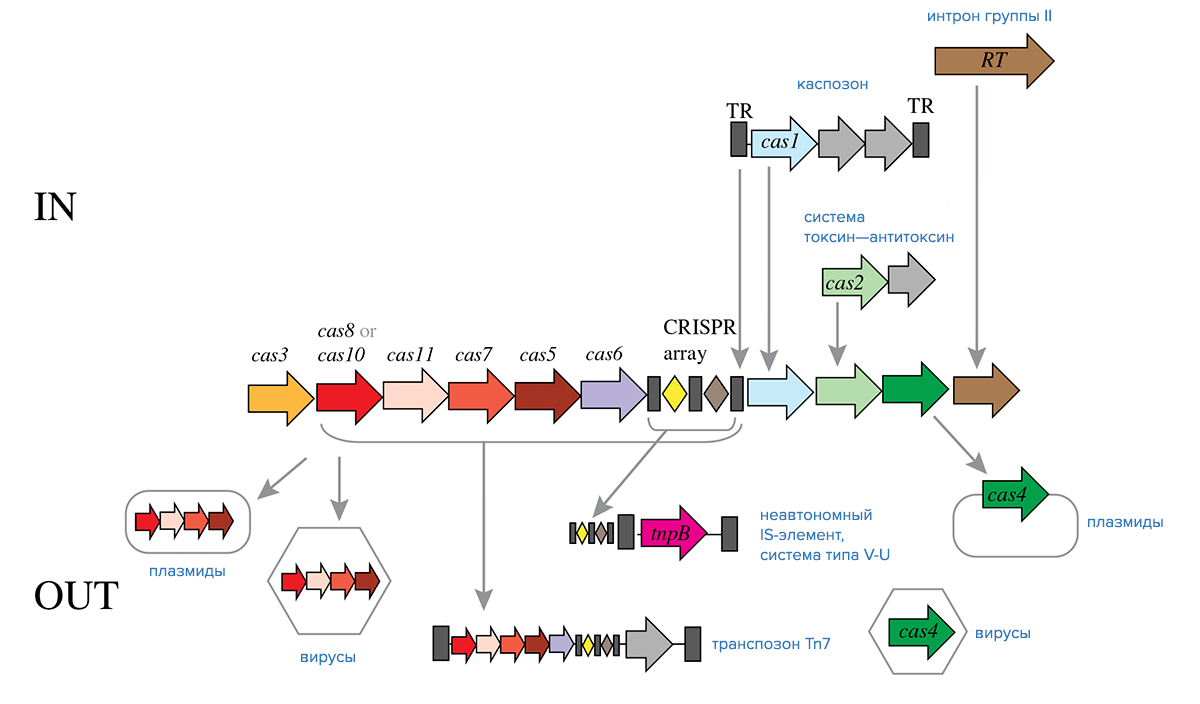
Рисунок 4. Обмен компонентами между системами CRISPR-Cas класса 1 и мобильными генетическими элементами в ходе эволюции. Условные обозначения: RT — обратная транскриптаза, TR — концевые повторы.
[3], рисунок адаптирован
Происхождение систем класса 2
Эффекторная компонента систем класса 2 радикальным образом отличается от таковой у систем класса 1. Все эффекторные функции выполняет не сложный многобелковый комплекс, а один большой белок со многими доменами. Удивительно, но все белки-эффекторы систем класса 2 гомологичны нуклеазам, которые кодируются разнообразными мобильными генетическими элементами. Эффекторные белки систем типов II и V — Cas9 и Cas12 соответственно — имеют общий домен с эндонуклеазами семейства RuvC (RuvC-подобный домен). Собственно, этот домен — единственное, что объединяет Cas9 и Cas12: их аминокислотные последовательности совершенно различны, и эти белки, по-видимому, не гомологичны друг другу [3].
Если не брать в расчет белки Cas, ближайшие родственники белков Cas9 и Cas12 — это белки TnpB, также содержащие домен RuvC и принадлежащие IS-элементам семейства IS605 и их родственникам (рис. 5). Интересно, что гены tnpB — одни из самых многочисленных генов в геномах бактерий и архей. Судя по всему, Cas9, Cas12 и TnpB имеют общее эволюционное происхождение. Системы CRISPR/Cas подтипа V-U вообще не имеют генов адаптационного модуля и содержат, помимо локуса CRISPR, только несколько генов белков, гомологичных TnpB, которые, вероятнее всего, и являются эффекторными. Предполагают, что подтип V-U появился совсем недавно по эволюционным меркам и представляет собой модель предковой формы систем типов II и V [3].
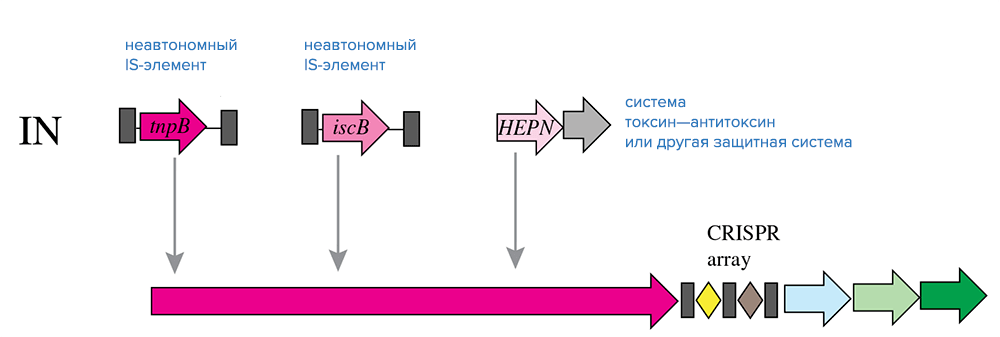
Рисунок 5. Обмен компонентами между системами CRISPR-Cas класса 2 и мобильными генетическими элементами в ходе эволюции. Условные обозначения: HEPN — РНКаза.
[3], рисунок адаптирован
Появление систем II и V типов связывают со случайной вставкой TnpB-содержащих IS-элементов рядом с локусом CRISPR (рис. 5). Дальнейший ход эволюции сопровождался независимым преобразованием белков TnpB в эффекторные белки путем получения дополнительных доменов (правда, не совсем понятно, откуда: возможно, источниками дополнительных доменов стали внутренние акты дупликации и рекомбинации) [3].
Помимо разнообразных мобильных элементов, важную роль в эволюции систем CRISPR/Cas, по-видимому, сыграли системы токсин—антитоксин: показано, что многие белки-токсины из этих семейств родственны некоторым белкам Cas (а именно Cas2) [3].
Системы типа VI, подробно рассматриваемые в следующем разделе, отличаются тем, что распознают исключительно РНК-мишени. Системы этого типа почти наверняка связаны с системами токсин—антитоксин, поскольку в их белках Cas часто обнаруживают домен HEPN, имеющийся у многих белков-токсинов, действующих как РНКазы (рис. 5) [3].
Системы CRISPR/Cas, распознающие РНК
Как мы уже отмечали, некоторые системы CRISPR/Cas III типа класса 1 распознают мишени, представленные не молекулами ДНК, а молекулами РНК. Как правило, РНК-мишенями выступают транскрипты, считанные с чужеродного ДНК-генома. Для того чтобы извлечь из них спейсеры, пригодные для вставки в CRISPR, работает особая обратная транскриптаза, ассоциированная с CRISPR. Очень часто этот фермент слит в единую молекулу с белком Cas1 или же кодируется геном, расположенным рядом с геном cas1. Как показал филогенетический анализ, обратные трансриптазы, работающие с CRISPR, наиболее близки к обратным транскриптазам интронов II группы . Вероятно, системы CRISPR/Cas приобрели обратную транскриптазу, когда интрон II группы случайно вставился рядом с локусами CRISPR/Cas (рис. 4) [3].
Подробнее об обратной транскрипции можно прочесть в статье «Мода на ретро. Где встречается обратная транскрипция, и как она эволюционировала» [7].
Недавно открытые системы VI типа класса 2 с эффекторным белком Cas13 действуют исключительно на РНК-мишени. Белок Cas13 представляет собой РНК-направляемую РНКазу и эффективно защищает, например, кишечную палочку Escherichia coli от бактериофага MS2, чей геном представлен РНК [8]. Интересной особенностью, ранее не описанной для систем CRISPR/Cas, обладает эффекторный белок подтипа VI-B (Cas13b): его активность может повышаться или понижаться под действием вспомогательных белков Cas.
Когда Cas13 активируется РНК-мишенью, он становится неспецифичной РНКазой, которая режет все РНК, до которых сможет «дотянуться». Разумеется, такое действие токсично для клетки и приводит либо к ее программируемой смерти, либо переводит клетку в покоящееся состояние. Как мы уже отмечали, подобное действие на клетку оказывает активированный белок Cas10 систем III типа класса 1. Поскольку РНК-содержащие бактерифаги весьма немногочисленны, вероятнее всего, системы VI типа разрушают транскрипты, считанные с вирусных ДНК-геномов, и запускают гибель клетки или переводят ее в состояние покоя в ответ на интенсивную экспрессию вирусных генов [9].
Минималистичные варианты систем CRISPR/Cas
Наряду с возникновением сложно организованных систем CRISPR/Cas, в состав которых входят разнообразные белки и РНК, есть и обратный «тренд» — появление минималистичных систем CRISPR/Cas с уменьшенными возможностями по адаптации или интерференции. Часто такие «урезанные» варианты CRISPR/Cas переносятся транспозонами и плазмидами. Так, у Tn7-подобных транспозонов имеется уменьшенный вариант системы I-F или I-B (рис. 4). Показано, что систему I-F транспозоны «подхватили» один раз, а вот приобретение ими системы I-B происходило дважды. В этих системах отсутствует адаптационный модуль и белок Cas3, необходимый для разрушения мишени, поэтому они неспособны ни к адаптации, ни к интерференции. Однако они содержат все компоненты, необходимые для процессинга пре-crРНК, поэтому теоретически могут образовывать crРНК и распознавать ДНК-мишень. Экспериментально эти системы еще не изучены, однако предполагают, что они могут способствовать вставке транспозонов за счет образования в сайтах-мишенях R-петель (участков ДНК, на которых цепи разъединены, при чем одна образует комплементарный гибрид с РНК, а вторая остается свободной) [3].
Еще один пример минималистичных систем CRISPR/Cas — системы IV типа класса 1. Как правило, они содержатся в плазмидах или профагах в геномах различных бактерий. За редкими исключениями, в системах IV типа нет адаптационного модуля, но есть гены cas5, cas7, cas8 и, в редких случаях, cas6 (рис. 4). Самих локусов CRISPR в системах IV типа, как правило, нет. Поскольку эти системы встречаются почти исключительно на плазмидах, они могут каким-то образом обеспечивать их мобильность и стабильность [3].
Систематический скрининг прокариотических геномов позволил найти вариант системы подтипа I-E, лишенный гена cas3 и потому неспособный разрушать мишень, однако, судя по входящим в его состав генам, он может быть задействован в сигнальных путях, в частности, приводящих к запуску программируемой клеточной гибели [3].
Заключение
Общая тема, то и дело возникающая при обсуждении эволюции и происхождения систем CRISPR/Cas, — это взаимодействие с мобильными генетическими элементами (рис. 4 и 5). Напомним, по меньшей мере четыре их группы внесли вклад в эволюцию систем CRISPR/Cas. В числе таких элементов:
- каспозоны, от которых произошел адаптационный модуль;
- интроны II группы, «подарившие» обратную транскриптазу некоторым системам III типа;
- IS-элементы семейства IS605 и близкие к ним, ставшие предками эффекторных модулей систем II и V типов;
- системы токсин—антитоксин, внесшие вклад в эволюцию белка Cas2.
Во всех этих случаях мобильные элементы встраивались по соседству с локусами CRISPR/Cas и постепенно вовлекались в работу защитных систем [3].
Еще один «тренд» в эволюции систем CRISPR/Cas — утрата тех или иных компонентов, необходимых для полноценного функционирования системы. Во многих случаях такие «урезанные» системы стали принимать участие в совершенно других процессах [3]. Что стало причиной такого «переключения» пока, опять же, остается неясным. Будем надеяться, что дальнейший прогресс в геномике прокариот позволит ответить на эти вопросы о системах CRISPR/Cas.
Литература
- Просто о сложном: CRISPR/Cas;
- CRISPR-системы: иммунизация прокариот;
- Eugene V. Koonin, Kira S. Makarova. (2019). Origins and evolution of CRISPR-Cas systems. Phil. Trans. R. Soc. B. 374, 20180087;
- Анти-CRISPR: ответ вирусов;
- Как CRISPR/Cas работает не по специальности;
- Мобильные генетические элементы прокариот: стратификация «общества» бродяжек и домоседов;
- Мода на ретро. Где встречается обратная транскрипция, и как она эволюционировала;
- Omar O. Abudayyeh, Jonathan S. Gootenberg, Silvana Konermann, Julia Joung, Ian M. Slaymaker, et. al.. (2016). C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353, aaf5573;
- Eugene V Koonin, Kira S Makarova, Feng Zhang. (2017). Diversity, classification and evolution of CRISPR-Cas systems. Current Opinion in Microbiology. 37, 67-78;
- Prarthana Mohanraju, Kira S. Makarova, Bernd Zetsche, Feng Zhang, Eugene V. Koonin, John van der Oost. (2016). Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science. 353, aad5147.