Биоинженеры научились получать ДНК-структуры, сборкой и разборкой которых можно управлять
14 мая 2015
Биоинженеры научились получать ДНК-структуры, сборкой и разборкой которых можно управлять
- 1231
- 0
- 5
«Эволюция» ДНК-роботов. Теперь их научили поднимать и опускать руки, распадаться на детали и собираться вновь. «Мир в ожидании Терминатора!» (тизер).
Рисунок в полном размере.
-
Автор
-
Редакторы
Немецкие ученые расширили возможности молекулярной инженерии, применив для сборки структур из ДНК стэкинг концов двойных спиралей. С помощью этих относительно слабых взаимодействий можно соединять отдельные блоки из ДНК, и, что важно, такие соединения будут обратимыми. Используя лабильные контакты для соединения отдельных блоков, можно управлять их сборкой и манипулировать формой конструкции.
Относительно недавно появилась область инженерии, внешне очень напоминающая забаву для интеллектуалов, — конструирование из молекул ДНК . Природные ДНК — это спирали, состоящие из двух налипших друг на друга нитей (цепочек), в последовательности нуклеотидов которых закодирована генетическая информация. Каждый нуклеотид одной нити имеет пару в другой, а «слипаются» они за счёт нековалентных водородных связей (парные нуклеотиды называются комплементарными) [1]. Если две цепочки ДНК комплементарны друг другу, то они свяжутся водородными связями и образуют спираль, которая стабилизируется еще и благодаря независящим от последовательности нуклеотидов гидрофобным взаимодействиям и стэкингу. В случае ДНК стэкинг представляет собой нековалентное взаимодействие (взаимопритяжение) ароматических колец в составе азотистых оснований соседних пар комплементарных нуклеотидов. Эти основания располагаются почти перпендикулярно оси спирали и параллельно друг другу, образуя подобие стопки монет, — получается, что условно нижняя «монетка» взаимодействует с условно верхней и так далее (рис. 1).
Помимо использования «живых» молекул в конструировании, активно развиваются различные небиологические инженерные технологии, инспирированные разумностью устройства природных систем. В частности — механизмами самосборки, укладки и функционирования ДНК. Об упругой архитектуре и бионическом конструкторе талантливого российского изобретателя читайте в статье «Бионический конструктор Эльпюль» [2]. — Ред.
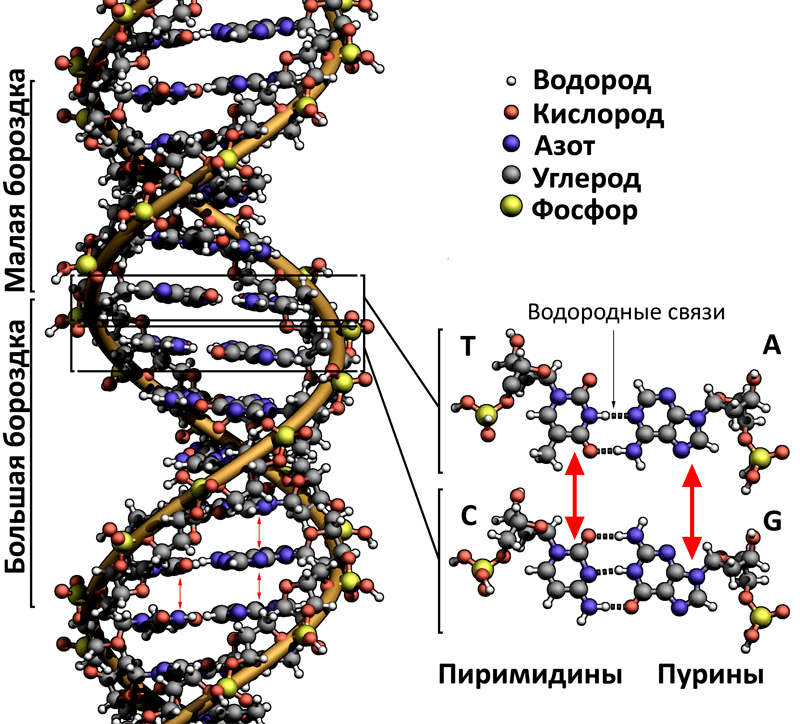
Рисунок 1. Структура спирали двухцепочечной ДНК. Красными стрелками показаны стэкинг-взаимодействия между азотистыми основаниями соседних нуклеотидов.
Для конструирования фигур из ДНК используют как искусственно синтезированную длинную молекулу с произвольной последовательностью, так и нуклеиновые кислоты из живых организмов. Раздобыв такую длинную цепочку, можно синтезировать короткие «ДНК-скрепки» , которые будут соединять отдельные участки основной цепи между собой, придавая ей определенную форму . Этот метод называется ДНК-оригами. Однако он имеет ряд недостатков — структуры на основе одной длинной молекулы собираются долго и с низким выходом.
Разумеется, синтез ДНК обычно обслуживает другие цели — так сказать, более приземленные (например, некоторые виды генно-инженерных работ), оригами же — новое и довольно экзотическое назначение синтеза. Тем не менее задачи ДНК-оригами были серьезными изначально — помощь при проведении рентгеновской кристаллографии. Со временем спектр потенциальных применений самостоятельно собирающихся по заранее разработанной биоинженером схеме ДНК-наноконструкций существенно расширился. Например, из них можно создавать рабочую часть сенсорных биочипов, способных диагностировать инфекционные, генетические и онкологические заболевания по единичным молекулам: «ДНК-оригами: путь от гравюры до нанороботов длиной в 30 лет» [3]. У технологии есть потенциал зайти еще дальше — покуситься на «умную» терапию: в прошлом году сообщили о создании ДНК-нанороботов, способных выполнять несложные логические операции прямо в организме таракана: «Голактеко опасносте: ДНК-роботы в живом организме» [4]. До полноценного ДНК-компьютера еще далеко, но ведь для решения простых задач типа обнаружения в организме заданного набора молекул и высвобождения-невысвобождения по результатам диагностики лекарства (антисенс-олигонуклеотида или обычного «химиката») и не требуется высокая производительность — Ред.

Рисунок 2. Созданная из ДНК модель шаттла (справа) и ее аналог из конструктора «Лего».
Другой метод конструирования из ДНК — это построение модульных структур из более коротких молекул. Хорошо продумав форму отдельного модуля, можно собирать из ДНК довольно сложные трехмерные конструкции с полостями и выступами (рис. 2), например, контейнеры для адресной доставки лекарств [5], [6].
Серьезное ограничение, которое мешало развивать ДНК-конструирование — это статичность таких структур. Подавляющее большинство из них можно было разобрать только один раз (попросту говоря, сломать), и только немногие выдерживали некоторое количество циклов сборки-разборки, в каждом из которых сложная конструкция немного портилась. Поэтому до сих пор конструирование из ДНК было похоже на склеивание моделей, а не на сборку конструктора. Модели могли получаться забавными, но практической пользы от них было мало. Со статичными структурами и близко нельзя было подойти к природному богатству молекулярного арсенала — например, делать комплексы, которые меняют форму, разбираются и собираются при изменении условий без вреда для компонентов. В общем, биоинженерам хотелось добавить жизни монолитным ДНК-конструкциям. И вот недавно немецкие ученые придумали, как это можно сделать [7].
Исследователи решили, что нужно расширять репертуар свойств ДНК, которые используются при конструировании. Взаимодействие комплементарных нуклеотидов — это не единственное, что может влиять на форму ДНК. Помимо сил, которые держат вместе нити двойной спирали, между нуклеотидами возникают и другие, более слабые взаимодействия. Особенно активно природа применяет эти способы формирования структуры в отношении других нуклеиновых кислот — РНК. В отличие от ДНК, РНК — это одна цепь нуклеотидов, которая не обязана поддерживать форму двойной спирали. Однако молекулы РНК иногда складываются в причудливые формы, в образовании которых участвуют не только водородные связи, но и более слабые взаимодействия. Одно из них — описанный выше стэкинг. Благодаря такому взаимодействию могут притягиваться друг к другу торцы двойных спиралей ДНК. Немецкие ученые решили применить его, чтобы соединять отдельные блоки своего ДНК-конструктора. Стэкинг оснований ДНК чувствителен к условиям среды, поэтому собранные уже известными методами прочные блоки можно было бы соединять и разъединять этими более лабильными связями, лишь модулируя условия.
При использовании стэкинга для соединения отдельных блоков из ДНК нужно проектировать их таким образом, чтобы в месте их взаимодействия располагались торцы двойных спиралей. Можно создавать блоки с разными структурами из выступающих спиралей и выемок на торцах. Тогда только те детали, у которых такие выступы и углубления совпадают, будут соединяться между собой — как в пазле (рис. 3).
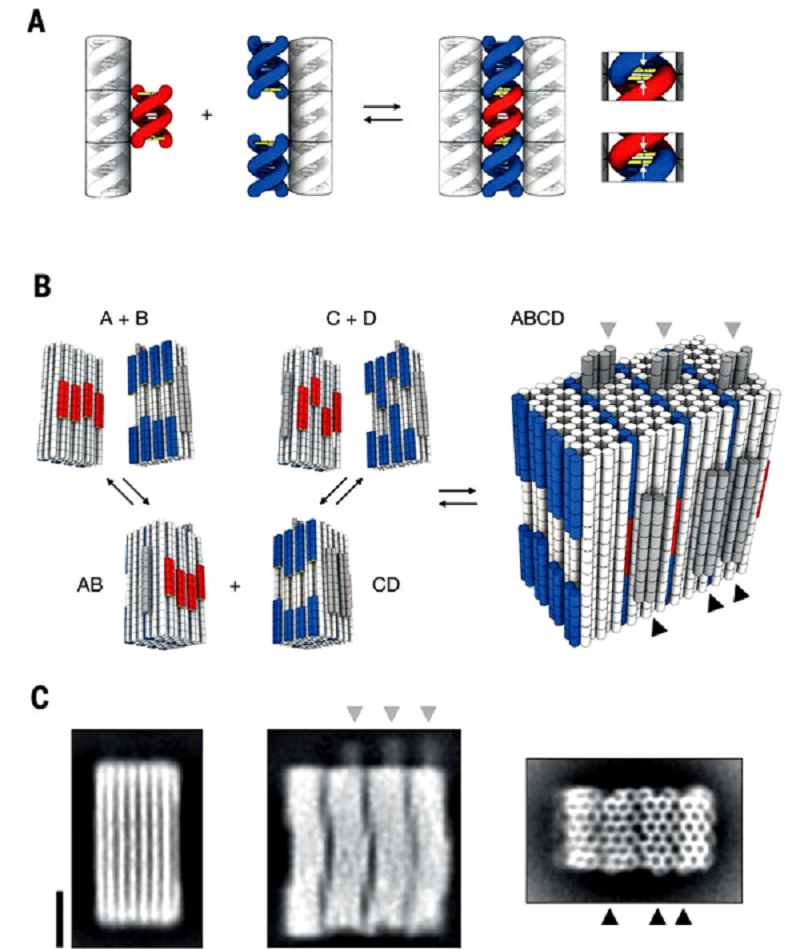
Рисунок 3. Стэкинг-опосредованное конструирование синтетических ДНК-объектов. A — Принцип соединения ДНК-блоков с помощью стэкинга (слабого притяжения) торцов двойных спиралей. B — Более сложные структуры, собранные из деталей с подходящими интерфейсами (главное — их можно собирать-разбирать многократно). C — Микрофотографии деталей. Применяли просвечивающую электронную микроскопию (ПЭМ), масштабная линейка — 20 нм.
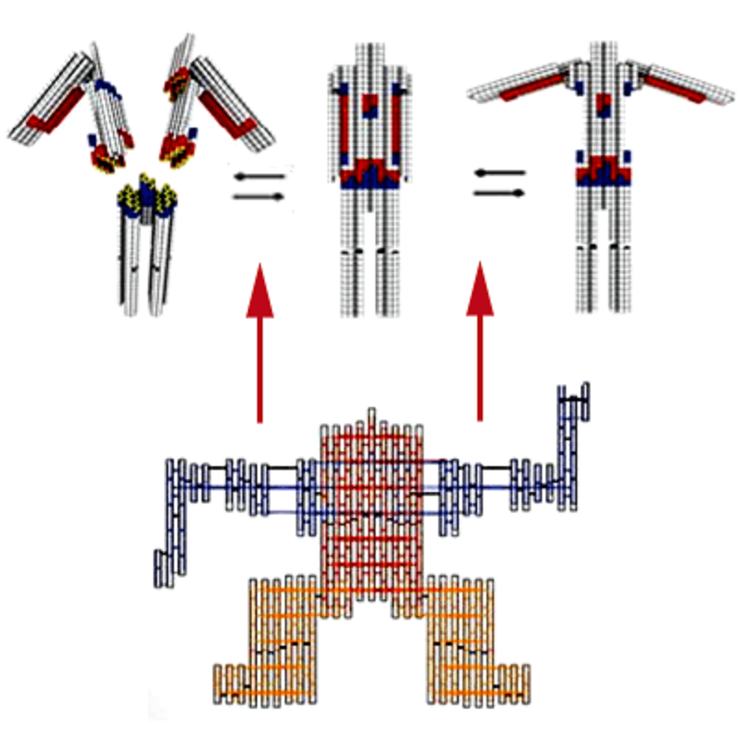
Можно встраивать такие подходящие друг к другу интерфейсы и в одну конструкцию — тогда ее части можно будет соединять и разъединять. Например, ученым удалось получить фигуру, похожую на ножницы, которые могли раскрываться и закрываться. Собрали даже похожего на человека «робота», который поднимал и опускал руки (рис. 4).
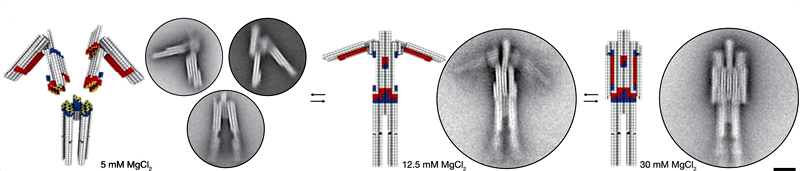
Рисунок 4. «Робот» из ДНК, поднимающий и опускающий руки. Концентрации катионов магния, необходимые для таких манипуляций, показаны под соответствующими рисунками. Масштабная линейка на ПЭМ-изображениях — 25 нм.
Управлять соединением и разъединением блоков из ДНК можно, меняя температуру или концентрацию катионов в растворе. Кроме того, ученые попробовали применять специальные «замки» — цепочки, которые мешали взаимодействиям интерфейсов деталей. «Замки» можно было открыть, добавляя комплементарные им цепочки нуклеотидов. Все эти методы позволяли обратимо менять форму конструкций. Ученые проверили свои структуры на прочность, заставив их циклически менять состояние в течение четырех суток. Молекулярные конструкции выдержали такое испытание.
Теперь, когда мы научились получать из ДНК модули, сборкой которых можно управлять, пришло время поразмыслить над конструированием из ДНК аналогов элементов клеточного скелета (актиновых и тубулиновых филаментов), а также заняться сборкой всяческих открывающихся контейнеров для лекарств. А уж возможности для сборки из ДНК миниатюрных боевых роботов и прочих забавных вещиц just for fun у нас теперь практически безграничные...
Литература
- Роль слабых взаимодействий в биополимерах;
- Бионический конструктор Эльпюль;
- ДНК-оригами: путь от гравюры до нанороботов длиной в 30 лет;
- Голактеко опасносте: ДНК-роботы в живом организме;
- Yonggang Ke, Luvena L. Ong, William M. Shih, Peng Yin. (2012). Three-Dimensional Structures Self-Assembled from DNA Bricks. Science. 338, 1177-1183;
- Элементы: «Наноструктуры из ДНК можно собирать по принципу конструктора “Лего”»;
- Thomas Gerling, Klaus F. Wagenbauer, Andrea M. Neuner, Hendrik Dietz. (2015). Dynamic DNA devices and assemblies formed by shape-complementary, non–base pairing 3D components. Science. 347, 1446-1452.