
CRISPR-системы: иммунизация прокариот
14 ноября 2014
CRISPR-системы: иммунизация прокариот
- 12790
- 6
- 14
Деградация чужеродной ДНК эффекторными комплексами CRISPR-систем.
-
Авторы
-
Редакторы
Статья на конкурс «био/мол/текст»: CRISPR — это система специфического иммунитета прокариот, характерная как для бактерий, так и для архей. CRISPR-структуры впервые были описаны почти 30 лет назад, однако их функция долгое время оставалась загадкой. CRISPR-системы состоят из геномных кассет, в которые записывается информация о вирусных или плазмидных инвазиях, и Cas-белков, обеспечивающих молекулярный механизм иммунитета. В ответ на инфекцию клетка с CRISPR вырезает из чужеродного генома небольшой фрагмент и встраивает его в кассету. Благодаря такому механизму роста, CRISPR-системы являются уникальным примером эволюции «по Ламарку». Высокоэффективное узнавание ДНК, лежащее в основе работы CRISPR, оказалось привлекательно и для практического использования, и сейчас CRISPR-системы служат для точных манипуляций с самыми различными геномами, в том числе и с геномом человека.
Конкурс «био/мол/текст»-2014
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2014 в номинации «Биоинформатика и молекулярная эволюция».
Главный спонсор конкурса — дальновидная компания Генотек.
Конкурс поддержан ОАО «РВК».
Спонсором номинации «Биоинформатика» является Институт биоинформатики.
Спонсором приза зрительских симпатий выступила фирма Helicon.
Свой приз также вручает Фонд поддержки передовых биотехнологий.
Мир микробов жесток. Чтобы выжить, бактерии должны размножаться как можно быстрее. Основные ограничения на скорость деления клетки накладывает размер генома: чем он компактнее, тем быстрее деление. У прокариот он действительно относительно невелик — на несколько порядков меньше, чем у эукариот. Средний размер генома бактерии составляет 3 млн. пар оснований, и все функции, необходимые и достаточные для их успешной и эффективной жизни, кодируются 3 тыс. генов. Избыточность генетического материала для бактерий непозволительна, однако их геномы помимо уникальных последовательностей содержат и небольшое число повторяющихся. Одна из самых загадочных структур — это короткие прямые повторы, регулярно расположенные группами. Между повторами располагаются уникальные последовательности примерно той же длины — спейсеры. Такая структура была названа CRISPR (от англ. Clustered Regularly Interspaced Short Palindromic Repeats — короткие палиндромные повторы, регулярно расположенные группами). Число повторяющихся единиц (повтор + спейсер) может достигать нескольких сотен, а CRISPR-локусы часто встречаются в стремящихся к минимализму геномах бактерий. Почему бактерии терпят такую непозволительную избыточность генетического материала? Зачем нужны повторы и вся CRISPR-структура?
Портрет CRISPR
Впервые повторяющиеся последовательности равной длины, перемежающиеся уникальными участками, были описаны в 1987 г. в геноме основного модельного организма микробиологии и молекулярной биологии — кишечной палочки (Escherichia coli) [1]. Позже похожие структуры нашли и у многих других прокариот: как у филогенетически очень далеких друг от друга видов бактерий, так и у архей. Среди них, например, архея Haloferax mediterranei, живущая в очень соленой воде Мертвого и Средиземного морей; цианобактерии рода Anabaena; патогенные бактерии Mycobacterium tuberculosis — возбудитель туберкулеза; Streptococcus pyogenes, вызывающая различные заболевания — от ангины до воспалений кожи и мышц у человека и других млекопитающих [2–5]. CRISPR-структуры были найдены у 45% всех известных бактерий и 85% архей [6]. В геномах эукариот и вирусов ничего подобного обнаружено не было. Со временем стало понятно, что CRISPR — это сложная многокомпонентная система, а повторы — только малая часть «айсберга». Из чего же состоит этот айсберг?
В первую очередь — из кассет, содержащих повторы и спейсеры. Число составных звеньев в CRISPR-кассете может варьировать от двух до нескольких сотен. Пока рекордсменом в этом признана морская протеобактерия Haliangium ochraceum с 587 спейсерами в кассете [6]. Часто в геномах встречается более одной кассеты. Больше всего их обнаружено в ДНК метаногенной термофильной археи Methanocaldococcus jannaschii — 18 кассет занимают почти 1% ее генома.
СRISPR-структуры чаще располагаются на основной хромосоме (нуклеоиде), но иногда входят в состав плазмид (маленьких кольцевых, автономно реплицирующихся молекул ДНК). Повторы в кассете, как правило, строго совпадают между собой по длине и последовательности нуклеотидов. Лишь последний повтор иногда отличается от канонической последовательности остальных, да и то лишь в нескольких концевых позициях.
Для повторов характерна частичная диадная симметрия — участки последовательности в начале и в конце повтора обратно комплементарны. Благодаря этому концы палиндромных повторов могут взаимодействовать и образовывать устойчивые вторичные структуры, прежде всего шпильки.
Спейсеры в кассете часто равны по длине, но отличаются по последовательности нуклеотидов. Наборы спейсеров у штаммов одного вида тоже могут сильно различаться. Благодаря высокой вариабельности, задолго до того, как выяснилась роль CRISPR в клетках прокариот, их стали использовать для быстрого типирования штаммов известных бактерий: возбудителей чумы (Yersinia pestis) [7], дифтерии (Corynebacterium diphtheriae) [8], гастроэнтерита (Campylobacter jejuni) [9], а также уже упоминавшихся Mycobacterium tuberculosis [2] и Streptococcus pyogenes [4].
Чередующиеся повторы и спейсеры образуют «тело» CRISPR-кассеты. «Головой» служит лидерная последовательность; она задает направление транскрипции кассеты (рис. 1). Средняя длина лидерной последовательности около 400 пар нуклеотидов; она не содержит открытых рамок считывания, т.е. не кодирует белки.
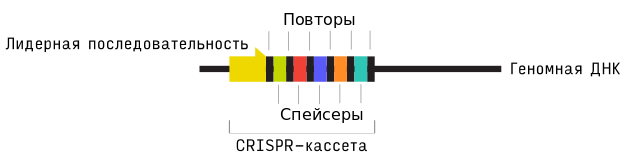
Рисунок 1. Структура CRISPR-кассеты.
Лидерные последовательности отличаются высоким содержание аденина (A) и тимина (T). Уотсон-криковская пара A-Т удерживается только двумя водородными связями, а G-С — тремя, поэтому двухцепочечную ДНК в AT-богатых участках легче расплести и превратить в одноцепочечную. В AT-богатых регионах малая бороздка ДНК более узкая — такая топология служит характерным местом посадки для многих белков, взаимодействующих с ДНК. AT-богатые участки часто встречаются в регуляторных последовательностях и точках начала репликации. Предполагают, что лидерная последовательность регулирует транскрипцию CRISPR-кассеты, а значит и функционирование всей системы [10]. Для некоторых организмов промоторы в лидерной последовательности обнаружены экспериментально.
Спейсеры, повторы, лидерная последовательность: таков обобщенный портрет геномной CRISPR-структуры (рис. 1). Но можно ли, глядя на него, предположить какую роль эта структура играет в клетке? С момента открытия CRISPR-кассет возникло несколько гипотез о её функции.
Гипотетические функции
Гипотеза 1: сегрегация хромосом. Для выживания прокариотам важно делиться не только быстро, но и качественно — так, чтобы генетический материал родительской клетки правильно удваивался и равномерно распределялся между потомками. Распределение хромосом между дочерними клетками контролируют системы сегрегации. У эукариот правильное расхождение хромосом обеспечивают центромеры — участки с особенной последовательностью и структурой, соединяющие сестринские хроматиды. Прокариоты значительно проще по организации целого ряда клеточных процессов; в частности, центромер у них нет. Однако у бактериальных плазмид P и F1 правильное расхождение двух копий плазмиды после репликации контролирует система par [11]. Важный элемент par-системы — итероны, короткие тандемные повторы, собранные в область под названием parC. С parC связывается белок parR, а тот, в свою очередь, с parM и другими белками системы. То есть, parC-область — это что-то вроде центра кипения, на котором, как пузырьки в чайнике, собираются клеточные белки системы сегрегации. Важной особенностью этой области считаются тандемно расположенные повторы, что очень напоминает CRISPR-кассеты.
В геномах двух видов архей — Haloferax volcanii и H. mediterranei — содержится несколько CRISPR-кассет общей протяженностью до 1600 пар нуклеотидов [6]. Что произойдет, если удалить их, или, наоборот, удвоить? Оказалось, что при введении дополнительных копий CRISPR-кассет у H.volcanii часто наблюдались отклонения в распределении генетического материала при делении клеток и снижалась их жизнеспособность [3]. CRISPR-кассеты H. volcanii и H. mediterranei находятся в мегаплазмидах и собственно хромосомной ДНК, т.е. наиболее крупных репликонах (молекулах ДНК, копирование которых начинается с одной точки). О чем это может говорить? Если CRISPR действительно работает системой сегрегации, то в основном и отвечает за правильное распределение по дочерним клеткам важных генов крупных репликонов, а расхождение мелких плазмид с несущественными генами происходит более или менее случайно. Такой гипотетический сценарий соответствует тому, что мы сейчас знаем и думаем (или думаем, что знаем) о системах сегрегации репликонов у прокариот. Эти наблюдения свидетельствуют в пользу участия CRISPR в расхождении хромосом.
Гипотеза 2: рекомбинация по повторам. Вторая гипотеза тоже опирается, в основном, на повторы CRISPR-системы, но учитывает не их тандемное расположение, а идентичность. Часто повторы служат горячими точками для геномных перестроек. Они происходят благодаря рекомбинации, гомологичной или гетерологичной. В некотором смысле участки повторов в двухцепочечной молекуле ДНК напоминают застежки-липучки: одна цепь играет роль крючков, а другая — петелек. В случае CRISPR-кассеты крючки одного повтора могут зацепиться за петельки другого, при этом образуются структуры, напоминающие крест — структуры Холлидея. Эти cтруктуры нельзя распутать, а можно только разрезать, в результате чего формируются рекомбинантные молекулы ДНК. В ходе рекомбинации часто происходят потери, перевороты и удвоения протяженных участков генома. Такие крупные перестройки не всегда вредны: иногда они служат источником необходимой изменчивости для эволюции и адаптации к меняющимся условиям окружающей среды [12]. Тандемно расположенные повторы CRISPR-кассет могут служить мишенями для рекомбинации в геномах бактерий и архей. Часто наблюдаемые делеции целых групп последовательно идущих спейсеров из середины кассеты служат этому примером.
Гипотеза 3: система репарации ДНК. Прокариотический геном относительно невелик, но очень структурирован и чем-то напоминает книжный шкаф аккуратного человека, где книги по астрономии стоят на одной полке, а по садоводству — на другой. Точно так же гены прокариот, отвечающие за одну биологическую функцию (например, защиту от теплового шока), часто расположены рядом. Это удобно для клетки. Во-первых, такое соседство гарантирует, что белки, которые совместно выполняют некоторую функцию, сразу после трансляции будут находиться поблизости в клетке. Во-вторых, это облегчает регуляцию экспрессии функционально связанных генов. Кроме того, это позволяет некоторым функциональным системам распространяться в процессе горизонтального переноса. Благодаря генной колокализации о функции гена зачастую можно судить по его ближайшим соседям. Такой подход сейчас используется для аннотации геномов, т.е. для установления функций генов в геноме.
Давайте по этому принципу попробуем проанализировать функции генов, лежащих рядом с CRISPR-кассетами. У организмов с кассетами в непосредственной близости от CRISPR-локуса располагаются гены, которых нет у организмов без CRISPR-кассет, — это cas-гены (от англ. CRISPR-associated — сцепленные с CRISPR). Филогенетическая сцепленность и близость расположения свидетельствуют в пользу функциональной взаимосвязи cas-генов и CRISPR-кассет.
Набор cas-генов у разных организмов сильно различается, однако у всех организмов с CRISPR есть ген cas1, его можно считать универсальным маркером CRISPR-систем. Какова функция белков, кодируемых cas-генами? Сas1 имеет выраженный положительный заряд и может электростатически взаимодействовать с отрицательно заряженным сахаро-фосфатным остовом ДНК. По свойствам Cas1 напоминает ДНК-связывающие белки, например, гистоны эукариот. Этот белок нарезает одноцепочечную и двухцепочечную ДНК на фрагменты длиной около 80 нуклеотидов [13]. Cas2 расщепляет молекулы РНК [14]. Cas3 относится к хеликазам, которые расплетают цепи двухцепочечной ДНК. Такие ферменты участвуют в репарации (починке) ДНК, регуляции транскрипции генов, сегрегации хромосом и перестройках хроматина. Cas4 — экзонуклеаза, отщепляющая концевые нуклеотиды молекул ДНК и РНК. Экзонуклеазы — компоненты систем рекомбинации и часто входят в состав ДНК-полимеразных комплексов [15].
Определение функций cas-генов позволило только понять, что CRISPR как-то связаны с перестройками ДНК. Предположили, что CRISPR может работать системой репарации, т.е. чинить повреждения ДНК, у термофильных бактерий и архей, живущих в экстремальных условиях — при температурах более 100 °С [16]. Термофилы поразительно устойчивы к действию факторов, повреждающих ДНК: ионизирующему и ультрафиолетовому излучениям, химическим мутагенам. Термофилы должны обладать очень эффективной системой репарации ДНК, однако ничего подобного у них не найдено. На роль такой системы была выдвинута CRISPR со всеми Cas-белками. В пользу гипотезы, в первую очередь, говорит сходство некоторых Сas-белков с эндонуклеазами RecB подсемейства RecBCD — основной системе рекомбинационной репарации E.coli. У других Сas-белков обнаружены домены, похожие на каталитические домены ДНК- и РНК-полимераз, а также хеликаз, которые тоже пригодились бы для репарации ДНК. Однако остается еще ряд белков, совершенно точно связанных с CRISPR, функция которых пока неизвестна. Они были названы RAMP (от англ. Repeat-Associated Mysterious Protein — загадочный белок, ассоциированный с повторами). Возможно, эти загадочные белки могут служить дополнительными регуляторными ДНК-связывающими субъединицами в составе репарационных комплексов, или из RAMP построены «скользящие зажимы» для ДНК-полимераз. Таким образом, по набору функций CRISPR/cas-система, казалось бы, удовлетворяет минимальным требованиям для системы репарации ДНК.
Гипотеза 4: противовирусный иммунитет. В 2005 г. сразу несколько независимых исследовательских групп проанализировали спейсеры уже известных кассет с целью понять их происхождение [17–19]. Они сравнили спейсеры со всеми известными последовательностями ДНК (доступными в базе данных GenBank — основном хранилище такой информации). Оказалось, что спейсеры CRISPR-кассет у Streptococcus thermophilus и S. vestibularis часто совпадают с участками генов бактериофагов (вирусов бактерий), специфичных к стрепококкам, или плазмид S. thermophilus и Lactococcus lactis [18]. Некоторые спейсеры оказались идентичны последовательностям бактериальных геномов: такие спейсеры часто попадают на последовательности профагов — геномов умеренных бактериофагов, интегрированных в ДНК бактерий. Складывалось впечатление, что спейсеры — это что-то вроде обломков чужеродных генетических элементов, которые вторглись в прокариотическую клетку на каком-то этапе ее эволюционной истории.
Вскоре и у многих других архей и бактерий нашли спейсеры внехромосомного происхождения. Участки вирусных и плазмидных геномов, совпадающие со спейсерами, назвали протоспейсерами. Продукты генов, содержащих протоспейсеры, участвуют в репликации ДНК, сборке вирусных частиц, защите ДНК от рестрикции (разрезания ДНК эндонуклеазами), переводе вирусов в спящее (интегрированное в геном хозяина) состояние и обратной их активации, сегрегации репликонов. Эти функции важны вирусам и плазмидам для проникновения в клетки прокариот, размножения и дальнейшего распространения.
Установление происхождения спейсеров принесло больше вопросов, чем ответов: спейсеры — это просто кусочки вирусных геномов, застрявшие в геномах прокариот, или эта структура несет какую-то функцию? Если да, то в чем заключается эта функция и зачем нужны повторы, лидерная последовательность и cas-белки? Помимо происхождения спейсеров к 2005 г. стало известно, что: устойчивость к фаговым инфекциям (у Streptococcus thermophilus) зависит от числа спейсеров в CRISPR-кассете [18]; механизм появления CRISPR-кассет тесно связан с cas-генами [20]; CRISPR-кассеты транскрибируются (у Archeoglobus fulgidus и Sulfolobus sulfoctaricиus) c образованием малых РНК [21].
Все эти факты навели на мысль о том, что CRISPR защищает прокариот от вирусов и плазмид [18]. Согласно такой гипотезе, включение участков геномов мобильных элементов в виде спейсеров — не что иное, как иммунная память, которая может передаваться по наследству. Такая система должна давать своим хозяевам существенный выигрыш в приспособленности, что могло бы объяснять широкое распространение CRISPR-систем в мире прокариот.
Так какая же из гипотез о роли CRISPR оказалась верна?
Функция CRISPR
Люди научились одомашнивать не только коров и собак, но и бактерий. Streptococcus thermophilus — одна из ключевых одомашненных микробных культур — используется для получения сыров, йогуртов и других молочнокислых продуктов. Но бактерии, даже самые полезные, тоже болеют. Молочная промышленность постоянно обеспокоена подверженностью S. thermophilus различным вирусным инфекциям. Неслучайно именно на этом модельном организме проведены ключевые эксперименты по уточнению функции CRISPR-систем.
Если заразить живую культуру S. thermophilus бактериофагами, то большинство бактерий погибнет, но очень небольшая часть выживет. Чем же выжившие отличаются от изначальной культуры? Оказалось, что их геном стал длиннее на 0.01% за счет того, что в CRISPR-кассету добавились 1–4 новых спейсера [22]. При повторном заражении этой культуры теми же бактериофагами все клоны выживали. Как будто, переболев вирусной инфекцией, бактерия стала немного опытнее и записала себе в «медицинскую карту» что-то важное об этом вирусе, и такая инфекция ей теперь не страшна. Действительно, новые спейсеры оказались комплементарны участкам генома заразившего ее вируса. Получается, что CRISPR-кассета — это история болезней бактерии, а спейсеры соответствуют записям об отдельных инфекциях. Если теперь искусственно вырезать из вирусного генома небольшие фрагменты и вставить их в виде новых спейсеров, то клетка окажется невосприимчива к исходному вирусу, даже если никогда раньше с ним не встречалась. Наблюдение оказалось верным и в обратную сторону: при удалении из CRISPR-кассет спейсеров, подобных последовательностям данного бактриофага, исчезала и устойчивость к нему [22]. Эта серия простых и изящных экспериментов на S. thermophilus полностью подтвердила гипотезу об иммунной функции CRISPR-систем. Благодаря полярной манере включения новых спейсеров, CRISPR-кассеты можно буквально читать как историю взаимоотношений прокариот и их вирусов на определённом эволюционном промежутке. CRISPR — это не только иммунитет, но ещё и память о недавних победах прокариотической клетки.
Что же происходит дальше с выжившей бактерией и вновь встроенным спейсером? По своему происхождению он полностью совпадает с участком фагового генома (протоспейсером) и обеспечивает полную защиту. Если клеток с таким спейсером много, то вирус вряд ли преуспеет в заражении большого числа бактерий и не сможет размножиться в массовых количествах, а, значит, вымрет. Не случись повторного заражения тем же вирусом, спейсер в кассете долго не задержится: со временем он будет утерян в результате рекомбинации между повторами кассеты.
Заметим, что вирус тоже не бездействует — он умеет «убегать» от CRISPR-иммунитета, накапливая точечные мутации в последовательности протоспейсера. Интересно, что единичные замены в некоторых из них сразу лишают бактерию устойчивости к вирусу, замены в других лишь несколько ослабляют ее. В любом случае, когда протоспейсер теряет сходство с бактериальным спейсером, как злоумышленник, сделавший серию пластических операций, становится непохож на себя, пропадает и устойчивость. Теперь клетку при заражения спасет только встраивание нового спейсера.
Механизм работы
Какие молекулярные механизмы обеспечивают CRISPR-опосредованный иммунитет? Правильнее говорить о работе CRISPR/Cas-системы. Ведь только за счет сложных белковых машин из Cas-белков CRISPR-кассетам и удается реализовать защитную функцию. CRISPR/Cas-система работает в три такта: 1) адаптация, или приобретение новых спейсеров; 2) созревание эффекторных комплексов; 3) иммунный ответ, то есть разрушение чужеродной ДНК.
Адаптация. Когда клетку с CRISPR-системой заражает вирус, из его генома вырезается небольшой фрагмент и встраивается в кассету в качестве спейсера [22]. При повторных заражениях одним и тем же агентом включение первого спейсера, особенно если он из-за случайных мутаций не обеспечивает полной защиты, ускоряет приобретение дополнительных спейсеров против того же самого чужеродного репликона. Это явление назвали праймированием [23]. Несколько спейсеров повышают шансы справиться с инфицирующим агентом даже при неполном совпадении спейсеров и протоспейсеров. Лидерная последовательность — это «точка роста» CRISPR-кассеты. С ней взаимодействуют белковые машины, отвечающие за включение новых спейсеров. Без лидерной последовательности кассеты не могут расти [24] (рис. 2).
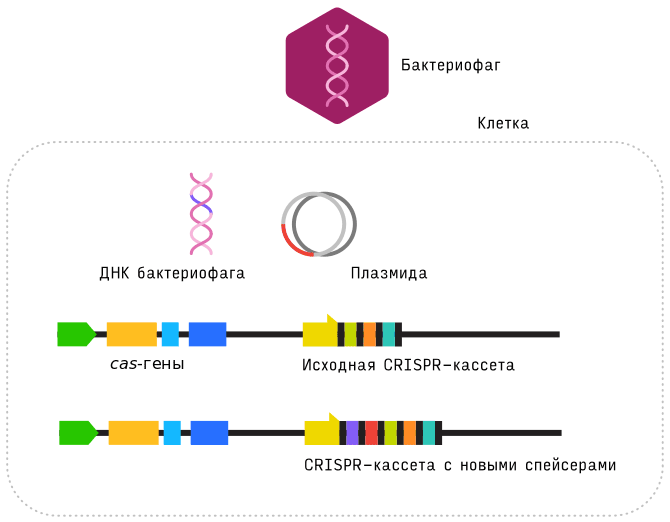
Рисунок 2. Включение новых спейсеров в кассету.
Привилегированных вирусных и плазмидных генов, служащих источником протоспейсеров, нет, однако это не совсем случайные последовательности. В вирусных геномах рядом с протоспейсерами находятся короткие, обычно длиной в два-три нуклеотида, PAM-мотивы (от. англ. protospacer adjacent motif) [25]. Это участки связывания белков, важных для выщепления протоспейсера и своеобразные подсказки для CRISPR-системы, что найденный спейсером участок относится к чужеродной ДНК.
Ключевые игроки на стадии приобретения новых спейсеров — белки Cas1 и Cas2 [26]. В некоторых случаях они даже слиты для удобства в один белок. Такая белковая машина работает последовательно: сканирует чужеродную ДНК в поисках PAM-последовательности; вырезает протоспейсер; распознает лидерную последовательность и вставляет новый спейсер с дополнительной копией повтора рядом с ней (рис. 2).
Созревание эффекторных комплексов. Спейсеры — своего рода пассивная иммунная память. Для защиты от интервентов нужно перевести ее в активный иммунный ответ. Это тоже происходит в несколько стадий. Сначала СRISPR-кассета экспрессируется: она служит матрицей для синтеза длинной молекулы первичного РНК-предшественника. Экспрессия запускается благодаря регуляторной области в составе лидерной последовательности. Одновременно начинают синтезироваться Сas-белки. Из них строится скелет активного эффекторного комплекса. У кишечной палочки такой комплекс, названный Cascade [27], состоит из молекул пяти белков и по форме напоминает морского конька (рис. 3).
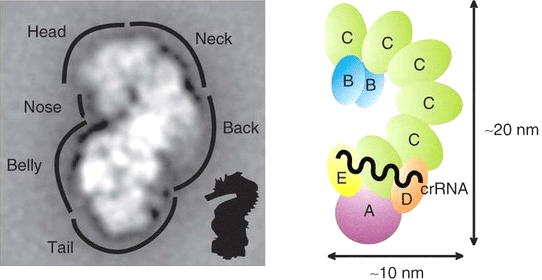
Рисунок 3. Структура Cascade-комплекса.
Комплекс из Cas-белков нарезает длинный первичный РНК-предшественник на короткие РНК (CRISPR-РНК или, кратко, crРНК), соответствующие последовательности одного спейсера, обрамленной неравными по длине участками повторов [27] (рис. 4). Субъединицы белкового комплекса расположены в пространстве так, что между ними образуется спиральная бороздка, в которую вкладывается crРНК. Cas-белки с намотанной на них crРНК составляют готовый к сражениям эффекторный комплекс.

Рисунок 4. Созревание эффекторных комплексов.
Иммунный ответ. Правильно сориентированная сrРНК в составе комплекса может комплементарно взаимодействовать с протоспейсером в вирусной или плазмидной ДНК. Только этого мало: нужно не просто узнать чужеродную ДНК, но и уничтожить ее. У E. coli за это отвечает эндонуклеаза Cas3. Она может резать как одноцепочечную, так и двухцепочечную ДНК. Cas3 разрезает ДНК очень эффективно, но совсем не специфично. Эффекторные Cas-crРНК-комплексы, напротив, очень специфично узнают последовательности, комплементарные спейсеру, изменяют свою конформацию и только после этого рекрутируют и правильным образом усаживают на ДНК белок Cas3 [29]. Особенно важны для такого узнавания семь нуклеотидов на границе протоспейсера и PAM-мотива — якорный участок. Если между ним и сrРНК есть различия хотя бы в один нуклеотид, то CRISPR-иммунитет не сработает. Единичные отличия crРНК от протоспейсера вне этой области допускаются и уже не так драматически влияют на успех CRISPR-иммунитета. По мере накопления одноцепочечных разрывов в ДНК-мишени, сродство эффекторного комплекса к ней снижается: он отсоединяется и распадается на отдельные субъединицы. Cas3 вносит большое число разрывов, начиная с места посадки и далее в более или менее случайной манере, кроша ДНК-интервента (рис. 5). Возможно, короткие фрагменты ДНК, образованные в результате работы Cas3, служат заготовками для новых спейсеров. Такая преемственность между циклами работы CRISPR/Cas-систем могла бы объяснять эффект праймирования [23]. Описанный молекулярный сценарий позволяет CRISPR-системам нейтрализовать вирусные инфекции, предотвращать трансформацию плазмидами, нарушать процесс лизогенизации (перехода вирусов в покоящееся состояние за счет встраивания своего генома в геном прокариота-хозяина), а также препятствовать индукции провирусов (переходу покоящихся вирусов, интегрированных в геном, в активное состояние). Изначально считалось, что CRISPR-системы уничтожают только ДНК. Однако позднее открыли архейные CRISPR/Cas-системы, эффективные против вирусов с РНК-геномами [30].
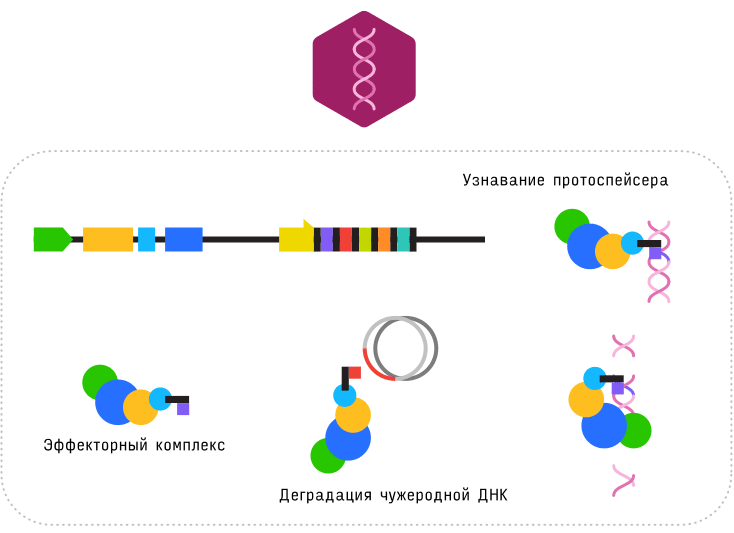
Рисунок 5. Деградация чужеродной ДНК эффекторными комплексами CRISPR-систем.
Ошибки CRISPR. Живое ошибается. Время от времени полимеразы путают нуклеотиды, эндонуклеазы разрезают неправильные последовательности, а рибосомы неверно узнают антикодоны тРНК и включают неправильные аминокислоты в белки. Ошибаются и CRISPR-системы.
В кассетах некоторых организмов присутствуют спейсеры, совпадающие с участками собственного генома клетки; на первый взгляд, это страшно. Такие спейсеры заставляют CRISPR работать по аутоиммунному сценарию, т.е. разрушать собственную ДНК.
Автоспейсеры — это не такое уж редкое явление. У 18% организмов, имеющих CRISPR-системы, есть по меньшей мере одна кассета хотя бы с одним таким спейсером-предателем [31]. Автоспейсеры возникают на стадии адаптации из-за ошибок работы Cas1 и Cas2. Часто эти спейсеры располагаются в начале кассеты, а значит, появились недавно. Судя по всему, автоспейсеры в активных CRISPR-кассетах живут очень недолго: либо они успевают оказать негативное воздействие на клетку, либо еще до этого удаляются из кассеты.
При попадании в кассету автоспейсеров, нужно немедленно инактивировать CRISPR-систему. У Lactobacillus acidophilus обнаружен автоспейсер к гену 16S рРНК, а это одна из трех ключевых рибосомальных РНК, формирующих структуру рибосомы [31]. Такой спейсер в составе активной кассеты означает немедленную гибель клетки. Однако, хотя кассета в клетке есть, клетка остается в живых. В чем здесь секрет? У L. acidophilus практически полностью отсутствуют cas-гены, т.е. CRISPR-система, судя по всему, нефункциональна. Возможно, именно из-за негативных аутоиммунных эффектов так часто находят дегенеративные, очевидно неработающие CRISPR-кассеты.
Эволюция CRISPR-систем
Ламарк верил во врожденное стремление организмов к самосовершенствованию. По Ламарку, эволюция происходит посредством «упражнения или неупражнения органов»: постоянное использование органа приводит к его постепенному улучшению. Жираф тянется к листьям на дальних ветках — и его шея понемногу удлиняется. Такое изменение наследуется. По Дарвину, основные факторы эволюции — это случайные, ненаправленные мутации и отбор.
С позиций современного ламаркизма, адаптивная реакция на определенный фактор внешней среды должна быть опосредована молекулярным механизмом, вносящим изменения в геном. Напротив, согласно парадигме дарвиновской эволюции, внешняя среда рассматривается не как первопричина, а, скорее, как селективная сила, которая может благоприятствовать закреплению определенных случайных изменений, если они оказываются адаптивными в текущих условиях.
Посмотрим теперь на CRISPR-систему [32]: фактор окружающей среды (вирус) непосредственно задействован в модификации генома; модификация (вставка уникального спейсера) напрямую воздействует на фактор окружающей среды, послуживший ее причиной; модификация очевидно адаптивна и передаётся по наследству. Все отвечает ламарковскому сценарию.
Практическое использование
CRISPR-системы привлекательны высокой эффективностью, программируемостью и наследуемостью не только для бактерий и архей, но и для человека. Где же CRISPR могут пригодиться?
Маркировка и типирование штаммов. Жизнь каждого бактериального штамма неповторима и уникальна, как и череда перенесенных им инфекций, записанная языком спейсеров. Индивидуальный рисунок CRISPR-кассеты служит естественной генетической маркировкой запатентованных коммерческих штаммов. Типирование штаммов на основании множества спейсеров CRISPR-кассет давно применяется для определения эпидемиологической разновидности разных видов болезнетворных бактерий.
Сейчас при помощи молекулярного клонирования можно внести функциональные CRISPR/Cas-системы в клетки очень широкого круга организмов [33]. Для чего их можно использовать?
Прививки для бактерий. Бактериофаги представляют серьезную угрозу для культур промышленных бактерий. CRISPR — это отличная настраиваемая платформа. На ее основе можно собрать нужный профиль устойчивости только за счет добавления спейсеров к соответствующим бактериофагам. Благодаря CRISPR-системам можно увеличивать продолжительность жизни ценных штаммов.
Ограничение распространения нежелательных генов, локализованных на плазмидах. Если исследователь хочет застраховать свой штамм от приобретения плазмид, несущих нежелательные гены, ему на помощь тоже приходят CRISPR. Так можно ограничивать распространение генов резистентности к антибиотикам и даже целых островков патогенности.
Прививки от бактерий, фаготерапия. Бактерии ответственны за множество болезней человека. А существенная часть бактерий, населяющих тело человека, в том числе, болезнетворных, содержит CRISPR-системы [34]. Если научиться правильно на них влиять и эффективно обходить CRISPR-иммунитет, то можно будет бороться с бактериями с помощью их естественных врагов, бактериофагов — именно этим занимается фаготерапия, но пока, к сожалению, недостаточно успешно для массового применения. Находясь в условиях непрекращающейся гонки вооружений с бактериями, фаги научились красть их оружие, CRISPR-системы, и использовать для избегания клеточного иммунитета [35]. В ближайшем будущем с помощью CRISPR можно будет сконструировать суперфаги, очень специфичные и высоко эффективные против тяжелых бактериальных инфекций человека.
Генетические ножницы на основе CRISPR. Последовательности-мишени специфически узнаются с помощью crРНК в составе эффекторного комплекса. Эти crРНК можно модифицировать, а значит менять мишень для эффекторного комплекса. Таким образом, CRISPR-системы можно легко перепрограммировать и использовать как удобный инструмент направленной манипуляции с геномами: для встраивания, удаления и модификации структурных генов и регуляторных последовательностей. Хотя CRISPR-система — это исключительно прокариотическая придумка, она может быть успешно перенесена в клетки человека и многих других эукариот без потери функциональности и без негативного эффекта для реципиента [33]. Такая технология обладает большим потенциалом для генной терапии, лечения генетических заболеваний путем замены/удаления поврежденных участков генов, и создания высоко специфичных противовирусных вакцин [36], [37].
Статья написана в соавторстве с Анной Гоглевой, аспиранткой группы биоинформатики отдела вычислительной системной биологии Института общей генетики им. Н.И. Вавилова РАН (ИОГен).
Исходно статья опубликована в двух частях в журнале «Природа» [38], [39].
Литература
- Y Ishino, H Shinagawa, K Makino, M Amemura, A Nakata. (1987). Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product.. J. Bacteriol.. 169, 5429-5433;
- Peter M. A. Groenen, Annelies E. Bunschoten, Dick van Soolingen, Jan D. A. van Errtbden. (1993). Nature of DNA polymorphism in the direct repeat cluster of Mycobacterium tuberculosis; application for strain differentiation by a novel typing method. Mol Microbiol. 10, 1057-1065;
- F.J.M. Mojica, C. Ferrer, G. Juez, F. Rodríguez-Valera. (1995). Long stretches of short tandem repeats are present in the largest replicons of the Archaea Haloferax mediterranei and Haloferax volcanii and could be involved in replicon partitioning. Mol Microbiol. 17, 85-93;
- Nancy P. Hoe, Kazumitsu Nakashima, Slawomir Lukomski, Diana Grigsby, Mengyao Liu, et. al.. (1999). Rapid selection of complement-inhibiting protein variants in group A Streptococcus epidemic waves. Nat Med. 5, 924-929;
- Bernd Masepohl, Kirsten Görlitz, Herbert Böhme. (1996). Long tandemly repeated repetitive (LTRR) sequences in the filamentous cyanobacterium Anabaena sp. PCC 7120. Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression. 1307, 26-30;
- Ibtissem Grissa, Gilles Vergnaud, Christine Pourcel. (2007). The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats. BMC Bioinformatics. 8, 172;
- Gilles Vergnaud, Dongsheng Zhou, Mikhail E. Platonov, Christine Pourcel, Ruifu Yang, et. al.. (2007). Analysis of the Three Yersinia pestis CRISPR Loci Provides New Tools for Phylogenetic Studies and Possibly for the Investigation of Ancient DNA. Advances In Experimental Medicine And Biology. 327-338;
- Igor Mokrousov, Elena Limeschenko, Anna Vyazovaya, Olga Narvskaya. (2007). Corynebacterium diphtheriae spoligotyping based on combined use of two CRISPR loci. Biotechnol. J.. 2, 901-906;
- L. M. Schouls, S. Reulen, B. Duim, J. A. Wagenaar, R. J. L. Willems, et. al.. (2003). Comparative Genotyping of Campylobacter jejuni by Amplified Fragment Length Polymorphism, Multilocus Sequence Typing, and Short Repeat Sequencing: Strain Diversity, Host Range, and Recombination. Journal of Clinical Microbiology. 41, 15-26;
- Rotem Sorek, Victor Kunin, Philip Hugenholtz. (2008). CRISPR — a widespread system that provides acquired resistance against phages in bacteria and archaea. Nat Rev Micro. 6, 181-186;
- Maria A. Schumacher. (2008). Structural biology of plasmid partition: uncovering the molecular mechanisms of DNA segregation. Biochem. J.. 412, 1-18;
- D. J. Wilson, E. Gabriel, A. J.H. Leatherbarrow, J. Cheesbrough, S. Gee, et. al.. (2009). Rapid Evolution and the Importance of Recombination to the Gastroenteric Pathogen Campylobacter jejuni. Molecular Biology and Evolution. 26, 385-397;
- Blake Wiedenheft, Kaihong Zhou, Martin Jinek, Scott M. Coyle, Wendy Ma, Jennifer A. Doudna. (2009). Structural Basis for DNase Activity of a Conserved Protein Implicated in CRISPR-Mediated Genome Defense. Structure. 17, 904-912;
- Ki Hyun Nam, Fran Ding, Charles Haitjema, Qingqiu Huang, Matthew P. DeLisa, Ailong Ke. (2012). Double-stranded Endonuclease Activity inBacillus haloduransClustered Regularly Interspaced Short Palindromic Repeats (CRISPR)-associated Cas2 Protein. J. Biol. Chem.. 287, 35943-35952;
- Daniel H. Haft, Jeremy Selengut, Emmanuel F. Mongodin, Karen E. Nelson. (2005). A Guild of 45 CRISPR-Associated (Cas) Protein Families and Multiple CRISPR/Cas Subtypes Exist in Prokaryotic Genomes. PLoS Comp Biol. 1, e60;
- K. S. Makarova. (2002). A DNA repair system specific for thermophilic Archaea and bacteria predicted by genomic context analysis. Nucleic Acids Research. 30, 482-496;
- . (2005). CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 151, 653-663;
- . (2005). Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology. 151, 2551-2561;
- Francisco J.M. Mojica, Chc)sar D�ez-Villase�or, Jes�s Garc�a-Mart�nez, Elena Soria. (2005). Intervening Sequences of Regularly Spaced Prokaryotic Repeats Derive from Foreign Genetic Elements. J Mol Evol. 60, 174-182;
- Ruud. Jansen, Jan. D. A. van Embden, Wim. Gaastra, Leo. M. Schouls. (2002). Identification of genes that are associated with DNA repeats in prokaryotes. Mol Microbiol. 43, 1565-1575;
- Thean-Hock Tang, Norbert Polacek, Marek Zywicki, Harald Huber, Kim Brugger, et. al.. (2004). Identification of novel non-coding RNAs as potential antisense regulators in the archaeon Sulfolobus solfataricus. Molecular Microbiology. 55, 469-481;
- R. Barrangou, C. Fremaux, H. Deveau, M. Richards, P. Boyaval, et. al.. (2007). CRISPR Provides Acquired Resistance Against Viruses in Prokaryotes. Science. 315, 1709-1712;
- Kirill A. Datsenko, Ksenia Pougach, Anton Tikhonov, Barry L. Wanner, Konstantin Severinov, Ekaterina Semenova. (2012). Molecular memory of prior infections activates the CRISPR/Cas adaptive bacterial immunity system. Nat Commun. 3;
- Reidun K. Lillestøl, Shiraz A. Shah, Kim Brügger, Peter Redder, Hien Phan, et. al.. (2009). CRISPR families of the crenarchaeal genus Sulfolobus: bidirectional transcription and dynamic properties. Molecular Microbiology. 72, 259-272;
- . (2009). Short motif sequences determine the targets of the prokaryotic CRISPR defence system. Microbiology. 155, 733-740;
- Ido Yosef, Moran G. Goren, Udi Qimron. (2012). Proteins and DNA elements essential for the CRISPR adaptation process in Escherichia coli. Nucleic Acids Research. 40, 5569-5576;
- Judith Reeks, James H. Naismith, Malcolm F. White. (2013). CRISPR interference: a structural perspective. Biochem. J.. 453, 155-166;
- Matthijs M Jore, Magnus Lundgren, Esther van Duijn, Jelle B Bultema, Edze R Westra, et. al.. (2011). Structural basis for CRISPR RNA-guided DNA recognition by Cascade. Nat Struct Mol Biol. 18, 529-536;
- Edze R. Westra, Paul B.G. van Erp, Tim Künne, Shi Pey Wong, Raymond H.J. Staals, et. al.. (2012). CRISPR Immunity Relies on the Consecutive Binding and Degradation of Negatively Supercoiled Invader DNA by Cascade and Cas3. Molecular Cell. 46, 595-605;
- Caryn R. Hale, Peng Zhao, Sara Olson, Michael O. Duff, Brenton R. Graveley, et. al.. (2009). RNA-Guided RNA Cleavage by a CRISPR RNA-Cas Protein Complex. Cell. 139, 945-956;
- Adi Stern, Leeat Keren, Omri Wurtzel, Gil Amitai, Rotem Sorek. (2010). Self-targeting by CRISPR: gene regulation or autoimmunity?. Trends in Genetics. 26, 335-340;
- Eugene V Koonin, Yuri I Wolf. (2009). Is evolution Darwinian or/and Lamarckian?. Biology Direct. 4, 42;
- Jeffry D Sander, J Keith Joung. (2014). CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol. 32, 347-355;
- Anna A Gogleva, Mikhail S Gelfand, Irena I Artamonova. (2014). Comparative analysis of CRISPR cassettes from the human gut metagenomic contigs. BMC Genomics. 15, 202;
- Kimberley D. Seed, David W. Lazinski, Stephen B. Calderwood, Andrew Camilli. (2013). A bacteriophage encodes its own CRISPR/Cas adaptive response to evade host innate immunity. Nature. 494, 489-491;
- А не замахнуться ли нам на... изменение генома?;
- Элементы: «Прокариотическая система иммунитета поможет редактировать геном»;
- Гоглева А.А. и Артамонова И.И. (2014). CRISPR-системы: структура и гипотетические функции. Природа. 6, 16–21;
- Гоглева А.А. и Артамонова И.И. (2014). CRISPR-системы: механизм действия и применения. Природа. 7, 3–9.








