Ферменты haut couture
19 августа 2007
Ферменты haut couture
- 1048
- 4
- 3
Поиск и получение новых ферментов представляет огромный интерес для синтеза химикатов и лекарств, а также создания инструментов для молекулярной биологии
-
Автор
-
Редакторы
Тысячи ферментов — приемуществено белковых катализаторов биохимических реакций в живой клетке — созданы природой за миллионы лет в процессе эволюции. По сравнению с ней попытки учёных, занимающихся дизайном новых ферментов в лаборатории, выглядят пока, мягко говоря, примитивно. Залогом их успеха является использование тактики, присущей самой эволюции.
Отставание учёных легко объяснимо — у них нет в запасе многих миллионов лет, бывших в распоряжении эволюции. Создание новых ферментов, основанное на наших знаниях структуры белковых молекул и механизмов химических реакций, пока приводит к довольно скромным результатам. Принципы, по которым аминокислотная последовательность белков определяет их структуру и каталитические свойства, не понятны нам до конца, и поэтому, все ещё не могут применяться на практике должным образом. Впрочем, существует возможность обойти эту трудность, симулируя эволюцию in vitro. В последнем номере журнала Nature исследователи Зеелиг (Burkhard Seelig) и Шостак (Jack W. Szostak) описывают подобный подход, который позволил им получить новый фермент, соединяющий вместе концы двух фрагментов РНК [1].
Вообще говоря, получение новых ферментов с нужными свойствами — задача большой практической важности. Такие высокоэффективные катализаторы, каковыми являются ферменты, всегда востребованы современной биотехнологией, химической и фармацевтической промышленностью. Например, «дизайнерский» фермент может катализировать реакцию синтеза нужного лекарства.
Существует несколько методов получения новых ферментов. Один подход состоит во внесении случайных изменений в аминокислотную последовательность существующих природных ферментов (рандомизация) с последующим «искусственным отбором» (селекцией) in vitro. Этот подход приносит неплохие результаты, но ограничивается относительно небольшим числом возможных исходных вариантов (106–108, при желаемом 1012).
Альтернативный метод использует иммунную систему организма, создающую начальное разнообразие и осуществляющую естественный отбор, и, тем самым, создающую каталитические антитела (абзимы, от англ. abzyme, antibody и enzyme). Один из механизмов работы катализаторов, ферментов в частности — связывание и стабилизация переходного состояния (уже не субстрат, но ещё не продукт) катализируемой химической реакции. Таким образом, если антитело связывает молекулу с пространственной структурой, похожей на структуру переходного состояния, то оно может катализировать и соответствующую реакцию. Но для получения каталитических антител необходимо детальное знание механизма реакции, а также возможность синтезировать вещество, имитирующее переходное состояние — условия, выполняющиеся не часто.
Абзимы можно считать искусственно полученными ферментами, поскольку их создание опирается на знание механизма реакции. Но само «изготовление» конкретных каталитический антител происходит полностью in vivo, в результате чисто генетических перестроек, дающих огромное количество исходных вариантов антител, и процесса иммунной селекции. Абзимы обычно способны увеличивать скорость химических реакций в 104–106 раз, но для природных ферментов это весьма скромный показатель. Дело в том, что стабилизация переходного состояния — это лишь один из нескольких способов катализа, используемых ферментами.
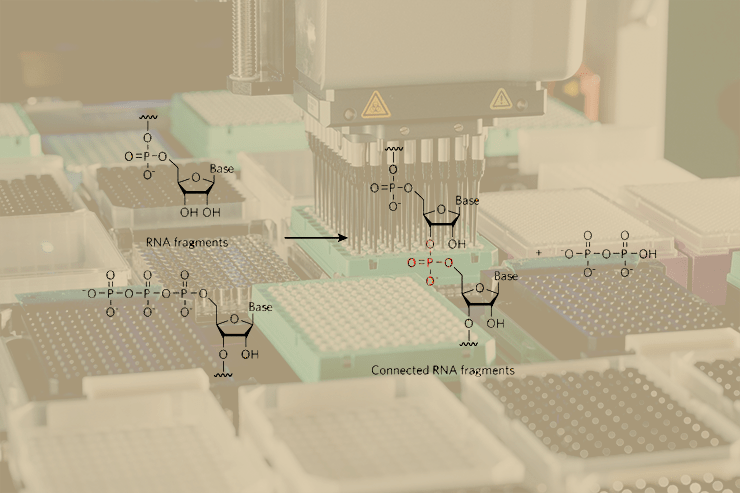
В отличие от искусственно созданных белков-катализаторов, природные ферменты возникли в процессе эволюции в результате естественного отбора, который напрямую опирается на взаимодействие фермента с субстратом и продуктом реакции, и лишь опосредованно — с переходным состоянием. В статье Зеелига и Шостака как раз и применён подход, имитирующий естественный отбор, позволивший этим учёным получить новый фермент, катализирующий реакцию образования ковалентной связи между двумя фрагментами РНК — РНК-лигазу.
Метод, который они использовали, носит название «мРНК дисплей» [2]. Главная особенность метода заключается в том, что белки, проверяемые на наличие каталитической активности, ковалентно связаны каждый с мРНК, которая их и кодирует. Отбор функциональных белков осуществляется из in vitro транскрибируемой и транслируемой библиотеки кДНК, содержащей более 1012 различных последовательностей. В данном случае использовалась синтетическая кДНК-библиотека, состоящая из последовательностей домена человеческого рецептора ретиноидной кислоты (RXR альфа), в котором две петли были заменены на сегменты по 12 и 9 случайных аминокислотных остатков. Домен содержит два «цинковых пальца», мотив, содержащийся в белках, связывающих нуклеиновые кислоты.
Библиотека кДНК была транскрибирована in vitro и тем самым превращена в набор соответствующих молекул мРНК, которые затем транслировались in vitro с образованием мРНК-белковых гибридных молекул. Эти молекулы использовались в качестве матриц для реакции обратной транскрипции с затравкой (праймером), слитой с 3′-концом 5′-трифосфорилированного фрагмента РНК (PPP-субстратом). В результате чего получали библиотеку мРНК-кДНК дуплексов с ковалентно связанным белком и субстратом. Далее, эта библиотека инкубировалась в присутствии биотинилированного олигорибонуклеотида (HO-субстрата) и комплементарного обеим субстратам олигонуклеотида (молекулярной «шины»). Белки, которые были способны катализировать образование связи между двумя субстратами (лигирование РНК), ковалентно присоединяли остаток биотина к собственной комплементарной ДНК, которая отделялась от всего остального при отмывке за счёт взаимодействия биотина со стрептавидином, иммобилизованном на твердофазной агарозной матрице. После отмывки, кДНК снималась с матрицы после расщепления светочувствительной вставки между HO-субстратом и остатком биотина. На последнем этапе селекционного цикла, кДНК амплифицировали при помощи ПЦР (полимеразной цепной реакциии). Полученный материал далее использовался в качестве исходного для следующего цикла.
Чтобы имитировать изменчивость за счёт случайных мутаций и перестроек, как это происходит в природе, после 8-го раунда селекции кДНК подвергли рекомбинации (расщепление эндонуклеазой рестрикции с последующим случайным соединением фрагментов ДНК-лигазой), а затем, «склонной к ошибкам» ПЦР-амплификации (вместо обычной, берётся ДНК-полимераза, неточно копирующая молекулы ДНК).
После этого раунды селекции были продолжены до 17-ого цикла, в ходе которых давление отбора постепенно усиливалось за счёт уменьшения времени реакции образования связи, катализируемой отбираемыми белками.
В результате, авторы статьи [3] создали и изолировали новый фермент — РНК-лигазу. Не известно ни одного природного фермента, способного катализировать лигирование 5′-трифосфорилированного РНК-олигонуклеотида и 3′-концевой гидрокси-группы другой молекулы РНК — реакцию описанную в данной статье. Например, фермент, катализирующий сходную реакцию, РНК-лигаза фага Т4, соединяет 3′-гидрокси-группу и 5′-монофосфорилированную РНК, с одновременным превращением АТФ в АМФ и неорганический фосфат и образованием промежуточного соединения — АМФ, ковалентно связанного в активном центре лигазы. Реакция, катализируемая «дизайнерской» лигазой, скорее напоминает удлинения цепи на один нуклеотид во время РНК-полимеризации: в обоих случаях растущая цепь и трифосфат-содержащий субстрат спариваются азотистыми основаниями с матрицей; 3′-гидроксил растущей цепи атакует альфа-фосфат 5′-трифосфатной группы; наконец, в результате образования связи высвобождается пирофосфат.
Хотя белки и оказались более приспособлены для того, чтобы стать превосходными ферментами в ходе естественного отбора, миллиарды лет назад жизнь, возможно, начиналась с РНК-ферментов, рибозимов (от англ. ribozyme, ribonucleic acid и enzyme), составляющих гипотетический мир РНК, предшествовавший современному миру белков и ДНК. Реакция лигирования РНК занимает особое место для тех, кто изучает эволюцию жизни из мира РНК, поскольку она является существенным этапом процесса саморепликации РНК. Шостак и Дэвид Бартэл (David Bartel) были первыми, кто в 1993 году получили рибозим лигазу методом искусственного отбора in vitro [4]. Тот подход послужил прототипом метода in vitro эволюции рибозимов, и был адаптирован для белковых ферментов в последней статье Зеелига и Шостака.
Литература
- Burckhard Seelig, Jack W. Szostak. (2007). Selection and evolution of enzymes from a partially randomized non-catalytic scaffold. Nature. 448, 828-831;
- R. W. Roberts, J. W. Szostak. (1997). RNA-peptide fusions for the in vitro selection of peptides and proteins. Proceedings of the National Academy of Sciences. 94, 12297-12302;
- Burckhard Seelig, Jack W. Szostak. (2007). Selection and evolution of enzymes from a partially randomized non-catalytic scaffold. Nature. 448, 828-831;
- D. Bartel, J. Szostak. (1993). Isolation of new ribozymes from a large pool of random sequences [see comment]. Science. 261, 1411-1418.