Фолдинг «воочию»
18 февраля 2008
Фолдинг «воочию»
- 2307
- 6
- 3
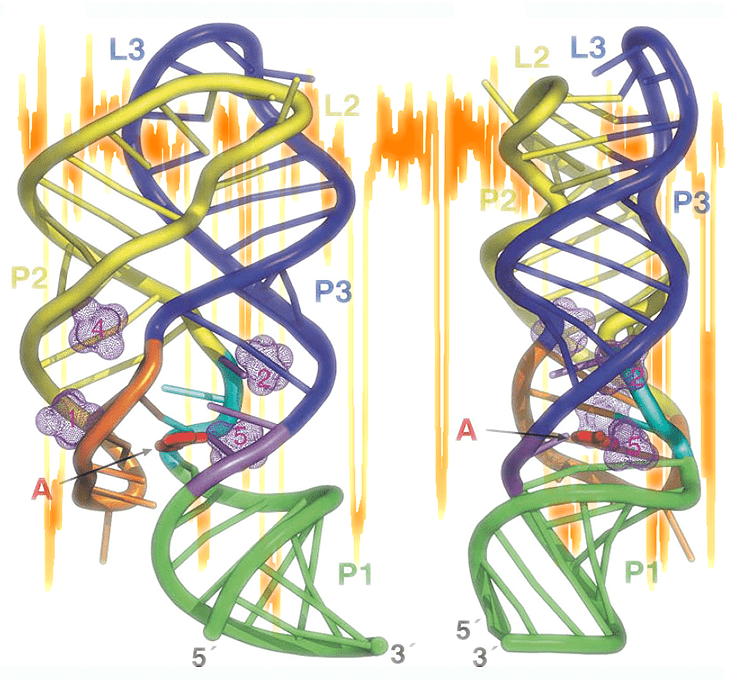
мРНК, способные связывать низкомолекулярные соединения, и, меняя при этом пространственную укладку, влиять на интенсивность собственной трансляции, называются рибоселекторами (riboswitch). Активным центром таких мРНК являются аптамеры — структурные модули, непосредственно связывающие лиганды. На рисунке показана пространственная структура аденин-чувствительного рибоселектора, схема которого дана на рисунке 1.
-
Автор
-
Редакторы
Фолдинг — физический процесс пространственной укладки биополимеров — одна из наиболее актуальных проблем современной физико-химической биологии. До сих пор никому ещё не удавалось проследить за сворачиванием одной-единственной молекулы в реальном времени. И вот, исследователи из Стэнфорда сконструировали оригинальный молекулярный манипулятор, позволивший им «растянуть» молекулу мРНК, содержащую аденин-чувствительный рибоселектор (riboswitch), и дать ей свернуться вновь. Анализ «профилей сил», возникающих при таком растягивании, позволил впервые «увидеть» последовательные стадии фолдинга молекулы и даже отследить событие связывания аденина этой мРНК.
Процесс сворачивания биомакромолекул в уникальную, только им свойственную форму, часто называют фолдингом (от англ. to fold — сворачивать, складывать). Физические явления, лежащие в основе фолдинга (а значит, и всей физики белка), до сих пор до конца не поняты. Вследствие этого, теоретическое предсказание пространственной структуры белков или нуклеиновых кислот — в общем случае неразрешённая задача, где лишь на отдельных примерах удаётся достигнуть определённого успеха. С другой стороны, экспериментально полученные структуры биологических молекул дают нам уже результат фолдинга и мало что говорят о процессах, управляющих пространственной укладкой. Судить же о самом процессе сворачивания до сих пор можно было лишь по косвенным биохимическим или микрокалориметрическим данным, не позволяющим понять детали этого явления.
Исследователи из Стэндорда под началом Стивена Блока (Steven Block) докладывают о весьма значимом результате: им впервые удалось в реальном времени проследить за сворачиванием достаточно сложной единичной молекулы [2]. Такое наблюдение удалось проделать с помощью оригинального молекулярного манипулятора, позволяющего «вытягивать» молекулу из «нативного» (полностью свёрнутого) в полностью распрямлённое состояние и фиксировать степень «вытянутости» в зависимости от приложенной силы («силовой профиль вытягивания»). Анализ этих профилей позволил судить и о стадиях фолдинга и о промежуточных состояниях, в которых одни структурные элементы уже «расплелись», а другие ещё нет, и даже о связывании низкомолекулярных соединений, влияющих на энергетику молекулы наравне с образованием элементов вторичной структуры.
Молекулой, для которой впервые пронаблюдали последовательные стадии сворачивания, стала матричная РНК (мРНК), содержащая в 5′-нетранслируемой области аденин-чувствительный рибоселектор (riboswitch) — особым образом упакованный участок молекулы РНК, способный, подобно рецептору, связывать низкомолекулярные соединения (в этом случае — аденин). Связывание происходит в «ядре» рибоселектора — аптамере, меняющем при этом взаимодействии конформацию, что влияет на интенсивность трансляции всей мРНК и, следовательно, активность соответствующего гена. Например, если среда богата аденином, активность гена, содержащего адениновый рибоселектор, снижается — так, в частности, регулируется метаболизм нуклеотидов. (По аналогии с природными аптамерами с помощью in vitro-селекции можно получать искусственные ДНК или РНК, связывающие малые молекулы, но это уже совсем другая история.)
Прибор, с помощью которого производились манипуляции с РНК и измерялись силы, образно называется «лазерным пинцетом» — из-за его способности перемещать субмикронные объекты с помощью сфокусированного лазерного луча. В области фокусировки образуется своеобразная «оптическая ловушка» — из-за действующих на «пойманный» объект сил, порождаемых рассеянием света. Перемещая луч, можно заставить двигаться и объект в «ловушке», одновременно по динамике изменения положения рассчитывая действующие на него силы. «Пинцет» позволяет развивать (и, соответственно, измерять) усилия порядка пиканьютон (пН) — характерных значений «тяги» молекулярных машин, таких как кинезин, перемещающийся по микротрубочкам или РНК-полимераза, продвигающаяся по ДНК (кстати, эти объекты тоже исследуются в лаборатории Блока).
Поскольку синтез и сворачивание биополимеров неразделимы, изучение фолдинга мРНК проводилось котранскрипционно (рис. 1). Отдельная молекула РНК-полимеразы (РНК-Пол) из E. coli, синтезировавшая in situ по подготовленному ДНК-шаблону (рис. 1а) молекулу мРНК, была прикреплена к пластиковой бусине, находящейся в одной из «оптических ловушек» экспериментальной установки (рис. 1б). Первичный РНК-транскрипт, появляющийся с РНК-полимеразы, комплементарен «липкому концу» 3-кб двуцепочечной ДНК, противоположным концом присоединённой к другой бусине, зафиксированной во второй «оптической ловушке». Таким образом получается фигура, напоминающая гантелю, ДНК-«рукоятка» которой служит для того, чтобы увеличить расстояние между бусинами, без неё бывшее бы слишком маленьким. Управляя положением бусин в «ловушках», исследователи могли увеличивать расстояние между молекулой РНК-Пол и 5′-концом мРНК, заставляя молекулу «расплетаться», а потом сворачиваться вновь. Записываемые при этом «профили растяжения» являлись основным источником информации, на основании которой делали выводы о последовательностях событий, сопровождающих фолдинг/дефолдинг. (Другими словами, исследователи, конечно, не могли «видеть», как сворачивается и разворачивается отдельная молекула — они судили об этом, наблюдая в микроскоп за движением микронного размера шариков, управляемых лазером.)
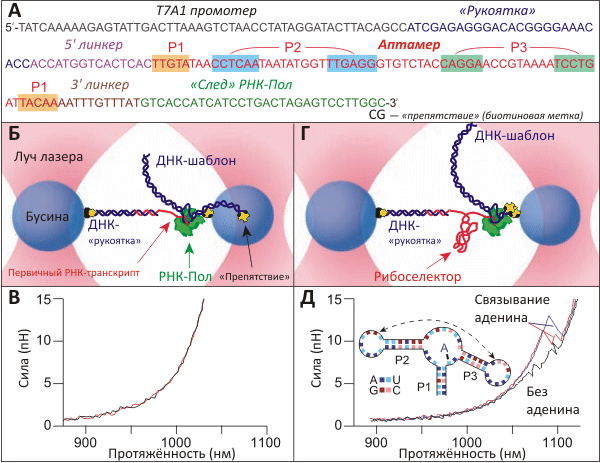
Рисунок 1. Изучение стадий и характеристик сворачивания аденинового рибоселектора. А. ДНК-шаблон, с которого происходит транскрипция изучаемой мРНК. Показаны: нетранскрибируемый промотер, 25-остаточный участок, гибридизующийся с ДНК-«рукояткой» (см. Б), pbuE рибоселекторный аптамер из Bacillus subtilis (спаривающиеся остатки подсвечены) с парой линкеров с обоих концов. Б. Эксперимент проводился в паре «оптических ловушек», созданных сфокусированными лучами лазера. Левая бусина заключена в более «тугую» ловушку и может считаться неподвижной; к ней прикреплена ДНК-«рукоятка», служащая для увеличения расстояния между двумя бусинами, суммарный радиус которых намного превышает размер изучаемой молекулы РНК. Свободный конец ДНК гибридизуется с 5′-последовательностью мРНК, синтезируемой РНК-полимеразой (РНК-Пол) по ДНК-матрице. В. Два «силовых профиля», полученных при «растягивании» мРНК на стадии, пока транскрибируемый аптамер ещё не образовал выраженных вторичной и третичной структуры. Г. На этой стадии эксперимента ДНК-шаблон полностью транскрибирован, и РНК-полимераза блокирована молекулой стрептавидина, связанного биотиновой меткой-«препятствием». Транскрипт представляет собой полностью уложенный рибоселектор. Д. «Силовые профили растяжения» полностью синтезированной и упакованной молекулы рибоселектора указывают на определённую последовательность событий при дефолдинге аптамера. Без добавления аденина в среду на профиле (чёрная кривая) заметно два события — расплетение «шпилек» P2 и P3 (структура аптамера дана на врезке). Аденин, связываемый аптамером, изменяет картину дефолдинга (красная и синяя кривые).
«Силовой профиль растяжения», характеризующий распрямление молекулы в зависимости от приложенной силы (когда «ловушки» разводятся в разные стороны с постоянной скоростью), показал, что первичный транскрипт, представляющий собой лишь часть аптамера, неструктурирован (рис. 1в). В то же время, на профиле, записанном сразу после окончания транскрипции аптамера, заметны характерные пики (рис. 1д), говорящие о том, что мРНК образовала определённые элементы вторичной структуры. В отсутствие аденина профиль искажают два маленьких пика, положение которых позволяет заключить, что они относятся к расплетению «шпилек» P3 и P2 (на рис. 1д показаны соответствующие профили и схема строения аптамера). Если же эксперимент проводили с добавлением аденина, который связывался аптамером, профиль искажался значительно сильнее, свидетельствуя о стабилизации структуры аптамера.
Детальный анализ 800 «профилей растяжения» молекулы позволил рассчитать спектр сил, действующих при фолдинге/дефолдинге аптамера и выявить четыре стадии этого процесса (рис. 2). Как видно из реконструированного «ландшафта» фолдинга, связывание аденина существенно стабилизирует аптамер, увеличивая энергетический разрыв между новым стабилизированным состоянием и промежуточными формами.
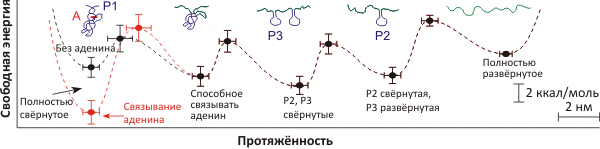
Рисунок 2. Состояния аптамера и их энергетические характеристики, изученные в процессе дефолдинга под действием постоянной силы. Энергетический «ландшафт» фолдинга аптамера, получен на основе анализа «профиля растяжения», записанного при 6,5 пН в присутствии (красная кривая) или в отсутствии (чёрная кривая) аденина. Схематично показаны пять вероятных состояний аптамера.
«В чём мы действительно заинтересованы, так это в фундаментальном понимании того, как сворачиваются биомолекулы, и как они выполняют свойственные им функции. Никому ещё не удавалось подробно описать процесс формирования третичной структуры. А ведь аптамерные РНК, в отличие от тех, что просто кодируют информацию, обязательно обладают довольно сложной третичной структурой!» — поясняет свою работу Блок [3].
Используя силу «лазерного пинцета», Блок и коллеги не просто описали энергию, заключающуюся в каждой «складке» мРНК, но и энергию, необходимую для перехода из одного состояния в другое, а также частоту образования/расплетения отдельных элементовструктуры в реальном времени. Учёные, пронаблюдав под микроскопом за движением крохотных пластиковых бусин, напоминающих пару жемчужин в мантии фантастического моллюска, приблизились к пониманию того, как молекула, обладающая уникальным трёхмерным строением, ведёт себя «в жизни». «Взять настоящую молекулу и крутить её как хочешь — это просто клёво», — подводит итог Блок.
Литература
- Alexander Serganov, Yu-Ren Yuan, Olga Pikovskaya, Anna Polonskaia, Lucy Malinina, et. al.. (2004). Structural Basis for Discriminative Regulation of Gene Expression by Adenine- and Guanine-Sensing mRNAs. Chemistry & Biology. 11, 1729-1741;
- W. J. Greenleaf, K. L. Frieda, D. A. N. Foster, M. T. Woodside, S. M. Block. (2008). Direct Observation of Hierarchical Folding in Single Riboswitch Aptamers. Science. 319, 630-633;
- First direct observation of 3D molecule folding in real time. (2008). ScienceDaily.