Как транспозоны CRISPR/Cas приручили
30 июля 2019
Как транспозоны CRISPR/Cas приручили
- 1493
- 0
- 7
Транспозон Tn6677 холерного вибриона при перемещениях по геному направляется РНК
-
Автор
-
Редактор
Уже давно известно, что системы CRISPR/Cas, защищающие бактерий и архей от вирусов, иногда обнаруживают в составе бактериофагов и транспозонов. Как правило, такие системы CRISPR/Cas неполные и не кодируют нуклеаз. Каково же функциональное предназначение этих «демо-версий» CRISPR/Cas? Недавно на страницах Nature американские исследователи сообщили, что бактериальные Tn7-подобные транспозоны используют свои системы CRISPR/Cas для РНК-направленной интеграции в геном. Более того, ученые предполагают, что транспозоны, которые содержат систему CRISPR/Cas и интегрируются в то место ДНК, которое комплементарно направляющей РНК, могут стать новым инструментом для редактирования генома. Наша статья посвящена этому интереснейшему открытию.
Эволюция систем адаптивного иммунитета бактерий и архей CRISPR/Cas тесно связана с транспозонами. В частности, считается, что гены ключевых белков, необходимых для вставки спейсеров (Cas1) и РНК-направленного разрушения ДНК (Cas9 и Cas12), произошли от генов транспозонов . Кроме того, транспозоны, содержащие собственные (пусть и неполные) системы CRISPR/Cas, вовсе не редкость. Однако лишь совсем недавно удалось экспериментально показать, что системы CRISPR/Cas могут быть нужны транспозонам для интеграции в строго определенное место генома, определяемое направляющей РНК [1].
О работе системы CRISPR/Cas рассказано в статье «Просто о сложном: CRISPR/ Cas» [2], а о разнообразии и эволюции этих систем — в статье «Разнообразие и эволюция систем CRISPR/ Cas» [3].
Пёстрый мир мобильных генетических элементов раскрывает статья «Мобильные генетические элементы прокариот: стратификация “общества” бродяжек и домоседов» [4].
Авторы недавней публикации в Nature работали с транспозоном, родственным транспозону Tn7 кишечной палочки (Escherichia coli), который уже довольно неплохо изучен. Этот транспозон необычен тем, что для перемещения по геному может использовать два взаимно исключающих механизма: он может неспецифично встраиваться в отстающую цепь в ходе репликации или же осуществлять сайт-специфическую интеграцию рядом с консервативной геномной последовательностью. Транспозоны, родственные Tn7, часто несут собственные системы CRISPR/Cas, которые, однако, не содержат генов, кодирующих нуклеазы. Исследователи предположили, что Tn7-подобные транспозоны стали использовать свои системы CRISPR/Cas для совершенно иной цели, нежели разрушение инородной ДНК, а именно, для сайт-специфической интеграции в бактериальный геном.
Транспозон Tn7 содержит характерные последовательности на левом и правом концах, необходимые для транспозиции, и пять собственных генов, обозначаемых tnsA—tnsE [5]. Белковые продукты этих генов вместе образуют сложно организованную транспозазу, катализирующую перемещение транспозона по геному: TnsA и TnsB осуществляют вырезание транспозона, а TnsB, кроме того, катализирует его интеграцию; TnsD и TnsE — взаимно исключающие факторы, определяют место интеграции транспозона в геном; TnsC — АТФаза, обеспечивает энергией процесс транспозиции.
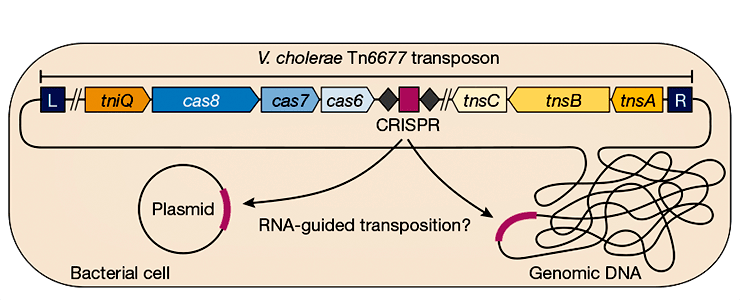
Объектом изучения американских ученых стал транспозон Tn6677, родственный Tn7 и перемещающийся по геному холерного вибриона Vibrio cholerae. Этот транспозон содержит собственную систему CRISPR/Cas, характерные концевые последовательности, как у Tn7, и оперон tns, содержащий гены tnsA, tnsB и tnsC. Любопытно, что гомолог гена tnsD E. coli, обозначаемый tniQ, входит в состав оперона cas — составной части системы CRISPR/Cas, а не tns. Система CRISPR/Cas Tn6677 кодирует комплекс Cascade, который, в отличие от аналогичных комплексов бактериальных систем, не содержит нуклеаз. Сам локус CRISPR включает четыре повтора и три разделяющих их спейсера (см. [2]).
Исследователи ввели в клетки E. coli компоненты Tn6677 в составе отдельных плазмид, а его локус CRISPR изменили таким образом, чтобы он кодировал направляющую РНК, комплементарную участку генома ниже гена glmS. С помощью ПЦР с праймерами, специфичными к транспозону, ученые показали, что транспозон действительно интегрировался в бактериальный геном в нужное место, причем транспозиция произошла в обеих ориентациях (то есть транспозон мог вставляться в геном и в положении «задом наперед»), хотя и с неодинаковой частотой.
Хотя в природе транспозон Tn6677 никогда не интегрируется ниже локуса glmS, выбранного исследователями, и в контрольном эксперименте без направляющей РНК он никогда не появлялся в этом месте, ученые решили дополнительно удостовериться, что специфичность интеграции обеспечена именно направляющей РНК. Для этого они провели аналогичный эксперимент по интеграции транспозона с двумя другими направляющими РНК, нацеленными на другой ген — lacZ. Результат был тот же: в месте, соответствующем направляющим РНК, происходила вставка транспозона, причем в обеих ориентациях.
Но какие именно белки необходимы для сайт-специфической транспозиции, опосредованной РНК? Ученые по очереди вносили мутации или делетировали белки, кодируемые транспозоном, и выяснили, что для транспозиции необходимы белки TnsB, TnsC и Cas6, который осуществляет процессинг направляющей РНК. Более того, оказалось, что белок TniQ (гомолог белка TnsD, участвующего в определении сайта интеграции Tn7) непосредственно взаимодействует с комплексом Cascade через белок Cas6, который и связывает направляющую РНК. Таким образом, эффекторный комплекс Cascade непосредственно взаимодействует с компонентом транспозазы, что еще раз подтверждает РНК-зависимую природу транспозиции Tn6677 (рис. 1).
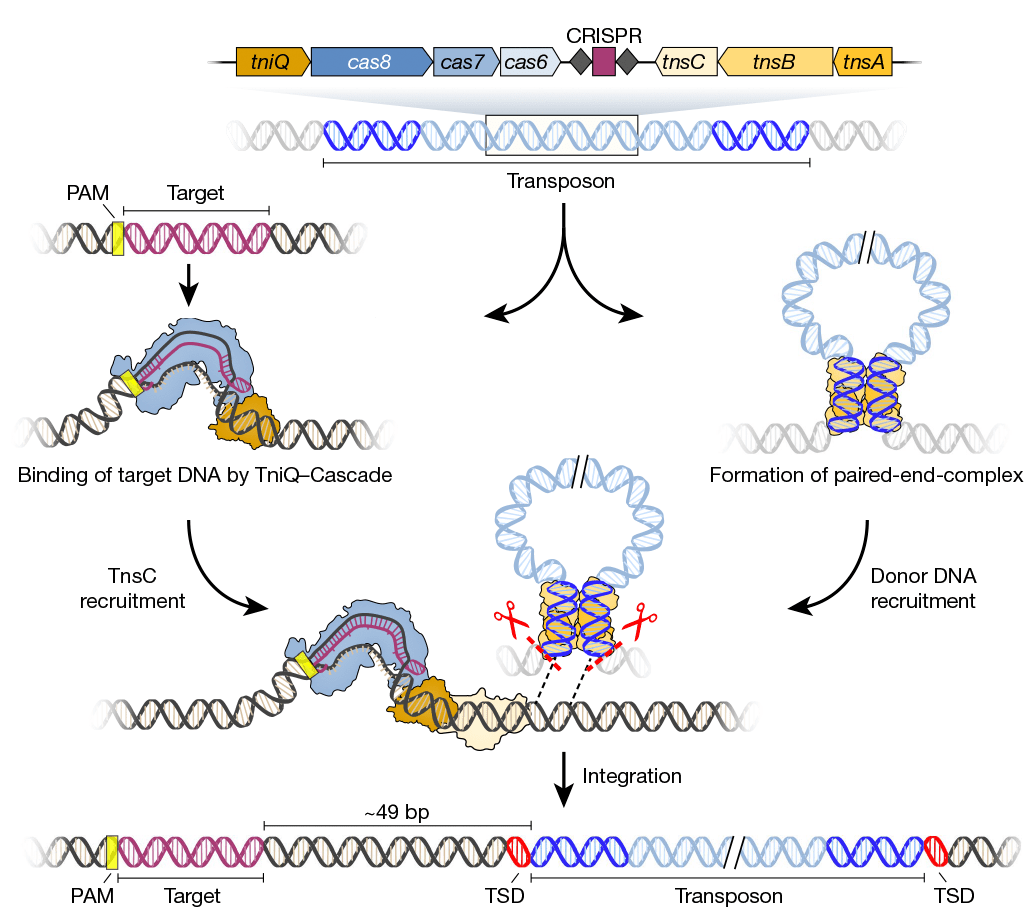
Рисунок 1. Предполагаемый механизм РНК-зависимой транспозиции Tn6677
Чтобы получить детальную картину работы системы, исследователи провели серию дополнительных экспериментов по уточнению значений тех или иных характеристик транспозона, необходимых для его эффективного перемещения. Они установили, что наиболее эффективно транспозиция происходит в случае транспозона, состоящего из 775 пар нуклеотидов (п.н.), а для эффективного перемещения совершенно необходимы особые последовательности, состоящие из 105 п.н. на левом конце транспозона и 47 п.н. на правом. Эти значения, несомненно, пригодятся при разработке технологии РНК-программируемой вставки ДНК в геном на основе транспозона Tn6677.
Насколько же специфична РНК-программируемая транспозиция? Может ли транспозон интегрироваться не только в тот сайт, на который ему «указывает» направляющая РНК, но и попутно в другие участки генома? Для ответа на этот вопрос ученые применили инструмент Tn-seq, изначально разработанный для поиска мест интеграции транспозонов группы mariner в геноме. Оказалось, что неспецифической интеграции Tn6677 почти не происходит, и в 99% случаев транспозон интегрируется строго туда, куда ему и нужно, — ниже локуса glmS.
Описанная система интересна не только с чисто фундаментальной точки зрения как новый механизм транспозиции, но и как возможная основа для новой системы редактирования генома без необходимости внесения двуцепочечных разрывов. Однако реализуется ли этот оптимистичный прогноз на практике, покажет время.
Литература
- Sanne E. Klompe, Phuc L. H. Vo, Tyler S. Halpin-Healy, Samuel H. Sternberg. (2019). Transposon-encoded CRISPR–Cas systems direct RNA-guided DNA integration. Nature. 571, 219-225;
- Просто о сложном: CRISPR/Cas;
- Разнообразие и эволюция систем CRISPR/Cas;
- Мобильные генетические элементы прокариот: стратификация «общества» бродяжек и домоседов;
- N. L. Craig. (1991). Tn7: a target site-specific transposon. Mol Microbiol. 5, 2569-2573.