
Новый метод CETCh-seq может за одну метку поймать много результатов
20 ноября 2015
Новый метод CETCh-seq может за одну метку поймать много результатов
- 897
- 0
- 7
Наживка одна — добычи много. Благодаря новейшим методам редактирования генома, универсальные антитела могут использоваться при работе с большим количеством белков.
Статья на конкурс «био/мол/текст»: На сегодняшний день редактирование генома при помощи системы CRISPR/Cas9 революционно упрощает многие методы, применяемые в молекулярно-биологических исследованиях. Одним из таких методов является анализ ChIP-seq, бодро шагающий по страницам ведущих научных журналов. Давайте посмотрим на его новую модификацию.
Конкурс «био/мол/текст»-2015
Эта работа опубликована в номинации «Лучшее новостное сообщение» конкурса «био/мол/текст»-2015.
Спонсором номинации «Лучшая статья о механизмах старения и долголетия» является фонд «Наука за продление жизни». Спонсором приза зрительских симпатий выступила фирма Helicon.
Спонсоры конкурса: лаборатория биотехнологических исследований 3D Bioprinting Solutions и студия научной графики, анимации и моделирования Visual Science.
ChIP-seq: сшивай, разделяй и секвенируй
В клетках каждого живого организма находятся белки, которые управляют работой генов, — транскрипционные факторы (ТФ). Для того чтобы как-то повлиять на ген, ТФ должен присоединиться к определенной последовательности ДНК. Но как понять, с каким участком генома он связывается? Для этого существует метод, который называется иммунопреципитация хроматина с последующим секвенированием — ChIP-seq (от англ. Chromatin ImmunoPrecipitation Sequencing) (рис. 1). Суть этого метода в том, что ДНК, химически сшитая со связанными с ней белками, «узнается» специально подготовленными антителами. Антитела вырабатывает иммунная система млекопитающих в ответ на присутствие в крови инородного фактора — антигена [1], [2].
Антитела помещаются на хроматографическую колонку со специальными гранулами, затем туда же добавляют коктейль из ДНК и белков. В итоге на гранулах остаются антитела, связанные с исследуемым белком, который, в свою очередь, связан с определенными фрагментами ДНК, а вся остальная смесь проходит сквозь колонку, ни с чем не взаимодействуя. Потом происходит определение последовательности ДНК, с которой был связан наш белок. Таким образом мы можем узнать, с какими генами и хромосомами связывается тот или иной транскрипционный фактор, вплоть до нуклеотида. ChIP-seq, однако!
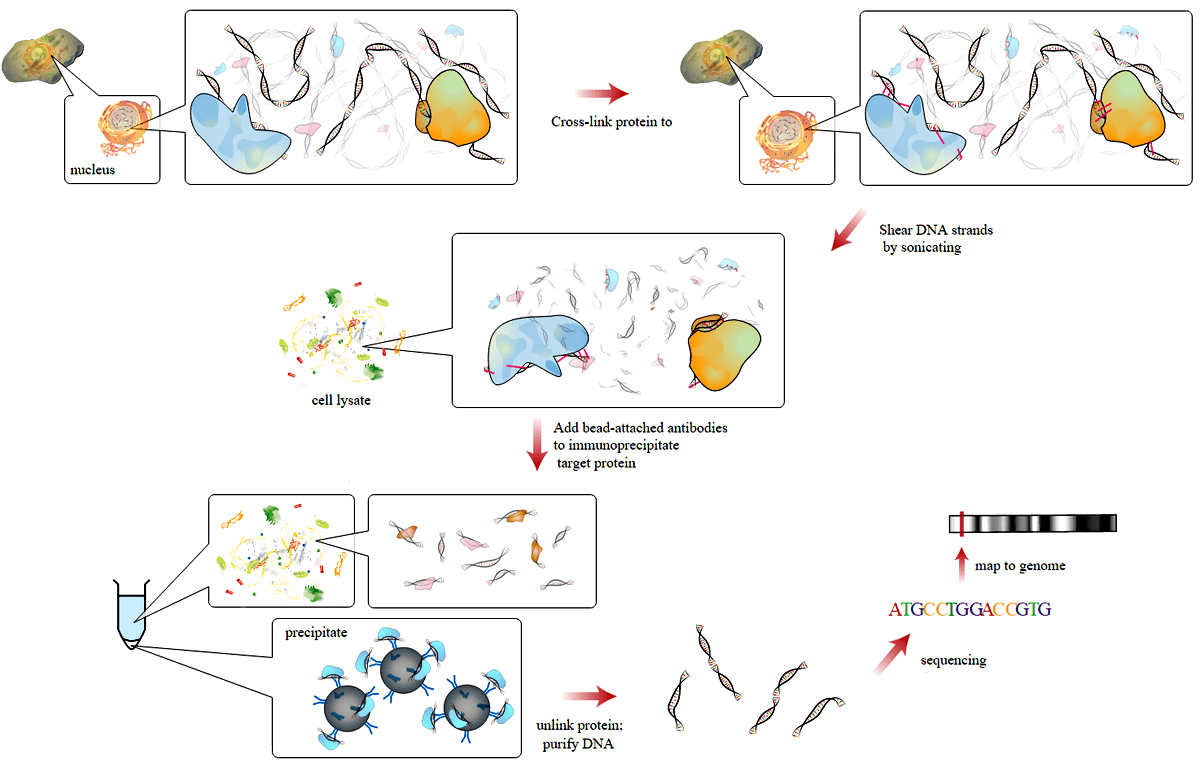
Рисунок 1. Метод ChIP-seq.
Однако метод ChIP-seq достаточно трудоемкий. Один из самых сложных его этапов — это изготовление высокоспецифичных антител. Для того чтобы получить антитела к белку, используется млекопитающее, которому несколько раз вкалывают небольшое количество этого белка с разницей примерно в месяц. Затем у животного берут несколько миллилитров крови, в сыворотке которой содержатся уже выработанные антитела. Из крови удаляют форменные элементы, очищают в несколько этапов, и только после этого антитела готовы к использованию (рис. 2). Процесс изготовления антител занимает несколько месяцев, но, что самое забавное, получаемый на выходе результат непредсказуем. Антитела получаются далеко не ко всем белкам и далеко не с первого раза.
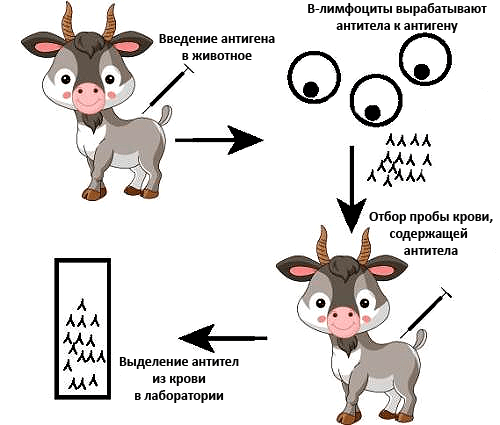
Рисунок 2. Получение антител.
CETCh-seq — тот же ChIP-seq, только лучше
О том, как решить эту проблему, нам рассказали американские исследователи из Алабамы в своей новой статье [3]. Они предлагают не делать каждый раз новые антитела, а предварительно присоединять к исследуемому транскрипционному фактору специальную белковую метку — тэг (от англ. tag — этикетка), — качественные и специфичные антитела к которой давно существуют.
Для испытаний нового метода использовали культуру клеток гепатокарциномы человека (HepG2). В качестве опытных транскрипционных факторов был выбран ряд белков таким образом, чтобы уровень их синтеза был максимально разнообразен.
Для того чтобы встроить тэг в каждый из этих белков, необходимо отредактировать геном клеточной культуры. И вот для этих-то целей исследователи применили «генные ножницы» — систему CRISPR/Cas9, одолженную у бактерий (рис. 3) [4–6]. Эта система состоит из молекулы РНК, комплементарной тому участку ДНК, вблизи которого нужно сделать разрез, и фермента-нуклеазы Cas9. Когда РНК соединяется с ДНК, именно эта нуклеаза режет выбранный участок гена. В честь этой системы авторы назвали свой метод CETCh-seq (от англ. CRISPR Epitope Tagging ChIP-seq).
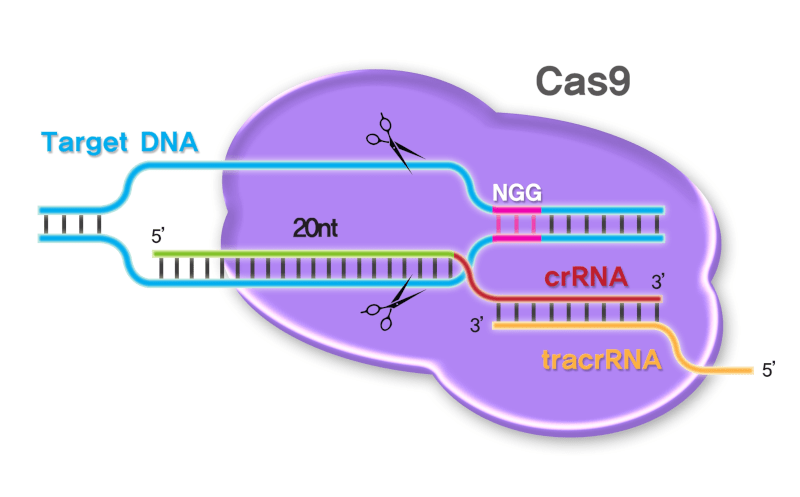
Рисунок 3. CRISPR/Cas9-система.
Для осуществления анализа CETCh-seq в клетки HepG2 поместили две генетические конструкции. Первая несла в себе CRISPR/Cas9-систему, а вторая — встраиваемый тэг. Кроме тэга во второй конструкции находился также ген устойчивости к антибиотику неомицину. Такой ген позволяет проводить селективный отбор — на среде с неомицином вырастают только те клетки, в которые конструкция успешно встроилась. Далее следовал обычный анализ ChIP-seq, но уже с антителами к тэгу, а не к самому белку (рис. 4).
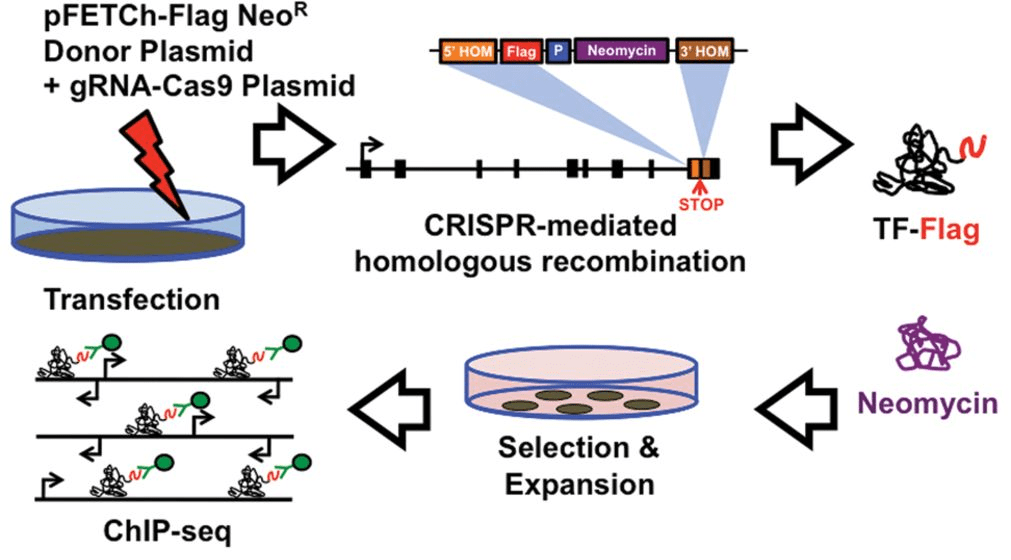
Рисунок 4. Метод CETCh-seq. Клетки трансфецируют плазмидой, несущей: нуклеазу Cas9, молекулу gRNA, специальную метку (Flag), особую линкерную последовательность и ген устойчивости к неомицину. Это приводит к встраиванию всей конструкции в ген интересующего нас транскрипционного фактора на 3’-конце, перед стоп-кодоном. В результате появляется возможность провести скрининг колоний с применением неомицина для выбора тех из них, где встраивание произошло успешно. В отобранных клетках ТФ синтезируется вместе с меткой, к которой в ходе анализа ChIP-seq присоединяются необходимые антитела.
Прежде всего перед исследователями встает вопрос: будет ли встроенная конструкция давать какие-либо побочные эффекты? Редактирование генома может привести к изменению экспрессии генов, и такие клетки будут отличаться от немодифицированных, а значит, будут непригодными для исследований. Поэтому авторы сравнили экспрессию генов до и после встройки конструкции, и принципиальных отличий найдено не было. Следовательно, можно приступать непосредственно к эксперименту.
Вначале для демонстрации достоверности нового метода авторы исследовали методом CETCh-seq те белки, для которых уже существовали данные ChIP-seq. И действительно, для каждого белка при использовании универсальных антител получились такие же результаты, как и при старом подходе. А если не видно разницы, то зачем платить больше (рис. 5)?
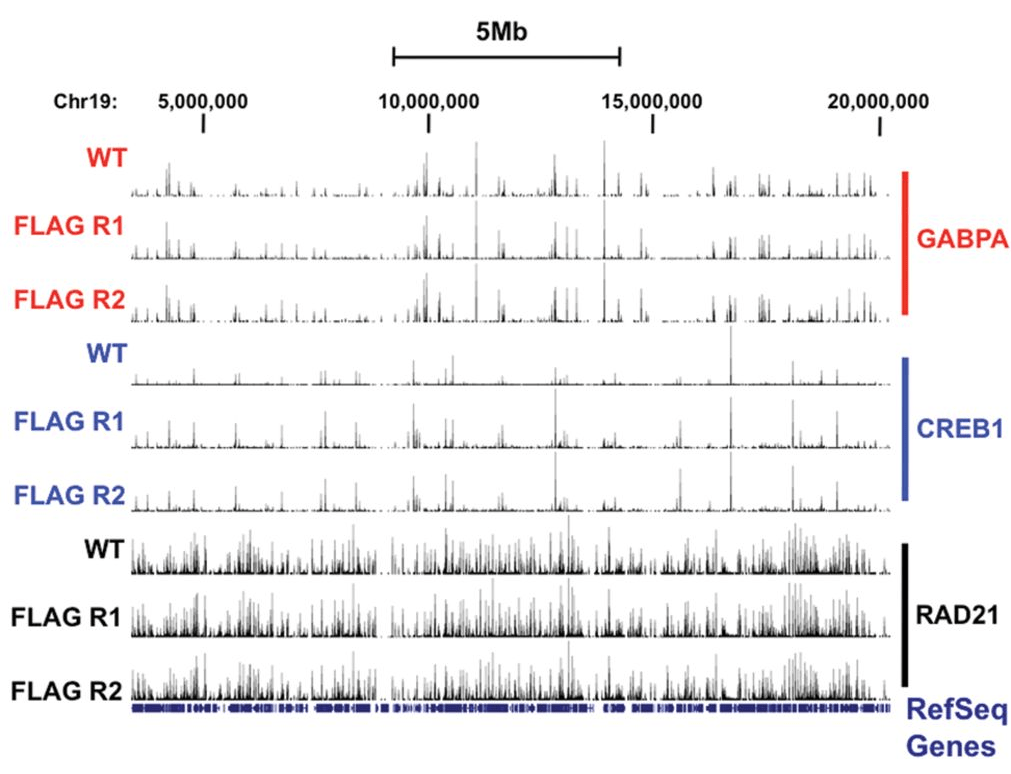
Рисунок 5. Данные CETCh-seq белков GABPA, CREB1 и RAD21 соответствуют данным ChIP-seq. На рисунке изображен участок генома 19 хромосомы. Черные пики маркируют присутствие белка на ДНК. Для каждого из белков расположение и величина таких пиков практически совпадают. Wt — СhIP-seq с антителами к белкам и FLAG R1 и R2 — две повторности CETCh-seq с антителами к тэгу.
Первый эксперимент показал, что метод работает. Поэтому далее авторы применили метод CETCh-seq к белку, для которого не существует данных ChIP-seq в клетках HepG2. Две повторности этого эксперимента показали воспроизводимый результат. Это означает, что новый метод работает. По крайней мере, для клеток гепатокрациномы. Но подойдет ли новый метод для других клеточных культур?
Для ответа на этот вопрос авторы провели схожие эксперименты на клетках аденокарциномы груди MCF7. В этой культуре метод также сработал. Кроме того, сработал этот метод и на эмбриональных клетках мыши.
Эта работа показала, что метод CETCh-seq отличается от своего предшественника простотой исполнения, а результаты получаются аналогичные. Поэтому в будущем ожидается его широкое распространение в исследовании транскрипционных факторов. За это исследование вряд ли дадут Нобелевскую премию, но подобного рода «технические» работы весьма часто влекут за собой важные фундаментальные открытия.
Литература
- От рака вылечит... верблюд!;
- Моноклональные антитела;
- Saviс D., Patridge E.C., Newberry K.M., Smith S.B., Meadows S.K., Roberts B.S. et al. (2015). CETCh-seq: CRISPR epitope tagging ChIP-seq of DNA-binding proteins. Genome Res. 25, 1581–1589;
- А не замахнуться ли нам на... изменение генома?;
- Джагаров Д. (2014). Умные ножницы для ДНК. «Элементы»;
- CRISPR-системы: иммунизация прокариот.






