
Огурцы-убийцы, или Как встретились Джим Уотсон и Гордон Мур
18 августа 2011
Огурцы-убийцы, или Как встретились Джим Уотсон и Гордон Мур
- 1669
- 4
- 1
Полупроводниковая технология прочтения последовательности ДНК, основанная на микрочипах, обещает очередную революцию в полногеномном секвенировании. Кто знает, может быть, на этот раз революция доберется и до здравоохранения?
-
Автор
-
Редакторы
Пожалуй, одной из самых стремительно развивающихся технологий на вооружении современной молекулярной биологии является высокопроизводительное секвенирование ДНК. Гонка за геномом ценой в $1000, набирающая все больше участников из числа крупнейших биотехнологических и фармацевтических корпораций с многомиллионными R&D-бюджетами, а равно и небольших динамично развивающихся «гаражных» компаний из университетских кампусов, уже подарила ученым не одно поколение систем, способных читать человеческие геномы с высокой точностью, в рекордно сжатые сроки и по баснословно низкой (в сравнении с проектом «Геном человека») цене. Недавно были опубликованы результаты работ, в которых молодая и многообещающая технология секвенирования компании Ion Torrent (ныне часть корпорации Life Technologies), основанная на полупроводниковых микрочипах, была успешно использована для расшифровки нескольких важных геномов.
«Биомолекула» уже не раз писала про секвенирование генетической информации (см., например, «454-секвенирование (высокопроизводительное пиросеквенирование ДНК)» [1] и «Перевалило за тысячу: третья фаза геномики человека» [2], а также [3–5]), равно как и про проект «Геном человека» [6]. Заинтересованного, но не осведомленного в этих вопросах читателя мы напутствуем сначала обратиться к этим материалам.

Рисунок 1. Полупроводниковый микрочип для секвенирования ДНК Ion 316
Первым образцом, исследованным с помощью инновационной технологии Ion Torrent, стал геном бактерии Escherichia coli, а точнее ее патогенного энтерогеморрагического штамма O104:H4 [7] (имеет ген потенциально смертоносного Shiga-токсина), мгновенно распространившегося по Европе не без помощи печально известных испанских огурцов-убийц (killer-gurken aus Spanien). Эта вспышка, унесшая 40 жизней и отправившая в больницы тысячи людей, была взята под контроль эпидемиологами во многом благодаря беспрецедентной скорости, с которой была получена последовательность ДНК E. coli O104:H4. Знание генома патогена было необходимо врачам для разработки действенных методов диагностики и лечения инфекционного заболевания. Как только последовательность ДНК была получена, были разработаны тест-системы для выявления штамма с помощью количественной ПЦР. В 8:30 утра 30 мая был запущен секвенатор Ion Torrent PGMTM в лаборатории корпорации Life Technologies в Дармштате, а уже в 19:29 в University Hospital Muenster была завершена разработка тест-систем. Спустя несколько дней геном был независимо прочитан на таком же приборе в лаборатории Пекинского геномного института, а затем был доработан и уточнен с использованием данных, полученных на более мощных (но не столь скоростных) платформах Roche 454 и Illumina HiSeq. Микрочип, используемый в новом поколении машин для секвенирования ДНК, изображен на рис. 1.
Вторым геномом, который был прочитан с использованием полупроводниковой технологии Ion Torrent, стал геном Гордона Мура (рис. 2), пионера компьютерной индустрии и основателя корпорации Intel [8]. Помимо всего прочего, Гордон Мур является соучредителем компании Ion Torrent, а также автором известного «закона Мура», описывающего долгосрочную тенденцию развития производительности компьютеров. Производительность прямо пропорциональна количеству транзисторов, которые можно разместить на микропроцессоре компьютера; одна из формулировок закона Мура гласит, что это число удваивается каждые два года (рис. 3).

Рисунок 2. Пионеры микроэлектроники и молекулярной биологии. Слева: Гордон Мур, пионер компьютерной индустрии и основатель корпорации Intel. Справа: Джим Уотсон, один из первооткрывателей структуры ДНК и отцов-основателей молекулярной биологии.
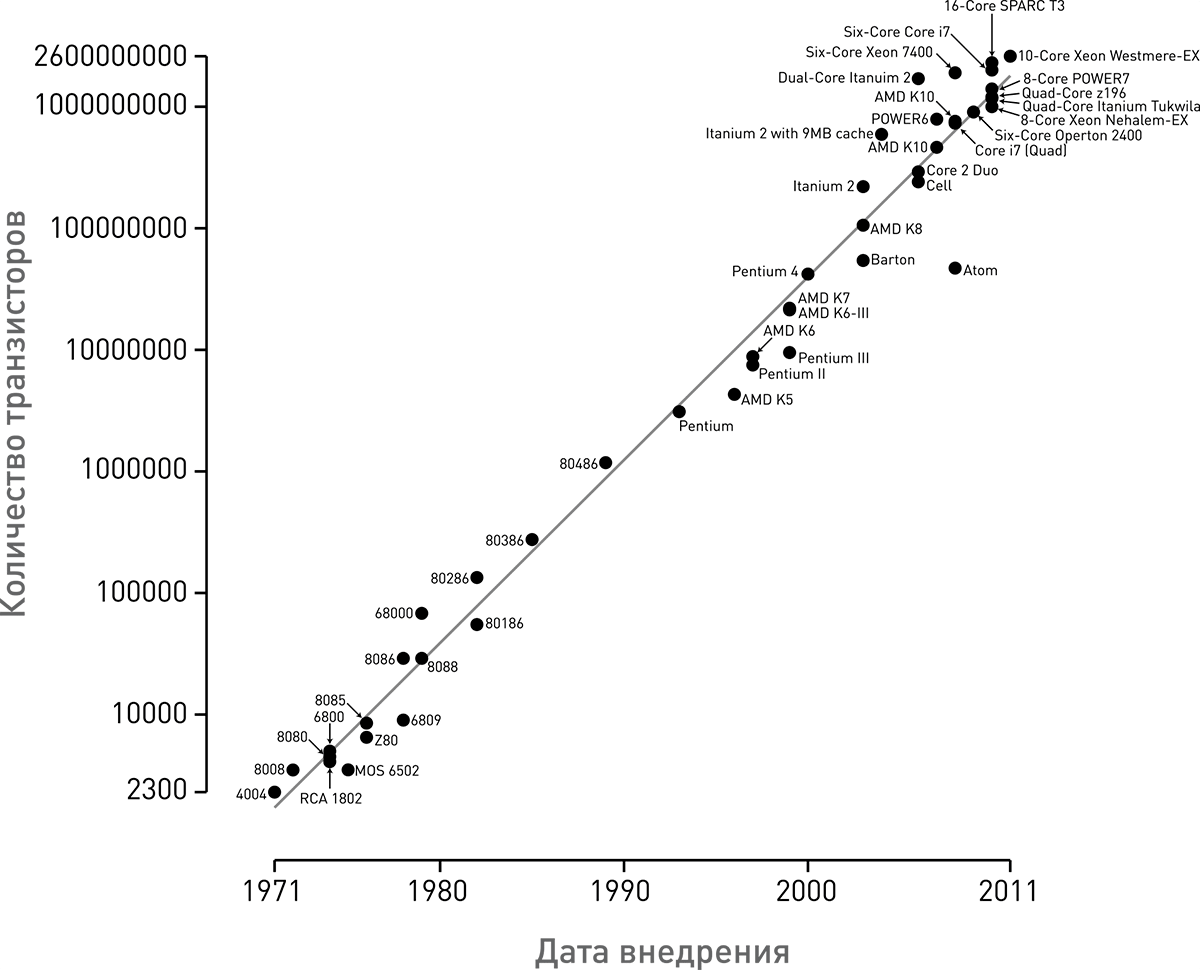
Рисунок 3. Закон Мура. График из статьи о законе Мура наглядно демонстрирует, что число транзисторов на чипе каждые два года удваивается. Обратите внимание, что шкала на оси ординат логарифмическая.
Удивительно то, что развитие технологий секвенирования ДНК по скорости сегодня обгоняет прогресс в области микропроцессоров. Это наглядно демонстрирует график, опубликованный на сайте Национального Института исследований генома человека (рис. 4).
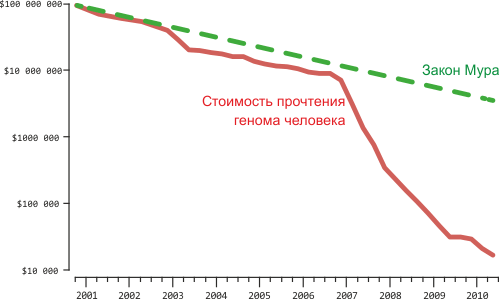
Рисунок 4. Стоимость секвенирования генома человека. Эта зависимость отражает скорее стоимость генома, по размерам совпадающего с геномом человека, чем стоимость конкретного проекта. Здесь учитываются: затраты на человеческий труд, менеджмент и администрирование, реактивы, вспомогательные и расходные материалы; приборы для секвенирования с учетом амортизации в течение трех лет; работа по хранению и упорядочиванию лабораторных данных, а также их биоинформатический анализ; создание геномных библиотек (необходимых для проведения секвенирования); отправка и размещение полученных результатов в общественных базах данных, а также непрямые, но связанные с указанными выше, расходы. Качество прочтения генома должно соответствовать или превосходить стандарты, принятые для данных, полученных методом Сэнгера. Другими словами, мера «качества» прочитанной последовательности должна быть не ниже 20 (Phred Quality Score ≥ Q20). Чтобы рассчитать стоимость секвенирования произвольно взятого генома, нужно знать его размер и необходимое покрытие, которое позволит осуществить приемлемую «сборку» последовательностей из отдельных фрагментов. Тут нужно ориентироваться на то, какая аппаратная платформа используется — это определяет длину индивидуальных прочтений. Для данного графика были использованы следующие допущения: размер человеческого генома: 3 млрд. пар нуклеотидных оснований (п.н.); необходимое покрытие: 6-кратное (6×) для секвенирования по Сэнгеру (длина прочтения 400–600 п.н.); 10× для платформы 454 (длина прочтения 300–400 п.н.); 30× для платформ SOLiD или Illumina (длина прочтения 50–100 п.н.). Для данных, полученных после января 2008 года (с применением платформ «второго поколения»), график отражает стоимость «ре-секвенирования» (повторного прочтения), когда уже известная последовательность генома человека используется в качестве эталонной («референсной»). Необходимая степень покрытия будет выше для геномов, прочитываемых впервые (de novo assembly). См. также: «Код жизни: прочесть не значит понять» [5].
Возможно, именно это и побудило авторов статьи [8] выбрать геном Гордона Мура в качестве образца для демонстрации потенциала новой полупроводниковой технологии секвенирования ДНК. Первые полгода с момента выхода этой технологии на массовый рынок продемонстрировали ее редкую способность к совершенствованию. За это время производительность чипов, используемых для секвенирования, увеличилась на порядок. 314 — чип первого поколения — позволяет «на потоке» прочитывать последовательности от 10 млн. пар нуклеотидных оснований (п.н.); он имеет около 1,2 млн. сенсоров (индивидуальных реакторов, в которых проходит секвенирование отдельных фрагментов). 316 — чип второго поколения — несет уже 6,1 млн. сенсоров и позволяет получать от 100 млн. п.н. За это же время длина индивидуальных прочтений (пока в лаборатории компании) возросла вдвое (с 100 до 200 п.н.). Представители Ion Torrent обещают к концу 2011 года еще в 10 раз увеличить производительность системы за счет чипа третьего поколения (318), который позволит получать от 1 млрд. оснований и будет нести 11 млн. сенсоров. Помимо этого, Ion Torrent обещает удвоение длины индивидуальных прочтений до 400 п.н. в 2012 году.
И хотя для расшифровки генома Гордона Мура потребовалось 1000 полупроводниковых микрочипов (314), что соответствует около 1 млрд. сенсоров, и стоило это во много раз дороже, чем при использовании высокопроизводительных платформ, таких как SOLiD или Illumina, работа сотрудников Ion Torrent представляет собой важную веху в развитии технологий секвенирования. Она демонстрирует, как комбинация полупроводниковых микрочипов и природной биохимии дает поразительный результат — простой, быстрый, и дешевый способ секвенирования ДНК. Способ, который позволяет напрямую, посредством электронных сенсоров, регистрировать молекулярно-биологические события (встраивание комплементарных нуклеотидов полимеразой в растущую цепь ДНК). Так Уотсон встречается с Муром.
Стоит отметить, что на сегодняшний день это единственная технология секвенирования, никоим образом не задействующая свет. Все другие современные технологии в основе своей опираются на флуоресценцию (Сэнгер, SOLiD, Illumina, Pacific Biosciences, Нelicos) или люминесценцию (Roche 454, пиросеквенирование [1]). Потенциально это многократно снижает накладные расходы (не требуются дорогостоящие флуоресцентные реактивы), что и является одним из главных козырей технологии.
В статье [8] также показано, как технология производства микрочипов позволяет создать чип для секвенирования с 1 млрд. сенсоров, что сделает полупроводниковое секвенирование человеческих геномов рутиной, — подобно тому, как рутиной стал, например, синтез белка, несколько десятилетий назад бывший передним краем биохимических наук (рис. 5).

Рисунок 5. Прогресс биохимических технологий. Синтез белка, в конце 20 века еще представлявший головную боль и одновременно сложнейшую задачу для биохимиков, в наши дни стал практически рутинной операцией, требующей лишь доступа к современному оборудованию. Фотографии слайдов из заключительного слова В.Т. Иванова (директора Института биоорганической химии РАН) на 5-м Российском симпозиуме «Белки и пептиды» (Петрозаводск, август 2011 г.).
Литература
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК);
- Перевалило за тысячу: третья фаза геномики человека;
- Геном Нобелевского лауреата Джеймса Уотсона скоро будет расшифрован;
Скверный анекдот: негр, китаец и Крейг Вентер...;- Код жизни: прочесть не значит понять;
- Геном человека: как это было и как это будет;
- Alexander Mellmann, Dag Harmsen, Craig A. Cummings, Emily B. Zentz, Shana R. Leopold, et. al.. (2011). Prospective Genomic Characterization of the German Enterohemorrhagic Escherichia coli O104:H4 Outbreak by Rapid Next Generation Sequencing Technology. PLoS ONE. 6, e22751;
- Jonathan M. Rothberg, Wolfgang Hinz, Todd M. Rearick, Jonathan Schultz, William Mileski, et. al.. (2011). An integrated semiconductor device enabling non-optical genome sequencing. Nature. 475, 348-352.






