
Откуда появились человеческие коронавирусы?
25 августа 2020
Откуда появились человеческие коронавирусы?
- 10231
- 12
- 2
Межвидовые контакты приводят к зоонозам
коллаж автора статьи (изображения из открытых источников)
-
Автор
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: Недавно среди людей распространилась новая коронавирусная инфекция. В результате началась крупнейшая за последнее время пандемия. Судя по всему, это явление было вызвано случайным, но в то же время закономерным попаданием в нашу популяцию из мира животных уже седьмого по счету коронавируса. Часто ли такое происходит и почему?
Конкурс «Био/Мол/Текст»-2020/2021
Эта работа опубликована в номинации «Вирусы и микроорганизмы» конкурса «Био/Мол/Текст»-2020/2021.

Генеральный партнер конкурса — ежегодная биотехнологическая конференция BiotechClub, организованная международной инновационной биотехнологической компанией BIOCAD.

Спонсор конкурса — компания SkyGen: передовой дистрибьютор продукции для life science на российском рынке.

Спонсор конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Надо признаться: мы не знаем, сколько вирусов существует в природе. Сейчас известно 6590 видов этих облигатных внутриклеточных паразитов. Но, по некоторым осторожным оценкам, только среди млекопитающих могут циркулировать сотни тысяч пока не описанных видов вирусов [1]. Отмечу, что разнообразие живого мира, мягко говоря, не ограничивается одним классом позвоночных животных. Безусловно, неизвестные вирусы способны вызывать заболевания человека. Самое грустное в том, что даже при наличии известной геномной последовательности (а чаще всего сиквенса нет — объект-то неизвестный!) невозможно сказать, насколько опасен тот или иной вирус. Таким образом, неизвестно даже примерное число вирусов, потенциально способных приводить к эпидемиям или пандемиям.
Чем чаще и тяжелее протекает инфекция, тем больше ресурсов вкладывают в изучение аспектов взаимодействия патогена с организмом человека. Из понимания этих деталей возникают идеи для разработки лекарств. Сейчас эффективно и специфично можно вылечить или предотвратить примерно 20 вирусных заболеваний, от которых погибало или погибает много людей (например полиомиелит и бешенство). Но наше знание даже, казалось бы, хорошо изученных объектов весьма обрывочно. Например, десятки, если не сотни, научных групп много лет активно изучают вирус полиомиелита. Структуру генома и вирусные белки описали десятилетия назад. А в 2019 году внезапно нашли еще один белок, облегчающий распространение вируса в клетках кишечного эпителия [2].
Сейчас активно разрабатывают методы специфической терапии еще примерно 20 болезней, которые вызывают вирусы (например ВИЧ или SARS-CoV-2). Но это лишь верхушка айсберга: около 200 других вирусов (например лиссавирус Иркут [3] или тоготовирус Бурбон [4]) приводят к заболеваниям человека разной степени тяжести. Про них по большей части можно сказать только то, что:
- нуклеотидная последовательность известна;
- это опасно.
Более того, есть страшная статистика. Когда человек умирает от вирусного энцефалита (воспаления головного мозга, вызванного вирусной инфекцией), в 60% случаев конкретный возбудитель заболевания остается неизвестным [5].
Один и тот же вирус у разных видов приводит к разным состояниям организма. Смертельно опасный для кошек патоген может либо никак не размножаться в собаках, либо распространиться в собачьей популяции, не вызывая тяжелых симптомов. При этом если в среднем по популяции инфекция не передается следующему носителю до «освобождения от патогена», то болезнь самоискореняется. А в обратном случае — вирус закрепляется в новой экологической нише, то есть в новом виде. После межвидового перескока популяции одного вируса оказываются изолированными друг от друга. Рано или поздно это приводит к образованию из одного предкового варианта отличающихся потомков, адаптированных к распространению в разных животных. Таким образом, у каждой группы животных в каждой временнóй точке есть десятки, а скорее сотни «своих» адаптированных вирусов. И такая картина не является постоянной — периодически происходят заносы новых инфекций, а старые могут исчезнуть. На самом деле всё еще сложнее, потому что для каждой изолированной группы одного вида есть свой уникальный набор адаптированных патогенов, которые могут быть смертельно опасными для другой изолированной группы того же вида.
Возможность межвидовой передачи вирусов зависит от интенсивности контактов между разными животными [6]. Например, число контактов между людьми и летучими мышами считается небольшим: летучих мышей, как правило, не содержат в качестве домашних животных и не разводят для употребления в пищу. Тем не менее в некоторых регионах мира этих животных едят. В рационе почти половины жителей деревень на юге Камеруна присутствуют летучие мыши [7]. Летучие мыши этого региона — естественные резервуары филовирусов и хенипавирусов, вызывающих такие опасные заболевания, как лихорадка Эбола [8] и инфекция Нипах [9]. Таким образом, прямая передача вируса от летучих мышей к людям возможна, что периодически и происходит в разных уголках земного шара.
Инфекции «зоологического» происхождения, то есть попадающие в нашу популяцию от животных, называют зоонозами [10]. Если не все, то, пожалуй, большинство вирусных заболеваний человека либо когда-то были зоонозами, начавшими передаваться от человека к человеку (например, гриппом [11] человек может заразиться от другого человека), либо остаются зоонозами, которыми можно заболеть в результате контакта с животным (а еще грипп может передаться человеку от свиньи или утки). Иногда инфекция совершает межвидовой перескок не напрямую, в результате контакта человека с «естественным резервуаром», а через «промежуточного хозяина» инфекции. Так, дикие водоплавающие птицы — это естественный резервуар всех вирусов гриппа типа А [12]. Замечу, что иногда разводимые человеком животные контактируют со своими дикими сородичами. Например, домашняя птица зачастую находится в свободном выгуле, при котором возможно взаимодействие с дикими утками. В результате зараженная домашняя утка может инфицировать других сельскохозяйственных животных, которых мы едим и разводим. Вирусом гриппа друг от друга могут заражаться люди, свиньи, лошади, норки, тюлени, собаки, курицы и другие позвоночные животные [13]. В 2009 году произошла пандемия, вызванная вирусом гриппа А H1N1. Этот патоген попал в человеческую популяцию от свиней [14].
Не всегда заражение человека новым вирусом приводит к развитию пандемии. Если никакой болезни нет, то из-за отсутствия жалоб и симптомов никто и не узнает, что случилась «неудачная», или абортивная инфекция. Но иногда люди заражаются зоонозами, которые затем циркулируют без участия животных (грипп, ВИЧ-инфекция, лихорадка Эбола, COVID-19).
Возможность распространения патогена зависит от многих факторов. Например, вирус бешенства передается при ослюнении раневой поверхности. Такой способ делает возможным циркуляцию бешенства среди лисиц [15]. Но заражение человека бешенством от другого человека в литературе не описано — у людей в норме не принято кусать друг друга. По этой причине бешенство было и будет оставаться классическим примером зооноза для людей. Отмечу, что эта болезнь не всегда циркулировала в популяции плотоядных животных.
Хуже обстоит ситуация с респираторными инфекциями, которые могут передаваться при чихании, кашле или даже простом разговоре. Возможность возникновения эпидемии зависит от влияния среды обитания патогена на распространение инфекции. Под «средой обитания патогена» подразумевают совокупность большого числа социальных, географических и эпидемиологических факторов. Например, в различных сообществах люди предпочитают соблюдать разную дистанцию при разговоре. Это влияет на распространение инфекций, передающихся воздушно-капельным путем. Климатические условия в разных регионах неодинаковы. Сезонность в распространении гриппа, судя по всему, обусловлена колебаниями абсолютной влажности [16]. Кроме того, на вероятность передачи инфекции влияют принятые регуляторными организациями меры эпидемиологического контроля. Набор таких факторов уникален для каждого отдельного региона, что приводит к разной эффективности распространения инфекции.
Какие бывают коронавирусы и все ли они опасны для человека?
Cемейство Coronaviridae включает в себя два подсемейства. Подсемейство Letovirinae состоит из единственного вида Microhyla letovirus 1, недавно обнаруженного в лягушках [17]. Подсемейство Orthocoronavirinae состоит из четырех родов: Alphacoronavirus (19 видов), Betacoronavirus (14 видов), Deltacoronavirus (7 видов), Gammacoronavirus (5 видов) (рис. 1). До введения греческих букв в качестве приставок (альфа-, бета-, гамма-) рода называли классификационными группами номер 1, 2 и 3 соответственно [18]. После пересмотра номенклатурных деталей описали четвертый род вирусов, который по аналогии назвали дельтакоронавирусами. Коронавирусы могут поражать разных позвоночных животных (куриц, индеек, собак, свиней, дельфинов, китов, грызунов, летучих мышей, верблюдов и других).
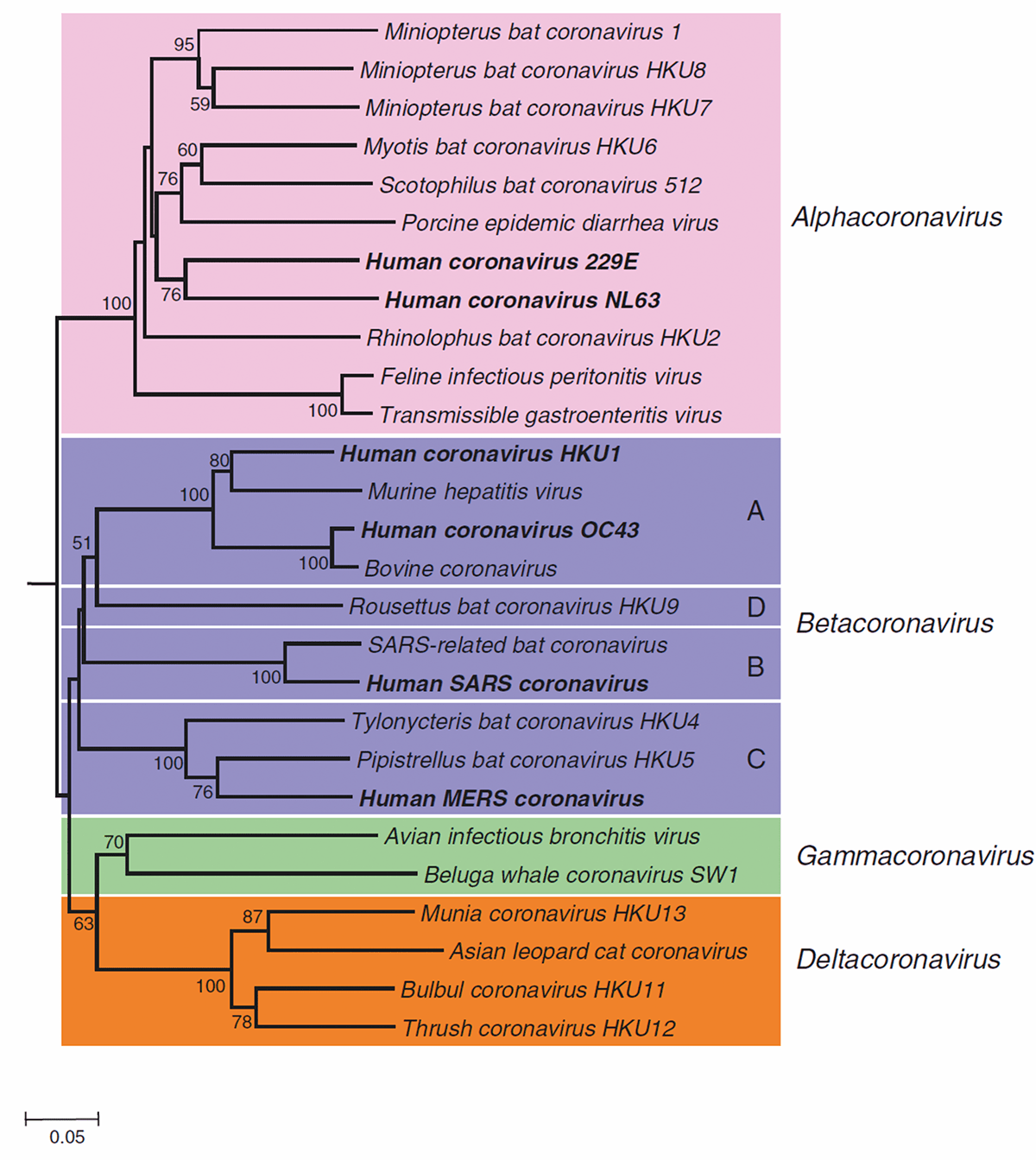
Рисунок 1. Филогенетические взаимоотношения избранных представителей подсемейства Orthocoronavirinae. Названия вирусов, описанных у человека, выделены жирным шрифтом.
Неизвестно, какие из коронавирусов потенциально способны распространиться в нашей популяции, а какие — нет. Более того, непонятна даже доля уже обнаруженных коронавирусов: тут можно предположить любое значение в диапазоне между 0 и 100 процентами. При этом даже родственные коронавирусы могут распространяться между людьми с разной эффективностью. Например, SARS-CoV и SARS-CoV-2 принадлежат к одному виду коронавирусов [19]. SARS-CoV — это аббревиатура от Severe Acute Respiratory Syndrome CоronaVirus, то есть вызывающий тяжелый острый респираторный синдром коронвирус (ТОРС-КоВ). После вспышки атипичной пневмонии 2002–2004 годов у диких животных обнаружили сотни вирусов, которые, согласно филогенетическому анализу, принадлежали к этому же виду. Совокупность таких патогенов обозначили как родственные SARS-CoV. К февралю 2020 года стало понятно, что ранее неизвестный представитель SARS-related coronavirus вызывает человеческую респираторную инфекцию. Всемирная организация здравоохранения и международный комитет по таксономии вирусов предложили назвать коронавирусную инфекцию, начавшуюся в 2019 году, аббревиатурой COVID-19 (Coronavirus disease 2019), а возбудителя болезни — SARS-CoV-2 соответственно. Два человеческих SARS-коронавируса (то есть два варианта одного вида) приводят к разным заболеваниям. В летучих мышах циркулируют другие представители этого вида, случаи заражения человека которыми пока не описали. Пандемический потенциал этих вызывающих SARS коронавирусов неясен, но вызывает серьезные опасения. Сейчас известно, что люди заражались коронавирусами животных как минимум семь раз.
Альфакоронавирусы NL63 и 229E, а также бетакоронавирусы HKU1 и OC43 вызывают до 30% случаев острых респираторных заболеваний (ОРЗ) человека [20]. Чаще всего связанные с этими четырьмя коронавирусами болезни протекают легко и без серьезных последствий. К сожалению, иногда происходят осложнения. В качестве примера можно привести внутрибольничную вспышку бетакоронавируса OC43 летом 2003 года в Канаде, в результате которой погибло 8% пожилых пациентов [21]. В другом случае альфакоронавирус NL63 вызвал тяжелые пневмонии и острые бронхиты у детей в Китае, что привело к госпитализации с благополучным исходом [22]. После подобных сообщений становится непонятно, насколько все-таки безопасны «обычные» человеческие коронавирусы. Возможно, что это просто малоизученная тема. Кроме того, прямо сейчас кто-то из нас может быть носителем пока не описанного пятого «обычного» коронавируса. Но так как серьезных симптомов нет, то носитель не побежит обращаться в больницу, а просто передаст заразу следующему человеку. А даже если вирусосодержащий материал каким-то чудом попадет в клинико-диагностическую лабораторию, то результаты анализа будут отрицательными — стандартизированные наборы реагентов детектируют лишь известные вирусы. Открытие же новых вирусов возможно только при кооперации врачей, грамотно распознающих вирусосодержащий материал, и исследователей, способных секвенировать неизвестные вирусы. Например, HKU1, попавший в человеческую популяцию примерно 70 лет назад, обнаружили только в 2005 году [23].
Современные методы филогенетического анализа позволяют датировать происхождение той или иной группы вирусов. Грубо говоря, время появления вируса в новом виде животного зашифровано в количестве и качестве возникших изменений в нуклеотидной последовательности патогена в сравнении с вирусом промежуточного хозяина. Последний общий предок вызывающих ОРЗ человека коронавирусов NL63, 229E, OC43 и HKU1 существовал примерно 650, 200, 130 и 70 лет назад соответственно [24]. Это значит, что ориентировочно в это время новые коронавирусы попадали в популяцию наших пращуров. В результате могли возникать пандемии (см. врезку) заболеваний. Напомню, что молекулярные методы диагностики вошли в широкую практику относительно недавно. До этого момента установить точную причину той или иной «серой хвори» было практически невозможно. При этом надо понимать, что варианты вируса, которые существовали, например, 130 лет назад, отличались от знакомых нам сейчас потомков этих предковых форм. А разные варианты вирусов могут восприниматься организмом по-разному.
Естественным резервуаром предков бетакоронавирусов HKU1 и OC43 были грызуны, а предков альфакоронавирусов NL63 и 229E — летучие мыши (рис. 2) [32]. Промежуточными хозяевами OC43 считаются коровы, а 229E — альпаки [25]. Такие выводы получают при сравнении нуклеотидных последовательностей патогенов. Практически идентичные последовательности геномов вирусов, выделенных из разных видов животных, показывают недавнюю межвидовую передачу вируса. Отсутствие же очень похожих последовательностей вирусов в разных видах говорит лишь о незнании реального распространения патогена в окружающей среде.
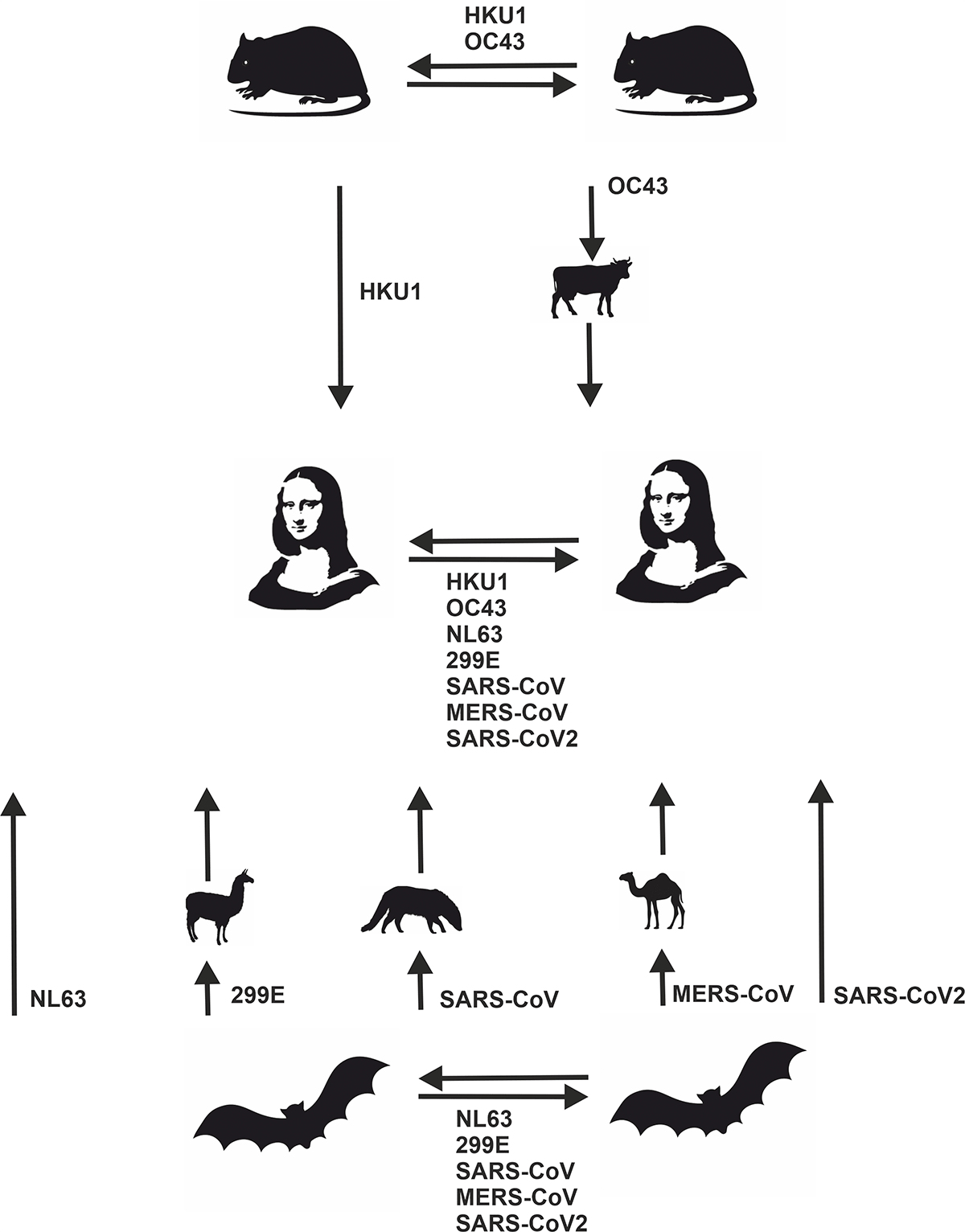
Рисунок 2. Летучие мыши — это естественные резервуары NL63, 299E, SARS-CoV, MERS-CoV, SARS-CoV-2, а грызуны — естественные резервуары HKU1 и OC43. Коровы, альпаки, циветы и верблюды — промежуточные хозяева OC43, 229E, SARS-CoV и MERS-CoV соответственно. Промежуточные хозяева HKU1, NL63 и SARS-CoV-2 неизвестны из-за неполноты знаний экологии коронавирусов.
рисунок автора статьи
В XXI веке произошло три случая заражения человека коронавирусами животных, в результате которых инфекция начала циркулировать в нашей популяции. Все три вируса относятся к бетакоронавирусам.
SARS-CoV
В 2002–2004 годах в Китае случилась вспышка атипичной пневмонии. Это заболевание назвали SARS. Эпидемия началась в ноябре 2002 года в южной провинции Гуандун, откуда быстро распространилась на соседние территории. Последний случай первой вспышки SARS зафиксировали в июне 2003-го. Всего заболело примерно 8000 человек, 9% погибло [33]. Следует отметить, что в конце 2003 года, спустя полгода после завершения эпидемии, в Китае произошли новые заражения SARS [33]. Вторую вспышку быстро локализовали, заболели всего четыре человека. Природным резервуаром SARS-CoV оказались летучие мыши. От летучих мышей заразились циветы — промежуточные хозяева коронавирусной инфекции, через контакт с которыми SARS-CoV попал в человеческую популяцию [32].
MERS-CoV
Второй случай возникновения способного к передаче от человека к человеку коронавируса произошел на Аравийском полуострове. Инфекцию назвали MERS, то есть Middle East Respiratory Syndrome, или ближневосточный респираторный синдром. Эту болезнь вызывает коронавирус MERS-CoV. Конкретное время начала эпидемии остается загадкой: называют сроки от ноября 2009 года до апреля 2012 года [34]. Всего, по данным ВОЗ, на 31 января 2020 года были лабораторно подтверждены 2506 случаев в 27 странах. Максимальное число заражений произошло в 2013–2015 годах, однако эпидемия продолжается до сих пор. Заболевание протекает как бессимптомно, так и с развитием тяжелой пневмонии, септическим шоком и полиорганной недостаточностью, что приводит к смерти примерно в 36% случаев [35]. Естественным резервуаром предковых форм MERS-CoV оказались летучие мыши, а промежуточными хозяевами — верблюды. Антитела к MERS-CoV у верблюдов обнаружили в архивном биологическом материале, собранном в 1983 году. Это значит, что не позднее 1983 года вирус попал в популяцию верблюдов, которые стали промежуточными хозяевами [32]. Заражение человека от верблюда вирусом MERS-CoV происходило много раз, то есть MERS продолжает оставаться инфекцией зоологического происхождения (зоонозом). Передача вируса от человека к человеку тоже возможна, но считается недостаточно эффективной для развития пандемии [35]. Тем не менее при нарушении эпидемиологических норм возможно успешное распространение MERS-CoV в человеческой популяции. Например, в 2015 году гражданин Южной Кореи путешествовал по странам Аравийского полуострова. После возвращения домой у пациента поднялась температура и появился кашель. Больной посетил три больницы, где находился в переполненных помещениях, ожидая своей очереди к врачу [36]. Всего в результате единственного завоза MERS-CoV в Южную Корею заболели 186 человек, 38 из них погибли. Эпидемия продлилась два месяца. Вспышку удалось локализовать за счет составления общей сети распространения инфекции, выявления возможных контактов и последующего карантина двух десятков тысяч человек [37].
SARS-CoV-2
Согласно филогенетическому анализу, SARS-CoV-2 попал в человеческую популяцию в конце ноября — начале декабря 2019 года [38], [39]. Судя по всему, это было единичное случайное событие. SARS-CoV-2 вызывает COVID-19 [40]. SARS-коронавирусы чаще всего циркулируют в летучих мышах, которые являются естественными резервуарами этих патогенов. Пандемический потенциал других SARS-коронавирусов неясен, но вызывает серьезные опасения.
Филогенетически ближайший к SARS-CoV-2 коронавирус RaTG13 обнаружили у летучей мыши в китайской провинции Юннань [41]. Число идентичных нуклеотидов между геномами этих двух вирусов составляет приблизительно 96%. Четыре процента различий — это довольно много. Последний общий предок SARS-CoV-2 и RaTG13 существовал десятки лет назад: за один год в геноме возникает примерно 0,08% мутаций. Некоторые участки поверхностного белка SARS-CoV-2 больше похожи на соответствующие регионы коронавируса, выделенного из панголинов [39]. Это говорит лишь о том, что сейчас не известны практически идентичные SARS-CoV-2 последовательности геномов вирусов, выделенных не из человека. Значит, промежуточный хозяин SARS-CoV-2, от которого заразился нулевой пациент, пока неизвестен. Отметим, что геномы коронавирусов, выделенных из цивет и верблюдов, практически идентичны геномам SARS-CoV и MERS-CoV соответственно. В результате промежуточный хозяин двух предыдущих человеческих коронавирусов был быстро определен. Есть надежда, что секвенирование вирома животных того региона, где началась пандемия, покажет промежуточного хозяина SARS-CoV-2 [39].
По разным оценкам, в результате предыдущей пандемии (гриппа в 2009 году) погибли десятки [42] или сотни [43] тысяч человек. А от все еще продолжающейся пандемии COVID-19 по данным на июль 2020 года умерли сотни тысяч пациентов. К сожалению, пока не наступило то время, когда можно было бы оценить итоговый урон, нанесенный человечеству этой коронавирусной инфекцией. В текущей ситуации больше всего пугает неизвестность нового патогена. Аспекты взаимодействия SARS-CoV-2 с хозяином на молекулярном, клеточном, тканевом, организменном и популяционном уровнях остаются предметом активного изучения, которое, по сути, началось лишь несколько месяцев назад. Очень многие детали неясны. Например, NL63 можно повторно обнаружить в пациенте спустя несколько месяцев после первого выздоровления [44]. Непонятно, насколько подобная особенность характерна для других человеческих коронавирусов. Другая деталь — существует феномен антитело-зависимого усиления (antibody-dependent enhancement, ADE) инфекции, при котором болезнь протекает тяжелее, если в организме уже есть антитела к возбудителю. Эту особенность наблюдали для вирусов Эбола, Зика, Денге, SARS-CoV [45]. Роль ADE в патогенезе COVID-19 сейчас активно изучается. Кроме того, для HKU1 и OC43 показана сезонность в распространении инфекции [46]. Но для SARS-CoV-2 сейчас отсутствует понимание вклада этого важнейшего фактора, прошло слишком мало времени. Для ответа на эти и многие другие вопросы потребуются годы кропотливой работы тысяч исследователей. Но, несмотря на то, что очень многого мы пока не знаем, некоторые факты уже известны. Например, концентрация SARS-CoV-2 при COVID-19 в верхних дыхательных путях на несколько порядков выше, чем у SARS-CoV при SARS [47]. Значит, SARS-CoV-2 эффективнее реплицируется в глотке, что приводит к более интенсивному распространению респираторной инфекции.
Заключение
Набор вирусов, циркулирующих в каждом виде животных, уникален. Такая совокупность патогенов постоянно меняется со временем. Периодически происходит межвидовой перескок вируса, что может приводить к непредсказуемым последствиям. Люди не раз заражались коронавирусами от разных животных: например, четыре коронавируса, начавшие циркулировать в человеческой популяции давно, вызывают «обычные» ОРЗ. Но остается загадкой то, как происходила адаптация этих вирусов к циркуляции в новом виде много лет назад. Недавно три других коронавируса обнаружили в человеке. SARS-CoV вызвал быстро локализованную вспышку атипичной пневмонии в Китае. На Аравийском полуострове уже почти десять лет регистрируют случаи заражения человека MERS-CoV от верблюдов, что не приводит к развитию пандемии. Седьмой описанный человеческий коронавирус, SARS-CoV-2, вызвал пандемию, которая началась в конце 2019 года. Судя по всему, появление новых зоонозных инфекций, которые начинают распространяться между людьми, — это случайное, но в то же время закономерное событие.
Литература
- Simon J. Anthony, Jonathan H. Epstein, Kris A. Murray, Isamara Navarrete-Macias, Carlos M. Zambrana-Torrelio, et. al.. (2013). A Strategy To Estimate Unknown Viral Diversity in Mammals. mBio. 4;
- Valeria Lulla, Adam M. Dinan, Myra Hosmillo, Yasmin Chaudhry, Lee Sherry, et. al.. (2019). An upstream protein-coding region in enteroviruses modulates virus infection in gut epithelial cells. Nat Microbiol. 4, 280-292;
- Galina N., Larisa M., Sergei I., Ilya G., Natalya G., et. al.. (2013). The Fatal Case of Lyssavirus Encephalitis in the Russian Far East. Encephalitis;
- Olga I. Kosoy, Amy J. Lambert, Dana J. Hawkinson, Daniel M. Pastula, Cynthia S. Goldsmith, et. al.. (2015). Novel Thogotovirus Associated with Febrile Illness and Death, United States, 2014. Emerg. Infect. Dis.. 21;
- Peter Kennedy, Phenix-Lan Quan, W. Lipkin. (2017). Viral Encephalitis of Unknown Cause: Current Perspective and Recent Advances. Viruses. 9, 138;
- Colin R. Parrish, Edward C. Holmes, David M. Morens, Eun-Chung Park, Donald S. Burke, et. al.. (2008). Cross-Species Virus Transmission and the Emergence of New Epidemic Diseases. MMBR. 72, 457-470;
- Helene Baudel, Helene De Nys, Eitel Mpoudi Ngole, Martine Peeters, Alice Desclaux. (2019). Understanding Ebola virus and other zoonotic transmission risks through human–bat contacts: Exploratory study on knowledge, attitudes and practices in Southern Cameroon. Zoonoses Public Health. 66, 288-295;
- Eric M. Leroy, Brice Kumulungui, Xavier Pourrut, Pierre Rouquet, Alexandre Hassanin, et. al.. (2005). Fruit bats as reservoirs of Ebola virus. Nature. 438, 575-576;
- Jan Felix Drexler, Victor Max Corman, Florian Gloza-Rausch, Antje Seebens, Augustina Annan, et. al.. (2009). Henipavirus RNA in African Bats. PLoS ONE. 4, e6367;
- Zoonotic diseases. (2017). CDC;
- Гонки с вирусом: эпидемиология и экология вируса гриппа;
- Sun-Woo Yoon, Richard J. Webby, Robert G. Webster. (2014). Evolution and Ecology of Influenza A Viruses. Influenza Pathogenesis and Control - Volume I. 359-375;
- Colin R. Parrish, Pablo R. Murcia, Edward C. Holmes. (2015). Influenza Virus Reservoirs and Intermediate Hosts: Dogs, Horses, and New Possibilities for Influenza Virus Exposure of Humans. J. Virol.. 89, 2990-2994;
- Gavin J. D. Smith, Dhanasekaran Vijaykrishna, Justin Bahl, Samantha J. Lycett, Michael Worobey, et. al.. (2009). Origins and evolutionary genomics of the 2009 swine-origin H1N1 influenza A epidemic. Nature. 459, 1122-1125;
- Andrei A. Deviatkin, Alexander N. Lukashev, Elena M. Poleshchuk, Vladimir G. Dedkov, Sergey E. Tkachev, et. al.. (2017). The phylodynamics of the rabies virus in the Russian Federation. PLoS ONE. 12, e0171855;
- J. Shaman, M. Kohn. (2009). Absolute humidity modulates influenza survival, transmission, and seasonality. Proceedings of the National Academy of Sciences. 106, 3243-3248;
- Khulud Bukhari, Geraldine Mulley, Anastasia A. Gulyaeva, Lanying Zhao, Guocheng Shu, et. al.. (2018). Description and initial characterization of metatranscriptomic nidovirus-like genomes from the proposed new family Abyssoviridae, and from a sister group to the Coronavirinae, the proposed genus Alphaletovirus. Virology. 524, 160-171;
- Richard A. Kaslow, Lawrence R. Stanberry, James W. Le Duc Viral Infections of Humans — Springer US, 2014;
- Coronaviridae Study Group of the International Committee on Taxonomy of Viruses. (2020). The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol. 5, 536-544;
- Shuo Su, Gary Wong, Weifeng Shi, Jun Liu, Alexander C.K. Lai, et. al.. (2016). Epidemiology, Genetic Recombination, and Pathogenesis of Coronaviruses. Trends in Microbiology. 24, 490-502;
- David M Patrick, Martin Petric, Danuta M Skowronski, Roland Guasparini, Timothy F Booth, et. al.. (2006). An Outbreak of Human Coronavirus OC43 Infection and Serological Cross-Reactivity with SARS Coronavirus. Canadian Journal of Infectious Diseases and Medical Microbiology. 17, 330-336;
- Yanqun Wang, Xin Li, Wenkuan Liu, Mian Gan, Lu Zhang, et. al.. (2020). Discovery of a subgenotype of human coronavirus NL63 associated with severe lower respiratory tract infection in China, 2018. Emerging Microbes & Infections. 9, 246-255;
- Patrick C. Y. Woo, Susanna K. P. Lau, Chung-ming Chu, Kwok-hung Chan, Hoi-wah Tsoi, et. al.. (2005). Characterization and Complete Genome Sequence of a Novel Coronavirus, Coronavirus HKU1, from Patients with Pneumonia. JVI. 79, 884-895;
- Diego Forni, Rachele Cagliani, Mario Clerici, Manuela Sironi. (2017). Molecular Evolution of Human Coronavirus Genomes. Trends in Microbiology. 25, 35-48;
- Bogumiła Kempińska-Mirosławska, Agnieszka Woźniak-Kosek. (2013). The influenza epidemic of 1889–90 in selected European cities – a picture based on the reports of two Poznań daily newspapers from the second half of the nineteenth century. Med Sci Monit. 19, 1131-1141;
- Michael Bresalier. (2012). ‘A Most Protean Disease’: Aligning Medical Knowledge of Modern Influenza, 1890–1914. Med. Hist.. 56, 481-510;
- Mark Honigsbaum. (2011). The ‘Russian’ influenza in the UK: Lessons learned, opportunities missed. Vaccine. 29, B11-B15;
- J MULDER. (1958). PRE-EPIDEMIC ANTIBODY AGAINST 1957 STRAIN OF ASIATIC INFLUENZA IN SERUM OF OLDER PEOPLE LIVING IN THE NETHERLANDS. The Lancet. 271, 810-814;
- Walter R. Dowdle. (2006). Influenza Pandemic Periodicity, Virus Recycling, and the Art of Risk Assessment. Emerg. Infect. Dis.. 12, 34-39;
- Leen Vijgen, Els Keyaerts, Elien Moës, Inge Thoelen, Elke Wollants, et. al.. (2005). Complete Genomic Sequence of Human Coronavirus OC43: Molecular Clock Analysis Suggests a Relatively Recent Zoonotic Coronavirus Transmission Event. JVI. 79, 1595-1604;
- Nathalie Arbour, Robert Day, Jia Newcombe, Pierre J. Talbot. (2000). Neuroinvasion by Human Respiratory Coronaviruses. J. Virol.. 74, 8913-8921;
- Jie Cui, Fang Li, Zheng-Li Shi. (2019). Origin and evolution of pathogenic coronaviruses. Nat Rev Microbiol. 17, 181-192;
- Roy M. Anderson, Christophe Fraser, Azra C. Ghani, Christl A. Donnelly, Steven Riley, et. al.. (2004). Epidemiology, transmission dynamics and control of SARS: the 2002–2003 epidemic. Phil. Trans. R. Soc. Lond. B. 359, 1091-1105;
- The WHO MERS-CoV Research Group. (2013). State of Knowledge and Data Gaps of Middle East Respiratory Syndrome Coronavirus (MERS-CoV) in Humans. PLoS Curr;
- Alimuddin Zumla, David S Hui, Stanley Perlman. (2015). Middle East respiratory syndrome. The Lancet. 386, 995-1007;
- Sun Young Cho, Ji-Man Kang, Young Eun Ha, Ga Eun Park, Ji Yeon Lee, et. al.. (2016). MERS-CoV outbreak following a single patient exposure in an emergency room in South Korea: an epidemiological outbreak study. The Lancet. 388, 994-1001;
- Myoung-don Oh, Wan Beom Park, Sang-Won Park, Pyoeng Gyun Choe, Ji Hwan Bang, et. al.. (2018). Middle East respiratory syndrome: what we learned from the 2015 outbreak in the Republic of Korea. Korean J Intern Med. 33, 233-246;
- 2019-nCoV: очередной коронованный убийца?;
- Suzanne L. Miller, Julie Gill, Gavin R. Webb. (2007). The Proximal Origin of the Hamstrings and Surrounding Anatomy Encountered During Repair. The Journal of Bone & Joint Surgery. 89, 44-48;
- Coronaviridae Study Group of the International Committee on Taxonomy of Viruses. (2020). The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol. 5, 536-544;
- Peng Zhou, Xing-Lou Yang, Xian-Guang Wang, Ben Hu, Lei Zhang, et. al.. (2020). A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 579, 270-273;
- Eskild Petersen, Marion Koopmans, Unyeong Go, Davidson H Hamer, Nicola Petrosillo, et. al.. (2020). Comparing SARS-CoV-2 with SARS-CoV and influenza pandemics. The Lancet Infectious Diseases;
- 2009 H1N1 pandemic (H1N1pdm09 virus). (2009). CDC;
- Patience K Kiyuka, Charles N Agoti, Patrick K Munywoki, Regina Njeru, Anne Bett, et. al.. (2018). Human Coronavirus NL63 Molecular Epidemiology and Evolutionary Patterns in Rural Coastal Kenya. The Journal of Infectious Diseases. 217, 1728-1739;
- Nikolai Eroshenko, Taylor Gill, Marianna K. Keaveney, George M. Church, Jose M. Trevejo, Hannu Rajaniemi. (2020). Implications of antibody-dependent enhancement of infection for SARS-CoV-2 countermeasures. Nat Biotechnol. 38, 789-791;
- Stephen M. Kissler, Christine Tedijanto, Edward Goldstein, Yonatan H. Grad, Marc Lipsitch. (2020). Projecting the transmission dynamics of SARS-CoV-2 through the postpandemic period. Science. 368, 860-868;
- Roman Wölfel, Victor M. Corman, Wolfgang Guggemos, Michael Seilmaier, Sabine Zange, et. al.. (2020). Virological assessment of hospitalized patients with COVID-2019. Nature. 581, 465-469.







