
Разграничение видов при помощи генетических данных: пример ДНК-идентификации рыб из озера Плещеево
11 июля 2023
Разграничение видов при помощи генетических данных: пример ДНК-идентификации рыб из озера Плещеево
- 403
- 0
- 0
Плещеевская ряпушка — одна из героинь этой работы.
Рисунок в полном размере.
фото авторов
-
Авторы
-
Редакторы
Чем больше исследователи накапливают знаний об изменчивости последовательностей различных генов животных и растений, и чем больше появляется методов биоинформатической обработки генетических данных, тем менее возможным становится экспертное, умозрительное выделение того или иного вида. Все чаще этот процесс отдается на откуп формальным математическим методам. При этом у исследователей возникает соблазн выделить те или иные популяции или региональные группы в отдельные таксоны. Каким способом проводить делимитацию видов на основе генетических данных? В этой работе мы сравниваем разные методы автоматического разделения видов на примере ДНК-идентификации рыб из Плещеева озера, расположенного в Ярославской области России.
«Какая польза от того, что у них есть имена», — сказал Комар, — «если они не отзываются на них?»
«Для них — никакой пользы», — сказала Алиса, — «но это полезно людям, которые их называют, я полагаю. Иначе зачем вообще знать, как что называется?»
Льюис Кэрролл, «Алиса в Зазеркалье»
Развитие молекулярно-генетических методов открыло для биологов возможности для более точного, быстрого и подробного изучения видового богатства, чем при «классическом» подходе с многолетними наблюдениями и длительной камеральной обработкой полученного материала. Благодаря развитию международных проектов по ДНК-идентификации, в первую очередь так называемого «ДНК-баркодинга» = «штрихкодирования жизни», основанного на исследовании последовательностей гена, кодирующего первую субъединицу цитохромоксидазы с (COI), в настоящее время накоплен большой объем данных о последовательностях генов сотен тысяч видов живых организмов. Сейчас для ряда групп животных и растений имеется возможность в автоматическом режиме определять вид по последовательности его гена COI и некоторых других генов, наподобие того, как по небольшому штрихкоду в магазине можно узнать все о товаре — от его цены до места изготовления.
Однако чем больше исследователи накапливают знаний о генетической изменчивости и методах биоинформатической обработки, тем менее возможным становится «ручное» выделение того или иного вида, основанное на знаниях, опыте и, зачастую, субъективном видении ситуации экспертом по данной группе, обладающим для этого достаточным багажом знаний и опыта. Весь этот процесс все более автоматизируется и отдается на откуп формальным математическим методам. Ключевым в этом процессе является способ выделения (делимитации) «таксономических единиц» (так называемых mOTU) на основании молекулярных данных. Есть несколько математических подходов к выделению таких «таксономических единиц», реализованных в виде компьютерных программ, и результаты их применения к конкретному набору последовательностей не всегда совпадают.
Например, самый простой метод выделения таксономических единиц — это сравнительный анализ с референсными последовательностями в двух глобальных базах — NCBI GenBank и The Barcode of Life Data System. Но здесь же кроется и опасность неверного определения «эталонных образцов», причем подобные ошибки вовсе не редкость [2]. Более сложные методы связаны с делимитацией, выполняемой уже исходя из определенных математических моделей и вычислений в специализированных программах. Среди таких подходов есть как более простые, связанные с вычислением «разрывов» (минимальных генетических различиях, считающихся достаточными для разграничения видов), так и более сложные компьютерные реализации, основанные на теореме Байеса, коалесцентных моделях и т.д.
У современных исследователей, располагающих молекулярно-генетическими данными с гораздо большим разрешением по сравнению с морфологическими методами, чаще возникает соблазн выделить те или иные популяции или региональные группы в отдельные таксоны. Это выражается в настоящее время в массовом описании новых видов, что даже получило название «таксономическая инфляция» [3]. Последняя затрудняет работу как самих систематиков, так и тех ученых, которые лишь пользуются генетическим определением видов в их исследованиях, посвященных совсем иным проблемам — экологии, биогеографии и т.д. Мало того, тот или иной способ делимитации генетических данных зачастую применяется в зависимости от предпочтений того, кто ее проводит, в том числе от его «идеологии»: предпочитаемой концепции вида, взглядов на математический анализ и даже традиций подобных работ в той или иной группе современных организмов.
Поэтому важной задачей представляется проведение сравнения эффективности разных методов генетической делимитации на примере хорошо исследованной ситуации с неким макротаксоном в выбранном регионе, причем чем проще эта ситуация, тем более достоверно исследователь сможет понять, насколько тот или иной метод «врет».
В нашей работе, выполненной под руководством д.б.н., чл.-корр. РАН Алексея Алексеевича Котова [1], объединившей исследователей из научных организаций России (ИБВВ РАН и ИПЭЭ РАН) и Китая (Нанкинский Университет), мы провели ДНК-идентификацию рыб из озера Плещеево, расположенного в Ярославской области (рис. 1). Оригинальные последовательности, полученные в ходе наших работ, сравнивали с последовательностями ДНК выявленных видов из международных баз данных, а также исследовали эффективность делимитации видов рыб разными методами.

Рисунок 1. Озеро Плещеево — голубой бриллиант в Золотом кольце России.

Рисунок 2. Герб города Переславль-Залесский украшает знаменитая «царская селедка» — плещеевская ряпушка.
В целом, видовой состав озера характеризуется довольно малым разнообразием — за многие годы изучения водоема морфологическими методами здесь выявлено лишь 12 массовых видов рыб. Ихтиофауна Плещеева озера хорошо изучена, не в последнюю очередь благодаря «царской селедке» — уникальной плещеевской популяции европейской ряпушки, поставлявшейся к царскому столу еще с XV века — и даже попавшей на герб города Переславль-Залесский (рис. 2). В настоящее время переславская ряпушка — особо охраняемый вид, она внесена в Красную книгу Российской Федерации (рис. 3). Также в озере обитает лещ — излюбленный объект любительского рыболовства и очень крупная моллюскоядная форма плотвы. Для сбережения природных богатств здесь больше тридцати лет назад организован Национальный парк «Плещеево озеро», благодаря чему обеспечивается не только охрана природных ресурсов, но и проводится многолетний мониторинг экосистемы самого водоема и его окрестностей (рис. 4). Достаточная изученность ихтиофауны озера обусловила его выбор как модельного водоема. Однако при выполнении такой, казалось бы, рутинной работы мы решили использовать «слепой» метод, когда исследователь как бы не знает о видовой принадлежности образцов — и получили довольно интересные и неожиданные результаты.

Рисунок 3. Плещеевская ряпушка в конце 1970-х годов (слева) и в настоящее время (справа).
фото авторов

Рисунок 4. Сбор материала для генетических исследований — не самая легкая и чистая работа.
фото авторов
Мы сформировали набор из 125 последовательностей по гену цитохромоксидазы c, 24 из которых впервые получены для Плещеева озера. Последовательности анализировали 15 разными методами делимитации. Из 21 «классических» видов рыб, встречающихся в Плещеевом озере, и их ближайших родственников на основании генетических данных было получено от 16 до 43 mOTU (вероятных «молекулярных» вида, см. рис. 5). Отметим, что для хорошо изученных групп с относительно простой таксономической структурой наблюдается лучшее соответствие делимитации по разным алгоритмам, в то время как несоответствие одного или нескольких методов, используемых для разграничения видов, является сигналом о необходимости пристального внимания таксономистов к этой группе.
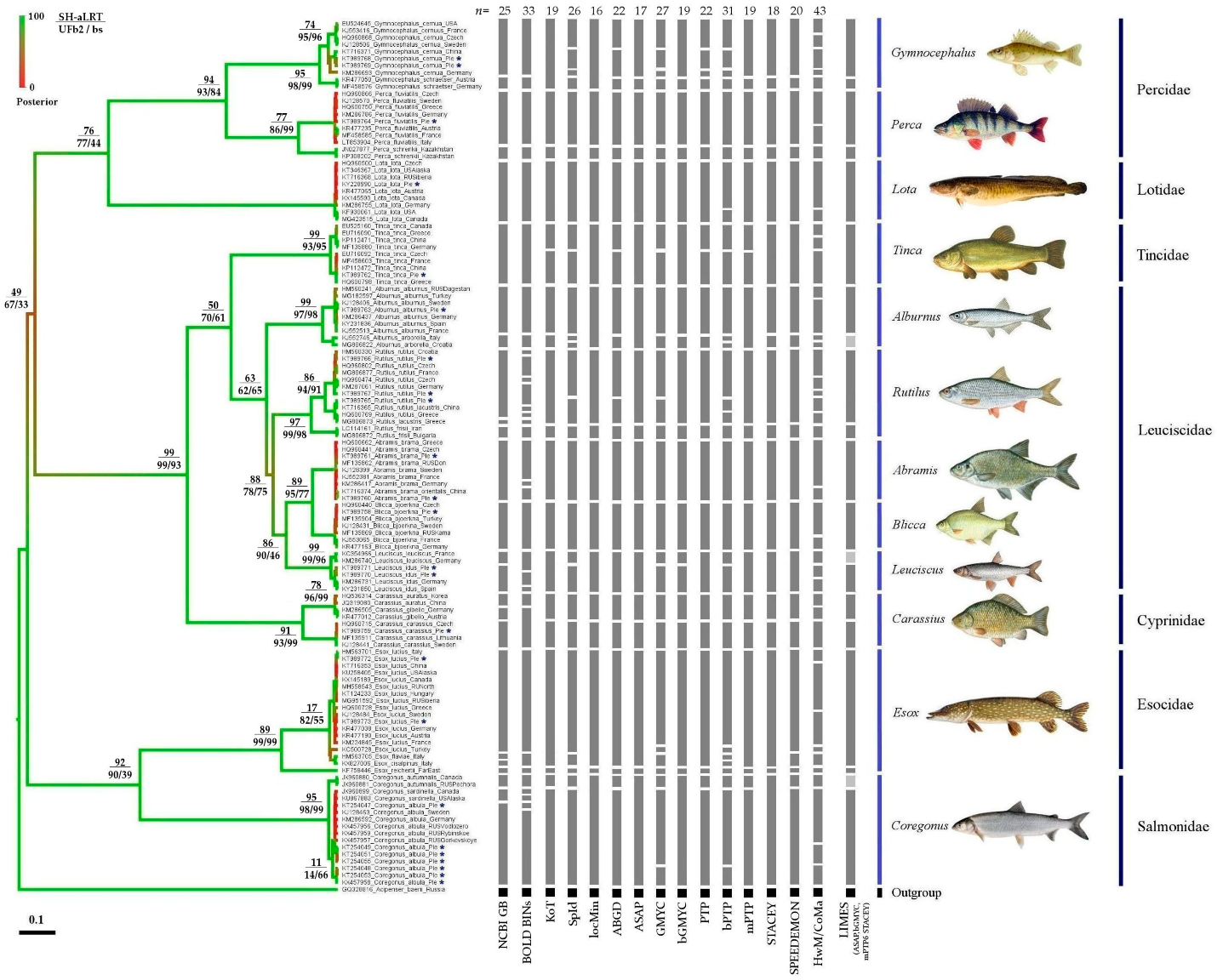
Рисунок 5. Результат делимитации ихтиофауны озера Плещеево разными методами на основании данных по нуклеотидному разнообразию локуса COI (ДНК-баркодинг). Результаты делимитации разными методами представлены в виде объединяющих столбиков. Число выделяемых mOTU («молекулярных» видов) представлено в строке n. Знаком * указаны уникальные последовательности образцов из озера Плещеево. В качестве примера приведена консенсусная таксономия LIMES, поддержка не менее чем 3 из 4 методов (обозначена темной заливкой).
иллюстрация авторов работы по [1]
Один из факторов, влияющих на эффективность методов делимитации видов — это таксономический ранг последовательностей в наборе данных. Для нашей выборки все методы делимитации демонстрируют высокую эффективность на уровне от рода до семейства. С другой стороны, на уровне видов и mOTU использование того или иного метода может значительно повлиять на предположение о видовом богатстве в представленной выборке. Даже наше небольшое исследование приводит к важному вопросу о соотношении OTU независимо от способа их выделения и видов. Увы, к настоящему времени имеется более тридцати концепций вида, часть из которых дополняют друг друга, а некоторые, наоборот — противоречат [4].
Хотя при рассмотрении природных данных лишь небольшое количество выделяемых генетических кластеров можно характеризовать в качестве отдельных видов, и в целом нельзя отождествлять mOTU и виды, но во многих работах они употребляются как синонимы (особенно в случае исследования так называемых «криптических» видов«, практически неразличимых морфологически), что может быть и правомерно, но требует отдельного рассмотрения. В общем же случае, исходя из самой популярной сейчас концепции «филогенетического вида», таковым можно будет считать любую популяцию негибридного происхождения [5].
Возможно, с «политической» точки зрения охраны биологических ресурсов для включения в «красные списки», как находящиеся под угрозой исчезновения, целесообразно было бы признать локальные популяции отдельными видами. Это может облегчить законодательное оформление и привлечение финансирования для их сохранения [6]. Исходя из такого подхода, можно было бы и ряпушку из Плещеева озера выделить в отдельный вид, чему формально не противоречат генетические данные. Однако такой подход не вполне научен, и совершенно недопустимо ставить судьбу находящихся под угрозой исчезновения популяций в зависимость от решений таксономистов, имеющих свои, часто субъективные, предпочтения. В целом, более разумно вместо охраны таксонов заниматься охраной локальных фаун, о чем еще больше 30 лет назад говорил Михаил Валентинович Мина — выдающийся отечественный ихтиолог-эволюционист [7]. В нашем случае, именно популяция ряпушки Плещеева озера и внесена для охраны в «Красную книгу Российской Федерации», тогда как на остальной территории этот вид охраняемым не является.
В заключение можно отметить, что, проведя сравнительный анализ, мы выделили пять наиболее адекватных метода делимитации видов на основании генетических данных, и для некоторых представили простые скрипты вычисления для среды R, которые может запустить даже неискушенный пользователь. По итогам нашей работы мы можем констатировать, что априорные причины использования того или иного метода на сегодняшний день недостаточно сформулированы, и для точной и всесторонней оценки биоразнообразия необходимо использовать комбинацию нескольких подходов для выделения mOTU.
Литература
- Dmitry P. Karabanov, Alexey A. Kotov, Elena A. Borovikova, Yulia V. Kodukhova, Xiaowei Zhang. (2023). Comparison of the Efficiency of Single-Locus Species Delimitation Methods: A Case Study of a Single Lake Fish Population in Comparison against the Barcodes from International Databases. Water. 15, 1851;
- Mikko Pentinsaari, Sujeevan Ratnasingham, Scott E. Miller, Paul D. N. Hebert. (2020). BOLD and GenBank revisited – Do identification errors arise in the lab or in the sequence libraries?. PLoS ONE. 15, e0231814;
- Frank E. Zachos. (2015). Taxonomic inflation, the Phylogenetic Species Concept and lineages in the Tree of Life - a cautionary comment on species splitting. J Zoolog Syst Evol Res. 53, 180-184;
- Zachos F. Species concepts in biology. Historical development, theoretical foundations and practical relevance. Springer International Publishing: Cham, 2016. — 232 p.;
- M. V. Mina, Yu. S. Reshetnikov, Yu. Yu. Dgebuadze. (2006). Taxonomic novelties and problems for users. J. Ichthyol.. 46, 476-480;
- Fenton P. D. Cotterill, Peter J. Taylor, Spartaco Gippoliti, Jacqueline M. Bishop, Colin P. Groves. (2014). Why One Century of Phenetics is Enough: Response to “Are There Really Twice As Many Bovid Species As We Thought?”. Systematic Biology. 63, 819-832;
- Mikhail V. Mina. (1991). Problems of Protection of Fish Faunas in the Ussr. Neth J Zool. 42, 200-213.





