
SHERLOCK — молекулярный сыщик спешит на помощь!
10 мая 2017
SHERLOCK — молекулярный сыщик спешит на помощь!
- 2415
- 0
- 11
рисунок Софи Найденовой
-
Автор
-
Редактор
Темы
Современная медицина неотделима от точных и быстрых анализов. Вирусы Эбола и Зика, медленно тлеющие эпидемии ВИЧ и гепатита С, массовое генотипирование и профилактика наследственных заболеваний требуют от исследователей умения легко находить исчезающе малые количества нуклеиновых кислот-мишеней в самых разных образцах. Новейший метод SHERLOCK дает уникальную возможность детектировать отдельные молекулы полинуклеотидов с помощью системы, помещающейся на ладони.
Не найдется молекулярного биолога, который хотя бы раз в жизни не сталкивался с поиском известной последовательности нуклеиновой кислоты в образце. Сегодня эта, наверное, самая частая и надоедливая задача в молекулярной биологии имеет множество решений. Чего только не выдумали молекулярные биологи за полвека. Тут вам и классический ПЦР-скрининг с гель-электрофорезом, отлично справляющийся с простыми качественными анализами, а заодно навевающий теплую ностальгию по уютным университетским практикумам. Есть и количественная ПЦР в реальном времени для задач посложнее. Ну, а для поиска отдельных молекул, затерянных среди миллиардов своих собратьев в сложных образцах, существуют разнообразные варианты анализа на ДНК-чипах и прочие новомодные капельные цифровые ПЦР [1], [2]. Всего не перечислишь .
Статья «Важнейшие методы молекулярной биологии и генной инженерии» [3] рассказывает о многих из них. — Ред.
Многие из этих методов обладают феноменальной точностью и могут найти буквально единичные молекулы нуклеиновых кислот [4]. К сожалению, все они требуют дорогих и громоздких приборов, а также долгого времени на анализ. Но искать нуклеиновые кислоты хочется быстро, дешево и просто — особенно, если ты исследователь-полевик, изучающий вспышку ящура среди оленей Чукотки или состав колбасы в сельском магазине. Система, пригодная для таких исследований, должна работать при мягких температурных условиях, например, при температуре тела и исключать сложное оборудование. Идеальный вариант, если она будет создана в виде тест-полоски.
В создании аналитической системы мечты нам может помочь метод, описанный авторами Science в недавней статье [5]. Из-за своей выдающейся способности быстро находить отдельные молекулы нуклеиновых кислот он получил броское название SHERLOCK. Как часто бывает в таких случаях, расшифровку подогнали под красивую аббревиатуру, но смысл она тем не менее не утратила: SHERLOCK = Specific High sensitivity Enzymatic Reporter unLOCKing.
Аналогично большинству аналитических методов, SHERLOCK начинается со стадии амплификации. Но в нем используется не обычная трехстадийная ПЦР, требующая высокотемпературного плавления ДНК, отжига праймеров и элонгации при 72 °С, а ее близкий, но гораздо менее капризный родственник — рекомбиназная полимеразная амплификация [6]. В этой реакции, протекающей в изотермических условиях с оптимумом в 37–42 °С, в обязательном порядке используют набор из трех белков (рекомбиназы, SSB-белка и вытесняющей ДНК-полимеразы) и пары праймеров к амплифицируемому фрагменту. Фермент рекомбиназа провоцирует спаривание олигонуклеотидного праймера с комплементарной последовательностью в двуцепочечной ДНК-матрице уже при комнатной температуре. Чтобы праймер не был вытеснен вернувшейся на свое место второй цепью матрицы, в смеси всегда находится избыток SSB-белков, стабилизирующих одноцепочечную ДНК. С двух праймеров, севших на матрицу с помощью упомянутых белков-помощников, начинается синтез ДНК, который ведет Bst-полимераза, вытесняющая по ходу движения вторую цепь из ДНК-дуплекса. Сталкивавшиеся с ней в работе люди знают, что ее оптимум — 65 °С [7], но и при 37 °С она все еще сохраняет свою активность.
Если мы имеем дело с РНК-матрицей, то описанную смесь реагентов дополняет обратная транскриптаза, превращающая реакцию в RT-RPA. После раунда амплификации, смесь олигонуклеотидов подвергается транскрипции с помощью T7 РНК-полимеразы, промотор которой содержится в праймерах, использованных в RPA. Так после двух или трех (в случае RT-RPA) реакций синтеза мы получаем смесь нуклеиновых кислот, обогащенную молекулами РНК искомой последовательности. И тут мы подходим к главной фишке метода SHERLOCK. В дело вступает уже порядком всех утомившая за последние годы система CRISPR-Cas [8].
Об истории и возможностях этой системы «биомолекула» рассказывала в статьях: «CRISPR-эпопея и ее герои» [9] и «Просто о сложном: CRISPR/Cas» [10]. — Ред.
Как вы уже догадались, в качестве зонда выступает crРНК, часть последовательности которой комплементарна искомой последовательности полинуклеотида-мишени. Образовавшийся РНК-дуплекс активирует фермент нуклеазу, но только не высокоспецифичную Cas9, а неспецифичную Cas13a из Leptotrichia wadei. Связавшись с дуплексом crРНК-мишени, Cas13a начинает неспецифично резать свободные нуклеиновые кислоты и рано или поздно добирается до флуоресцентно-меченых РНК-сенсоров, в избытке плавающих вокруг. Их расщепление легко детектируется флуориметром и свидетельствует о том, что SHERLOCK достиг цели (рис. 1).
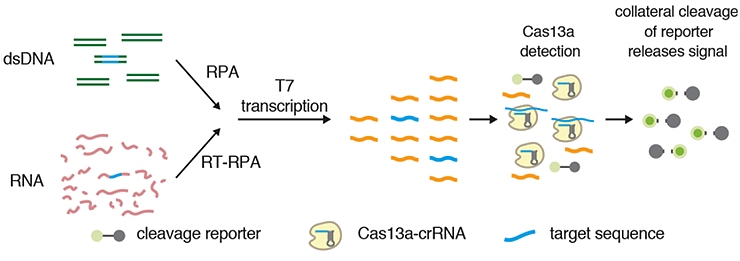
Рисунок 1. Схема работы метода SHERLOCK.
Перспективы метода выглядят многообещающе. SHERLOCK с успехом прошел множество безжалостных проверок. Он безропотно соглашался работать даже с «грязными» образцами, содержащими до 2% человеческой сыворотки. Оказалось, что он с успехом находит как ДНК-, так и РНК-мишени. И даже при совмещении всех его компонентов в одной реакционной смеси сохраняется аттомолярная чувствительность. Все эти достоинства сочетаются с высокой специфичностью: с помощью SHERLOCK удалось различить образцы близкородственных вирусов Зика и Денге с титром меньше 2 аттомоль!
Вдохновленные этим результатом исследователи решили опробовать метод в распознавании однонуклеотидных полиморфизмов (SNP) человеческого генома. Образцы геномной ДНК получили из слюны четырех добровольцев. Протокол выделения включал либо полноценную очистку ДНК на колонке, либо обычный нагрев образца с разрушением клеток в течение 5 минут. В обоих различающихся по чистоте образцах SHERLOCK идентифицировал искомые аллели с чувствительностью, достаточной для распознавания гомо- и гетерозиготного генотипов.
Для того чтобы доказать пригодность метода к использованию в виде тест-полосок, все его компоненты были подвергнуты сорбции на твердофазный носитель и лиофилизированы. Чувствительность метода несколько снизилась, но 20-аттомолярные концентрации вирусов оказались вполне по плечу такому «походно-полевому Шерлоку».
На первый взгляд кажется, что SHERLOCK имеет много общего с методом LAMP. «Биомолекула» уже писала об этом быстром способе детекции нуклеиновых кислот [11]. Ведущую роль в нем так же играет та же самая Bst-полимераза, а время реакции составляет 5–30 минут. Возможность смеси всех компонентов в одной пробирке и изотермическая амплификация так же делают этот метод крайне привлекательным как для ученых, так и для медиков [12]. Однако, чтобы детектировать нуклеиновую кислоту-мишень методом LAMP, амплификация должна пройти как можно более полно и с максимальным выходом, что вынуждает проводить ее при «комфортных» для Bst-полимеразы 65 °С, а не при «хулиганских» 37 °С, как у SHERLOCK. В результате LAMP постепенно завоевывает себе место в стенах лабораторий, но в виде тест-полосок он так и не появился (рис. 2). А вот у SHERLOCK, работающего при температуре человеческого тела, самые блестящие перспективы! Тем более что единственный прибор, необходимый для его работы, — флуориметр — легко можно сконструировать в карманном формате.

Рисунок 2. SHERLOCK и LAMP.
рисунок Софи Найденовой
Литература
- Marco Severgnini, Paola Cremonesi, Clarissa Consolandi, Gianluca De Bellis, Bianca Castiglioni. (2011). Advances in DNA Microarray Technology for the Detection of Foodborne Pathogens. Food Bioprocess Technol. 4, 936-953;
- Matthew C. Strain, Steven M. Lada, Tiffany Luong, Steffney E. Rought, Sara Gianella, et. al.. (2013). Highly Precise Measurement of HIV DNA by Droplet Digital PCR. PLoS ONE. 8, e55943;
- Важнейшие методы молекулярной биологии и генной инженерии;
- R. T. Hayden, Z. Gu, J. Ingersoll, D. Abdul-Ali, L. Shi, et. al.. (2013). Comparison of Droplet Digital PCR to Real-Time PCR for Quantitative Detection of Cytomegalovirus. Journal of Clinical Microbiology. 51, 540-546;
- Jonathan S. Gootenberg, Omar O. Abudayyeh, Jeong Wook Lee, Patrick Essletzbichler, Aaron J. Dy, et. al.. (2017). Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356, 438-442;
- Olaf Piepenburg, Colin H Williams, Derek L Stemple, Niall A Armes. (2006). DNA Detection Using Recombination Proteins. PLoS Biol. 4, e204;
- Bst DNA polymerase, large fragment. Сайт NEB;
- L. Cong, F. A. Ran, D. Cox, S. Lin, R. Barretto, et. al.. (2013). Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339, 819-823;
- CRISPR-эпопея и ее герои;
- Просто о сложном: CRISPR/Cas;
- Поиск иголки в стоге сена за 10 минут — подсвети себе LAMPой;
- Manmohan Parida, Santhosh Sannarangaiah, Paban Kumar Dash, P. V. L. Rao, Kouichi Morita. (2008). Loop mediated isothermal amplification (LAMP): a new generation of innovative gene amplification technique; perspectives in clinical diagnosis of infectious diseases. Rev. Med. Virol.. 18, 407-421.






