Длинные некодирующие РНК бактерий
26 марта 2019
Длинные некодирующие РНК бактерий
- 1755
- 0
- 5
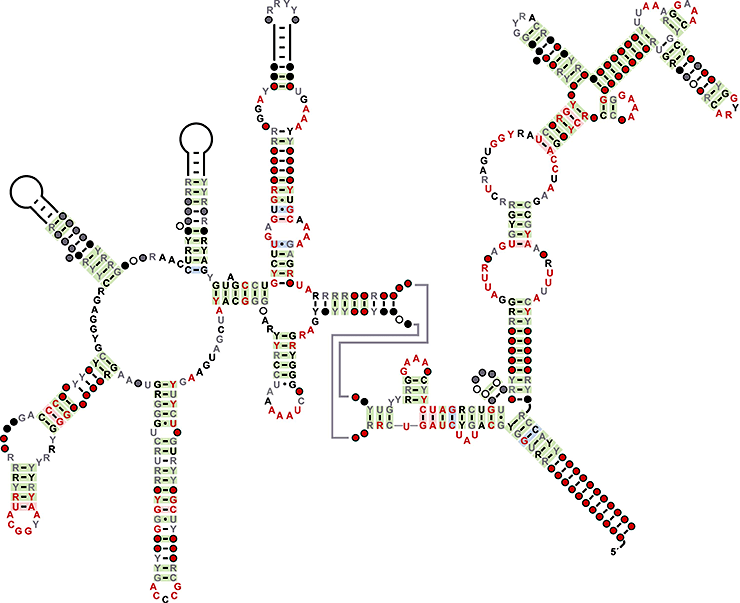
Консенсусная последовательность и вторичная структура OLE РНК, составленная на основе анализа 657 молекул. R — пуриновый нуклеотид, Y — пиримидиновый нуклеотид.
-
Автор
-
Редактор
Исследование длинных некодирующих РНК — одно из лидирующих направлений в современной молекулярной биологии и биоинформатике. В их изучении заинтересованы и медики, так как некоторые длинные некодирующие РНК являются биомаркерами определенных заболеваний. Однако обычно в поле зрения исследователей попадают длинные некодирующие РНК эукариот, а о прокариотах чаще всего забывают. Мы решили восстановить справедливость и рассказать об основных классах длинных некодирующих РНК бактерий, известных на сегодняшний день.
В последние годы длинные некодирующие РНК, или днкРНК (то есть РНК, достигающие более 200 нуклеотидов в длину и, как правило, не содержащие открытых рамок считывания), находятся на пике популярности: каждую неделю выходят свежие публикации, рассказывающие о новых функциях этих, как раньше считалось, бесполезных молекул [1]. Эти «темные лошадки» мира РНК оказались мощнейшими регуляторами экспрессии генов в эукариотических клетках (рис. 1). Заинтересовались ими и медики, так как некоторые длинные некодирующие РНК могут служить биомаркерами различных заболеваний, в том числе онкологических [2]. А есть ли длинные некодирующие РНК у бактерий? Как показывают последние исследования, в прокариотических клетках существует множество классов днкРНК, однако об их биологических функциях практически ничего не известно. В нашем обзоре мы расскажем о тех немногих классах длинных некодирующих РНК бактерий, о которых исследователям удалось что-то узнать.
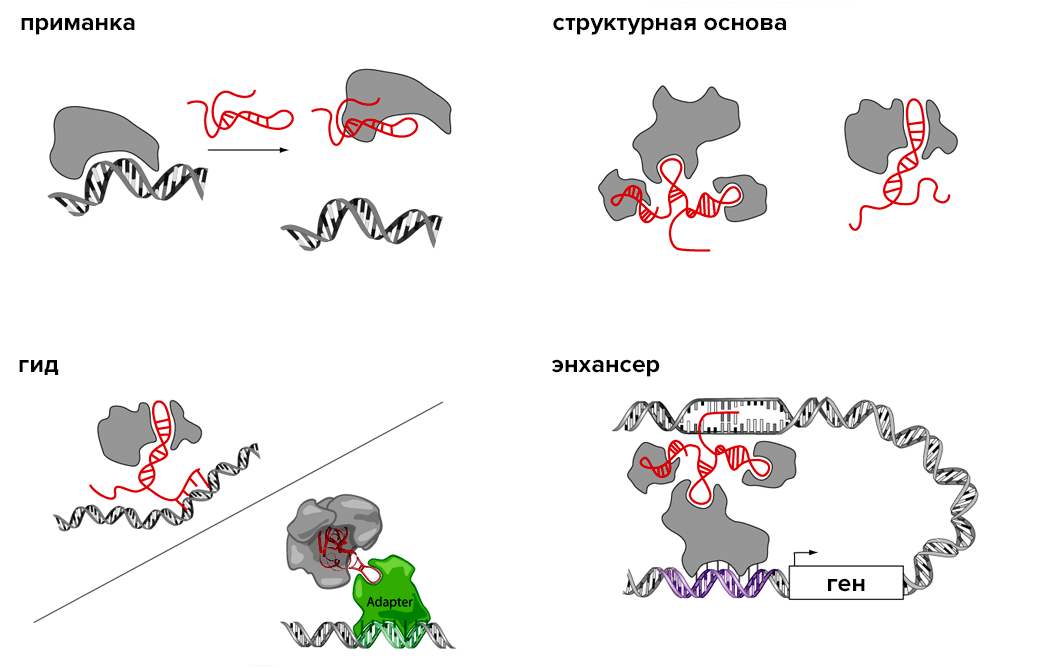
Рисунок 1. Основные модели функционирования длинных некодирующих РНК. Длинные некодирующие РНК могут связывать определенные белки, не давая им взаимодействовать со своими естественными мишенями и таким образом инактивируя («приманки»). Многие днкРНК выступают в роли структурной основы для сборки белковых комплексов. Другие направляют определенные белки (например, ферменты, модифицирующие гистоны) к ДНК. Наконец, длинные некодирующие РНК могут работать подобно энхансерам, вызывая выпетливание хромосомной ДНК и сближение удаленных друг от друга участков хромосом.
Новые классы длинных некодирующих РНК были идентифицированы с помощью биоинформатики, в частности, филогенетическим анализом. Эти РНК имеют длинные, довольно консервативные последовательности и практически не содержат повторов. Более того, они имеют сложную пространственную структуру. Большинство РНК выполняют свои функции, взаимодействуя с другими молекулами нуклеиновых кислот и белков или осуществляя катализ некоторых реакций, и кажется маловероятным, чтобы такие сложные молекулы, как бактериальные длинные некодирующие РНК, были задействованы только в столь простых процессах. Как правило, молекулы РНК со сложной пространственной структурой осуществляют реакции саморазрезания или работают как рибопереключатели, однако все такие молекулы, известные на данный момент, короче 200 нуклеотидов . Вполне возможно, что днкРНК бактерий выполняют функции, ранее не описанные для молекул РНК вообще. Пока же рассмотрим их классы, известные на сегодняшний день.
О роли малых некодирующих РНК в жизни бактерий рассказывается в статьях «Большие дела небольших молекул: как малые РНК дирижируют генами бактерий» [6] и «Малая РНК большого значения» [7].
OLE РНК
OLE (от англ. Ornate, Large, Extremophilic) РНК описаны в 2006 году при анализе 15 бактериальных геномов. На данный момент известно 657 молекул этой группы, причем все они найдены у экстремофильных бактерий или в образцах из окружающей среды. OLE РНК есть у многих представителей бактериального типа фирмикут, и они содержат довольно консервативную последовательность длиной 600 нуклеотидов, которая необходима для формирования сложной вторичной структуры, включающей несколько стеблей (см. заглавный рисунок). Сложная система выпячивания и петель, а также расположение консервативного участка свидетельствуют, что OLE РНК формируют развитую третичную структуру , необходимую для их функционирования [1].
О методах предсказания пространственной структуры РНК по нуклеотидной последовательности рассказывается в статье «Биоинформатика в мире РНК-структур» [8].
Так как OLE РНК широко распространены среди экстремофилов, кажется правдоподобным, что они как-то связаны с защитой этих микроорганизмов от агрессивных условий окружающей среды. Анализ транскриптома экстремофильной бактерии Bacillus halodurans показал, что ее ген ole, кодирующий OLE РНК, относится к числу наиболее интенсивно экспрессируемых. Более того, уровень его экспрессии повышается, когда бактерии подвергаются воздействию короткоцепочечных спиртов вроде этанола, которые образуются при росте бактерий в анаэробных условиях.
У многих бактерий ген ole входит в состав крупного оперона, кодирующего белки, участвующие в биосинтезе изопреноидов, репарации ДНК, метаболизме коферментов и регуляции транскрипции. Рядом с геном ole в опероне находится ген белка с неизвестной функцией, известного как OAP (от англ. OLE-associated protein), причем положение обоих генов в опероне относительно друг друга и других генов очень консервативно. Кроме того, OLE РНК локализуется вблизи клеточной мембраны, но только в присутствии OAP. Некоторые исследователи предполагают, что OLE РНК каким-то образом связаны с биосинтезом изопреноидов и, следовательно, влияют на биохимию клеточной мембраны, обеспечивая адаптацию клетки к экстремальным условиям. И как показали эксперименты с нокаутом генов, ни OLE РНК, ни OAP клеткам в нормальных условиях не нужны.
На данный момент OLE РНК составляют наиболее многочисленную группу бактериальных РНК длиной более 500 нуклеотидов, функции которых неизвестны. Более того, OLE РНК — одни из самых консервативных и сложно структурированных некодирующих РНК бактерий.
GOLLD РНК
GOLLD (от англ. Giant, Ornate, Lake- and Lactobacillales-Derived) РНК достигают более 800 нуклеотидов в длину, что делает их третьей по длине группой бактериальных РНК после 16S и 23S рРНК. Как и OLE РНК, GOLLD РНК, по всей видимости, имеют сложную третичную структуру. В большинстве случаев GOLLD РНК кодируются лизогенными бактериофагами, причем их гены нередко соседствуют с генами тРНК. Впрочем, у некоторых бактерий также есть GOLLD РНК, никак не связанные с бактериофагами. Вероятно, GOLLD РНК выполняют какую-то функцию, полезную и для вирусов, и для бактерий или же, напротив, представляют собой эгоистичные генетические элементы [1].
В пределах последовательности примерно 15% GOLLD РНК имеется тРНК. Смысл такого перекрывания неясен: предполагают, что GOLLD РНК могут каким-то образом участвовать в процессинге тРНК или регулировать их активность. Впрочем, GOLLD РНК и тРНК могут быть и совсем не связаны функционально, а их перекрывание просто позволяет получать сразу две некодирующие РНК из одного предшественника.
Эксперименты с Lactobacillus brevis, у которой ген GOLLD РНК (golld) находится в составе профага, показали, что уровень экспрессии этого гена связан с интенсивностью формирования вирусных частиц. Можно предположить, что GOLLD РНК каким-то образом способствует образованию новых вирионов, однако в клетках с нокаутированным геном golld образование вирусных частиц никак не нарушалось. Таким образом, функции GOLLD РНК остаются загадкой.
HEARO РНК
HEARO (от англ. HNH Endonuclease-Associated RNA and ORF) РНК представляют собой молекулы РНК длиной от 350 нуклеотидов с хорошо развитой пространственной структурой и открытой рамкой считывания. В большинстве случаев она кодирует предполагаемую эндонуклеазу HNH. По всей видимости, в некоторых случаях HEARO РНК транслируются, что было показано экспериментально для одной бактерии.
HEARO РНК обнаружены у бактерий из самых разных групп — фирмикут, протеобактерий, цианобактерий и актинобактерий. А недавно HEARO РНК нашли у археи Methanosarcina mazei. У некоторых бактерий имеется несколько десятков генов hearo. Столь широкое распространение, как полагают ученые, свидетельствует о том, что гены hearo представляют собой мобильные генетические элементы. Однако функции самой HEARO РНК на данный момент не выяснены [1].
Другие длинные некодирующие РНК бактерий
Для некоторых недавно описанных длинных некодирующих РНК бактерий все-таки удалось с высокой степенью достоверности установить биохимические функции. Примером может служить так называемая T-крупная (T-Large) РНК, которая напоминает самосплайсирующиеся интроны II типа, подвергшиеся круговой пермутации (то есть перевернутые «с ног на голову»). Т-крупная РНК в самом деле способна осуществлять реакции разрыва фосфодиэфирной связи, происходящие при нормальном сплайсинге РНК, и даже использует те же сайты сплайсинга. Однако из-за круговой пермутации сайты сплайсинга расположены в обратном порядке, поэтому экзоны, получающиеся при таком сплайсинге, имеют кольцевую, а не линейную форму, а интроны вырезаются не в виде лассо, а в виде разветвленной структуры, похожей на букву Т. Т-крупные РНК обнаружены у протеобактерий, цианобактерий и представителей типа Deinococcus-Thermus, однако биологическое значение осуществляемого ими необычного сплайсинга неизвестно.
IMES-1 (от англ. Identified in Marine Environmental Sequences) РНК имеют длину около 220 нуклеотидов, многие из которых очень консервативны, а во вторичной структуре образуют один псевдоузел и одно соединение нескольких стеблей. Метатранскриптомные данные, полученные для бактерий из Тихого океана, показали, что в их клетках IMES-1 РНК в пять раз многочисленнее 5S рРНК, однако причины столь высокого уровня экспрессии неясны. Изучение длинных некодирующих РНК этой группы осложняется еще и тем, что более 400 этих молекул обнаружены не в определенных клетках, а в метагеномных образцах.
В метагеноме рубца коров обнаружили еще одну длинную РНК бактериального происхождения — ROOL (от англ. Rumen-Originating, Ornate, Large). Известно 397 таких молекул, относящихся в основном к бактериям порядков Lactobacillales и Clostridiales. Согласно биоинформатическим предсказаниям, ROOL РНК являются одними из наиболее структурно сложных некодирующих РНК, хотя их последовательности не отличаются консервативностью. ROOL РНК немного напоминают GOLLD РНК: они также нередко тесно связаны с генами тРНК, обнаруживаются у бактериофагов и профагов. Можно предположить, что ROOL и GOLLD РНК выполняют схожие функции, однако значительные структурные различия делают эту гипотезу неправдоподобной.
У бактерий типов фирмикут и актинобактерий описано 1347 так называемых raiA РНК, гены которых очень часто находятся вблизи 5′-нетранслируемых областей генов raiA. Белок raiA связывается с рибосомами и замедляет трансляцию в условиях клеточного стресса. Гены raiA РНК иногда обнаруживают выше (upstream) генов, белковые продукты которых локализуются в периплазматическом пространстве и переносят различные вещества. Возможно, в обоих случаях мотив raiA функционирует как рибопереключатель или другой цис-регуляторный элемент, однако потенциальные лиганды, способные с ним взаимодействовать, пока неизвестны.
Сегодня описано около 20 классов бактериальных некодирующих РНК, функции которых еще не установлены. Мы рассмотрели лишь наиболее изученные и широко распространенные из них. Несомненно, дальнейший прогресс в биоинформатике (особенно метагеномике) позволит выявить еще немало групп этих загадочных молекул, а биохимия, структурная биология и биофизика помогут установить их функции в бактериальных клетках.
Литература
- Kimberly A. Harris, Ronald R. Breaker. (2018). Large Noncoding RNAs in Bacteria. Microbiology Spectrum. 6;
- Luka Bolha, Metka Ravnik-Glavač, Damjan Glavač. (2017). Long Noncoding RNAs as Biomarkers in Cancer. Disease Markers. 2017, 1-14;
- Обо всех РНК на свете, больших и малых;
- Кодирующие некодирующие РНК;
- Загадочное путешествие некодирующей РНК Xist по X-хромосоме;
- Большие дела небольших молекул: как малые РНК дирижируют генами бактерий;
- Малая РНК большого значения;
- Биоинформатика в мире РНК-структур;
- John L. Rinn, Howard Y. Chang. (2012). Genome Regulation by Long Noncoding RNAs. Annu. Rev. Biochem.. 81, 145-166.