Cut&Paste: от транспозонов к эпигеномике
22 февраля 2022
Cut&Paste: от транспозонов к эпигеномике
- 1996
- 0
- 4
Фермент Tn5-транспозаза — универсальный портной ДНК. Он может не только разрезать и сшить концы ДНК, но и вставить в нее нужные заплатки.
-
Автор
-
Редакторы
Темы
Статья на конкурс «Био/Мол/Текст»: Изучение того, как работают бактерии, необходимо не только врачам и инфекционистам. Молекулярные биологи подглядели умение бактерий разрушать чужие ДНК и превратили ферменты рестрикции и CRISPR/Cas9 в инструменты для генной инженерии. Без них уже сложно представить себе современные биологические исследования. Но можно ли, например, укротить транспозоны бактерий, которые могут «прыгать» по ДНК с помощью фермента транспозазы? Этот фермент и сам вырезает фрагмент ДНК, и сам его вставляет в другое место. История Tn5-транспозазы началась со случайного открытия транспозона Tn5 и привела к тому, что транспозаза попала во многие лаборатории, занимающиеся секвенированием нового поколения. С ее помощью можно не просто ускорить подготовку ДНК к секвенированию, но и упростить многие исследования, в том числе и эпигеномные.
Конкурс «Био/Мол/Текст»-2021/2022
 Эта работа заняла второе место в номинации «Академия и бизнес» конкурса «Био/Мол/Текст»-2021/2022.
Эта работа заняла второе место в номинации «Академия и бизнес» конкурса «Био/Мол/Текст»-2021/2022.
Партнер номинации — BIOCAD.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

Генеральный партнер конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
44% нашего генома занимают мобильные генетические элементы, в том числе транспозоны и их остатки. Эти участки ДНК, которые могут передвигаться из одной позиции генома в другую, сыграли огромную роль в эволюции живых организмов, увеличивая генетическое разнообразие как за счет хромосомных перестроек и вставок в геном, так и за счет влияния на экспрессию генов. Присутствие в геноме мобильных элементов, например, привело к появлению плаценты у млекопитающих [1].
Однако эволюционные процессы мы можем наблюдать и в течение нашей жизни. В микробных сообществах бактерии борются за место под солнцем (или ресурсы), выделяя антибиотики. Мы же боремся с болезнетворными бактериями, принимая их. Но бактерии сопротивляются: выживают те клетки, которым удалось либо разрушить антибиотик на подходе, либо не дать ему сработать в клетке. Возникают такие суперспособности благодаря генам устойчивости к антибиотикам.
Первый источник устойчивости — мутации в генах белков, на которые действует антибиотик. Второй же — более необычный — горизонтальный перенос генов. Именно в нем и участвуют мобильные элементы: плазмиды (небольшие кольцевые молекулы ДНК, способные переносить различные гены), бактериофаги (вирусы бактерий) и транспозоны [2], [20].
Транспозон Tn5 — неожиданное открытие
История открытия Tn5-транспозазы началась именно с изучения устойчивости бактерий к антибиотикам. Микробиологи Дуглас Берг и Джулиан Дейвис познакомились в 1974 году во время работы в Университете Женевы. Дуглас Берг изучал устойчивость кишечной палочки к бактериофагам в лаборатории Люсьена Каро. Джулиан Дейвис, который уже имел свою лабораторию в Висконсинском университете в Мэдисоне, приехал в творческий отпуск. Он исследовал феномен устойчивости к антибиотикам, R-факторы (плазмиды, несущие гены устойчивости) и их эволюцию. Берг часто беседовал с Дейвисом: его заинтересовала эта тема. Он задумал создать штаммы бактериофага λ, которые содержат гены устойчивости к антибиотикам: так бы ему было удобно отслеживать, как фаги инфицируют бактерии (если бактерии приобрели устойчивость к антибиотику — инфекция была успешной).
Однако результат его запланированного эксперимента оказался неожиданным [3].
Сейчас такую работу можно сделать довольно просто: вырезать ген устойчивости к антибиотикам с помощью ферментов рестрикции, ими же разрезать ДНК фага, а затем соединить концы последовательности гена и фага с помощью лигирования. Но в 1974 году в руках Берга еще не было таких инструментов, ему пришлось работать напрямую с бактериальными клетками.
Расскажем немного подробнее: λ-фаг может встраиваться в определенные последовательности ДНК — как геномной, так и плазмидной. Когда профаг активируется и начинает репликацию, новые фаговые частицы могут содержать гены бактерий, например гены устойчивости к антибиотикам. Получить такие частицы у Берга действительно получилось, но при анализе его ожидал сюрприз. Вставка произошла не в запланированных последовательностях. По бокам вставки нашлись два практически идентичных участка инвертированных повторов. Это очень напоминало недавно открытые транспозоны бактерий: годом ранее описали первый подобный транспозон, который нес устойчивость к ампициллину [4]. А транспозоны как раз могли перескакивать из одного участка ДНК в другой, в том числе и от плазмиды к фагу [5]. Транспозону, открытому Бергом, дали название Tn5.
Транспозаза Tn5
Транспозон Tn5 состоит из двух инвертированных последовательностей IS50 (IS, inverted sequences), которые окружают гены устойчивости к канамицину, блеомицину и стрептомицину [6]. Внешние (OE, outer end) и внутренние (IE, internal end) последовательности IS важны для работы транспозона, так как связываются с ферментом Tn5-транспозазой (Tnp), который закодирован в правой IS (IS50R) (рис. 1). Это единственный фермент, который нужен для активности транспозона.
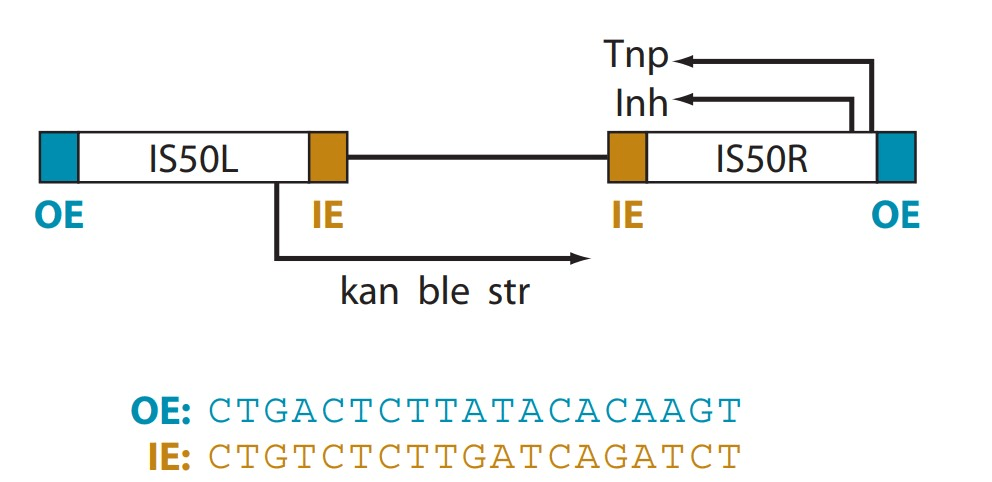
Рисунок 1. Строение транспозона Tn5. Транспозон Tn5 состоит из двух почти одинаковых инвертированных последовательностей: IS50L и IS50R, которые окружают три гена устойчивости к антибиотикам: канамицину (kan), блеомицину (ble) и стрептомицину (str). Каждая инвертированная последовательность имеет внешний (OE) и внутренний (IE) концы. В правой инвертированной последовательности закодирована транспозаза (Tnp), а также ингибитор транспозиции (Inh). В левой инвертированной последовательности закодированы усеченные варианты этих белков.
[7], рисунок c изменениями
Механизм работы транспозазы оказался очень простым (рис. 2): фермент разрезает ДНК, вырезая транспозон по инвертированным последовательностям, а потом вставляет транспозон туда, где присутствует девятинуклеотидная предпочтительная последовательность. Впрочем, последовательность имеет большое количество вариантов, поэтому встроиться транспозон может во многие места генома.
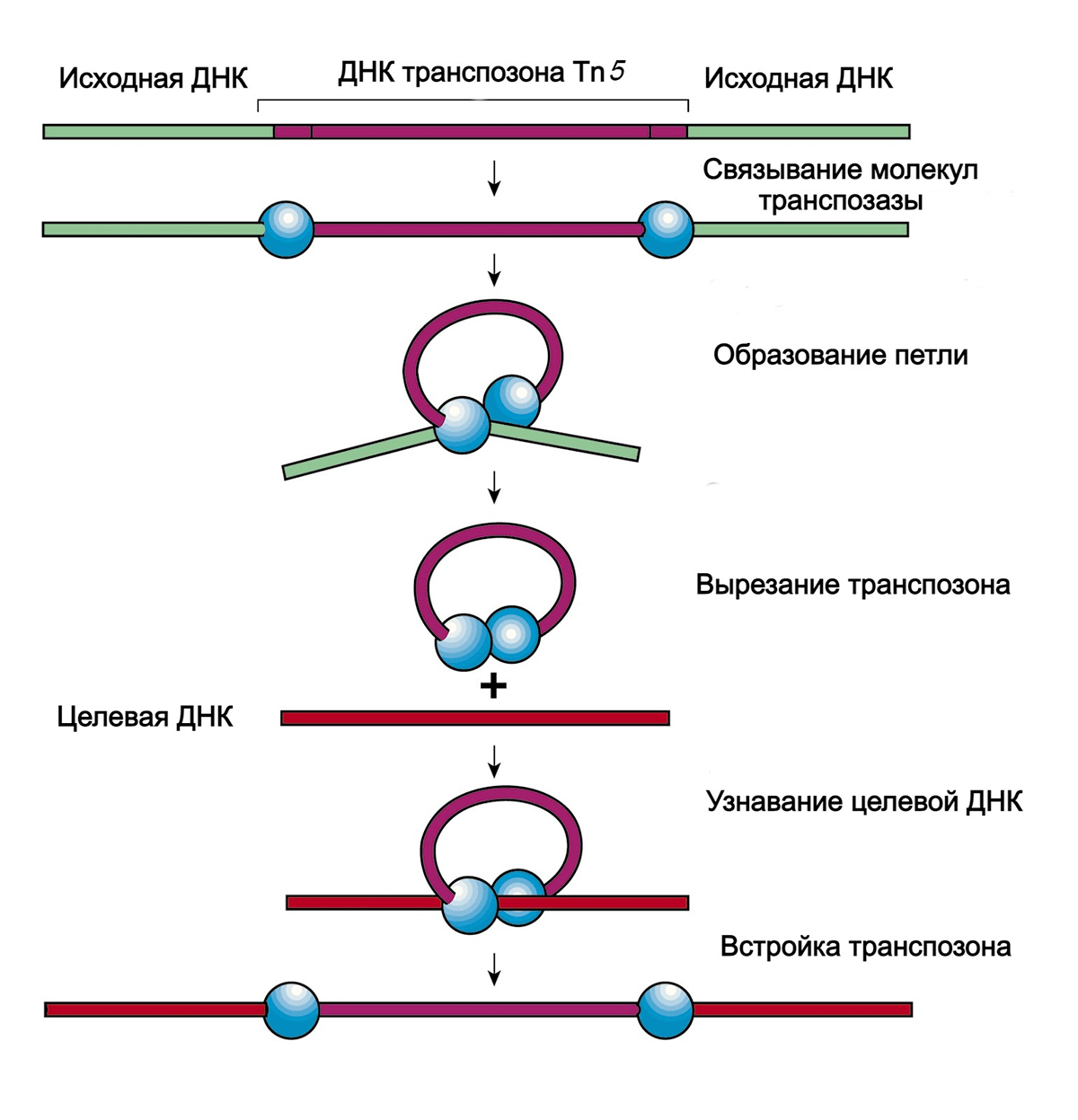
Рисунок 2. Механизм работы Tn5-транспозазы
сайт Висконсинского университета, рисунок с изменениями
Приручить Tn5
Дорогу Tn5 в индустрию дал Висконсинский университет в Мэдисоне, куда Дейвис вернулся после Женевы. Он пригласил Берга присоединиться и продолжить работу. Однако большую часть исследований Tn5, которые оказались важными для применения в биотехнологии, провел профессор Висконсинского университета Уильям Резникофф. Он познакомился с Бергом и его открытиями и заинтересовался изучением транспозонов бактерий. Вместе они охарактеризовали еще один транспозон — Tn10, а затем обнаружили Tn5-транспозазу, описали, как она работает и регулируется. Они также показали, что вставка транспозона Tn5, в целом, редкое явление. Причина этому простая: если бы транспозоны регулярно «прыгали» по геному, это увеличило бы мутационную нагрузку до неприемлемой степени. Частота транспозиции Tn5 составляет 10–5 событий на клетку на поколение (в целом, среднее значение для транспозонов E.coli [8]). Активность фермента подавляет ингибитор транспозазы — ее мутантная форма, которая считывается с той же рамки считывания и связывается с транспозазой и не дает ей разрезать ДНК.
Лаборатория Резникоффа занялась поиском более активных форм Tn5-транспозазы и к 1998 году пришла к заветной комбинации (рис. 3). Чтобы связаться между собой, молекулам транспозазы не всегда нужна общая ДНК. Замена лейцина на пролин в позиции 372 мешала образованию таких комплексов. Замена глутамата на лизин в позиции 54 усиливала связь фермента с ДНК транспозона. Наконец, аланин вместо метионина в позиции 56 предотвращал синтез ингибитора транспозазы в их с транспозазой общей рамке считывания. Первоначально исследователи просто хотели изучить, какие механизмы регулируют транспозицию и как один фермент обеспечивает весь этот сложный процесс. In vitro это было сделать проще, чем в бактериях [9]. Первые исследования включали только два основных компонента: очищенную активную транспозазу и целевую ДНК. Фермент успешно разрезал плазмиды на небольшие фрагменты, что и хотели от нее авторы. Но может ли такая активная транспозаза переносить участки ДНК?
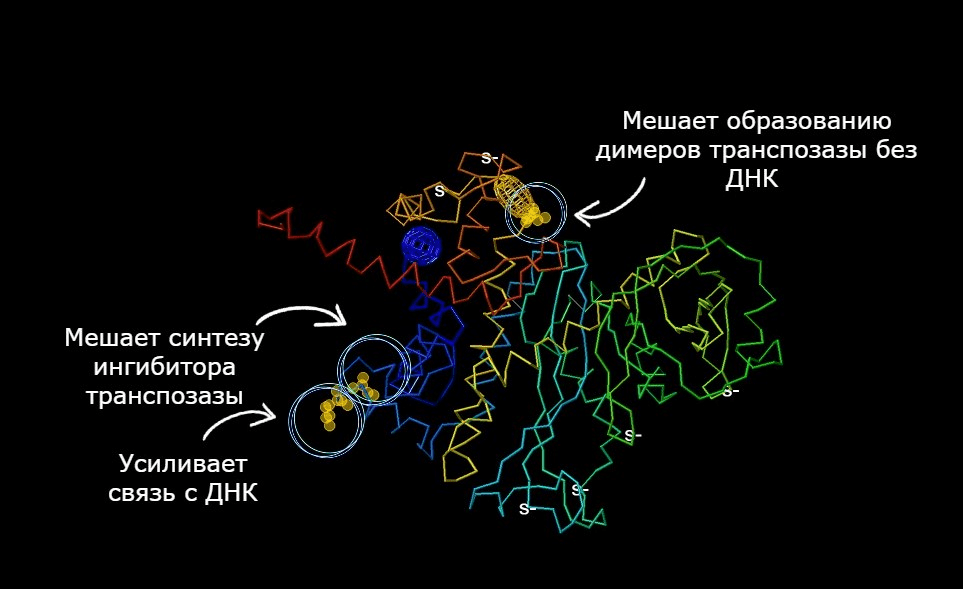
Рисунок 3. Мутации, усиливающие активность Tn5-транспозазы. Иллюстрация создана на основе PDB-структуры 1MM8 с помощью сервиса FirstGlance in Jmol. Кружками обозначены атомы аминокислот, которые были изменены в активной Tn5-транспозазе по сравнению с транспозазой дикого типа.
После открытия команды Резникоффа свойствами активной транспозазы заинтересовалась небольшая биотехнологическая компания Epicenter Technologies в Мэдисоне, основанная выпускниками Висконсинского университета. Она занималась продажей различных ферментов и реагентов для молекулярной биологии, но потенциал фермента с новыми функциями их заинтересовал. Ведь можно было бы использовать один фермент и для разрезания ДНК, и для вставки участков ДНК. Это заменяло бы уже привычный подход с рестрикцией и лигированием — тот самый, который еще не смог использовать Берг в своих экспериментах.
Совместно с лабораторией Резникоффа компания Epicenter Technologies разработала метод вставки нужных последовательностей ДНК, например, генов устойчивости к антибиотикам [10]. Они добавили в реакцию еще один компонент: ДНК-вставку. Чтобы она напоминала транспозон, помимо целевого гена вставка должна была содержать на концах повторы из 19 пар оснований, которые свяжутся с транспозазой. Однако они не эквивалентны внешним и внутренним последовательностям IS, а заменены на модифицированную, мозаичную последовательность. Сочетание этой последовательности и транспозазы с повышенной активностью увеличивают эффективность встраивания в 1000 раз по сравнению с обычной транспозазой. В 1999 году Epicenter Technologies выпустила несколько продуктов, которые основаны на работе Tn5-транспозазы под брендом EZ::TN.
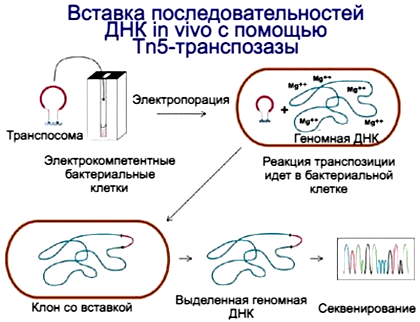
Рисунок 4. EZ::TN можно использовать для вставки ДНК в бактерии
Вставка последовательностей ДНК с помощью Tn5-транспозазы оказалась удобным инструментом для многих подходов. In vitro можно было вставлять в плазмиды гены устойчивости без работы с рестриктазами и лигированием. EZ::TN5 можно было использовать и для мутагенеза in vivo (рис. 4). Представьте, что некая бактерия мало изучена и мы хотим узнать, какие гены в ней отвечают за какие функции. Случайные вставки транспозона могут нарушить работу генов. Если определить, какой ген нарушен, и соотнести с фенотипом бактерий, то можно определить, для чего он нужен. Для такого подхода используют различные виды транспозонов бактерий, но в каждом случае процесс нужно оптимизировать, в том числе и под соответствующую группу бактерий. В случае Tn5-транспозазы сперва собирали ее комплекс с ДНК, содержащий чаще всего вставку гена устойчивости к антибиотикам (чтобы потом отобрать трансформированные клоны). Такой комплекс — транспосома — с помощью электрического тока переносится в бактерии. В дальнейшем место вставки можно определить с помощью секвенирования.
Но почему нельзя просто секвенировать ДНК неизвестных видов бактерий, как это делается сейчас? Можно, но только после того, как мы вступили в эру секвенирования нового поколения.
Tn5 ловит волну
К 2009 году технологии секвенирования ДНК нового поколения стали доступны многим. Компания Roche уже коммерциализировала технологию пиросеквенирования, а калифорнийская компания Illumina уже купила британскую компанию Solexa, которая разработала метод секвенирования с помощью флуоресцентно меченых нуклеотидов.
Подготовка ДНК к секвенированию нового поколения — многоступенчатый процесс, который включает в себя фрагментацию молекул ДНК, а также пришивание (лигирование) адаптерных последовательностей, которые связывают молекулы ДНК с поверхностью, где будет идти реакция. Полученный набор молекул называют библиотекой. Подробней об этом процессе можно прочитать в статье «Биомолекулы» «12 методов в картинках: секвенирование нуклеиновых кислот» [11].
Большой минус этого подхода — он занимает до двух дней. Как можно ускорить этот процесс? Ответом на этот вопрос стал новый продукт Epicenter Technologies под названием Nextera. Он основан на способности транспосом вставлять фрагменты ДНК in vitro. В этот раз вставляли именно те адаптерные последовательности, которые нужны для секвенирования на платформе Illuminа. Тагментирование (так назвали этот подход) заменяло и механическое фрагментирование ДНК, и реакцию лигирования адаптеров (рис. 5). Наборы Nextera позволяли снизить время подготовки библиотек до 90 минут (на практике, в зависимости от количества образцов и ДНК в них, — до 3,5 часов). На такой прорыв не могла не обратить внимание сама компания Illumina, и в 2011 году купила компанию Epicenter Technologies за 90 миллионов долларов, впрочем, оставив под ее контролем все продукты для работы с бактериями. Они по-прежнему продают наборы через компанию Lucigen под своим именем. Однако наборы для приготовления библиотек Illumina DNA Prep продаются напрямую через компанию Illumina.
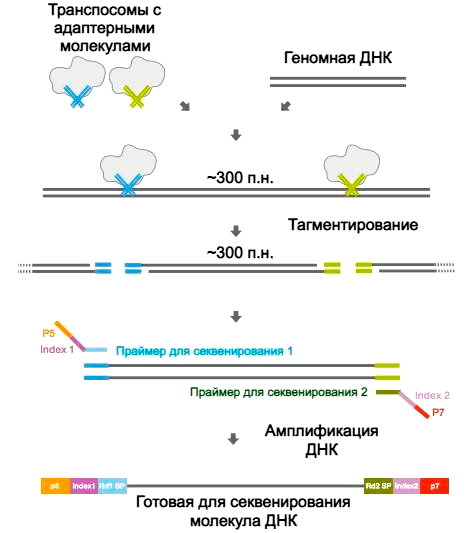
Рисунок 5. Подготовка библиотек с помощью наборов Nextera. Для подготовки библиотеки требуется ДНК (возможна работа с количествами от 1 до 500 нг) и транспосомы — комплекс транспозазы с двуцепочечными олигонуклеотидами. Одна цепь состоит из мозаичного повтора из 19 нуклеотидов, который обеспечит встраивание в геномную ДНК, а вторая комплементарна повтору, а также праймеру, необходимому для дальнейшей ПЦР-амплификации. При температуре 37 °C начинается реакция тагментации, при которой молекулы Tn5-транспозазы одновременно фрагментируют ДНК и вставляют олигонуклеотиды на концах каждого фрагмента. Реакцию останавливают и очищают реакционную смесь от транспозазы и буфера. Полученные молекулы ДНК используют для ПЦР-реакции с праймерами, необходимыми для секвенирования. В итоге получается готовая библиотека за минимальное количество времени.
сайт компании Illumina, рисунок с изменениями
Этап ПЦР — одна из основных проблем всех библиотек. Причина простая: разные фрагменты ДНК амплифицируются с разной эффективностью (хуже — GC-богатые). Но если под рукой у исследователей есть достаточное количество ДНК, то с помощью транспосом можно присоединить адаптер напрямую, без дальнейшей амплификации.
Одна из проблем приготовления библиотек с помощью метода Nextera — секвенирование линейных геномов (например, геномов фагов). Транспосома разрезает целевую ДНК, вставляя олигонуклеотиды, но добавить адаптер в самый конец исходной молекулы ДНК невозможно, останется участок, который не покрыт секвенированием. Поэтому получить точную последовательность концевых участков очень сложно.
Тем не менее приготовление библиотек с помощью тагментирования — это простой, удобный и быстрый метод. Кроме того, все шаги протокола проходят в одной пробирке, и это снижает риск потери образца, что ценно, если исходного материала очень мало.
Приготовить такие библиотеки можно как из геномной ДНК (в том числе из крови, слюны и тканей, подготовленных для гистологических исследований) и ПЦР-продуктов, так и из комплементарной ДНК (то есть РНК, обратно транскрибированной в ДНК), адаптировав протокол для РНК-секвенирования.
Транспосомы с соответствующими адаптерами можно использовать и в подготовке библиотек для других систем. Изначально Epicenter Technologies предлагали подготовку библиотек и для пиросеквенирования, но сейчас коммерческих решений для других платформ нет. Однако у компании Thermo Fisher Scientific, которая владеет технологией секвенирования Ion Torrent, есть своя система для подготовки библиотек. Она основана на транспозазе MuA бактериофага Мю, который встраивается в геном бактерий примерно таким же образом, что и транспозон Tn5.
Tn5-транспозазу можно купить и отдельно, причем это не эксклюзив компании Illumina, а секрет активной транспозазы — вовсе не секрет. Мутации и последовательность мозаичного повтора опубликованы, поэтому при желании можно производить белок самому, а также синтезировать необходимые для эксперимента олигонуклеотиды. На основе фермента есть и некоммерческие разработки, в том числе и для секвенирования ДНК одиночных клеток.
Чтобы обеспечить достаточное количество ДНК, геном клетки амплифицируют (то есть увеличивают количество копий с помощью ПЦР). Однако этот метод имеет проблему. Так как количество молекул увеличивается экспоненциально, да еще и с неизвестной степенью эффективности для каждого фрагмента ДНК, то и ошибки ПЦР тоже увеличиваются экспоненциально (рис. 6).
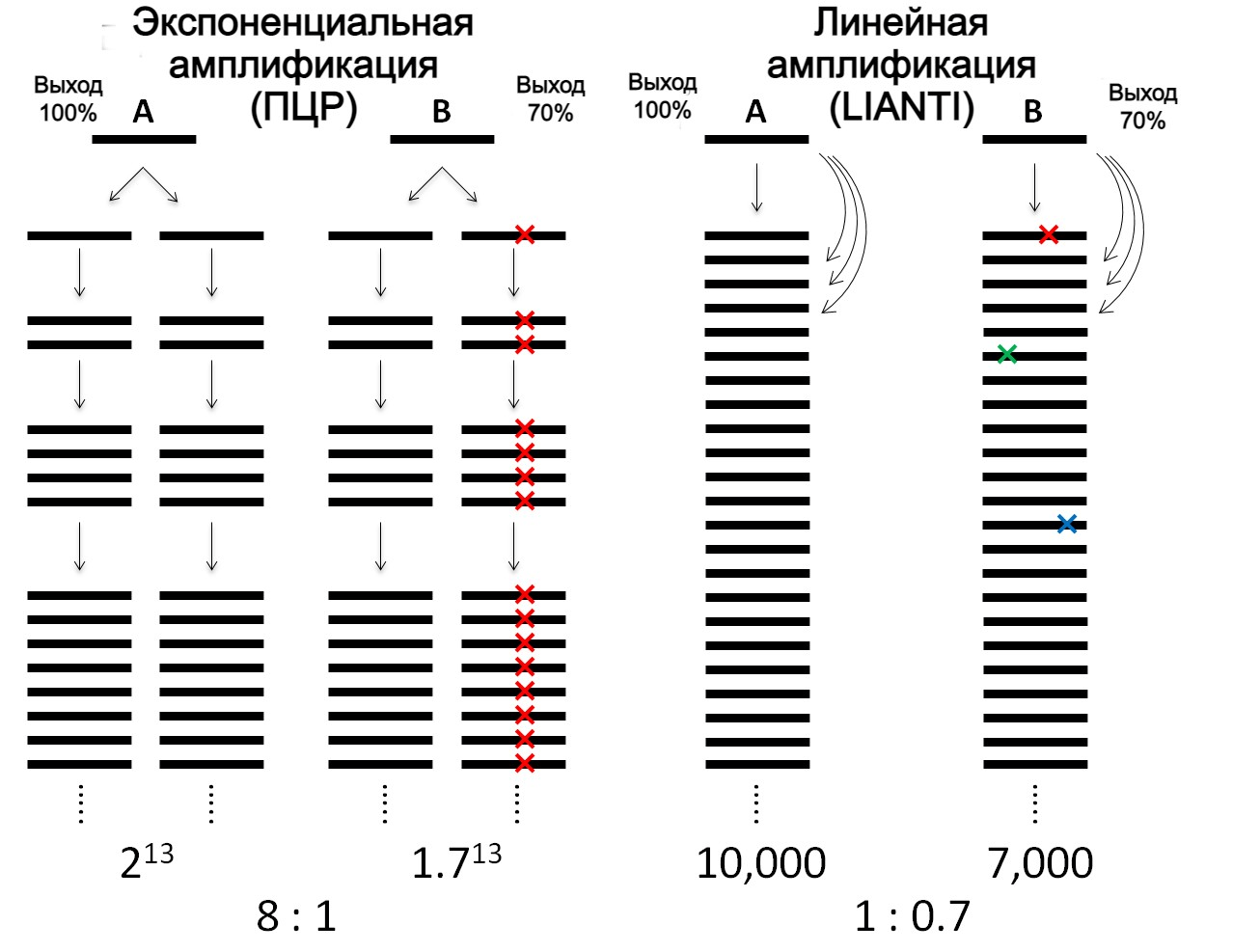
Рисунок 6. Сравнение экспоненциальной и линейной амплификаций. Для изучения генома одиночной клетки мы разбиваем его на фрагменты и амплифицируем. Фрагменты А и В имеют разную эффективность амплификации (например, из-за содержания GC-нуклеотидов) — 100% и 70%. Значит, их соотношение будет сильно различаться после завершения ПЦР, где амплификация экспоненциальная. Кроме того, даже самая лучшая ДНК-полимераза совершает ошибки. При частоте ошибок 10–7 при амплификации генома человека (3×109 нуклеотидов) появится около 300 ошибок только после первого цикла ПЦР. В каждом новом случае число ошибок увеличится. В итоге у нас получится группа молекул с «искусственным» однонуклеотидным полиморфизмом, вдобавок к настоящим. В случае линейной амплификации ошибки будут, но их количество не будет увеличиваться так же, как в случае экспоненциальной. Кроме того, ошибки будут возникать произвольно, и их можно будет отличить от настоящих полиморфизмов. А соотношение разных фрагментов будет линейно зависеть от эффективности амплификации.
сайт лаборатории Сяоляна Се (Xiaoliang Sunney Xie), рисунок с изменениями
Кроме того, фрагменты с разным числом копий генов, а также различающиеся одним нуклеотидом, плохо улавливаются. Избежать этого можно, используя линейную амплификацию: даже если у фрагментов ДНК будет разная эффективность амплификации, все потомки молекулы будут похожи (с поправкой на ошибки ПЦР).
Метод LIANTI (linear amplification via transposition insertion), основанный на использовании транспосом, разрешает эту проблему [12]. Транспосомы содержат олигонуклеотид, который помимо мозаичного повтора содержит T7-промотор. При тагментировании ДНК фрагментируется, а вставка промотора необходима для последующей реакции транскрипции in vitro, которая увеличит число копий фрагментов без ПЦР. После завершения реакции полученные молекулы РНК с помощью обратной транскрипции переводят опять в ДНК, а затем уже готовят библиотеки стандартными методами. Впрочем, пока этот метод еще не распространен.
Тагментирование и эпигенетика
Где еще можно использовать тагментированную ДНК? Эпигеномные исследования позволяют ответить нам на вопрос, как связаны активность генов и организация генома с различными функциями клеток и свойствами организма. Они также требуют секвенирования различных фрагментов ДНК [13]. Бисульфитное секвенирование и иммунопреципитация хроматина с последующим секвенированием (ChIP-seq) — самые распространенные методы изучения эпигенома.
С помощью бисульфитного секвенирования ДНК можно изучить распределение 5-метилцитозина (и других подобных модификаций) в геноме, чтобы узнать больше об активности генов в клетках. Для приготовления библиотек в таком эксперименте можно использовать специальные адаптеры с метилированными цитозинами в комплексе с Tn5-транспозазой. Библиотеку можно получить из 10 нг ДНК, что в 100 меньше, чем требуют стандартные методы [14]. А исследователи из Университета Чикаго смогли изучить профиль 5-гидроксиметилирования (продукта окисления 5-метилцитозина) в популяции из 1000 клеток [15]. Это колоссальная помощь при работе, где количество материала ограничено (например, с эмбрионами или участками головного мозга).
Помимо изучения модификаций ДНК, эпигеномные исследования исследуют распределение белков хроматина (например, гистонов и их модификаций, факторов транскрипции) с различными участками ДНК. Метод ChIPmentation — модификация стандартного метода ChIP-seq, который чаще всего используется для таких экспериментов [16]. Для этого хроматин исследуемых клеток фрагментируют (чаще всего ультразвуком), а затем очищают фрагменты, которые взаимодействуют с целевым белком. Различие между методами состоит в том, что при ChIPmentation адаптерные последовательности добавляются с помощью Tn5-транспозазы прямо к очищенным фрагментам хроматина, без необходимости сперва выделять ДНК (рис. 7). Это экономит время, потраченное на эксперимент. Единственный недостаток — так как реакция основана на работе фермента, нужно учитывать и количество ДНК, и размер исходных фрагментов хроматина. Слишком короткие фрагменты могут быть слишком фрагментированы, и библиотеки получатся плохого качества. Метод коммерциализирован компанией Diagenode.
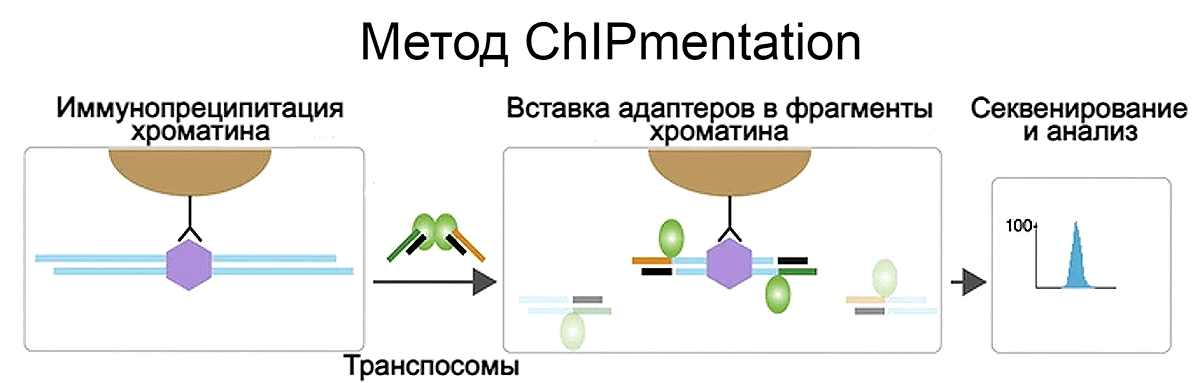
Рисунок 7. Схема изучения хроматина с помощью метода ChIPmentation
[16], рисунок с изменениями
Кроме того, появились новые методы, которые основаны исключительно на работе Tn5-транспозазы. Один из них — метод ATAC-seq (assay for transposase-accessible chromatin using sequencing, рис. 8) [17]. Tn5-транспозаза вставляет транспозоны туда, где ДНК бактерий слабо взаимодействует с белками нуклеоида. Авторы исследования решили проверить, может ли Tn5-транспозаза взаимодействовать с такими же участками в геноме эукариот: промоторами и регуляторными элементами, соответствующими открытому, активному хроматину. Раньше эти исследования можно было проводить только с помощью иммунопреципитации хроматина или, например, методом FAIRE-seq, но для них требуется большое количество материала (от 10 тысяч клеток).
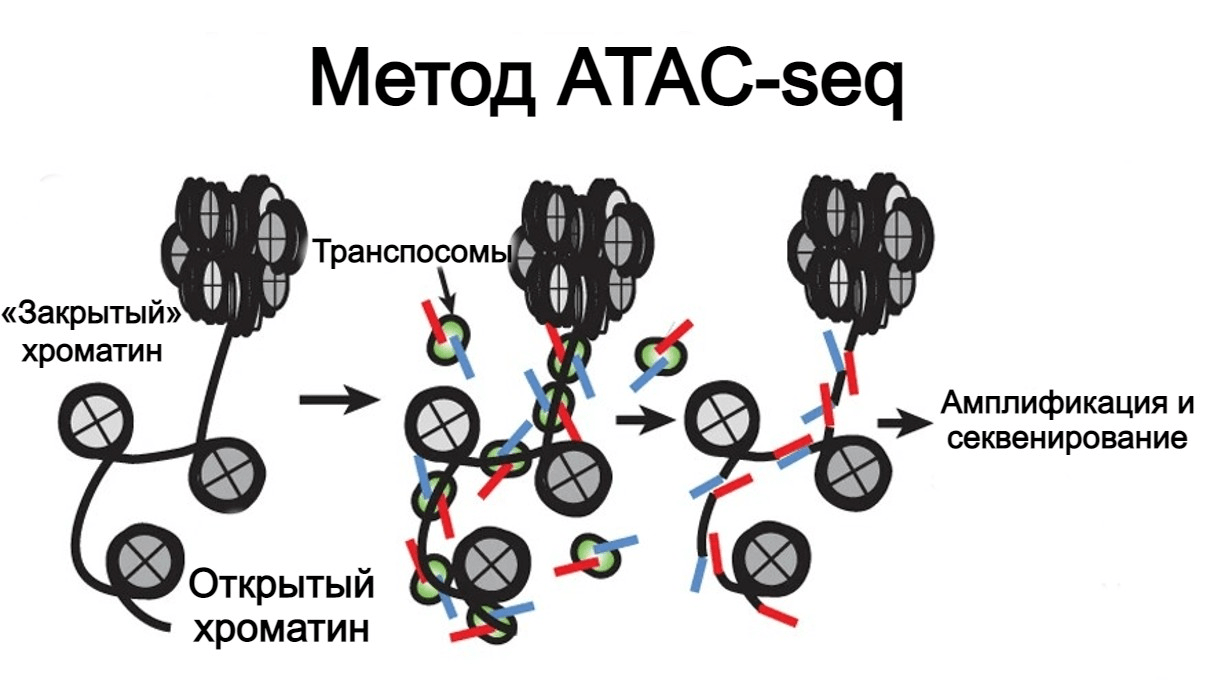
Рисунок 8. Схема изучения хроматина с помощью метода ATAC-seq
[17], рисунок с изменениями
Метод ATAC-seq прост: достаточно выделить ядра из исследуемых клеток и инкубировать их с транспосомами, содержащими адаптерные последовательности.
Транспосомы фрагментируют и метят доступные участки хроматина, которые затем очищают от белков и используют для приготовления библиотеки. Для этого метода требуется всего от 5000 клеток. Кроме того, метод адаптирован для исследования одиночных клеток [18]. Сейчас наборы для ATAC-seq предлагает несколько компаний, включая уже упомянутый Diagenode, Active Motif и других лидеров в продаже эпигенетических реагентов.
Метод CUT&Tag (Cleavage Under Targets and Tagmentation [19]) основан на модифицированной версии метода CUT&RUN (Cleavage Under Targets and Release Using Nuclease). Как и ChIP-seq, они позволяют изучить участки ДНК, которые связаны с определенными белками. Тем не менее они, в отличие от ChIP, не требуют подготовки хроматина. Оба протокола начинаются с узнавания антителами белков, которые интересны исследователю, в неповрежденных ядрах. Но следующая ступень — добавление нуклеазы (микрококковой нуклеазы в случае CUT&Run и Tn5-транспозазы в случае CUT&Tag). В обоих ситуациях используют фьюжн-белки с белком А (protein A), который связывается с антителом и обеспечивает направленное «разрезание» фрагментов ДНК, взаимодействующих с белками.
Последующие действия примерно такие же, как для ATAC-seq: транспосомы фрагментируют и метят доступные участки хроматина, которые затем очищают от белков и используют для приготовления библиотеки (рис. 9). Применение Tn5-транспозазы не только уменьшает время, потраченное на подготовку библиотеки, но и количество клеток, необходимых для эксперимента, из-за активности Tn5-транспозазы (5 тысяч клеток достаточно для одного эксперимента).
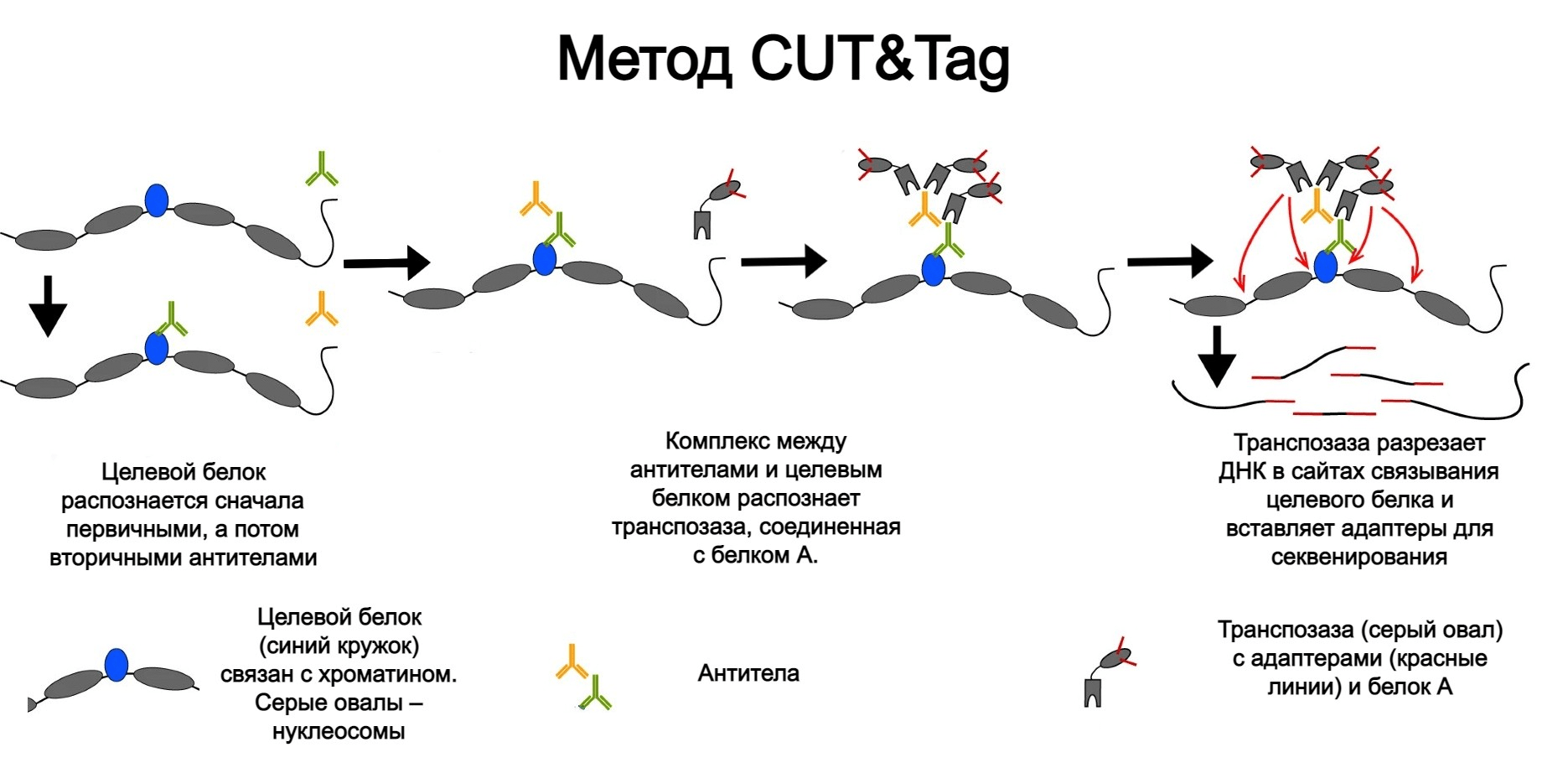
Рисунок 9. Схема изучения хроматина с помощью метода CUT&Tag
[19], рисунок с изменениями
Метод CUT&Tag, разработанный лабораторией Стивена Хенникофа, запатентован, и реагенты для него доступны для покупки. Впрочем, сама команда Хенникофа открыта для сотрудничества с лабораториями с целью оптимизации этого протокола.
Заключение
История Tn5-транспозазы началась с исследования устойчивости бактерий к фагам и продолжилась сначала упорным изучением механизмов ее работы лабораторией Уильяма Резникоффа, а потом успешной коммерциализацией этого открытия. Сейчас наборами для подготовки библиотек для секвенирования пользуются везде, где стоят секвенаторы Illumina. И учитывая, что эта компания не собирается сдавать свои позиции лидера рынка секвенирования, тагментирование останется важным методом для изучения генетических и эпигенетических процессов. А значит, история Tn5-транспозазы может продолжиться тысячами новых открытий, которые помогут изменить мир к лучшему.
Литература
- D. Emera, G. P. Wagner. (2012). Transposable element recruitments in the mammalian placenta: impacts and mechanisms. Briefings in Functional Genomics. 11, 267-276;
- Антибиотики vs Бактерии. «Война Бесконечности» или всему есть предел?;
- Douglas E Berg. (2017). Julian Davies and the discovery of kanamycin resistance transposon Tn5. J Antibiot. 70, 339-346;
- R. W. Hedges, A. E. Jacob. (1974). Transposition of ampicillin resistance from RP4 to other replicons. Molec. Gen. Genet.. 132, 31-40;
- D. E. Berg, J. Davies, B. Allet, J. D. Rochaix. (1975). Transposition of R factor genes to bacteriophage lambda.. Proceedings of the National Academy of Sciences. 72, 3628-3632;
- E.-A. Auerswald, G. Ludwig, H. Schaller. (1981). Structural Analysis of Tn5. Cold Spring Harbor Symposia on Quantitative Biology. 45, 107-113;
- William S. Reznikoff. (2008). Transposon Tn5. Annu. Rev. Genet.. 42, 269-286;
- Ana Sousa, Catarina Bourgard, Lindi M. Wahl, Isabel Gordo. (2013). Rates of transposition in Escherichia coli. Biol. Lett.. 9, 20130838;
- Igor Yu Goryshin, William S. Reznikoff. (1998). Tn5 in Vitro Transposition. Journal of Biological Chemistry. 273, 7367-7374;
- Igor Y. Goryshin, Jerry Jendrisak, Les M. Hoffman, Ronald Meis, William S. Reznikoff. (2000). Insertional transposon mutagenesis by electroporation of released Tn5 transposition complexes. Nat Biotechnol. 18, 97-100;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Chongyi Chen, Dong Xing, Longzhi Tan, Heng Li, Guangyu Zhou, et. al.. (2017). Single-cell whole-genome analyses by Linear Amplification via Transposon Insertion (LIANTI). Science. 356, 189-194;
- Молекулы и эпигеном ;
- Andrew Adey, Jay Shendure. (2012). Ultra-low-input, tagmentation-based whole-genome bisulfite sequencing. Genome Res.. 22, 1139-1143;
- Dali Han, Xingyu Lu, Alan H. Shih, Ji Nie, Qiancheng You, et. al.. (2016). A Highly Sensitive and Robust Method for Genome-wide 5hmC Profiling of Rare Cell Populations. Molecular Cell. 63, 711-719;
- Christian Schmidl, André F Rendeiro, Nathan C Sheffield, Christoph Bock. (2015). ChIPmentation: fast, robust, low-input ChIP-seq for histones and transcription factors. Nat Methods. 12, 963-965;
- Jason D Buenrostro, Paul G Giresi, Lisa C Zaba, Howard Y Chang, William J Greenleaf. (2013). Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10, 1213-1218;
- Jason D. Buenrostro, Beijing Wu, Ulrike M. Litzenburger, Dave Ruff, Michael L. Gonzales, et. al.. (2015). Single-cell chromatin accessibility reveals principles of regulatory variation. Nature. 523, 486-490;
- Hatice S. Kaya-Okur, Steven J. Wu, Christine A. Codomo, Erica S. Pledger, Terri D. Bryson, et. al.. (2019). CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun. 10;
- Мобильные генетические элементы прокариот: стратификация «общества» бродяжек и домоседов.