Молекулы и эпигеном
09 апреля 2021
Молекулы и эпигеном
- 7793
- 0
- 46
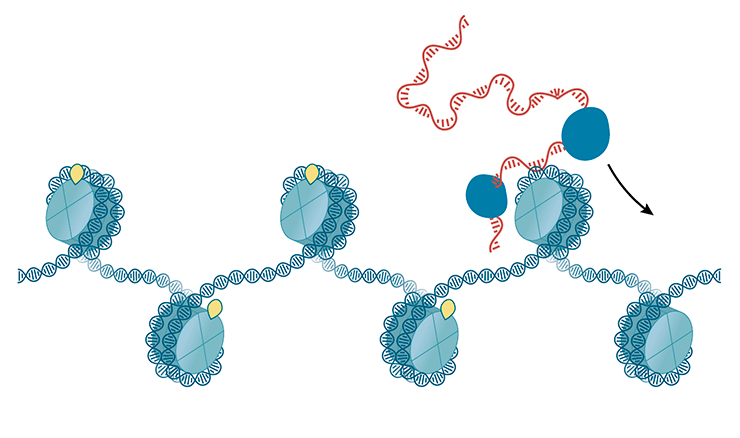
Белки в комплексе с длинной некодирующей РНК навешивают метки на гистоны
иллюстрация Михаила Гурьева
-
Автор
-
Редакторы
-
Иллюстратор
Упоминаний об эпигенетике вы не встретите в школьном учебнике биологии, а ведь эта наука рассказывает, как клетка реализует свой генетический потенциал, «вылепливая» из одного и того же «теста» (последовательности ДНК) совершенно разные «пироги»: клетки эпителия, легкого, нервной ткани и многие другие. Эпигенетика изучает хроматин: ДНК и ассоциированные с ней РНК и белки, а также взаимодействия между ними. В этой статье, которой мы открываем спецпроект по эпигенетике, вы познакомитесь с основными игроками эпигенетики — молекулами хроматина. Много внимания мы уделим методам его изучения — для более глубокого понимания того, как ученые делают открытия в этой области.
Эпигенетика
Эпигенетика — с одной стороны, молодая и бурно развивающаяся наука. С другой стороны, в этой науке уже сложился фундамент основных понятий и концепций. Цель спецпроекта — рассказать об основных концепциях эпигенетики просто, но в то же время со всех сторон. Читатель узнает, какие молекулы и структуры стоят за передачей наследственной информацией и какими механизмами осуществляется эта передача. Часть контента позаимствована из блога автора Friends of Chromatin.

Генеральный партнер спецпроекта — компания «Диаэм».
Это крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

Партнер этой статьи — «СкайДжин».
Пять причин выбрать SkyGen:
- Доступ к высококачественной продукции для молекулярной биологии.
- Высококвалифицированная научная поддержка.
- Быстрая логистика и складская программа.
- Удобное и взаимовыгодное сотрудничество.
- Адекватные цены.
Введение
Молекулы и клеточное ядро
По принятой биохимической классификации, в клетках есть молекулы четырех основных типов:
- нуклеиновые кислоты — дезоксирибонуклеиновая (ДНК) и рибонуклеиновая (РНК);
- белки;
- липиды [5];
- углеводы .
Об удивительных углеводах читайте спецпроект «Гликобиология». — Ред.
Кроме этого, клетки наполнены молекулами меньшего размера, и даже атомами (например ионами). Они выполняют две основные функции: регуляторов для основных молекул (увеличивая или уменьшая их химическую активность) и «строительных блоков» для них. Роль «строителей» играют белки — например, они создают двухцепочечную молекулу ДНК из дезоксирибонуклеотидных остатков. Но и сами белки тоже нужно строить: они создаются специальными «машинами» из белков и РНК — рибосомами, где роль главного строителя играет уже РНК.
Итак, молекулы большого размера состоят из меньших строительных блоков, которые часто состоят из еще меньших. ДНК состоит из дезоксирибонуклеотидных остатков, РНК — из рибонуклеотидных, белки — из аминокислот, углеводы — из моносахаридов (которые, кстати, входят и в состав нуклеотидов). Липиды довольно разнообразны и являются единственной химической категорией, которую классифицируют не по строению, а по свойствам: они не смешиваются с водой (на чем основан важный для молекулярной биологии гидрофобный эффект [6]).
Клетки содержат органеллы (как бы очень маленькие клеточные органы). Клеточное ядро — одна из важнейших органелл (чаще всего оно шарообразное или в форме эллипсоида), отделенная ядерной оболочкой от остальной части клетки — цитоплазмы. В ядре происходит часть процессов, лежащих в основе «центральной догмы» молекулярной биологии: ДНК ⇆ РНК → белок (рис. 1).
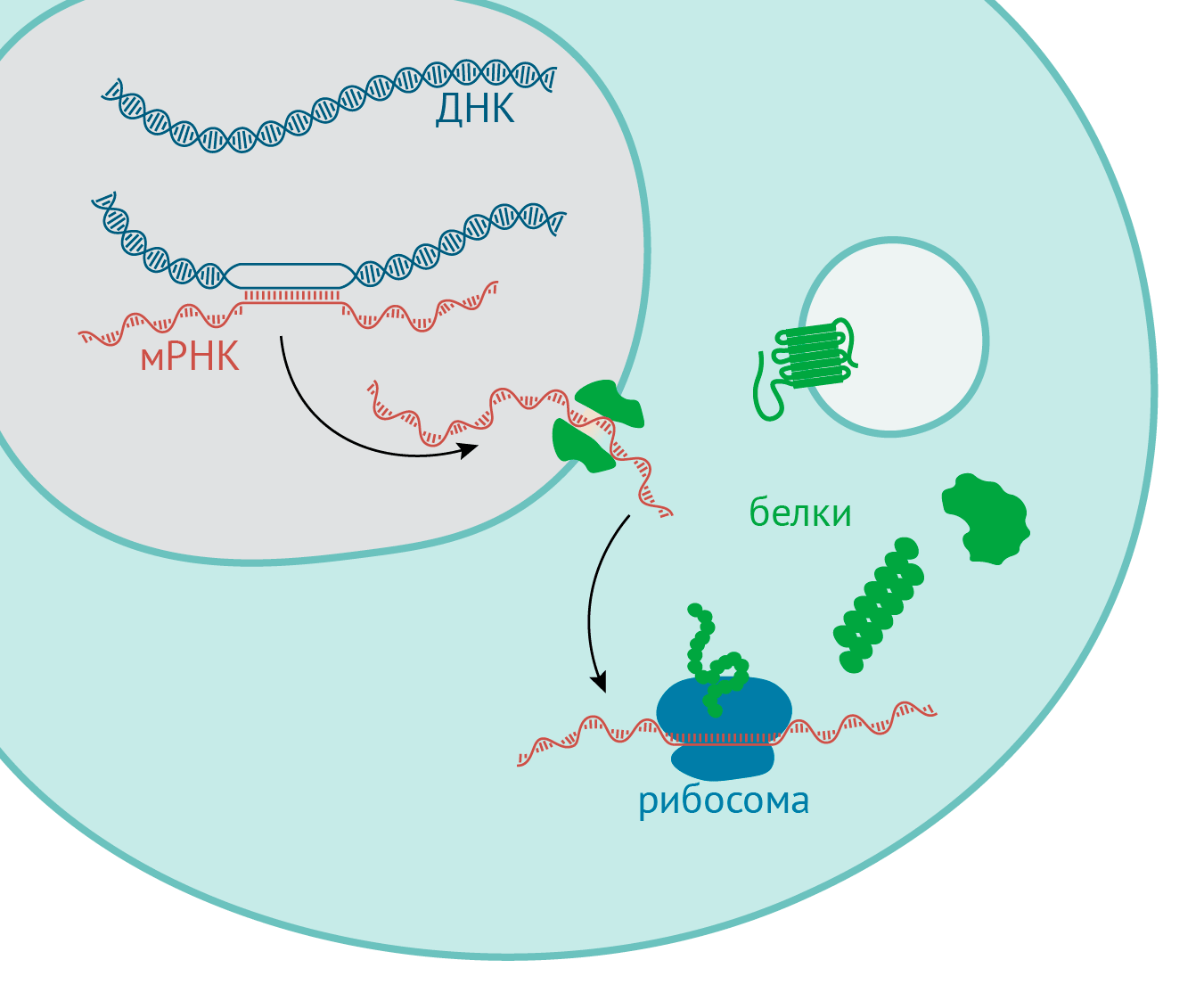
Рисунок 1. Центральная догма молекулярной биологии. Линейная последовательность ДНК переводится в сложный трехмерный белок через промежуточный продукт — РНК. Только часть этого процесса (транскрипция, или перевод ДНК в РНК) происходит в ядре. После этого РНК, если она кодирует белок (а может и не кодировать), переносится в цитоплазму и транслируется (переводится) в белок на рибосомах, причем каждые три нуклеотида кодируют одну аминокислоту. Рибосомная РНК синтезируется и начинает собираться вместе с другими белками в комплексы тоже в ядре. Интересно, что у некоторых вирусов и «прыгающих» элементов ДНК, в жизни которых есть стадия РНК, возможен обратный перевод РНК в ДНК (обратная транскрипция). Но обратного перевода белков в РНК пока не обнаружили.
иллюстрация Михаила Гурьева
ДНК и РНК
ДНК состоит из нуклеотидных остатков. Они, в свою очередь, строятся из трех фрагментов: остатков сахара (дезоксирибозы), трифосфата и азотистых оснований. Несмотря на поразительную длину человеческой ДНК (3,2 миллиарда пар нуклеотидов в каждой клетке, что в развернутом состоянии даст два метра длины!), типов азотистых оснований всего четыре: аденин (А), тимин (Т), цитозин (Ц) и гуанин (Г). ДНК находится в двухцепочечном состоянии, где нуклеотиды одной цепочки образуют водородные связи с нуклеотидами другой. И действует строгое правило: аденин всегда выстраивается напротив тимина, а цитозин — напротив гуанина. Это правило носит название комплементарности. РНК организована сходным образом, с двумя главными отличиями. Во-первых, на месте тимина всегда находится другое азотистое основание: урацил (У). Во-вторых, хотя иногда в клетке встречаются небольшие двухцепочечные РНК из двух комплементарных цепей, чаще всего РНК существует в виде одной цепочки, одни участки которой связаны с белками, а другие комплементарны внутри одной молекулы, образуя сложные пространственные укладки [7].
Комплементарность лежит в основе копирования ДНК, в ходе которой цепи расплетаются, и к каждой цепи специальным арсеналом белков по матричному принципу [8] достраивается комплементарная. Так наследственный материал удваивается, чтобы потом разойтись по дочерним клеткам в ходе клеточного деления. РНК тоже может удваиваться. Это происходит или с маленькими РНК-цепочками у растений, дрожжей и червячка C. elegans [9], или с цепочками большего размера у вирусов, у которых наследственная информация находится в форме РНК, а не ДНК.
Комплементарность также лежит в основе прямой и обратной транскрипции — перевода ДНК в РНК и обратно соответственно. При транскрипции двухцепочечный участок ДНК расплетается, и к цепи, которая кодирует ген, комплементарно достраивается участок РНК, который отделяется от ДНК и после завершения транскрипции живет своей жизнью в клетке.
Хромосомы и хроматин
У эукариот молекулы ДНК в клеточном ядре упакованы в хромосомы, в которых одна длинная молекула ДНК намотана на глобулы из белков-гистонов (как бусины на нитке) и сложена в компактную структуру вмeсте с другими молекулами белков и РНК. Всё вещество хромосом называют хроматином [3]. Еще в 1928 году Эмиль Хайтц заметил, что одни участки хроматина компактнее других, и разделил хромосомное вещество на развернутый эу- и компактный гетерохроматин [10].
Глобулы из гистонов носят название нуклеосом. Нуклеосомы устроены удивительно регулярно: каждая глобула имеет один и тот же план строения — восемь молекул гистонов (по две молекулы гистонов 2А (H2А), 2B (H2B), 3 (H3) и 4 (H4)) сложены в компактную структуру таким образом, что тетрамер (комплекс из четырех белков) из гистонов H3 и H4 оказывается «зажат» между двумя димерами (комплексами из двух белков) H2A и H2B. Вокруг одной глобулы обернуто 146 пар оснований молекулы ДНК (рис. 2) [11]. Такая упаковка позволяет уложить ДНК длиной в 2 метра в ядро размером в несколько микрометров, а также регулирует чтение генов.
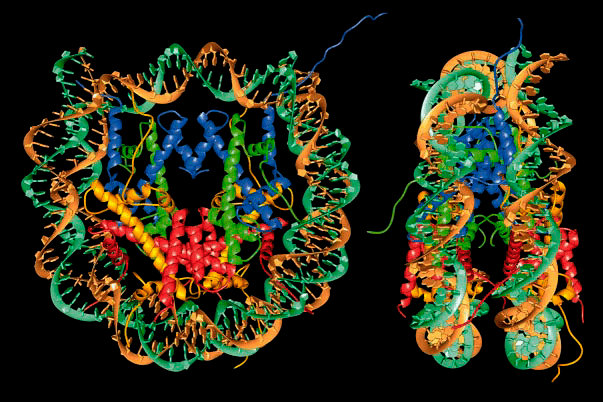
Рисунок 2. Кристаллическая структура нуклеосомы. 146 пар оснований ДНК обернуто вокруг октамера из гистонов.
Части гистонов выступают из нуклеосом и могут быть модифицированы — специальные белки-«писатели» пришивают к гистоновым «хвостам» разные химические группы: метильные, ацетильные, фосфатные, кротонильные, цитратные, серотонильные или же небольшие белки: убиквитин [12] и SUMO [13]. Другие белки — «читатели» — взаимодействуют с метками гистоновых «хвостов», причем в белках-«читателях» есть модули, специфично узнающие определенные метки. Еще есть специальные белки-«стиратели», снимающие метки с гистонов.
После открытий первых гистоновых модификаций Дэвид Аллис и Брайен Штраль выдвинули гипотезу «гистонового кода»: комбинации гистоновых меток являются кодом, который читается другими белками и локально определяет молекулярные процессы, происходящие в местах комбинаций меток [14]. Однако похоже, что картина сложнее, и далеко не только гистоновые метки управляют молекулярными процессами на хроматине. Кроме того, хотя метки и бывают взаимоисключающими, они действуют по отдельности, а не в комбинациях [15]. Белки-читатели узнают отдельные метки, а для регуляции процессов на хроматине иногда может быть достаточно всего одной модификации.
Интересно, что нуклеосомы могут отличаться друг от друга не только модификациями гистонов, но и самими гистонами. Есть так называемые варианты гистонов, которые могут заменять канонические гистоны в нуклеосомах. И структура нуклеосом с такими измененными гистонами слегка меняется.
Как устроено окружение нуклеосом с намотанной на них ДНК? С ними взаимодействуют тысячи белков и молекул РНК. Почти всегда белки и РНК собираются и находятся в больших комплексах. Иногда в одиночку — вне комплексов — они не могут выполнять свою функцию. Такие белковые комплексы — экзосомы и протеасомы [16] — разбирают синтезированные РНК и белки на нуклеотиды и цепочки из нескольких аминокислот соответственно. Большие комплексы из белка и РНК — сплайсосомы и комплексы полиаденилирования — осуществляют процессинг матричных РНК: вырезают из нее некодирующие участки и строят полиА-хвост на 3′-конце.
Короткие и длинные некодирующие РНК участвуют во многих процессах, происходящих в ядре, но в отличие от белков, редко обладают ферментативной активностью и, когда функционируют в комплексах, в основном играют структурную роль. Совсем короткие малые интерферирующие РНК (22 нуклеотидных остатка в длину) участвуют в образовании гетерохроматина и разрушении комплементарных РНК [7].
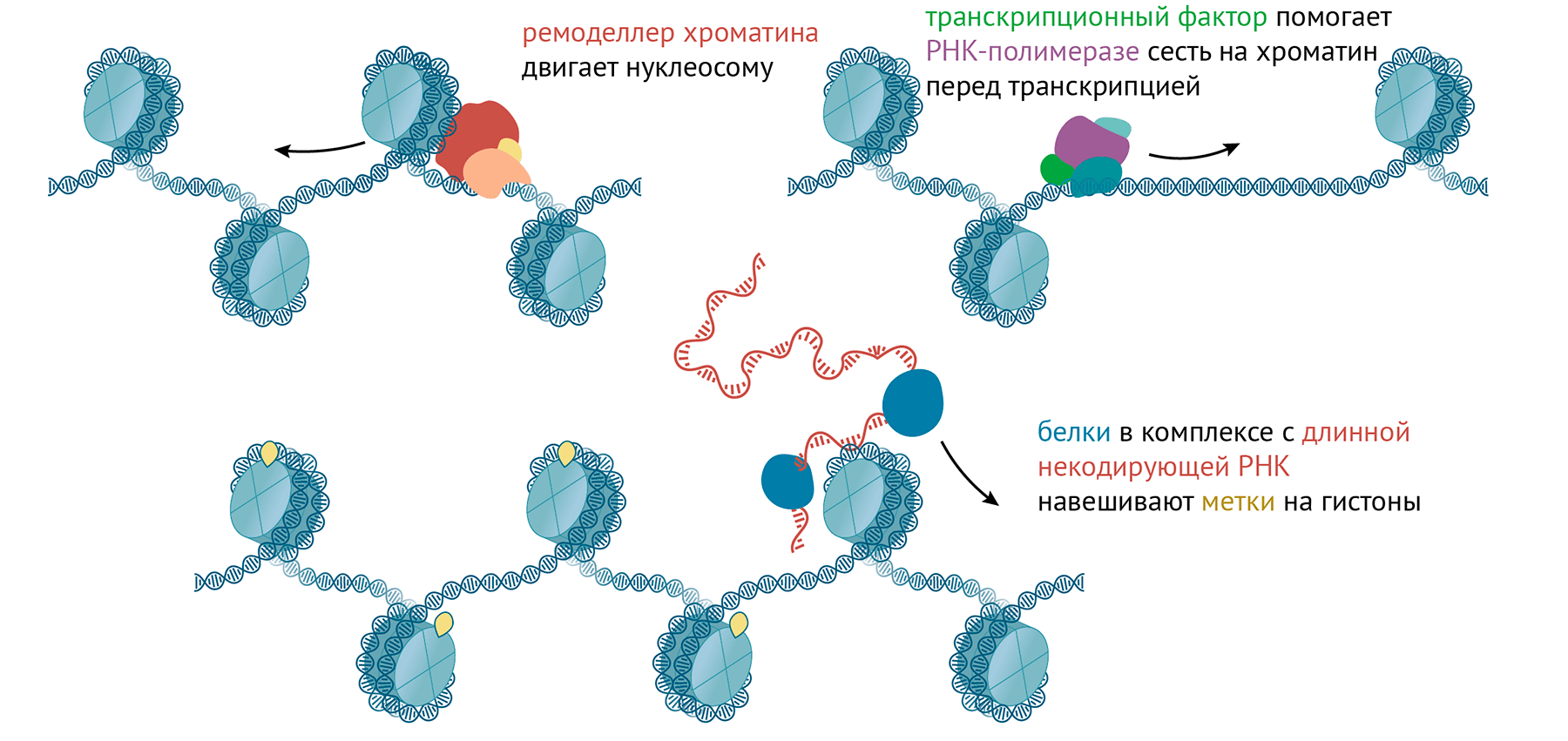
Рисунок 3. Хроматин на уровне нескольких нуклеосом. Кроме упомянутых белков-»читателей», «писателей» и «стирателей», взаимодействующие белки можно разделить по таким функциям:
- Транскрипционные факторы связываются с определенными последовательностями ДНК и помогают другим белкам связаться рядом. Например, они помогают ферменту РНК-полимеразе сесть на ДНК и начать транскрипцию — перевод последовательности ДНК в РНК. Или помогать связаться с ДНК белкам, которые строят гетерохроматин [17].
- Ремоделеры хроматина могут собирать, разбирать или двигать нуклеосомы взад-вперед по ДНК, а иногда накручивать ДНК на нуклеосомы и снимать с них двойную спираль, затрачивая при этом энергию АТФ.
- Хеликазы умеют расплетать двойную спираль ДНК и переплетать уже свернутые молекулы РНК.
- Топоизомеразы могут распутывать переплетенные или перезакрученные двойные спирали ДНК.
иллюстрация Михаила Гурьева
Линейные ДНК и РНК
Совокупность молекул ДНК образуют генóм. Ген — это фрагмент линейной последовательности ДНК, который может переводиться в РНК. Перед каждым гéном есть промотор — область, с которой связывается осуществляющий транскрипцию фермент (ДНК-зависимая РНК-полимераза). В конце гена находится терминатор — область, где РНК-полимераза отсоединяется от цепи ДНК, чтобы транскрипция закончилась. Интересно, что на транскрипцию гена могут влиять последовательности ДНК, отстоящие от него иногда на миллионы нуклеотидов, но расположенные близко к промотору в пространстве (об этом мы будем говорить гораздо подробнее дальше). Эти элементы называются энхансерами, если они увеличивают транскрипцию гена, или сайленсерами, если уменьшают. Некоторые элементы ДНК на линейной последовательности — инсуляторы — находятся между энхансером/сайленсером и промотором, не дают сблизиться им в 3D, и, соответственно, не дают повлиять на транскрипцию гена. Также инсуляторы могут ограничивать распространение разных типов хроматина по линейной ДНК.
У эукариот гены состоят из кодирующих и некодирующих участков, сменяющих друг друга на протяжении гена. Когда РНК с такого гена считывается, из нее вырезаются некодирующие участки (интроны), а кодирующие (экзоны) сшиваются. Такой процесс носит название сплайсинга. Иногда только некоторые кодирующие участки могут сшиваться, а другие деградируют (разбираются на строительные блоки). Это уже так называемый альтернативный сплайсинг.
Линейный хроматин
Как расположены белки и РНК на линейной последовательности ДНК? Во-первых, у каждого белка и по-своему модифицированного гистона свой неповторимый паттерн, или профиль связывания с ДНК (рис. 4). Даже когда профили связывания двух разных белков очень похожи, они немного отличаются друг от друга (рис. 5). Во-вторых, профили можно поделить на группы, а по этим группам профилей выделить типы хроматина. И получение профилей связывания, и выделение типов хроматина проводится с помощью компьютерных методов биоинформатики на основе данных ChIP-секвенирования и Dam-ID .
Зачастую эти биоинформатические методы очень сложны и основаны на математической теории; мы разбираем их в заключительной главе этой статьи.
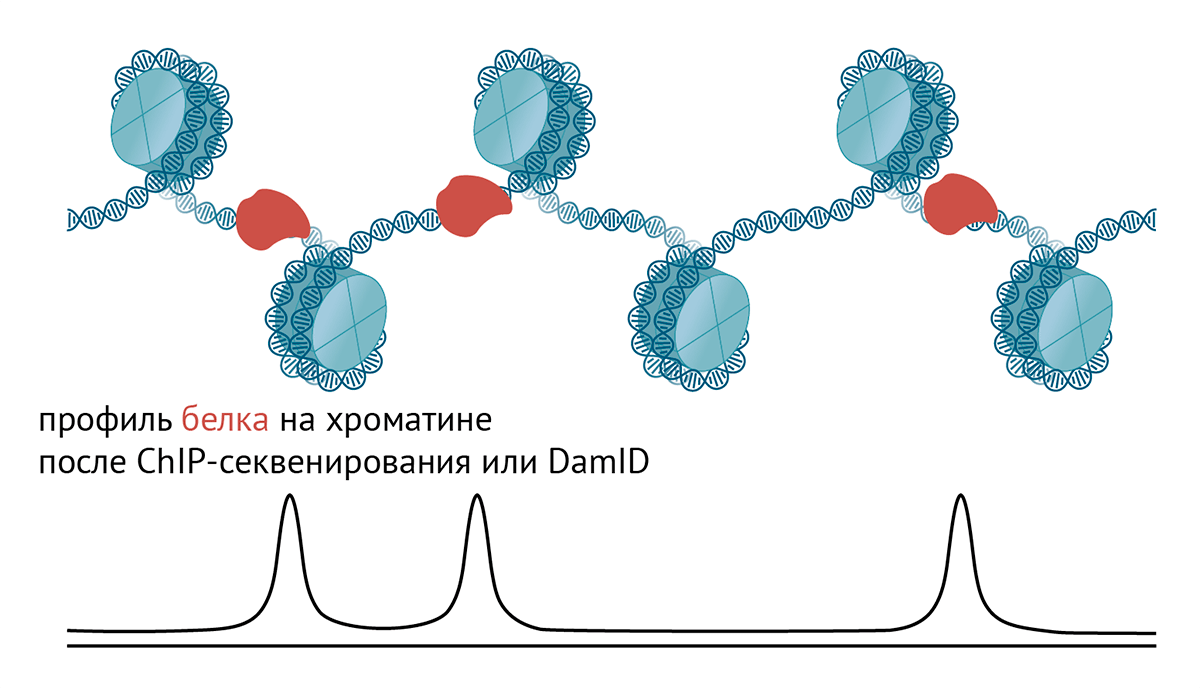
Рисунок 4. Профиль белка на хроматине, полученный после ChIP-seq или Dam-ID. По горизонтали отображена координата линейной ДНК, по вертикали — количество белка на данном участке ДНК.
иллюстрация Михаила Гурьева
Так, в одной из работ в клетках дрозофилы методом DamID определили профили связывания с ДНК 53-х белков, и на основе этого выделили пять типов хроматина, которые условно назвали «цветами» (рис. 5). Два цвета — красный и желтый — отнесли к разным типам активного хроматина, а еще три — синий, зеленый и черный — к разным типам неактивного.
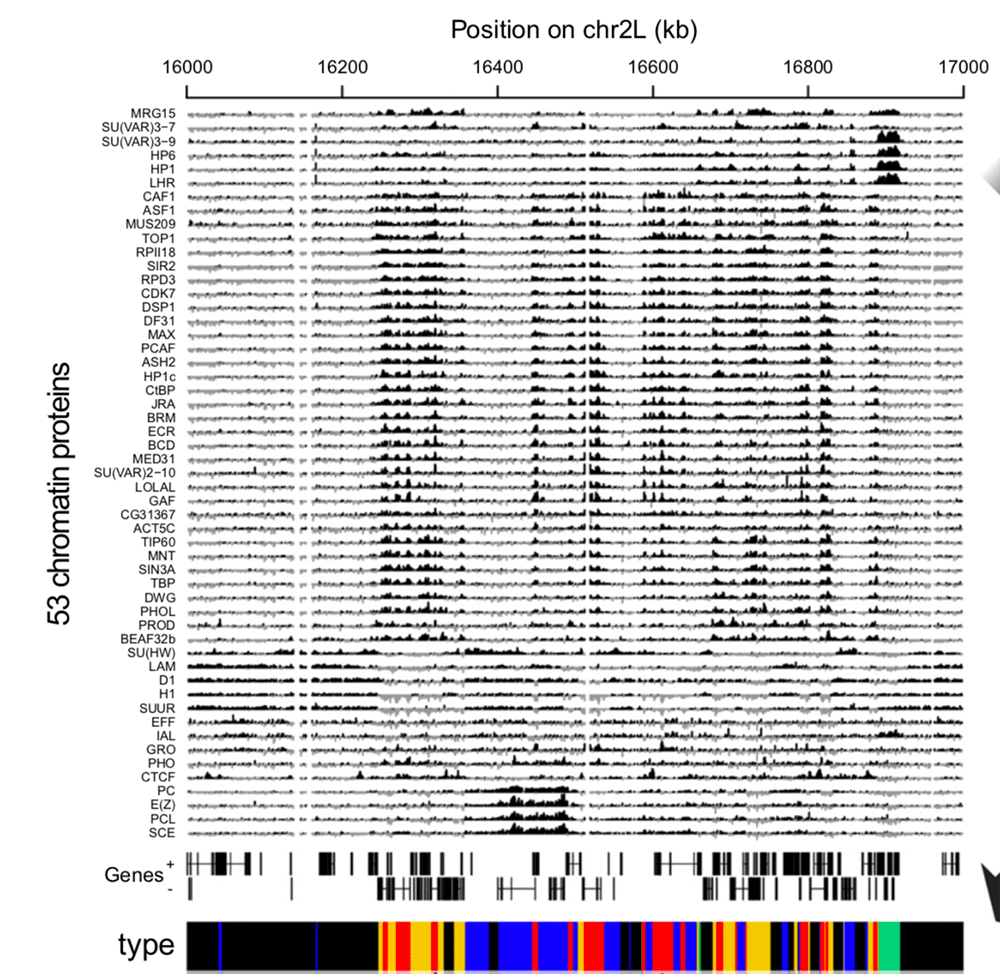
Рисунок 5. 53 профиля белков на хроматине после Dam-метилирования, объединенные в типы — «цвета» хроматина. Цвет, или тип хроматина, определяется в зависимости от того, какие белки преимущественно связываются с этими участками генома.
Чем отличаются активные цвета хроматина — красный и желтый? Когда ДНК удваивается, сначала удваиваются активные участки, а потом уже неактивные. Причем красные участки удваиваются раньше желтых. Кроме того, в желтом хроматине преимущественно расположены гены, которые активны всегда, а в красном — активные лишь при определенных условиях.
Два из трех неактивных цветов хроматина были известны и до этой работы. Зеленый тип хроматина — так называемый классический гетерохроматин, в котором присутствует белок HP1a (heterochromatic protein 1a, гетерохроматиновый белок 1а) и белки, которые взаимодействуют с ним (см. далее). На «синем» хроматине располагаются белки группы Polycomb, которые регулируют гены, важные для развития организма. Черный хроматин в этой работе открыли впервые. Выяснилось, что это примерно половина генома, и в нем сравнительно мало генов и транскрипции; если в нем и есть гены, то в основном неактивные [18].
В другой работе, основанной на ChiP-seq 18-и гистоновых модификаций, хроматин плодовой мушки дрозофилы поделили на девять типов [19]. Интересно, что многие (а возможно, и все) из них — разновидности уже знакомых нам пяти «цветов» [20]. После дрозофилы хроматин классифицировали на типы и в других организмах [21]: в 15 типах клеток мыши и шести человеческих клеточных линиях хроматин разделили на семь типов [22], а кроме того, похожее деление провели и для червячка C. elegans [23] и нескольких растений [24].
ДНК в 3D
Если взять ДНК из всех хромосом всего одной нашей клетки и вытянуть на полную длину, она составит целых два метра. Но диаметр клеточного ядра, в котором она уложена, — всего несколько микрометров. Следовательно, в ядре ДНК должна быть многократно свернута в пространстве.

Рисунок 6. Фибрилла 30 нм существует только in vitro
на основе модели фибриллы из [77]
Долгое время на основе экспериментов в пробирке, в которых собирали хроматиновые структуры из очищенной ДНК и очищенных гистонов, считалось, что ДНК, намотанная на нуклеосомы, упакована в фибриллу диаметром 30 нм (рис. 6). Однако недавно эксперименты по 3D электронной микроскопии на клетках (их удалось сделать благодаря специальной окраске ДНК) показали, что такой фибриллы нет, зато есть изогнутые цепи диаметром от 5 до 24 нм [25], [26].
Также еще до развития специальных методов для изучения пространственной укладки ДНК проводились эксперименты, показывающие, что ДНК в ядре организована в огромные петли. Эти петли можно было заметить с помощью электронной микроскопии после обработки ядер очень высоко концентрированной солью. Они были прикреплены к белковой основе ядра, оставшейся после того, как из ядра под действием соли уплыло много белков хроматина [27], [28].
Много новых открытий пришло с развитием методов Hi-C [3], [29] и Micro-C [30], о которых мы расскажем в последней главе этой статьи. Во-первых, выяснилось, что участки ДНК сворачиваются в небольшие глобулы (что-то наподобие шариков из скомканных нитей), которые, в свою очередь, сворачиваются в глобулы всё большего и большего размера (рис. 7) [29]. Глобулы из последовательностей ДНК размером от 100 тысяч до 1,5 миллионов нуклеотидных остатков получили название топологически ассоциированных доменов (ТАДов) [31]. Во-вторых, многие участки ДНК выпетливаются. Как соотносятся петли и глобулы? Похоже, что основания части петель совпадают с основанием глобул [32]! То есть сначала формируются петли, которые иногда «комкаются» в глобулы. Внутри больших петель могут быть петли поменьше. Правда, у плодовой мушки дрозофилы наличие петли зафиксировать на границе ТАДов пока не удалось, только внутри [33].
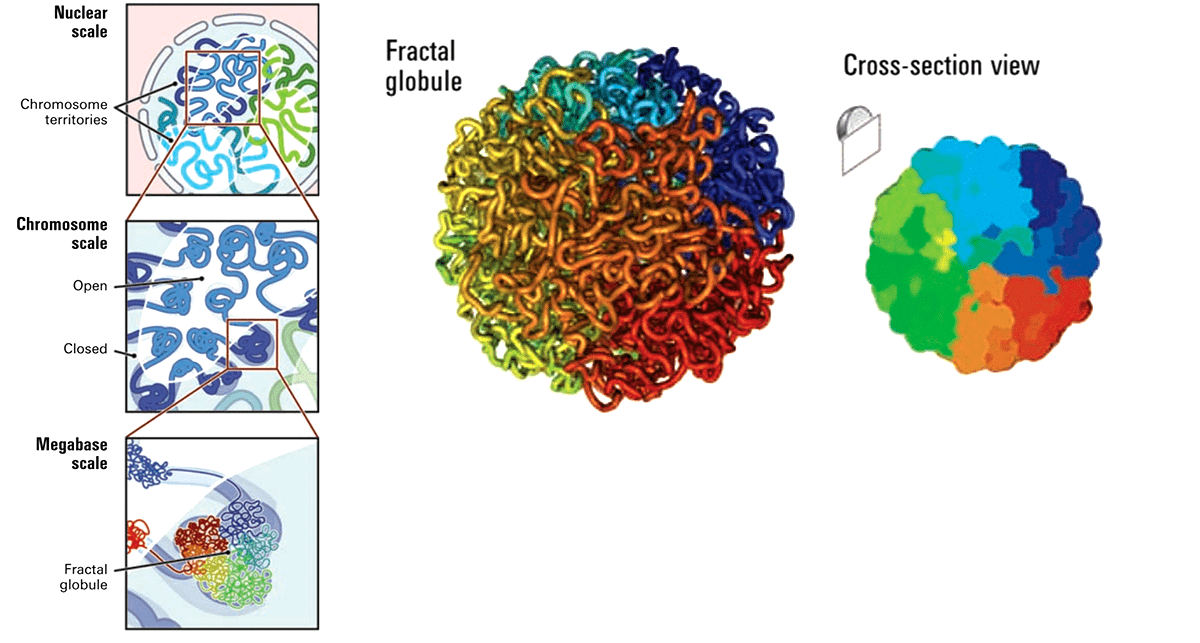
Рисунок 7. Пространственная упаковка ДНК. Левая панель: укладка хроматина во фрактальные глобулы. Хромосомные территории состоят из глобул поменьше, которые, в свою очередь, состоят из глобул еще поменьше. Правая панель: фрактальная глобула в объеме и в разрезе.
Интересно, что есть как глобулы с активными генами, так и глобулы, в которых гены замалчиваются. И всё вещество хромосом, весь хроматин, можно разделить на два компартмента — А и B, которые отличаются друг от друга тем, как часто контактируют в них участки ДНК. В B побольше, в A поменьше.
Цитологи уже давно подметили, что когда клетка делится, ee хромосомы гораздо компактнее. У многих организмов, в том числе у человека, есть половые и неполовые (соматические) хромосомы. Из всего набора хромосом к каждой неполовой хромосоме можно подобрать очень похожую на нее. Две половые хромосомы могут быть как похожими (например, две Х хромосомы у женщины), так и разными (Х и Y хромосомы у мужчины). Так, по набору половых хромосом у многих организмов, включая человека, в большинстве случаев можно определить пол. (Однако в целом пол — понятие куда более комплексное, чем присутствие определенного набора хромосом [34], [35].)
Две одинаковые по набору и порядку генов хромосомы, соединенные перетяжкой, образуют нечто похожее на букву X, которой часто обозначают хромосомы в школьных учебниках. Как свернуты хромосомы в такие буквы Х? По тем же принципам петель, как и хромосомы в неделящихся в данный момент клетках, только немного по-другому. В случае хромосом клеток, которые делятся в данный момент, часть ДНК закреплена на линейном каркасе из белков, из которого исходят петли ДНК. И это не те же самые петли, что и в неделящихся на данный момент клетках, а другие [36], [37].
Когезины и конденсины
Что меняет форму хромосом, когда клетка собирается делиться? Этим занимаются белки конденсины.
Интересно, что они устроены очень похоже на белки, которые поддерживают петли в ДНК — когезины. Все эти белки собраны в конструкции, подобные кольцу, маленькая часть которого открывается. Именно в это отверстие и продевается ДНК, после чего оно закрывается. В случае когезинов, две двухцепочечные цепи ДНК оказываются зажаты в основании петли и не могут разойтись (как всё это выглядит, см. на видео 1). В случае конденсинов процесс изучен хуже, но, похоже, происходит примерно то же самое, только, в отличие от когезинов, они держат другие петли — не те, что присутствуют в хромосомах, когда клетка не делится (см. раздел о том, как ДНК уложена в ядре). Интересно, что комплексы могут не только держать петли, но и делать их. Этот процесс пока плохо изучен, но похоже, что белки перемещаются по ДНК, выпетливая ее, пока не встречают особые белки, связанные с блокирующими последовательностями ДНК — инсуляторами, — где и застревают [38–42].
Видео 1. Выпетливание хроматина
анимация лаборатории Леонида Мирного
Хроматин в 3D
В одном из предыдущих разделов мы обсудили, что можно выделить разные типы («цвета») активного и неактивного хроматина в зависимости от того, какие белки он связывает. Возникает логичный вопрос: если ДНК свернута в пространстве, как организованы в 3D разные участки линейного хроматина? Если у хроматина можно выделить «цвета», то как покрашено клеточное ядро?
Основной принцип укладки хроматина можно сформулировать так: подобное часто, но не всегда, взаимодействует с подобным. Например, активные и неактивные участки хроматина взаимодействуют с участками такой же активности в пространстве [43]. Также давно известно, что если покрасить клетку на некоторые белки, которые сидят преимущественно в одном из цветов хроматина (например, тот же HP1a), то такие белки образуют отдельные структуры в ядре. HP1a-гетерохроматин, например, образует от одного до трех доменов в зависимости от того, собирается клетка делиться или нет [44]. Еще было показано, что многие домены от 10 до 500 тысяч нуклеотидных остатков длиной образуют кластеры. Их можно увидеть в клетках под микроскопом, если последовательности ДНК этих доменов в клетках спарить со светящейся комплементарной ДНК [45].
В общем-то, тот факт, что ДНК свернута в пространстве выпетливанием в отдельные домены (например, ТАДы), уже подразумевает, что участки хроматина, недалекие друг от друга на линейной последовательности ДНК, с приличной вероятностью будут располагаться недалеко друг от друга в пространстве. И действительно, линейные хроматиновые домены коррелируют, то есть похожи на пространственные домены ДНК [46]. Уже известны целые ТАДы, целиком покрашенные в отдельный цвет хроматина (например, синие Polycomb-ТАДы у дрозофилы [47]).
Как связаны между собой функции разных белков?
Каждый белок в клетке взаимодействует с другими белками или иными молекулами, и следовательно, у него есть какая-то функция. А взаимодействует с Б, структура Б меняется, взаимодействие Б с другими молекулами меняется, запускается каскад. Конечно, речь идет только о значимом взаимодействии А с Б, меняющим Б, а не о любом.
Часто таких функций несколько, причем на первый взгляд они могут быть не связаны. Например, как в ядре, так и в цитоплазме есть протеасома, одна из субъединиц которой участвует в ремоделинге хроматина [48]. Как одна функция может влиять на другую? Давайте разберем несколько примеров.
Например, и у человека, и у мушек Drosophila есть белок MOF (males absent on the first, самцы отсутствуют в первом <поколении>), и самцы мух с мутантным MOF не развиваются. Этот белок навешивает ацетильные метки на гистоны и вовлечен в систему дозовой компенсации у мушек. У самок дрозофилы две Х-хромосомы, а у самцов одна. И чтобы гены с этой хромосомы читались у обоих полов в одинаковом объеме, их транскрипция у самцов удвоена. Один белок — MSL2 (male specific lethal 2, только самцы летальны 2) — синтезируется только у самцов. Он собирает на Х-хромосоме самцов комплекс из белков, в который входит и MOF, окрашивая Х-хроматин в особенный «цвет» (о чем речь шла выше). MOF навешивает на гистоны ацетильные метки, заряженные отрицательно и потому отталкивающиеся друг от друга, что делает хроматин более доступным для белков, осуществляющих транскрипцию. Таким образом, гены, на которых MOF ацетилирует гистоны , читаются лучше [49] (а это и есть дозовая компенсация).
Недавно выяснилось, что MOF находится не только в ядре, но и в митохондриях, где регулирует транскрипцию генов, отвечающих за дыхание [50].
Теперь один пример про неактивный хроматин. Тройное метилирование по девятому лизину гистона (H3K9me3) — самая известная модификация такого хроматина. Чтобы навесить метильные метки на этот лизин, может потребоваться его деацетилировать. С H3K9me3 связывается один из главных белков гетерохроматина — HP1a (heterochromatin protein 1 alpha, гетерохроматиновый белок 1 альфа). Молекулы HP1a на соседних нуклеосомах прочно взаимодействуют между собой, и таким образом сближают нуклеосомы. Таким образом, определенные модификации гистонов (снятие ацетильных и навешивание метильных меток на H3K9) компактизируют хроматин, снижая его активность [51], [52].
Известно, что возле точки начала транскрипции гена на ДНК нуклеосомы расположены определенным образом. В дрожжах было показано, что, если выключить в клетке транскрипцию, то позиции нуклеосом меняются [53]. Как мы помним, именно ремоделеры хроматина двигают нуклеосомы взад-вперед по ДНК. Следовательно, транскрипция влияет на ремоделинг хроматина. Может ли быть обратное? Да, может. Например, немного подвинув нуклеосому, ремоделер хроматина может высвободить специальную последовательность ДНК — сайт связывания транскрипционного фактора (белка, помогающего РНК-полимеразе сесть и начать транскрипцию гена), — который с ней свяжется и запустит транскрипцию [54].
Модельный эксперимент эпигенетики
Сегодня, открыв PubMed, можно получить представление о типичной работе по молекулярной биологии. Там, например, может описываться новая функция какого-то белка. Чтобы установить его функции, обычно делают иммунопреципитацию и масс-спектрометрию (см. методы) и определяют, какие белки взаимодействуют с изучаемым. Чтобы проверить предполагаемую функцию, белок убирают из клетки (или значительно снижают его уровень) и смотрят на фенотип: что изменилось без него?
Представьте: мы изучаем белок А, выявляем список белков, с которыми он взаимодействует, и получаем, что они связаны с транскрипцией. Мы формулируем гипотезу, что наш белок A повышает или понижает уровень транскрипции. Для ее проверки нужно понизить в клетках уровень A и определить количество РНК отдельных генов в этих клетках, а также сравнить его с обычными (контрольными) клетками. Так мы выясним, на транскрипцию каких генов влияет А (и влияет ли).
Можно дальше сделать ChIP-секвенирование и определить, на каких последовательностях в геноме «сидит» А (то есть — построить профиль белка А; см. рис. 4 и 5). Может выясниться, что это происходит на промоторах тех генов, которые он регулирует. Дальше можно поинтересоваться, что общего у «его» промоторов? Например, располагаются ли они в определенных хроматиновых доменах, на определенных хромосомах, на тех же местах, где «сидят» другие белки с известной функцией? Допустим, мы нашли, что промоторы с А располагаются в активном хроматине, а сам он повышает транскрипцию «своих» генов. Но как белку A это удается?
Посмотрим внимательнее на список белков, с которыми взаимодействует А. Допустим, мы нашли среди них Б, который навешивает на хвосты гистонов ацетильные метки (которые, как мы помним, расталкиваясь, «разрыхляют» хроматин и делают его доступнее для белков транскрипции). Можно выдвинуть гипотезу, что наш белок не просто связывается с ДНК на промоторах в активном хроматине, а привлекает туда белок Б, ацетилирующий гистоны и повышающий транскрипцию. Можно понизить уровень А в клетке и сделать ChIP-секвенирование белка Б и навешиваемой им ацетильной метки гистонов. Так можно установить, что количество Б на хроматине тоже понижается, и гистоны под влиянием А теряют ацетильные метки. И так далее. Можно задавать всё новые и новые вопросы и отвечать на них, хотя, конечно не всегда эксперименты сработают и дадут однозначный ответ.
Почему данные могут быть ложными или противоречить друг другу? Иногда потому, что мы неправильно их интерпретируем. Например, если вернуться к списку белков — потенциальных партнеров белка А, — то может оказаться, что идентификация Б была неспецифической: например, из-за высокой концентрации в ядре (а это вполне возможно для белков, связанных с транскрипцией, или ремоделеров хроматина).
Иногда данные, полученные в пробирке, ничего не говорят о процессах в культуре клеток, а они, в свою очередь, кардинально отличаются от того, что происходит в живом организме. Потому что живые клетки — гораздо более сложные системы, чем те, что собраны из молекул в пробирке, а культуры клеток, которые культивируют в лабораториях уже долгие-долгие годы, эволюционировали, изменились и уже не совсем похожи на организмы, из которых их взяли. Как бы то ни было, полученные данные, пусть и противоречивые, всегда учат нас чему-то новому, и их важно публиковать, пусть и не в самых трендовых журналах. К сожалению, не все ученые это делают.
Какие научные работы делают ученые? Или все статьи важны
Пожалуй, все работы можно разделить на два типа. Первые практически не меняют сложившуюся парадигму, лишь добавляя детали в уже существующую картину. Например, работы, которые открывают новые молекулы, взаимодействующие с интересующей нас молекулой А, без связи функции А с совершенно другими функциями взаимодействующих молекул. Или работы, описывающие, как молекула А взаимодействует с молекулой Б, без описания принципиально новых связей между функциями А и Б (или значимо новых элементов строения или взаимодействия А и Б, если это работа по структурной биологии). Такие работы могут быть очень важны, например, для подтверждения результатов больших исследований, где изучаются взаимодействия между собой тысяч молекул. В случае таких масштабных работ очень велик риск ошибок, и каждое новое открытое взаимодействие рассматривается как потенциальное и требующее подтверждения. И статьи, которые детально подтверждают отдельные взаимодействия, очень важны.
Второй тип работ — те, которые вводят новые научные концепции. Они значительно меняют схему происходящего, которую мы можем представить себе исходя только из предшествующих данных. Например, работы, объясняющие связь разных функций молекул, скажем, транскрипции и ремоделинга хроматина. Или работы, описывающие принципиально новые методы, структуру укладки ДНК в ядре, классифицирующие разные типы («цвета») хроматина.
И хотя работы второго типа кажутся более привлекательными, и именно их старается делать большая часть ученых, не стоит недооценивать работы, которые на первый взгляд не отвечают на какой-либо очень важный вопрос. Нельзя угадать, возможно, через много лет выяснится, что какая-то из давно открытых молекул очень важна и ее нужно изучать. И тут-то и пригодятся данные про эту молекулу, открытые давным-давно и плохо цитируемые.
В следующей статье цикла мы рассмотрим, что представляют собой хроматиновые домены и какими они бывают. Но в заключение этого материала идет еще одна — последняя — глава, рассказывающая о методах, которыми исследуют строение ядра клетки и упаковку ДНК в нем.
Методы изучения ДНК и хроматина
Как получить много ДНК и как определить ее последовательность?
Теперь давайте разберем несколько методов, которые ученые используют в своих исследованиях. Увеличение количества (амплификация) интересующего фрагмента ДНК — ключевая химическая реакция в молекулярной биологии. Она называется полимеразной цепной реакцией (ПЦР) [55], [56]. Например, чтобы клонировать ген (вставить его в плазмиду — кольцевую последовательность ДНК, которая может размножаться в бактериях), нужно получить огромное количество копий гена и плазмиды. Много плазмиды можно размножить в бактериях. А что делать с геном? В отличие от плазмиды, линейная последовательность в бактериях удваиваться не будет. Химически синтезировать длинную цепочку ДНК очень дорого. Зато множество копий целевого гена можно «наработать» при помощи ПЦР. Также, например, во многих методах для определения последовательности ДНК — секвенирования — нужно большое количество молекул ДНК, и их, опять-таки, можно «наработать» ПЦР.
Для РНК реакции амплификации не разработано, несмотря на то, что в клетках существует процесс удвоения РНК. Проблема в том, что РНК-зависимая РНК-полимераза, осуществляющая процесс, не сможет работать при высоких температурах, а при низких комплементарные цепи РНК будут не расходиться, а слипаться. Но молекулярным биологам это и не нужно. Много РНК можно наработать транскрипцией с ДНК, а ДНК уже наработать при помощи ПЦР.
Как определить последовательность ДНК? Технологии секвенирования появились в середине прошлого века, сейчас их уже много, и они всё еще совершенствуются.
Рассмотрим один из самых используемых методов: секвенирование на платформе «Иллюмина» [56], [57]. Двухцепочечная ДНК, которую нужно секвенировать, расщепляется на фрагменты, после чего к их концам ДНК-лигазами пришиваются короткие двухцепочечные последовательности — адаптеры. После этого количество фрагментов увеличивают с помощью ПЦР. Раствор с такими фрагментами наносится на платформу, к которой пришиты праймеры, комплементарные адаптерам. На платформе фрагменты ДНК копируются таким образом, что скопировавшиеся фрагменты оказываются пришитыми к платформе.
После этого можно провести секвенирование. Тут тоже не обходится без ПЦР, только с особенностями. К нуклеотидам (как мы помним, это строительные блоки для ДНК) присоединены цветные метки. После пришивания такого меченого нуклеотида к цепочке ДНК-полимераза не способна сама пришить следующий. Это обеспечивает паузу, за время которой можно определить (по цвету флуоресценции), что это за нуклеотид. Определение проводится специальной камерой над платформой, к которой пришиты фрагменты ДНК, служащие матрицей для ПЦР. После этого флуоресцентную метку от нуклеотида отщепляют, делая возможным присоединение следующего нуклеотида; определяют его «цвет»; и так далее, пока ПЦР не закончится.
Так можно узнать последовательности коротких фрагментов — ридов (от англ. read — читать), или прочтений. Их длина в технологии «Иллюмина» не превышает 600 нуклеотидных остатков, а чаще — ≈200–300. Поскольку при фрагментации ДНК (которая происходит в самом начале процедуры) разные молекулы разрываются в разных местах, риды будут перекрываться, и, «выровняв» их на компьютере, можно собрать длинные исходные последовательности ДНК. К сожалению, такая сборка может давать сбои в участках ДНК, состоящих из повторяющихся последовательностей.
Для секвенирования РНК чаще всего начинают с обратной транскрипции — синтеза ДНК с цепи РНК. Фермент, который осуществляет это, называется РНК-зависимой ДНК-полимеразой. И полученную ДНК потом секвенируют.
Совсем недавно появившийся метод секвенирования ДНК и РНК — нанопоровое секвенирование [58], [59]. Его принцип крайне прост по сравнению с секвенированием на платформе «Иллюмина». Представьте себе небольшую камеру, заполненную раствором электролита и разделенную липидной мембраной, содержащей белковую пору. Под напряжением через пору пойдет электрический ток (поток ионов), а если в камере окажется молекула ДНК или РНК, она, подобно червячку, тоже начнет протискиваться через пору, и с прохождением каждого нуклеотидного остатка ток будет меняться, причем — в соответствии с тем, какой именно остаток проходит через пору в данный момент. Длина ридов в нанопоровом секвенировании — много тысяч пар оснований нуклеотидных остатков! Это очень много по сравнению с «Иллюминой». К тому же, такое секвенирование становится всё дешевле, а приборы — всё меньше в размерах. Можно секвенировать ДНК или РНК, просто загрузив ее в прибор немногим больше флешки. Правда, к сожалению, при нанопоровом секвенировании велика вероятность ошибок, что ограничивает его применение.
Методы секвенирования рутинно применяются для определения последовательностей геномов и уровня РНК в клетках, а также являются заключительным этапом таких методов как ChiP-seq, Dnase-seq, ATAC-seq и других.
Определение профилей белков на хроматине
Для выявления местоположения белков на ДНК — то есть определения ДНК-профилей (см. рис. 4 и 5) — был разработан метод иммунопреципитации хроматина и секвенирования (ChIP-sequencing, chromatin immunoprecipitation sequencing; рис. 8) [60], [61].
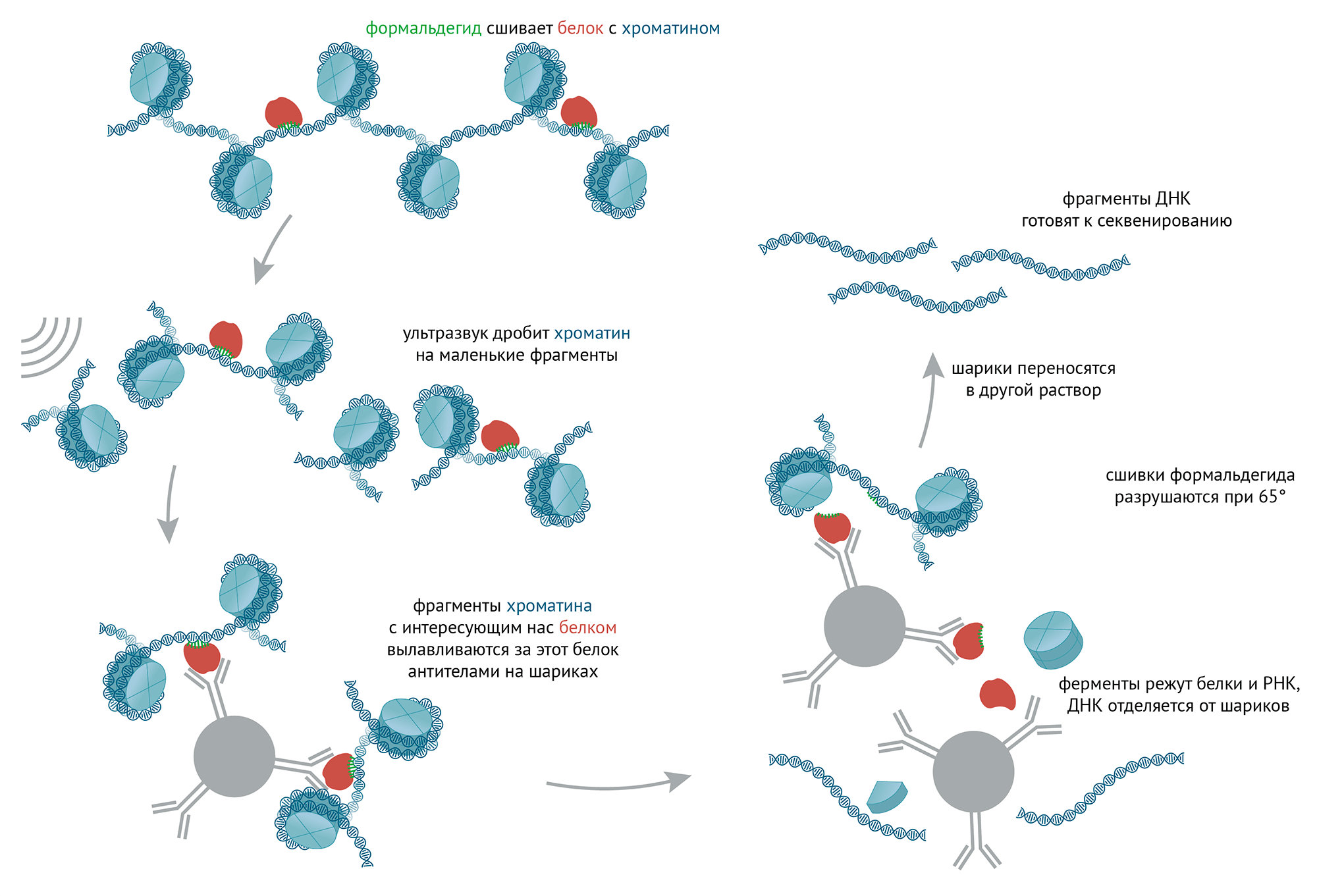
Рисунок 8. Схема ChiP-секвенирования. Клетки фиксируют формальдегидом, сшивающим многие молекулы между собой. В том числе молекулы белков пришиваются к ДНК, если связаны с ней. Потом хроматин дробят на фрагменты (200–300 нуклеотидов) ультразвуком или ферментами эндонуклеазами (действуют на ДНК как молекулярные ножницы [62]). Важно, что при нарезке ДНК молекулы всё еще сшиты между собой. Потом добавляют антитело — белок, который специфично взаимодействует только с нужным нам белком и ни с какими другими. Белки связываются с антителами, а их в свою очередь можно выловить из раствора специальными шариками, «уцепившись» за константные части молекул антител.
Итак, теперь у нас есть комплексы сшитых белков, за которые держатся антитела, за которые в свою очередь держатся шарики, которые можно отделить от раствора. Такие шарики с антителами и молекулярными комплексами отмывают специальными растворами от неспецифически налипших молекул, а потом шарики с антителами и молекулярными комплексами отделяют от раствора в отдельную пробирку. Туда добавляют раствор с ферментами, которые разрушают белки и РНК, и нагревают ее до 65 °C. При этом белки и РНК разрушаются, а формальдегидные сшивки между молекулами от нагрева исчезают. Теперь можно очистить фрагменты ДНК, с которыми взаимодействовал исследуемый белок, и секвенировать их. Результаты секвенирования покажут, с какими именно участками исходной ДНК взаимодействовал исследуемый белок.
иллюстрация Михаила Гурьева по [61]
Антитела — белки в форме буквы Y, которые вырабатывает наша иммунная система. Aнтитело можно сделать, введя животному в кровь белок, с которым потенциальное антитело должно взаимодействовать. Потом специальные иммунные клетки вырабатывают антитела в крови животного в ответ на инородный белок. Можно изолировать эти иммунные клетки и сделать так, чтобы они производили антитела в условиях лаборатории. Далее антитела легко почистить и использовать в методах молекулярной биологии. Подробнее о применении антител в исследованиях и медицине можно прочесть в спецпроекте «Биомолекулы» «Терапевтические антитела», например в статье «Краткая история открытия и применения антител» [63].
Похожий подход используется и для определения профилей определенных молекул РНК на ДНК [64]. Только для того, чтобы вытащить сшитые молекулярные комплексы из раствора, там используют не антитела, а молекулы РНК, комплементарные изучаемой. Эти РНК, в свою очередь, пришиты к биотину. Модифицированные биотином молекулы РНК можно соединить с шариками, «увешанными» молекулами белка стрептавидина: это прекрасная «удочка» для биотина, ведь взаимодействие этих двух молекул — одно из самых сильных в молекулярной биологии. Называется такой метод «аффинная очистка РНК» (RNA affinity purification, RAP) [65], [66].
Наконец, последний метод, который мы рассмотрим в этом разделе, является альтернативой ChiP-seq’у, но выгодно отличается тем, что для него нам не требуется специфическое антитело под нужный нам белок. Метод называется «идентификация ДНК-аденин-метилирования» (DNA adenine methyltransferase identification, DamID) по имени фермента метилтрансферазы Dam, которая метилирует ДНК по аденинам строго в последовательностях ГАТЦ (рис. 9) [67]. Такой метилирующий ДНК фермент в природе встречается только в бактериях, а в эукариотах ни фермента, ни метилированного аденозина нет. Поэтому можно отличить метилирование, протекающее в клетке в естественных условиях (по цитозинам), от метилирования, которое сделал этот фермент. Ген фермента Dam пришивают к гену нужного нам белка, и этот гибрид доставляют в клетку, где с него считывается гибридная мРНК, в результате чего транслируется гибридный белок. Он метилирует ГАТЦ-последовательности преимущественно в тех местах, где сидит его натуральный аналог. Но ведь и вторая часть гибридного белка — фермент Dam — тоже будет взаимодействовать с какими-то белками хроматина, и следовательно, будет притаскивать гибридный белок на участки ДНК, где натурального белка нет. Поэтому в качестве контроля берут клетки, куда доставляют только ген белка Dam. Белок, получающийся с РНК такого гена, будет метилировать только места ДНК, куда идет он сам. И сравнивая сайты метилирования гибридного белка и одиночного белка Dam, мы сможем отличить сайты связывания гибридного белка, которые специфичны для исследуемого белка, от сайтов, которые специфичны для Dam.
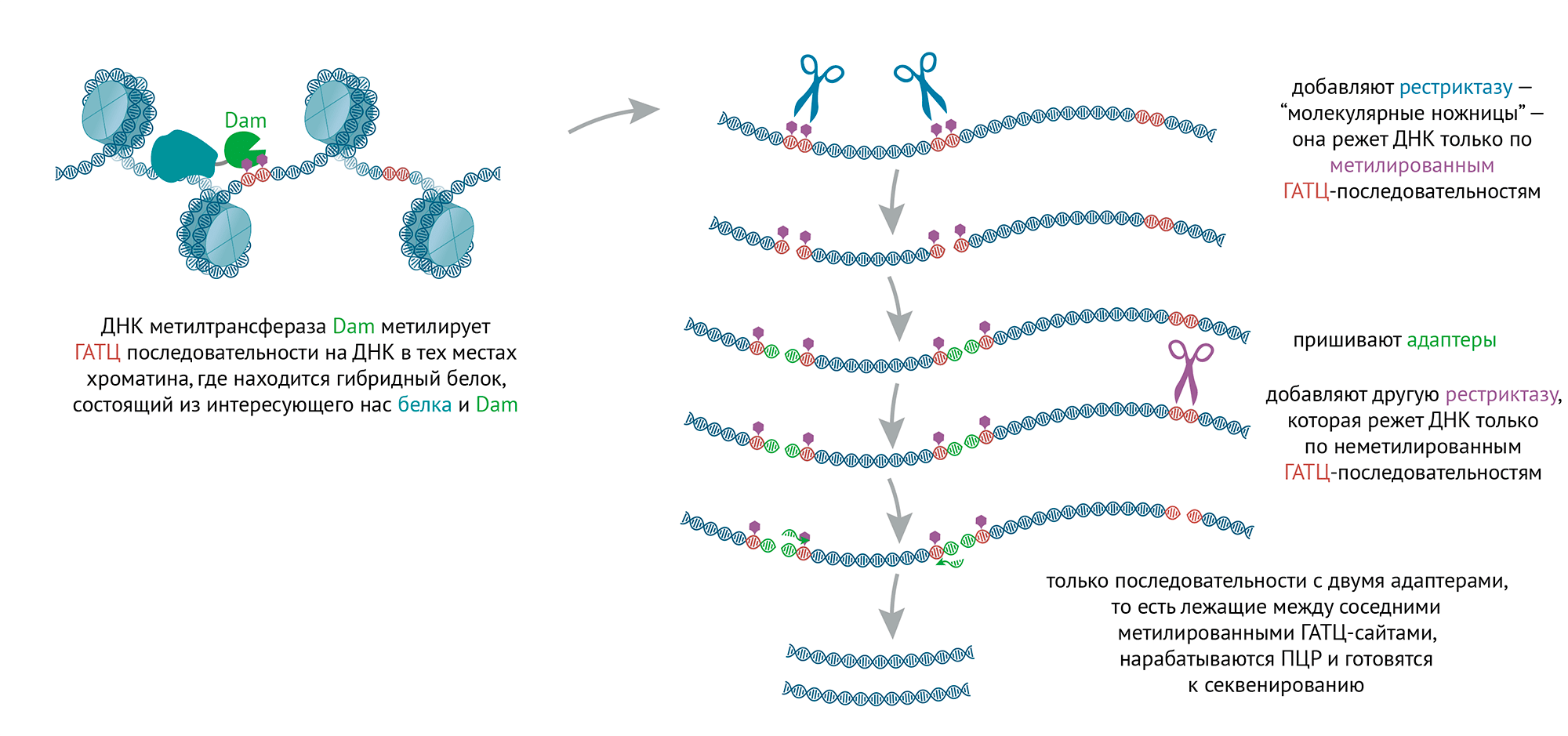
Рисунок 9. Схема Dam-метилирования. Как обнаружить сайты метилирования на очищенной ДНК? Для этого ДНК сначала нарезают рестриктазой DpnI, которая режет только метилированные по аденину ГАТЦ-последовательности. Затем к такой нарезанной ДНК пришивают адаптеры — короткие двухцепочечные последовательности ДНК. После чего нарезанную ДНК с пришитыми адаптерами нарезают второй раз рестриктазой DpnII, которая режет только неметилированные ГАТЦ-последовательности. При этом с двумя адаптерами по обе стороны фрагмента остаются только те кусочки ДНК, которые лежат между двумя соседними метилированными ГАТЦ-последовательностями. И такие фрагменты нарабатывают ПЦР, подбирая праймеры к последовательностям адаптеров.
иллюстрация Михаила Гурьева
С помощью методов ChIP-секвенирования и Dam-ID ученые получили множество профилей разных белков на ДНК. В том числе, эти данные классифицировали на типы, или «цвета», хроматина (см. раздел «Линейный хроматин»).
Определение доступности хроматина
Традиционно доступность хроматина определяли, подвергая ядра действию ДНКазы, которая режет только открытые участки хроматина и не пробирается к закрытым. На основе этого фермента разработали высокопроизводительный метод детекции ДНКазо-чувствительности хроматина — DNAse-seq. Сначала ядра обрабатываются ДНКазой. Затем к концам участков, которые она порезала, лигируют биотинилированный адаптер. Потом ядра обрабатывают рестриктазой, и образуются фрагменты с рестриктным сайтом на одном конце и адаптером на другом. К ним пришивают второй адаптер. Затем такие фрагменты с двумя адаптерами отделяют на стрептавидиновые шарики и секвенируют на платформе «Иллюмина» [68].
Позже появился более продвинутый метод ATAC-seq (Assay for Transposase-Accessible Chromatin using sequencing — метод для определения хроматина, доступного транспозазе, используя секвенирование). В нем ядра инкубируют с ферментом транспозазой. Она вносит двухцепочечные разрывы в ДНК в открытых участках хроматина и пришивает адаптеры в местах разрывов. Затем такие вырезанные участки с адаптерами секвенируют [69].
Определение контактов ДНК в пространстве
Как отследить взаимодействующие участки ДНК в ядре? Для ответа на этот вопрос было разработано несколько методов, в основе которых лежит один и тот же принцип (рис. 10) [3], [70], [71].
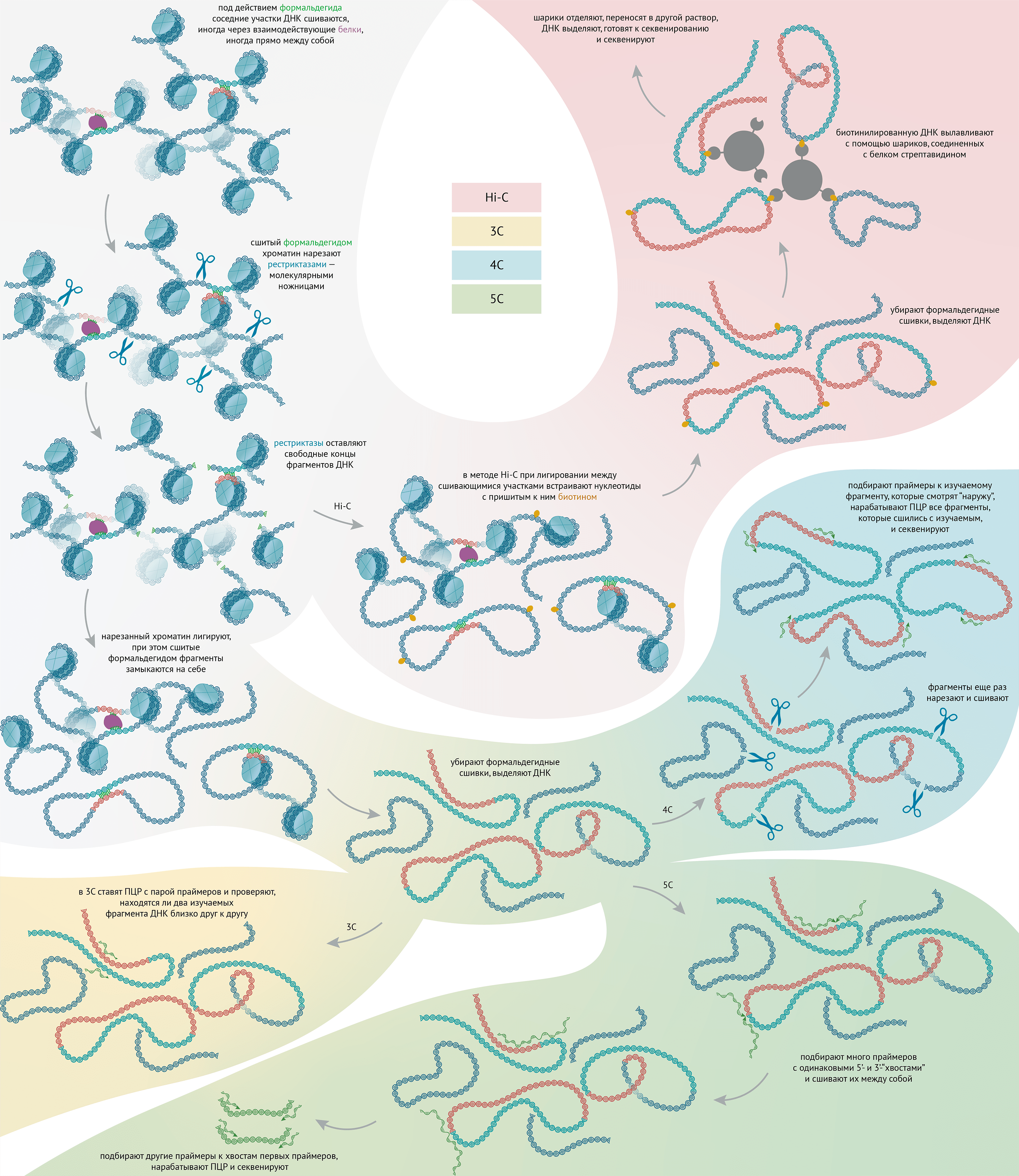
Рисунок 10. Схема С-методов. Представим себе два двухцепочечных участка ДНК, соединенные каким-нибудь белком. Сначала клетки фиксируют формальдегидом. При этом все молекулы в клетках химически сшиваются. В том числе фрагменты ДНК сшиваются с белками, которые их соединяют. Затем добавляют фермент рестриктазу, он действует как молекулярные ножницы и разрезает ДНК по определенным последовательностям. При этом разрезы вносятся в том числе и в фрагменты ДНК, соединенные с белком. После этого сшитые молекулы с порезанной ДНК разводят в большом объеме и сшивают ДНК заново ферментом лигазой. При этом пара фрагментов ДНК, находящихся близко друг к другу, сшиваются. Потом формальдегидные, но не ДНК-сшивки, химически удаляют, и белки, которые связывали близкие друг к другу участки ДНК, отделяются от уже гибридных участков ДНК. Теперь у нас имеется набор линейных и кольцевых молекул ДНК (в зависимости от того, сшилась одна или обе части фрагментов), включающие в себя фрагменты ДНК, которые находились рядом. В 3С, подобрав правильные праймеры, методом ПЦР проверяют, находятся ли два известных участка ДНК (например, промотор и энхансер) близко друг к другу [72].
Во втором методе из серии — 4С — гибридные молекулы ДНК, полученные по схеме 3С, разрезают рестриктазами и сшивают лигазами еще раз, при этом образовавшиеся после рестриктаз короткие фрагменты ДНК лигируются сами на себя, образуя кольцевые молекулы. Дальше ставят так называемую обратную ПЦР — праймеры подбирают к концам фрагмента, последовательность которого мы знаем, только с тем расчетом, чтобы ДНК синтезировалась не «внутрь», а «наружу», на последовательностях, которые мы не знаем. Таким образом, нарабатываются все фрагменты ДНК, сшившиеся и, следовательно, находившиеся рядом с изучаемым фрагментом ДНК [73], [74].
В методе 5С подбирают коллекцию двух типов праймеров, у которых один конец одинаков, а другой нет. Они покрывают своими 3′- или 5′-фрагментами выбранную часть молекулы ДНК. Можно комплементарно спарить эти праймеры со сшитыми линейными или кольцевыми фрагментами из 3С, и сшить между собой праймеры, которые окажутся рядом. A потом методом ПЦР увеличить количество этих сшивок, подобрав праймеры к 3′- или 5′-концам, одинаковым у всех предыдущих праймеров [75].
И, наконец, самый последний метод — Hi-C — позволяет секвенировать всё подряд и построить карту взаимодействий всех участков со всеми. Это достигается за счет того, что при лигировании добавляются и встраиваются на липкие концы гибридных молекул нуклеотидные остатки, к которым пришит биотин. Как мы помним, биотин очень прочно взаимодействует с белком стрептавидином, это одно из самых сильных взаимодействий в молекулярной биологии. Пользуясь таким сильным взаимодействием, можно выловить часть гибридных фрагментов ДНК, куда встроился нуклеотидный остаток с биотином, на шарики с пришитым на них стрептавидином, и секвенировать гибридные участки [29].
В последнее время получил распространение метод Micro-C, который похож на Hi-C, но в котором вместо рестриктазы используется микрококковая нуклеаза. Этот фермент режет ДНК гораздо чаще рестриктазы, внося разрывы между нуклеосомами, и результаты Micro-C имеют более высокое разрешение [76].
иллюстрация Михаила Гурьева
Методы Hi-C и Micro-C произвели настоящую революцию в биологии клеточного ядра. Благодаря им мы знаем, по каким принципам ДНК уложена в ядре, и какие архитектурные элементы и детали есть у генома в пространстве (см. «Хроматин в 3D»).
Иммунопреципитация, биотинилирование окрестности и масс-спектрометрия
Чтобы определить новые белки, вовлеченные в определенный молекулярный каскад (и, следовательно, выполняющие какую-то молекулярную функцию), можно выявить неизвестные белки, взаимодействующие с уже известными, задействованными в этом каскаде. Один из способов сделать это — иммунопреципитация [83].
Для этого разрушают мембраны клеток, а из содержимого выделяют нужные части (например, ядра или митохондрии) и дробят их на отдельные молекулярные комплексы. Затем к материалу добавляют антитела или комплементарную РНК (в зависимости от того, вылавливаем ли мы из раствора белки или РНК), соединенные с шариками, и перемешивают раствор. Через некоторое время нужные молекулы, налипшие на шарики, можно извлечь и отмыть от неспецифически связавшихся молекул. Дальше белки на шариках можно разрезать протеазами на пептиды — специальными молекулярными ножницами для белков. Какие именно пептиды плавают в растворе, можно определить масс-спектрометрией [84], а по этому набору пептидов определить, какие белки антитела вытащили из клетки. Если же нас интересует РНК на шариках, то можно ее отделить от белков и секвенировать.
Интересно, что специальным методом можно выявить не только белки, взаимодействующие с «нашим», но и расположенные неподалеку от него в пространстве! Такой метод появился относительно недавно и получил название «биотинилирование окрестности» (proximity labeling) [85], [86]. Суть его в том, что ген изучаемого белка соединяют с геном специального белка, биотинилирующего всё вокруг себя. Такую конструкцию доставляют в клетку, а дальше происходит следующее: часть этого химерного белка будет располагаться в том же месте клетки, что и нативный белок. А биотинилирующий всё вокруг фрагмент будет притаскиваться в это место в клетке и биотинилировать всё вокруг. Конечно, нужно учитывать, что гибридный белок будет идти и по путям биотинилирующего белка, но для этого ставят и отрицательный контроль: смотрят, что он биотинилирует в тех же клетках сам по себе, не в составе химеры. Потом биотинилированные белки можно связать со стрептавидиновыми шариками, почистить и определить масс-спектрометрией.
Недавно выяснилось, что один из используемых биотинилирующих белков может биотинилировать РНК [87], а также ДНК [88].
Вероятно, метод скоро будет применяться в качестве альтернативы ChIP-seq’у и DamID для белков, которые очень подвижны на ДНК и которые очень сложно исследовать двумя известными методами (например, некоторые ремоделеры хроматина). Дело в том, что биотинилирование одним из известных белков занимает всего одну минуту, а сшивка формальдегидом — минимум 5–10 минут, не говоря уже о часах, которые занимает DamID. Поэтому на профилях таких белков, полученных ChIP-seq’ом и DamID, сложно выделить пики — места, на которых сидит изучаемый белок. А метод с биотинилирующим белком мог бы эту проблему решить и дать пики, полученные одноминутной реакцией.
Данные, полученные иммунопреципитацией и биотинилированием окрестности, отвечают на разные вопросы и дополняют друг друга. В случае иммунопреципитации мы получаем список всех белков/РНК, которые прочно взаимодействуют с нашим белком, а в случае биотинилирования окрестности — список всего, что находится поблизости. Последнее может быть важно, поскольку некоторые важные взаимодействия очень слабые или короткоживущие, и иммунопреципитацией их не поймать.
Оба метода рутинно используются в типичных экспериментах по эпигенетике. Благодаря им были описаны составы многих белковых комплексов и органелл.
Литература
- От атомов к древу (рецензия);
- От атомов к древу (отрывок из книги);
- Путешествие внутрь клеточного ядра, или Системная биология хроматина;
- Молекулярная биология;
- Липидный фундамент жизни;
- Физическая водобоязнь;
- Обо всех РНК на свете, больших и малых;
- Инге-Вечтомов С.Г. (2003). Матричный принцип в биологии (прошлое, настоящее, будущее?). «Экологическая генетика». 1 (спецвыпуск), 3–15;
- Модельные организмы: нематода;
- Passarge E. (1979). Emil Heitz and the concept of heterochromatin: longitudinal chromosome differentiation was recognized fifty years ago. Am. J. Hum. Genet. 31 (2), 106–115;
- Karolin Luger, Armin W. Mäder, Robin K. Richmond, David F. Sargent, Timothy J. Richmond. (1997). Crystal structure of the nucleosome core particle at 2.8 Å resolution. Nature. 389, 251-260;
- Вездесущий убиквитин;
- SUMO: японская борьба или уникальная посттрансляционная модификация?;
- Brian D. Strahl, C. David Allis. (2000). The language of covalent histone modifications. Nature. 403, 41-45;
- Steven Henikoff, John M. Greally. (2016). Epigenetics, cellular memory and gene regulation. Current Biology. 26, R644-R648;
- Война и мир: как устроить белковую жизнь?;
- Aydan Bulut-Karslioglu, Valentina Perrera, Manuela Scaranaro, Inti Alberto de la Rosa-Velazquez, Suzanne van de Nobelen, et. al.. (2012). A transcription factor–based mechanism for mouse heterochromatin formation. Nat Struct Mol Biol. 19, 1023-1030;
- Guillaume J. Filion, Joke G. van Bemmel, Ulrich Braunschweig, Wendy Talhout, Jop Kind, et. al.. (2010). Systematic Protein Location Mapping Reveals Five Principal Chromatin Types in Drosophila Cells. Cell. 143, 212-224;
- Peter V. Kharchenko, Artyom A. Alekseyenko, Yuri B. Schwartz, Aki Minoda, Nicole C. Riddle, et. al.. (2011). Comprehensive analysis of the chromatin landscape in Drosophila melanogaster. Nature. 471, 480-485;
- Bas van Steensel. (2011). Chromatin: constructing the big picture. The EMBO Journal. 30, 1885-1895;
- Jason Ernst, Manolis Kellis. (2012). ChromHMM: automating chromatin-state discovery and characterization. Nat Methods. 9, 215-216;
- Feng Yue, The Mouse ENCODE Consortium, Yong Cheng, Alessandra Breschi, Jeff Vierstra, et. al.. (2014). A comparative encyclopedia of DNA elements in the mouse genome. Nature. 515, 355-364;
- Aaron C. Daugherty, Robin W. Yeo, Jason D. Buenrostro, William J. Greenleaf, Anshul Kundaje, Anne Brunet. (2017). Chromatin accessibility dynamics reveal novel functional enhancers in C. elegans. Genome Res.. 27, 2096-2107;
- Yue Liu, Tian Tian, Kang Zhang, Qi You, Hengyu Yan, et. al.. (2018). PCSD: a plant chromatin state database. Nucleic Acids Research. 46, D1157-D1167;
- Horng D. Ou, Sébastien Phan, Thomas J. Deerinck, Andrea Thor, Mark H. Ellisman, Clodagh C. O’Shea. (2017). ChromEMT: Visualizing 3D chromatin structure and compaction in interphase and mitotic cells. Science. 357, eaag0025;
- Организовать геном: запутанная история гипотез и экспериментов;
- Hancock R. and Hughes M.E. (1982). Organization of DNA in the eukaryotic nucleus. Biol. Cell. 44, 201–212;
- James R. Paulson, U.K. Laemmli. (1977). The structure of histone-depleted metaphase chromosomes. Cell. 12, 817-828;
- E. Lieberman-Aiden, N. L. van Berkum, L. Williams, M. Imakaev, T. Ragoczy, et. al.. (2009). Comprehensive Mapping of Long-Range Interactions Reveals Folding Principles of the Human Genome. Science. 326, 289-293;
- Tsung-Han S. Hsieh, Assaf Weiner, Bryan Lajoie, Job Dekker, Nir Friedman, Oliver J. Rando. (2015). Mapping Nucleosome Resolution Chromosome Folding in Yeast by Micro-C. Cell. 162, 108-119;
- Jesse R. Dixon, Siddarth Selvaraj, Feng Yue, Audrey Kim, Yan Li, et. al.. (2012). Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 485, 376-380;
- Suhas S.P. Rao, Miriam H. Huntley, Neva C. Durand, Elena K. Stamenova, Ivan D. Bochkov, et. al.. (2014). A 3D Map of the Human Genome at Kilobase Resolution Reveals Principles of Chromatin Looping. Cell. 159, 1665-1680;
- Kyle P. Eagen. (2018). Principles of Chromosome Architecture Revealed by Hi-C. Trends in Biochemical Sciences. 43, 469-478;
- Мальчик или девочка;
- Мальчики налево, девочки направо... А остальные?;
- Johan H. Gibcus, Kumiko Samejima, Anton Goloborodko, Itaru Samejima, Natalia Naumova, et. al.. (2018). A pathway for mitotic chromosome formation. Science. 359, eaao6135;
- N. Naumova, M. Imakaev, G. Fudenberg, Y. Zhan, B. R. Lajoie, et. al.. (2013). Organization of the Mitotic Chromosome. Science. 342, 948-953;
- Geoffrey Fudenberg, Nezar Abdennur, Maxim Imakaev, Anton Goloborodko, Leonid A. Mirny. (2017). Emerging Evidence of Chromosome Folding by Loop Extrusion. Cold Spring Harb Symp Quant Biol. 82, 45-55;
- Mahipal Ganji, Indra A. Shaltiel, Shveta Bisht, Eugene Kim, Ana Kalichava, et. al.. (2018). Real-time imaging of DNA loop extrusion by condensin. Science. 360, 102-105;
- Ryu J.-K., Rah S.-H., Janissen R., Kerssemakers J.W.J., Dekker C. (2020). Resolving the step size in condensing — driven DNA loop extrusion identifies ATP binding as the step — generating process. bioRxiv;
- Je-Kyung Ryu, Allard J. Katan, Eli O. van der Sluis, Thomas Wisse, Ralph de Groot, et. al.. (2020). The condensin holocomplex cycles dynamically between open and collapsed states. Nat Struct Mol Biol. 27, 1134-1141;
- Yan Li, Judith H. I. Haarhuis, Ángela Sedeño Cacciatore, Roel Oldenkamp, Marjon S. van Ruiten, et. al.. (2020). The structural basis for cohesin–CTCF-anchored loops. Nature. 578, 472-476;
- Wendy A. Bickmore, Bas van Steensel. (2013). Genome Architecture: Domain Organization of Interphase Chromosomes. Cell. 152, 1270-1284;
- Irene Chiolo, Aki Minoda, Serafin U. Colmenares, Aris Polyzos, Sylvain V. Costes, Gary H. Karpen. (2011). Double-Strand Breaks in Heterochromatin Move Outside of a Dynamic HP1a Domain to Complete Recombinational Repair. Cell. 144, 732-744;
- Alistair N. Boettiger, Bogdan Bintu, Jeffrey R. Moffitt, Siyuan Wang, Brian J. Beliveau, et. al.. (2016). Super-resolution imaging reveals distinct chromatin folding for different epigenetic states. Nature. 529, 418-422;
- Tom Sexton, Eitan Yaffe, Ephraim Kenigsberg, Frédéric Bantignies, Benjamin Leblanc, et. al.. (2012). Three-Dimensional Folding and Functional Organization Principles of the Drosophila Genome. Cell. 148, 458-472;
- Stefania del Prete, Pawel Mikulski, Daniel Schubert, Valérie Gaudin. (2015). One, Two, Three: Polycomb Proteins Hit All Dimensions of Gene Regulation. Genes. 6, 520-542;
- Tyler McCann, William Tansey. (2014). Functions of the Proteasome on Chromatin. Biomolecules. 4, 1026-1044;
- Tobias Straub, Peter B. Becker. (2007). Dosage compensation: the beginning and end of generalization. Nat Rev Genet. 8, 47-57;
- Aindrila Chatterjee, Janine Seyfferth, Jacopo Lucci, Ralf Gilsbach, Sebastian Preissl, et. al.. (2016). MOF Acetyl Transferase Regulates Transcription and Respiration in Mitochondria. Cell. 167, 722-738.e23;
- Joel C. Eissenberg, Sarah C.R. Elgin. (2014). HP1a: a structural chromosomal protein regulating transcription. Trends in Genetics. 30, 103-110;
- M. David Stewart, Jiwen Li, Jiemin Wong. (2005). Relationship between Histone H3 Lysine 9 Methylation, Transcription Repression, and Heterochromatin Protein 1 Recruitment. MCB. 25, 2525-2538;
- Singh A.K., Schauer T., Pfaller L., Straub T., Mueller-Planitz F. (2021). The biogenesis and function of nucleosome arrays. bioRxiv;
- Denis Tolkunov, Karl A. Zawadzki, Cara Singer, Nils Elfving, Alexandre V. Morozov, James R. Broach. (2011). Chromatin remodelers clear nucleosomes from intrinsically unfavorable sites to establish nucleosome-depleted regions at promoters. MBoC. 22, 2106-2118;
- 12 методов в картинках: полимеразная цепная реакция;
- Важнейшие методы молекулярной биологии и генной инженерии;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- D. Stoddart, A. J. Heron, E. Mikhailova, G. Maglia, H. Bayley. (2009). Single-nucleotide discrimination in immobilized DNA oligonucleotides with a biological nanopore. Proceedings of the National Academy of Sciences. 106, 7702-7707;
- Нанопоровое секвенирование: на пороге третьей геномной революции;
- Gordon Robertson, Martin Hirst, Matthew Bainbridge, Misha Bilenky, Yongjun Zhao, et. al.. (2007). Genome-wide profiles of STAT1 DNA association using chromatin immunoprecipitation and massively parallel sequencing. Nat Meth. 4, 651-657;
- Новый метод CETCh-seq может за одну метку поймать много результатов;
- Benjamin L Kidder, Gangqing Hu, Keji Zhao. (2011). ChIP-Seq: technical considerations for obtaining high-quality data. Nat Immunol. 12, 918-922;
- Краткая история открытия и применения антител;
- Как молекулы РНК общаются с хроматином;
- J. M. Engreitz, A. Pandya-Jones, P. McDonel, A. Shishkin, K. Sirokman, et. al.. (2013). The Xist lncRNA Exploits Three-Dimensional Genome Architecture to Spread Across the X Chromosome. Science. 341, 1237973-1237973;
- Загадочное путешествие некодирующей РНК Xist по X-хромосоме;
- Bas van Steensel, Steven Henikoff. (2000). Identification of in vivo DNA targets of chromatin proteins using tethered Dam methyltransferase. Nat Biotechnol. 18, 424-428;
- L. Song, G. E. Crawford. (2010). DNase-seq: A High-Resolution Technique for Mapping Active Gene Regulatory Elements across the Genome from Mammalian Cells. Cold Spring Harbor Protocols. 2010, pdb.prot5384-pdb.prot5384;
- Jason D. Buenrostro, Beijing Wu, Howard Y. Chang, William J. Greenleaf. (2015). ATAC‐seq: A Method for Assaying Chromatin Accessibility Genome‐Wide. Current Protocols in Molecular Biology. 109;
- Новый взгляд на геном: не просто цепочка генов, а трехмерная сеть, интегрирующая функциональные домены ядра;
- Пространственный контакт;
- J. Dekker. (2002). Capturing Chromosome Conformation. Science. 295, 1306-1311;
- Marieke Simonis, Petra Klous, Erik Splinter, Yuri Moshkin, Rob Willemsen, et. al.. (2006). Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture–on-chip (4C). Nat Genet. 38, 1348-1354;
- Zhihu Zhao, Gholamreza Tavoosidana, Mikael Sjölinder, Anita Göndör, Piero Mariano, et. al.. (2006). Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347;
- J. Dostie, T. A. Richmond, R. A. Arnaout, R. R. Selzer, W. L. Lee, et. al.. (2006). Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Research. 16, 1299-1309;
- Tsung-Han S. Hsieh, Assaf Weiner, Bryan Lajoie, Job Dekker, Nir Friedman, Oliver J. Rando. (2015). Mapping Nucleosome Resolution Chromosome Folding in Yeast by Micro-C. Cell. 162, 108-119;
- F. Song, P. Chen, D. Sun, M. Wang, L. Dong, et. al.. (2014). Cryo-EM Study of the Chromatin Fiber Reveals a Double Helix Twisted by Tetranucleosomal Units. Science. 344, 376-380;
- Jon-Matthew Belton, Rachel Patton McCord, Johan Harmen Gibcus, Natalia Naumova, Ye Zhan, Job Dekker. (2012). Hi–C: A comprehensive technique to capture the conformation of genomes. Methods. 58, 268-276;
- Alexey A. Gavrilov, Arkadiy K. Golov, Sergey V. Razin. (2013). Actual Ligation Frequencies in the Chromosome Conformation Capture Procedure. PLoS ONE. 8, e60403;
- Takashi Nagano, Yaniv Lubling, Eitan Yaffe, Steven W Wingett, Wendy Dean, et. al.. (2015). Single-cell Hi-C for genome-wide detection of chromatin interactions that occur simultaneously in a single cell. Nat Protoc. 10, 1986-2003;
- Houda Belaghzal, Job Dekker, Johan H. Gibcus. (2017). Hi-C 2.0: An optimized Hi-C procedure for high-resolution genome-wide mapping of chromosome conformation. Methods. 123, 56-65;
- Ulahannan N., Pendleton M., Deshpande A., Schwenk S., Behr J.M., Dai X. et al. (2019). Nanopore sequencing of DNA concatemers reveals higher-order features of chromatin structure. bioRxiv;
- Giuseppina Maccarrone, Juan Jose Bonfiglio, Susana Silberstein, Christoph W. Turck, Daniel Martins-de-Souza. (2017). Characterization of a Protein Interactome by Co-Immunoprecipitation and Shotgun Mass Spectrometry. Multiplex Biomarker Techniques. 223-234;
- 12 методов в картинках: протеомика;
- H.-W. Rhee, P. Zou, N. D. Udeshi, J. D. Martell, V. K. Mootha, et. al.. (2013). Proteomic Mapping of Mitochondria in Living Cells via Spatially Restricted Enzymatic Tagging. Science. 339, 1328-1331;
- Kyle J. Roux, Dae In Kim, Manfred Raida, Brian Burke. (2012). A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells. Journal of Cell Biology. 196, 801-810;
- Furqan M. Fazal, Shuo Han, Kevin R. Parker, Pornchai Kaewsapsak, Jin Xu, et. al.. (2019). Atlas of Subcellular RNA Localization Revealed by APEX-Seq. Cell. 178, 473-490.e26;
- Joseph R. Tran, Danielle I. Paulson, James J. Moresco, Stephen A. Adam, John R. Yates, et. al.. (2021). An APEX2 proximity ligation method for mapping interactions with the nuclear lamina. Journal of Cell Biology. 220.