
Face2Gene — фенотипирование нового поколения
10 января 2022
Face2Gene — фенотипирование нового поколения
- 1371
- 0
- 4
Логотип программного обеспечения Face2Gene для установки на персональный компьютер
-
Автор
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: На первый взгляд, арсенал современного врача-генетика кажется довольно обширным: генеалогический метод, анализ на биохимические маркеры заболевания, поиск мутаций в генах, ассоциированных с болезнью... Но достаточно ли этого? На какой кухне варится узкоспециализированный и немного загадочный специалист в сонме подопечных Гиппократа, и почему фенотипирование может вывести клиническую диагностику на новый уровень?
Конкурс «Био/Мол/Текст»-2021/2022
Эта работа опубликована в номинации «Академия & бизнес» конкурса «Био/Мол/Текст»-2021/2022.
Партнер номинации — BIOCAD.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

Генеральный партнер конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
У травматолога — рентген, а у врача-генетика — Face2Gene?
Еще несколько лет назад длительность постановки диагноза наследственной патологии могла занимать до пяти лет [1]. Предупредить развитие осложнений многих генетических заболеваний можно, быстро выявив его и приступив к лечению, но когда это занимает годы, риск возникновения неблагоприятных последствий существенно возрастает. В связи с этим исследователи и врачи долгое время искали наиболее эффективные подходы, комбинации методов, способные вовремя помочь пациентам.
В 2018 году американский врач-генетик Карен Грипп, в поисках диагноза наследственного заболевания своей маленькой пациентки, решила прибегнуть к помощи разработки команды (и Грипп в нее тоже входила) FDNA — программе Face2Gene, которая относится к методам фенотипирования нового поколения (NGP). Грипп использовала программу, чтобы подтвердить или опровергнуть наличие синдрома Видеманна—Штайнера у ребенка. Трудности в постановке диагноза возникли в связи с отсутствием у больной девочки многих физических проявлении заболевания, например, характерных черт лица. Однако кроме того, что ребенок потерял большую часть молочных зубов и приобрел несколько коренных в возрасте четырех лет, что стало тревожным звоночком для родителей и врачей, он не обладал привычной для таких больных внешностью.
Face2Gene работает благодаря искусственному интеллекту и позволяет, используя снимок лица пациента, обнаружить возможные генетические патологии, присущие изображенному на фотографии человеку. Карен Грипп еще до применения этого софта сделала предположение относительно диагноза, однако она решила проверить свою догадку. Face2Gene подтвердил ее, тогда врач воспользовалась более классическим молекулярно-генетическим методом анализа уже прицельно. По окончании работы с пациентом Карен Грипп была приятно удивлена: мало того, что Face2Gene верно определил заболевание, так еще и сэкономил время и реактивы (а значит, и деньги), сразу направив Карен в верном направлении [2]. После Грипп сравнит поиск в Face2Gene с поиском в Google, а тысячи врачей по всему миру начнут использовать футуристическую технологию на практике, тем самым совершенствуя ее...

Рисунок 1. Девочка с синдромом Ангельмана. Иногда определить наследственное заболевание очень сложно по фенотипу, но характерные особенности есть всегда, например, в данном случае: широкий рот, выдающийся вперед подбородок.
Я думаю, что у каждого педиатра и генетика будет такое приложение, и он будет использовать его, как свой стетоскоп [2].
Врач-генетик: наши дни
Для многих уже не секрет, чем жонглирует в мире науки генетик, а вот врач-генетик — это уже другая история. Не во всех крупных городах есть ординатура, подразумевающая серьезную подготовку специалиста данного профиля. Чаще всего дело ограничивается курсами профессиональной переподготовки. Но то, чем оперирует подобный специалист, охватывает очень много нюансов на стыке врачевания и науки. Давайте пройдемся по списку анализов и методов, которые используют врачи-генетики сегодня.
Во-первых, семейный анамнез, как самый безобидный способ «покопаться в генах» пациента. Генеалогический метод является косвенным, но позволяет довольно эффективно определить характер наследования и вероятность проявления заболевания [3]. Хороший вариант, но он как взгляд со стороны, который, к тому же, полагается на память пациента и его знания семейных заболеваний. Сегодня этап семейного анамнеза можно провести онлайн [4] — например, для жителей Москвы разработано мобильное приложение «ЕМИАС.ИНФО», при помощи которого в медицинскую карту можно внести данные по семейному анамнезу .
Во-вторых, биохимический подход, который в кругу врачей является более классическим и включает в себя выявление маркеров заболевания в крови пациента. Например, при подозрении на пропионовую ацидемию больной направляется на сдачу анализа для выяснения концентрации в крови ряда аминокислот. К сожалению, и он может давать на выходе ошибку — уж очень много факторов, которые влияют на биохимию крови: от питания, до того, выспался ли лаборант, который обрабатывал образец.
В-третьих, молекулярно-генетические методы, включающие в себя кариотипирование или пресловутую ПЦР, о которой сегодня слышат даже маленькие дети (о ней, кстати, на «Биомолекуле» выходила большая статья «12 методов в картинках: полимеразная цепная реакция» [5]). Ошибка не так велика, но определенно есть. Это может быть ошибка замены оснований полимеразы, повреждение ДНК в связи с воздействием высоких температур и ряд других несовершенств методики [6].
И в-четвертых, если клиника оснащена по первому слову техники, то могут быть использованы методы NGS, но тут в игру вступают бичи всех секвенаторов мира: загрязненные биологические образцы, наличие ингибиторов реакций, GC-богатые участки и шпильки, которые могут обрывать синтез, да и слегка рассеянных лаборантов тоже будет достаточно для того, чтобы что-то пошло не так. Показатели точности результата также не столь высоки, как хотелось бы для клинической диагностики. Например, доля ошибки при секвенировании коротких участков может варьировать от 5 до 20% для разных методов NGS [7]. До 55% клинических лабораторий используют секвенирование по Сэнгеру, ведь для получения результатов NGS-анализом необходимы не только специалисты в области «мокрой» биологии, но и биоинформатики.
Однако NGS до сих пор считается самой перспективной методикой для диагностики генетических заболеваний, ведь они считаются редкими и зачастую врачи, основываясь на осмотре и анализе крови, не могут поставить точный диагноз ввиду малого опыта лечения болезни и, возможно, избранного его проявления. Предполагается, что в недалеком будущем за точностью анализа будет следить искусственный интеллект, например он поможет формализовать некоторые этапы аннотирования, то есть определения в последовательности ДНК местонахождения и функции генов [7]. Сейчас возможности искусственного интеллекта адаптируют для нахождения потенциально патогенных мутаций. За совокупным использованием серьезных геномных анализов, наравне с более простыми генными, а также фенотипическими и биохимическими, будущее [4]. Стоит лишь привести в порядок тот хаос, который породил в мире Big Data быстрый скачок развития технологий, за которым клиническая диагностика никак не может поспеть [8].
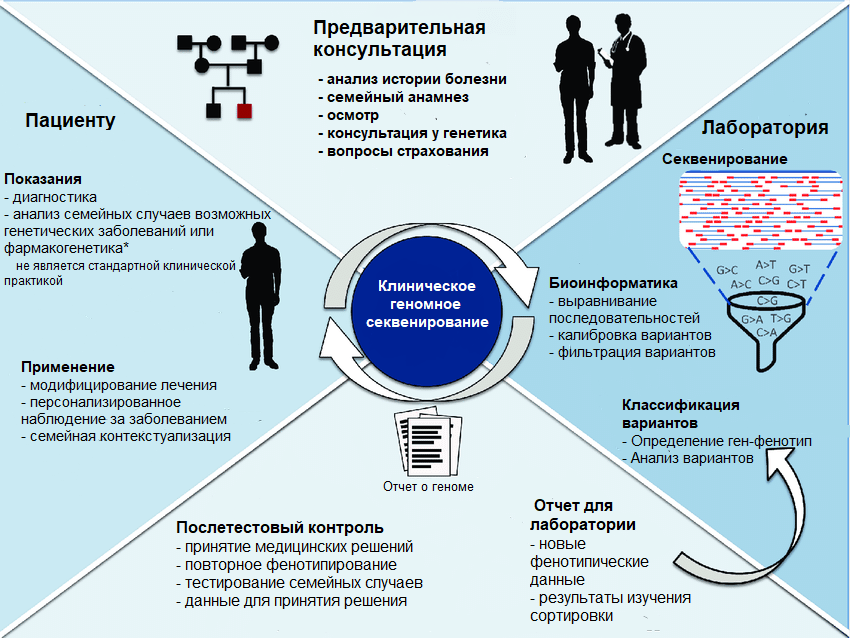
Рисунок 2. На схеме представлен план процесса клинического геномного секвенирования
Разберем-ка Face2Gene по винтикам
В самом начале 2019 года в журнале Nature Medicine вышла статья Identifying facial phenotypes of genetic disorders using deep learning («Идентификация лицевых фенотипов генетических заболеваний при помощи глубокого обучения»), в которой разработчики FDNA явили миру DeepGestalt, использующий алгоритмы глубокого обучения и компьютерное зрение. Именно DeepGestalt лег в основу Face2Gene [9]. Так как же работает этот яркий представитель фенотипирования нового поколения?
В основе Face2Gene лежит глубокая сверточная нейронная сеть. Немного подробнее о нейронных сетях вы можете прочесть в статье «Нейронные сети — будущие помощники врачей», опубликованной на «Биомолекуле» в прошлом году [10]. Фотографии, которые получает нейросеть, делятся на сегменты размером 100×100 пикселей, затем конвертируются в полутоновое изображение. После этого программа обрабатывает каждый полученный кусочек с определенной лицевой областью отдельно, чтобы затем объединить их обратно в один целостный образ для диагностического поиска на основе имеющейся базы данных Face2Gene. Формировалась эта база следующим образом: сперва в нее входили снимки из медицинских учебников с изображением лиц пациентов с генетическими заболеваниями, затем врачи стали добавлять снимки своих пациентов. Соответственно, с каждой новой фотографией процент точности программы повышается.
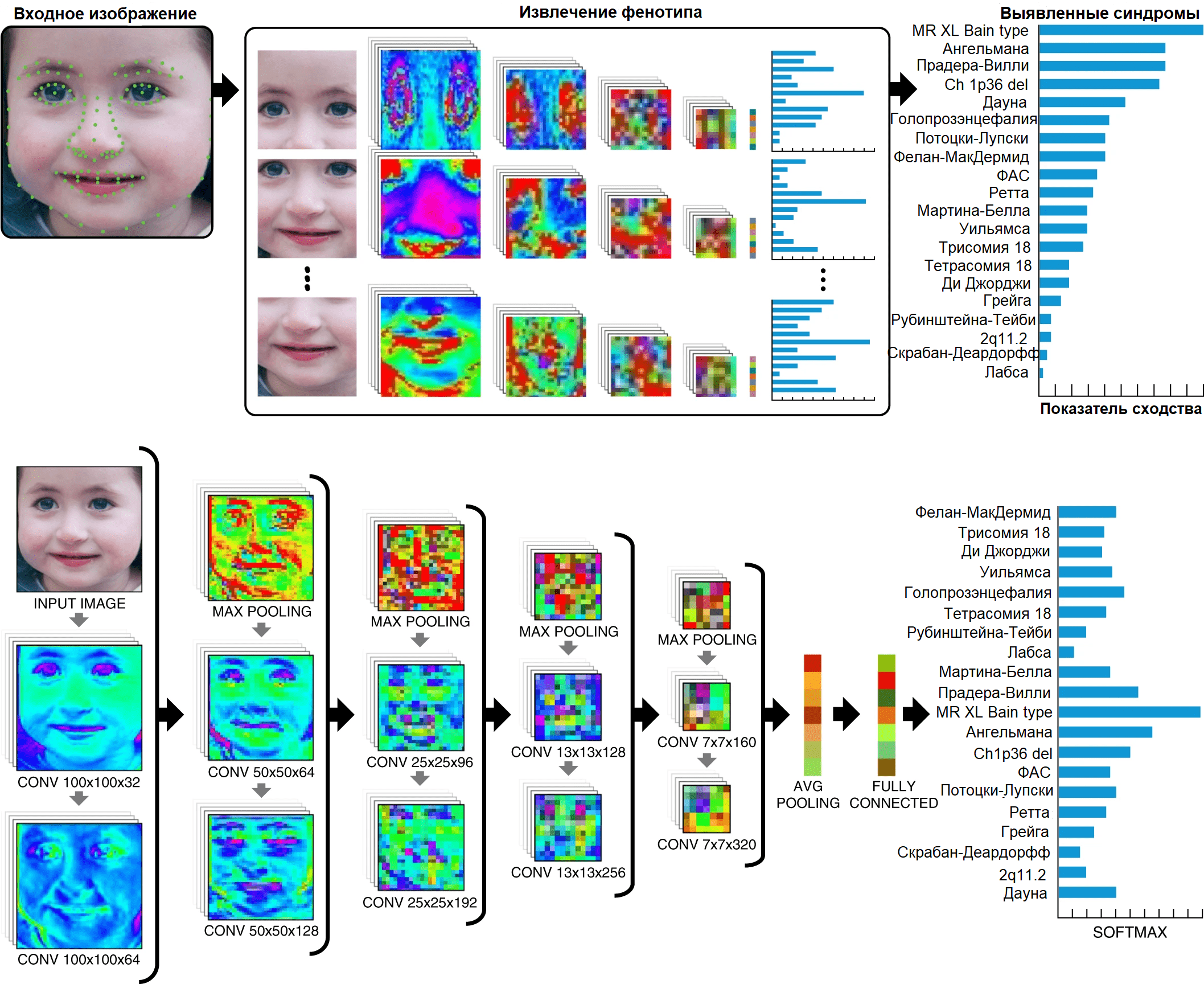
Рисунок 3. Этапы обработки изображения, подаваемого нейросети на вход. Видно, как сегментируется снимок для дальнейшего сравнения спектров со спектрами по отдельным синдромам из базы данных Face2Gene. Нейронная сеть состоит из 10 сверточных слоев, после каждой пары слоев следует слой объединения. Затем следует слой с выпадением, который необходим для удаления ненужных для выявления особенностей синдрома участков. К концу работы, как правило, характерные черты проявляются, а те, что были свойственны индивидуально пациенту, становятся незаметны. Последний слой softmax используется для задач классификации, когда количество возможных классов больше двух, после чего выходные данные сортируются для получения ранжированного списка генетических синдромов.
Также Face2Gene можно оптимизировать под определенные фенотипические характеристики. В самом начале проекта нейросеть была обучена на 17 106 снимках для 216 различных синдромов. Сегодня, благодаря регулярному пополнению базы данных, пользователь может проводить диагностику более чем 10 000 синдромов. А в основе этой диагностики лежат сотни тысяч снимков пациентов разных возрастов и этнической принадлежности.
Монополисты на рынке фенотипирования
FDNA, как создатели Face2Gene, уверенно занимают лидирующую позицию на рынке фенотипирования нового поколения (NGP). По данным, приведенным на официальном сайте, разработанную ими технологию используют 70% медицинских генетиков во всем мире (более чем в 130 странах). Благодаря тому, что разработку компании используют более чем в 2000 клинических центрах, база данных, которую программа берет за основу, постоянно пополняется и развивается, соответственно, увеличивая и достоверность диагностической оценки.
Сегодня подавляющее число больниц оснащено единой базой данных. Подобная тенденция способствовала развитию внутренних систем медицинских учреждений и поиску подходов к анализу данных, позволяющих помочь докторам в постановке диагноза и подборе терапии.
Ученые продолжают искать наиболее эффективные методы и алгоритмы для обработки данных, так к фенотипированию нового поколения относится не только технология Face2Gene, которая работает исключительно с изображением, но и разработки, анализирующие истории болезней из баз данных больниц. Например, Dr.Warehouse представляет собой систему полнотекстового поиска, способную выявить в компьютерном хранилище клиники похожие случаи (истории болезни). На вход программе поступают только записи врача с описанием. Подобное ПО позволяет быстро найти подобных больных, что оказывает существенное содействие при работе с носителями редких генетических заболеваний.
Аналогично разработчикам Face2Gene ряд исследователей работает над внедрением искусственного интеллекта в клиническую диагностику. Тогда как Face2Gene представляет собой развивающийся, но в то же время уже вполне обособленный и серьезный инструмент помощи врачам, способности нейросетей далеко не исчерпаны. Так, некоторые ученые анализируют способности компьютерного зрения в диагностике заболеваний [11].
На сегодняшний день в Face2Gene собраны данные по более чем 10 000 заболеваний, а полученные от пользователей снимки хранятся в общем безопасном хранилище, которое нейросеть использует для дальнейшего обучения.
Представители компании заявляют, что их основная цель — сделать фенотипирование нового поколения дополнительным стандартом к уже имеющимся секвенированию и биохимическому анализу в диагностике наследственных патологий с соблюдением конфиденциальности данных пациентов. Благодаря их разработке, страдающие от генетических заболеваний люди, которые теряют драгоценные дни во время постановки точного диагноза привычными методами, быстрее узнают диагноз и приступят к лечению.
Сегодня Face2Gene включает в себя следующие разделы:
- CLINIC — собственно, та самая часть приложения Face2Gene, которую врачи применяют в практике для диагностики наследственных патологий (загрузка и анализ изображения и выявление заболеваний);
- FORUMS — место, где можно оставлять и получать отзывы других пользователей и получать экспертную оценку своей работы;
- LIBRARY (London medical databases) — база данных синдромов для поиска интересующей информации и снимков;
- LABS — использование NGP для анализа данных NGS и изучения корреляции между двумя типами данных;
- RESEARCH — позволяет сравнивать и дополнять свои данные открытыми данными других ученых, включает в себя инструментарий для сравнительного анализа данных и его визуализации;
- ACADEMY — предоставляет возможность обучать студентов основным паттернам морфологии генетических патологий.
Рисунок 4. FDNA не собирается останавливаться на достигнутом: в перспективе они пополнят копилку методов, которые позволят дополнить данные, необходимые для клинической диагностики.
Число довольных пользователей нового девайса растет
В статье Evaluation of a computer-based facial dysmorphology analysis algorithm (Face2Gene) using standardized textbook photos («Оценка компьютерного алгоритма анализа дисморфологии лица (Face2Gene) с использованием стандартизированных фотографий из учебника») [12], опубликованной в журнале Nature, авторы отметили, что при помощи программы Face2Gene им удалось исключить ряд синдромов из предполагаемых при наличии неоднозначных клинических показателей, что подтвердило эффективность программы на практике.
Группа исследователей из Америки подтвердила эффективность применения Face2Gene для выявления редких наследственных патологий, даже в случае незначительных проявлений на уровне фенотипа [13]. Ученые во главе с Эмилией Дайкин, сотрудником Национальных институтов здоровья США, оценивают точность диагностики болезни Гоше при помощи Face2Gene в 90% [14].
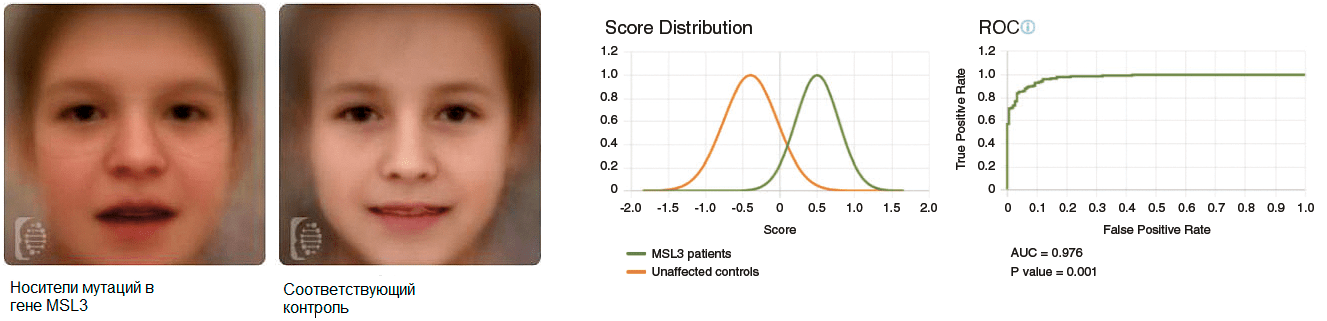
Рисунок 5. Из нескольких снимков лиц носителей патогенных мутаций в гене MSL3 Face2Gene конструирует составное изображение (крайнее слева), которое затем сравнивается с контрольным (второе слева). Размер площади под чертой AUC на графике ROC демонстрирует значительное отличие двух групп друг от друга (p < 0,005).
Группа ученых во главе с Томасом Лиром провела исследования по оценке эффективности фенотипирования нового поколения для пациентов с синдромом кошачьего глаза. Данный синдром характеризуется наличием в кариотипе дополнительной хромосомы и в клинической практике обычно подтверждается цитогенетическим анализом. С пылу с жару следующий вывод: «Несмотря на то, что сегодня технологии секвенирования следующего поколения с интерпретацией вариантов ДНК явно “в моде”, они не в состоянии решить все проблемы медицинской генетики. Технологии NGP, такие как Face2Gene, — это еще один новый игрок в цитогенетике, которому следует уделить больше внимания» [15].
Рост количества новых генетических синдромов, открываемых в связи с возросшим количеством лабораторий по всему миру и активным изучением человеческого генома в разных популяциях, поставил врачей в трудное положение. Им явно нужен «третий глаз», способный помочь в поисках верного диагноза. Исследователи отмечают также то, что этническая принадлежность пациентов может затруднять применение Face2Gene в качестве подобного пятого элемента в арсенале врача-генетика. Возможно, со временем пополнение библиотеки снимков сможет решить данную проблему.
Как пример можно привести исследование, в котором группа ученых из Японии проверила эффективность Face2Gene на примере групп пациентов, набранных на территории страны. Так, для пациентов с диагнозом синдром Дауна Face2Gene верно определяла патологию как наиболее вероятную в 82,2% случаев и как вторую по вероятности в 100% [16].
Как на фенотипировании «сделать деньги»
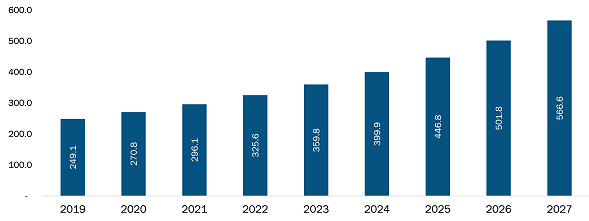
Рисунок 6. Показатели настоящей и будущей выручки рынка генетического тестирования всех типов в Мексике (по оси Y указана примерная сумма выручки в млн долларов США).
Доходы компаний, чья деятельность связана с генетическим анализом, растет с каждым годом. В то время как стоимость полногеномного секвенирования падает, просвещенность населения и интерес к собственной «базе данных» (то есть к ДНК) продолжает расти. Благодаря генетическому тестированию можно определить риски по многим заболеваниям, выяснить, откуда пришли ваши предки, проверить есть ли у вас непереносимость каких-либо продуктов. Интересными бонусами подобных анализов являются подробности, касающиеся персональной и наиболее оптимальной диеты для пациента, а также получение рекомендаций по выбору вида спорта, занятия которым будут вам полезны.
На Google Play и App Store приложение Face2Gene доступно для скачивания бесплатно, это сделано для того, чтобы в ускоренном темпе пополнялась база данных, что в свою очередь будет работать на улучшение прогностических способностей программы. Однако использовать приложение могут только специалисты: для регистрации необходимо указать должность и место работы. Помимо этого на платформах для скачивания можно наблюдать низкий рейтинг Face2Gene, но подавляющее большинство недовольных пользователей в жалобах ссылаются на проблемы и ошибки при регистрации. Вероятнее всего, это связано с несовершенством приложения, и в скором времени разработчики решат проблему.
На сайте Face2Gene можно увидеть следующие цены за использование ресурсов: за 10 долларов в месяц (цена без акции равна 70 долларам в месяц) можно получить доступ к LIBRARY.
По оценкам Growjo (сервиса для определения и прогнозирования роста мировых компаний), доход FDNA составляет 8,1 млн долларов в год. При этом официально в компании работает 52 сотрудника, таким образом, доход на одного человека равен 155 тысяч долларов в год. Согласитесь, работают сотрудники FDNA вполне успешно. Кстати, имеется одна вакансия...
По некоторым общим оценкам рынка генетического тестирования, ожидается, что в 2024 году его объем составит 22 миллиарда долларов США [7]. Поживем — увидим, какую долю из этой суммы перетянет на себя Face2Gene...
С некоторыми редкими заболеваниями врачи-генетики могут столкнуться минимальное количество раз за всю свою практику. Быстрый и неинвазивный анализ, для применения которого необходимы только фотографии пациента, поможет определить направление поиска, подтвердить или опровергнуть предварительный диагноз. Для носителей многих генетических патологий именно время может стать решающим фактором для подбора эффективной терапии и наиболее благоприятного прогноза. По всей видимости, технология фенотипирования нового поколения, такая как Face2Gene, в скором времени станет неотъемлемой частью клинической диагностики генетических заболеваний, дополняя более привычное генетическое тестирование и биохимический анализ наличия маркеров в крови.
Литература
- Joanna S. Amberger, Carol A. Bocchini, François Schiettecatte, Alan F. Scott, Ada Hamosh. (2015). OMIM.org: Online Mendelian Inheritance in Man (OMIM®), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798;
- Elie Dolgin. (2019). AI face-scanning app spots signs of rare genetic disorders. Nature;
- Бочков Н.П., Пузырев В.П., Смирнихина С.А. Клиническая генетика. М.: «ГЭОТАР-Медиа», 2018. — 592 с.;
- Yasmin Bylstra, Sonia Davila, Weng Khong Lim, Ryanne Wu, Jing Xian Teo, et. al.. (2019). Implementation of genomics in medical practice to deliver precision medicine for an Asian population. npj Genom. Med.. 4;
- 12 методов в картинках: полимеразная цепная реакция;
- Vladimir Potapov, Jennifer L. Ong. (2017). Examining Sources of Error in PCR by Single-Molecule Sequencing. PLoS ONE. 12, e0169774;
- Zhichao Liu, Liyuan Zhu, Ruth Roberts, Weida Tong. (2019). Toward Clinical Implementation of Next-Generation Sequencing-Based Genetic Testing in Rare Diseases: Where Are We?. Trends in Genetics. 35, 852-867;
- Krier J.B., Kalia S.S., Green R.C. (2016). Genomic sequencing in clinical practice: applications, challenges, and opportunities. Dialogues in clinical neuroscience. 18, 299–312;
- Yaron Gurovich, Yair Hanani, Omri Bar, Guy Nadav, Nicole Fleischer, et. al.. (2019). Identifying facial phenotypes of genetic disorders using deep learning. Nat Med. 25, 60-64;
- Нейронные сети — будущие помощники врачей;
- Roos van der Donk, Sandra Jansen, Janneke H.M. Schuurs-Hoeijmakers, David A. Koolen, Lia C.M.J. Goltstein, et. al.. (2019). Next-generation phenotyping using computer vision algorithms in rare genomic neurodevelopmental disorders. Genetics in Medicine. 21, 1719-1725;
- Matthew J. Javitt, Elizabeth A. Vanner, Alana L. Grajewski, Ta C. Chang. (2021). Evaluation of a computer-based facial dysmorphology analysis algorithm (Face2Gene) using standardized textbook photos. Eye;
- Bryan C. Mak, Rossana Sanchez Russo, Michael J. Gambello, Nicole Fleischer, Emily D. Black, et. al.. (2021). Craniofacial features of 3q29 deletion syndrome: Application of next‐generation phenotyping technology. Am J Med Genet. 185, 2094-2101;
- Emily Daykin, Nicole Fleischer, Magy Abdelwahab, Nehal Hassib, Raphael Schiffmann, et. al.. (2021). Investigation of a dysmorphic facial phenotype in patients with Gaucher disease types 2 and 3. Molecular Genetics and Metabolism. 134, 274-280;
- Thomas Liehr, Nicole Fleischer, Ahmed Al‐Rikabi. (2021). Next‐generation phenotyping in cat‐eye syndrome based on computer‐aided facial dysmorphology analysis of normal photographs. Mol Genet Genomic Med. 9;
- Hiroyuki Mishima, Hisato Suzuki, Michiko Doi, Mutsuko Miyazaki, Satoshi Watanabe, et. al.. (2019). Evaluation of Face2Gene using facial images of patients with congenital dysmorphic syndromes recruited in Japan. J Hum Genet. 64, 789-794;
- J Clayton-Smith, M E Pembrey. (1992). Angelman syndrome.. Journal of Medical Genetics. 29, 412-415;
- Theresa Brunet, Kirsty McWalter, Katharina Mayerhanser, Grace M. Anbouba, Amy Armstrong-Javors, et. al.. (2021). Defining the genotypic and phenotypic spectrum of X-linked MSL3-related disorder. Genetics in Medicine. 23, 384-395.







