12 методов в картинках: полимеразная цепная реакция
01 сентября 2017
12 методов в картинках: полимеразная цепная реакция
- 145848
- 17
- 110
рисунок Ольги Пташник
-
Авторы
-
Редакторы
-
Иллюстратор
Темы
Полимеразная цепная реакция почти для каждого из нас стала обыденностью, даже если этот каждый никогда и слов таких не слышал. Медицинские центры наперебой предлагают диагностировать у вас все мыслимые болезни с помощью «ПЦР-анализа». Но задумывались ли вы о том, что это за анализ? как там всё работает? для чего еще применяют ПЦР? и есть ли какие-то альтернативные, менее дорогие, трудоёмкие и, может быть, более эффективные методы анализа? Нет? А мы вам всё равно об этом расскажем...
12 биологических методов в картинках

Генеральный партнер цикла — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
Партнер этой статьи — Bio-Rad

Компания Bio-Rad Laboratories, Inc. USA («Био-Рад», США) является одним из мировых лидеров производства оборудования и реагентов для научных исследований. В рамках взаимодействия с научными, медицинскими, биотехнологическими и образовательными организациями «Био-Рад» предлагает современные технологии, оборудование и реагенты.
Одна из главных миссий «Биомолекулы» — докопаться до самых корней. Мы не просто рассказываем, какие новые факты обнаружили исследователи — мы говорим о том, как они их обнаружили, стараемся объяснить принципы биологических методик. Как вытащить ген из одного организма и вставить в другой? Как проследить в огромной клетке за судьбой нескольких крошечных молекул? Как возбудить одну крохотную группу нейронов в огромном мозге?
И вот мы решили рассказать о лабораторных методах более системно, собрать воедино в одной рубрике самые главные, самые современные биологические методики. Чтоб было интереснее и нагляднее, мы густо проиллюстрировали статьи и даже кое-где добавили анимации. Мы хотим, чтобы статьи новой рубрики были интересны и понятны даже случайному прохожему. И с другой стороны — чтобы они были так подробны, что даже профессионал мог бы обнаружить в них что-то новое. Мы собрали методики в 12 больших групп и собираемся сделать на их основе биометодический календарь. Ждите обновлений!
Полимеразная цепная реакция (ПЦР, PCR) — метод молекулярной биологии, позволяющий создать копии определенного фрагмента ДНК из исходного образца, повысив его содержание в пробе на несколько порядков.
Изобретение ПЦР полностью и безвозвратно изменило медицину, науку и нашу жизнь в целом. Появилась возможность быстро и эффективно диагностировать наследственные заболевания и инфекции, определять личность преступников по одному волоску и свободно манипулировать генами. Не будь этого уникального метода, человечество вряд ли оказалось бы на пороге эпохи генной терапии.
История метода
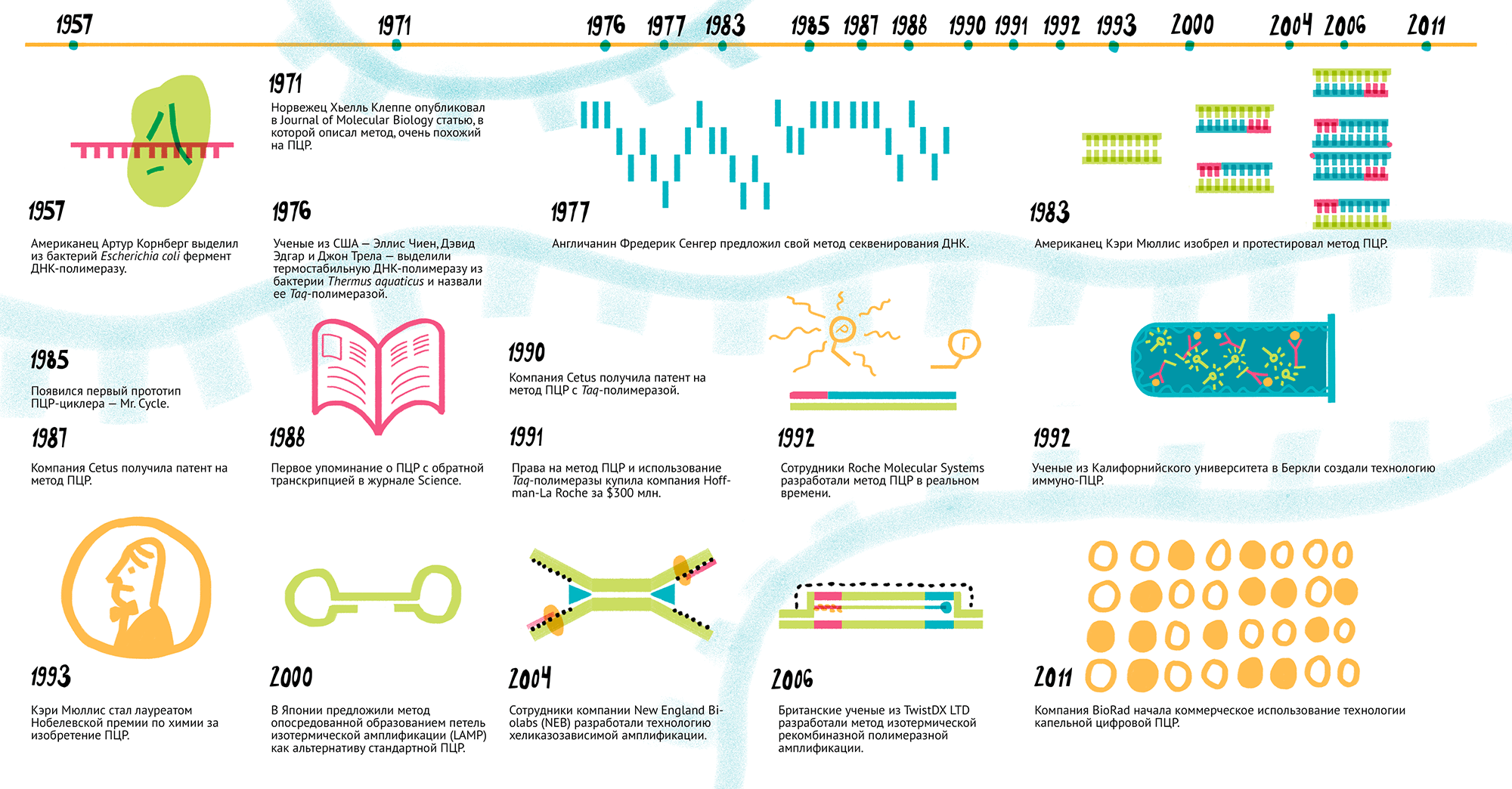
Рисунок 1. История развития метода ПЦР. 1957 г. Американец Артур Корнберг выделил из бактерий Escherichia coli фермент ДНК-полимеразу. 1971 г. Норвежец Хьелль Клеппе опубликовал в Journal of Molecular Biology статью, в которой описал метод, очень похожий на ПЦР. 1976 г. Ученые из США — Эллис Чиен, Дэвид Эдгар и Джон Трела — выделили термостабильную ДНК-полимеразу из бактерии Thermus aquaticus и назвали ее Taq-полимеразой. 1977 г. Англичанин Фредерик Сенгер предложил свой метод секвенирования ДНК. 1983 г. Американец Кэри Мюллис изобрел и протестировал метод ПЦР. 1985 г. Появился первый прототип ПЦР-циклера — Mr. Cycle. 1987 г. Компания Cetus получила патент на метод ПЦР. 1988 г. Первое упоминание о ПЦР с обратной транскрипцией в журнале Science. 1990 г. Компания Cetus получила патент на метод ПЦР с Taq-полимеразой. 1991 г. Права на метод ПЦР и использование Taq-полимеразы купила компания Hoffman-La Roche за $300 млн. 1992 г. Сотрудники Roche Molecular Systems разработали метод ПЦР в реальном времени. 1992 г. Ученые из Калифорнийского университета в Беркли создали технологию иммуно-ПЦР. 1993 г. Кэри Мюллис стал лауреатом Нобелевской премии по химии за изобретение ПЦР. 2000 г. В Японии предложили метод опосредованной образованием петель изотермической амплификации (LAMP) как альтернативу стандартной ПЦР. 2004 г. Сотрудники компании New England Biolabs (NEB) разработали технологию хеликазозависимой амплификации. 2006 г. Британские ученые из TwistDX LTD разработали метод изотермической рекомбиназной полимеразной амплификации. 2011 г. Компания BioRad начала коммерческое использование технологии капельной цифровой ПЦР.
Чтобы увидеть рисунок в полном размере, нажмите на него.
рисунок Ольги Пташник
1957 г. Американец Артур Корнберг впервые выделил из бактерий Escherichia coli фермент, который назвал ДНК-полимеразой [1]. Статьи с описанием работы он отправил в Journal of Biological Chemistry, где их отвергли... из-за названия фермента: рецензенты считали, что нужно использовать более точный термин «полидезоксирибонуклеотидполимераза» и что вообще «ДНК» в названии указывает на «генетическую активность» (сущность, составляющую?) фермента, а раз ее нет, то и называть фермент так нельзя [2]. Однако в 1958 году в журнале сменился главный редактор, и статьи наконец увидели свет [3]. А уже в 1959 году Артура Корнберга удостоили Нобелевской премии по физиологии и медицине.
1971 г. Норвежский биохимик Хьелль Клеппе (рис. 2) опубликовал в Journal of Molecular Biology статью, в которой описал метод, очень похожий на ПЦР [4]. С 1968 по 1970 годы Клеппе работал постдоком в Университете Висконсина, в лаборатории Хара Гобинда Кораны — нобелевского лауреата 1968 года за расшифровку генетического кода. Именно в лаборатории знаменитого индийца чуть раньше разработали методики синтеза олигонуклеотидных праймеров — «затравок», необходимых для работы ДНК-полимеразы.

Рисунок 2. Хьелль Клеппе.
Статья, написанная Клеппе в соавторстве с Кораной и посвященная репарационной репликации с помощью ДНК-полимеразы, содержала такие строки: «Можно ожидать, что после охлаждения получатся две структуры, каждая из которых содержит полноразмерную матричную цепь, подобающим образом связанную с праймером. Для завершения процесса репаративной репликации нужно будет добавить ДНК-полимеразу. В результате получатся уже две молекулы исходного дуплекса. Цикл можно повторять, каждый раз добавляя свежую порцию фермента».
К сожалению, эта гипотеза так гипотезой и осталась. Может, Клеппе даже и проводил какие-то эксперименты, но результаты не публиковал.
1976 г. Ученые из США, Эллис Чиен, Дэвид Эдгар и Джон Трела, выделили термостабильную ДНК-полимеразу из бактерии Thermus aquaticus и назвали ее Taq-полимеразой [5]. Этот фермент сохранял активность даже при температурах выше 75 °С.
1977 г. Фредерик Сенгер, английский биохимик и лауреат Нобелевской премии 1958 года за работы по структуре белков [6], предложил метод секвенирования ДНК, сейчас известный как метод Сенгера [7], [8]. Этим он заработал еще одну нобелевскую медаль, в 1980-м, и стал единственным ученым в истории, получившим две «химических» премии [9].
1983 г. Руководитель лаборатории синтеза ДНК в Cetus Corporation (США) Кэри Мюллис (рис. 3) апрельской ночью ехал вдоль побережья из Сан-Франциско в Мендосино, в свой загородный дом. Долгой трехчасовой дорогой он обдумывал отнюдь не проведение выходных, а предстоящий эксперимент по секвенированию ДНК. И тут (по словам Мюллиса) его озарило: он ясно представил процесс амплификации (преумножения) генов, который позже получит название полимеразной цепной реакции.

До работы в Cetus Кэри Мюллис изучал химию в Технологическом институте Джорджии, затем биохимию в Калифорнийском университете в Беркли, а после работал постдоком в Калифорнийском университете Сан-Франциско по направлению «фармацевтическая химия». В 1979 году его пригласили в Cetus синтезировать праймеры для секвенирования, а уже через два года Мюллис возглавил лабораторию синтеза ДНК и автоматизировал производство олигонуклеотидов [10].

Рисунок 3. Кэри Мюллис.
сайт www.playbuzz.com
В 1983 году Мюллис участвовал в проекте по изучению серповидноклеточной анемии. Чтобы проанализировать мутации, биологи проводили секвенирование по Сэнгеру, где используется один праймер для синтеза по одной из цепей ДНК. На серпантине по пути в Мендосино Мюллис предположил, что данные будут точнее, если использовать два праймера — для синтеза одновременно по двум цепям. Тогда можно будет сравнить получившиеся фрагменты и исключить неточности. И вот тут-то неожиданное прозрение заставило его вздрогнуть: если повторить цикл несколько раз, то можно получить множество копий нужного фрагмента строго определенной длины — она будет ограничена праймерами, от концов которых навстречу друг другу и будут строиться новые цепи ДНК [11]!
Вернувшись в понедельник в Cetus, Кэри Мюллис сразу направился в библиотеку, где попросил одного из сотрудников найти всю литературу о ДНК-полимеразе. В результате он не обнаружил ничего, касающегося амплификации. Это утверждение — самое слабое место во всей истории, так как в собранной библиотекарем стопке статей просто не могло не быть работы Хьелля Клеппе.
За следующие полгода Мюллис провел два эксперимента для проверки своей гипотезы, но безуспешно. Тогда он предположил, что отрицательный результат связан с большим размером ДНК-матрицы, используемой в опытах, и решил продолжить работу с маленьким вектором pBR322, в который вставил намножаемый ген. И 16 декабря 1983 года Мюллис впервые увидел вожделенные, хоть и слабые, полосы в геле для детекции. Однако другие сотрудники и руководство Cetus не разделили радости Мюллиса: их всё это попросту не интересовало [10].
1984 г. На ежегодной конференции корпорации Cetus в калифорнийском Монтерее Кэри Мюллис представил плакат, рассказывающий об амплификации гена β-глобина. К удивлению автора, и на этот раз его работу обошли вниманием [10].
Чуть позже Мюллису удалось кое-как убедить корпоративных боссов в важности его экспериментов: Cetus, как и многие начинающие компании, вкладывала ресурсы только в те проекты, что сулили прибыль в краткосрочной перспективе. Его освободили от обязанностей главы лаборатории и дали год на исследования ПЦР. И эти эксперименты завершились успешно [10].
1985 г. Мюллис и его группа разработчиков подали заявку на патент, который утвердили 28 июля 1987 года. В том же 1985-м в Cetus начали использовать для ПЦР термостабильную Taq-полимеразу, что значительно упростило работу: раньше перед каждым новым синтетическим циклом в смесь надо было добавлять новую порцию фермента, потому что он быстро выходил из строя от высоких температур. В декабре 1985 года журнал Science опубликовал первую статью Кэри Мюллиса о ПЦР [12].
Тогда же открылось совместное предприятие PerkinElmer Cetus Instruments (PECI), которое выпустило первый прототип ПЦР-циклера — Mr. Cycle (рис. 4). И только в 1987 году в продажу поступил первый общедоступный прибор, PCR-1000 Thermal Cycler.

Рисунок 4. Mr. Cycle — первый прототип ПЦР-амплификатора.
1986 г. Корпорация Cetus выплатила Кэри Мюллису премию $10 000. Остальные члены его группы получили по символическому доллару. Это обострило и так напряженные отношения в коллективе. Осенью Мюллис покинул компанию [13].
После ухода из Cetus он два года возглавлял молекулярно-биологический отдел в Xytronyx, а в 1992-м открыл компанию по продаже ювелирных изделий с амплифицированной ДНК знаменитостей — Элвиса Пресли, Мэрилин Монро и т.п. [14].
В 1993 году Кэри Мюллис стал лауреатом Нобелевской премии по химии за изобретение ПЦР. Его награждение — до сих пор больной вопрос для норвежского научного сообщества, где первооткрывателем метода считают Хьелля Клеппе.
Сейчас Мюллису 72 года, он работает научным сотрудником Научно-исследовательского института детской больницы Окленда и с 2011 года возглавляет предприятие Altermune LLC, занимающееся изучением иммунитета [14].
Кэри Мюллис нередко выступает на конференциях, рассказывает о своей жизни, работе и, конечно, своем открытии (см. видео 1). Он — прекрасный рассказчик, его всегда интересно слушать.
Видео 1. Выступление Кэри Мюллиса на конференции TED2002
Время от времени Мюллис принимает ЛСД. Однажды он рассказал о ночной встрече со светящимся зеленым енотом в лесу рядом со своим загородным домом. А Альберт Хоффман («отец» ЛСД) утверждал, что, по словам Мюллиса, ЛСД помог ему развить идею ПЦР [14].
В 1998 году вышла книга Кэри Мюллиса «Танец обнаженного разума» (Dancing naked in the mind field), где в обрамлении автобиографических историй ученый высказывает своё мнение о глобальном потеплении, СПИДе и других волнующих общество вопросах. Мюллис верит в астрологию и считает, что значительного изменения климата не происходит, между ВИЧ и СПИДом нет никакой связи, а все исследования, говорящие об обратном, — плоды заговора ученых-карьеристов с правительствами их стран.
1987 г. Компания Cetus подала патентную заявку на метод ПЦР с Taq-полимеразой, и ее одобрили в октябре 1990 года.
1989 г. Журнал Science объявил Taq-полимеразу молекулой года, а статья сотрудников Cetus о ПЦР с ее использованием [15] несколько лет поддерживала статус самой цитируемой биологической публикации [16].
В августе химический гигант DuPont подал против Cetus иск, в котором утверждал, что патенты на ПЦР получены неправомерно, поскольку этот процесс еще в 1970-х описал Хьелль Клеппе. В ответ на иск Ведомство по патентам и товарным знакам США (USPTO) решило переосвидетельствовать патенты. Но через год объявило, что они останутся действительными: комиссия нашла метод, описанный в работе Клеппе, слишком «неопределенным и сомнительным». К тому же там не упоминалась возможность экспоненциальной репликации — отличительной черты ПЦР. В суде представители DuPont так и не смогли доказать вторичность изобретения Мюллиса. 28 февраля 1991 года, после двух дней работы, суд вынес решение в пользу Cetus [10].
1991 г. Выигранный суд не помог корпорации Cetus: к ‘91-му году ее убытки превысили $60 млн, и в июле было объявлено о слиянии с биотехнологической компанией Chiron. А права на метод ПЦР и использование Taq-полимеразы в декабре продали компании Hoffman-La Roche за $300 млн [10]. С этой сделки Cetus выплатила Кэри Мюллису неоправданно малую сумму, фактически ограбив его.
С тех пор Hoffman-La Roche и ее «дочка» Roche Molecular Systems развивают метод полимеразной цепной реакции: у них уже более 1000 связанных с ПЦР патентов и заявок.
Принцип метода
Все мы знаем, что ДНК — это двухцепочечная молекула, где каждая цепочка состоит из звеньев-нуклеотидов. Нуклеотиды составлены из трех молекул: остатка фосфорной кислоты, сахара и азотистого основания. Если сахар и фосфат одинаковы у всех нуклеотидов в ДНК (в РНК сахар другой), то азотистых оснований четыре (если не считать редкие модификации): аденин, тимин, цитозин и гуанин, обозначаемые А, Т, Ц и Г соответственно. В молекулах РНК тимин заменен урацилом. Нуклеотиды соединяются в цепочку, образуя связи между фосфатной группой одного нуклеотида и гидроксильной — другого. В результате на одном конце каждой цепи ДНК «висит» фосфатная группа (5ˊ-конец), а на другом — гидроксильная (3ˊ-конец). Две цепи нуклеотидов расположены в молекуле ДНК антипараллельно, то есть напротив 3ˊ-конца одной находится 5ˊ-конец другой. Чтобы молекула была стабильной, цепочки должны как-то взаимодействовать друг с другом. Это обеспечивают водородные связи, образующиеся между азотистыми основаниями противоположных цепей по принципу комплементарности: А соединяется только с Т (или У в РНК), а Г — с Ц (рис. 5, видео 2). И поэтому, имея одну цепь ДНК, в соответствии с этим правилом легко построить ее пару. Собственно, на этом и основана ПЦР.
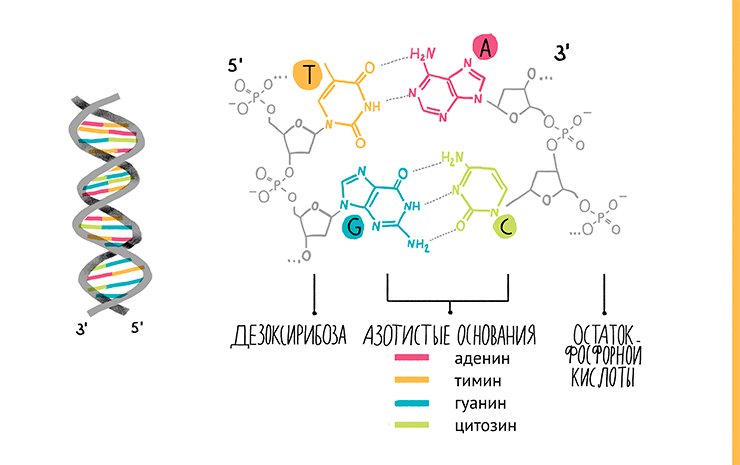
Рисунок 5. Строение ДНК.
рисунок Ольги Пташник
Видео 2. Строение ДНК.
Типичная реакционная смесь
- Анализируемая ДНК. Это может быть как отдельный кусочек молекулы, так и плазмида, хромосома или геном клетки полностью. Для грубой оценки сойдет даже суспензия клеток. ДНК служит матрицей для многократного копирования нужного участка.
- Праймеры.Праймер — это искусственно синтезированная короткая цепочка нуклеотидов (15–30 штук), комплементарная выбранному участку одной из цепей анализируемой ДНК. Один из праймеров обычно соответствует началу амплифицируемого отрезка, другой — его концу, но на противоположной цепи. У праймеров, как и у любого олиго- или полинуклеотида, есть 3ˊ- и 5ˊ-концы.
- Нуклеотиды. А точнее, дезоксинуклеотидтрифосфаты — четыре вида «кирпичиков» для строительства цепей ДНК: дАТФ, дТТФ, дЦТФ и дГТФ.
- ДНК-полимераза. Фермент, строящий комплементарную матричной цепь ДНК. Он может начинать синтез только от 3ˊ-конца праймера. Обычно используют термостабильные полимеразы, изначально выделенные из термофильных бактерий и архей: Thermus aquaticus (Taq-полимераза), Pyrococcus furiosus (Pfu-полимераза) и Pyrococcus woesei (Pwo-полимераза). Первая — самая производительная, а две другие — более точные.
- Буфер. Раствор, содержащий различные ионы для поддержания нужного рН, соли магния, необходимые для работы полимеразы, и неионный детергент Tween-20 в сочетании с BSA (бычьим сывороточным альбумином) для предотвращения налипания компонентов реакции на стенки пробирки. В случае ГЦ-богатых матриц в смесь часто добавляют энхансер — ДМСО (диметилсульфоксид), предотвращающий нежелательные взаимодействия между комплементарными участками матрицы.
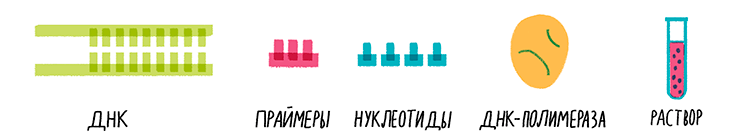
Рисунок 6. Состав смеси для ПЦР.
рисунок Ольги Пташник
Все компоненты смешивают в нужном объеме деионизованной воды в специальных пробирках для ПЦР и помещают в амплификатор (или ПЦР-циклер) (рис. 7, видео 3).
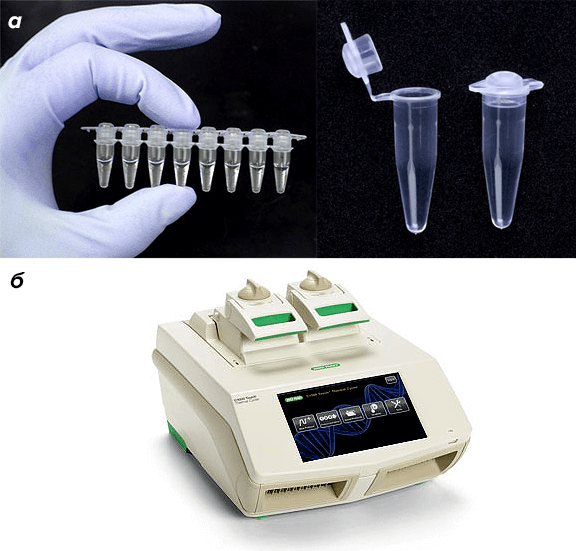
Рисунок 7. Расходные материалы и оборудование для ПЦР. а — ПЦР-пробирки. б — Амплификатор C1000 Touch™ производства Bio-Rad.
Видео 3. Приготовление смеси для ПЦР.
Этапы реакции
Цель ПЦР — получить множество одинаковых двухцепочечных кусочков ДНК строго определенной длины (обычно не более 2–3 тысяч пар нуклеотидов, т.п.н.). Для этого проводят 20–30 циклов реакции. Каждый цикл состоит из трех этапов.
1. Денатурация
Чтобы полимераза могла работать, две цепи ДНК-матрицы нужно разъединить. Для этого реакционную смесь нагревают до 94–98 °С. В таких условиях разрушаются водородные связи между азотистыми основаниями параллельных цепей.
2. Отжиг праймеров
На этом этапе праймеры специфично присоединяются к освободившимся цепям ДНК-матрицы с разных сторон копируемого участка 3ˊ-концами друг к другу (рис. 8, видео 4). Чтобы праймеры могли комплементарно связаться (отжечься) только с нужными участками, при их конструировании необходимо учитывать такую важную характеристику, как температура плавления (Тm). Это расчетная температура, при которой половина праймеров присоединяется к целевому участку ДНК. Отжиг проводят при температуре на 1–5 °С ниже Tm, но не выше оптимальной температуры работы полимеразы, то есть в пределах 40–72 °С [17].
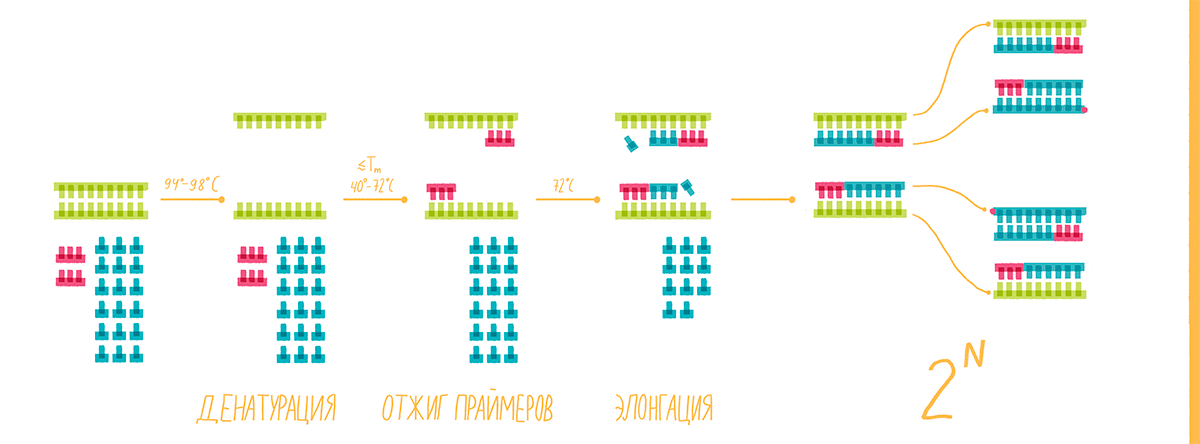
Рисунок 8. Полимеразная цепная реакция. На стадии денатурации цепи ДНК разъединяются, на следующем этапе (отжиг) к ним присоединяются праймеры, а далее полимераза начинает свою работу — синтез новых цепей ДНК (элонгация). И такой цикл повторяется многократно.
Чтобы увидеть рисунок в полном размере, нажмите на него.
рисунок Ольги Пташник
Видео 4. Полимеразная цепная реакция.
В идеале праймеры должны соответствовать следующим критериям:
- температуры плавления двух праймеров не должны различаться более чем на 5 °С;
- ГЦ-состав их должен уложиться в интервал 40–60%;
- в структуре олигонуклеотидов не должно быть шпилек (участков, комплементарных друг другу);
- праймеры не должны образовывать дуплексы (спариваться) друг с другом.
Еще лучше, если на 3ˊ-конце праймера будет гуанин или цитозин: они образуют с комплементарными основаниями три водородные связи (между А и Т образуются две), что делает комплекс праймер—матрица более стабильным.
В реальности редко получается соблюсти все условия из-за множества причин. Однако чем больше критериев соблюдено при создании праймеров, тем выше вероятность правильной их работы.
Чтобы разработать эффективные праймеры, необходимо знать последовательность ДНК у концов целевого участка, и, руководствуясь упомянутыми критериями, выбрать подходящие фрагменты, которым будут комплементарны будущие праймеры. Всё это удобно делать в специальных компьютерных программах — например, PrimerSelect: они и Тm рассчитают, и всякие спаривания изобразят, и вообще вынесут вердикт, удачная это пара праймеров или нет.
3. Элонгация, или синтез ДНК
Однажды ведущий ПЦР-специалист одного ветеринарного диагностического центра, показывая студентам постановку реакции, объяснила, что она потому называется полимеразной, потому что ее результаты наблюдают в полимерном геле. Возможно, есть и другие приверженцы этой гипотезы, однако сразу отметим, что она не верна. Полимеразная эта реакция от того, что в ее ходе фермент ДНК-полимераза последовательно выстраивает цепь ДНК (полимер) из нуклеотидов (мономеров), то есть полимеризует их. И делает она это на третьем этапе ПЦР.
Этот этап чаще проводят при температуре 72 °С — оптимальной для работы Taq-полимеразы. Фермент присоединяется к комплексам праймер—матрица и, выхватывая из раствора нуклеотиды, начинает по принципу комплементарности прилаживать их к 3ˊ-концу праймера (рис. 7). Удлинение, или элонгация, новой цепи ДНК идет с максимальной скоростью 50–60 нуклеотидов в секунду (то есть около 3000 в минуту). Однако при программировании ПЦР-циклера задают время с запасом: по минуте на каждую тысячу пар нуклеотидов.
Каждая вновь синтезированная цепочка ДНК становится, наравне со старой, матрицей для синтеза в следующем цикле. Таким образом, количество нужного продукта в процессе реакции возрастает экспоненциально. После прохождения всех циклов в реакционной смеси образуется столько специфических двухцепочечных продуктов, что их «массив» можно увидеть невооруженным глазом — проведя гель-электрофорез, о котором расскажем ниже.
К сожалению, экспоненциальная амплификация не может длиться вечно. Через 25–30 циклов количество функциональных молекул полимеразы в реакционной смеси истощается. Но чтобы добиться еще большего выхода продукта, содержимое пробирки можно разбавить, например, в 1000 раз и снова использовать для амплификации с уже новыми рабочими компонентами [18].
Визуализация продуктов ПЦР
Чтобы увидеть, намножились ли нужные участки ДНК, после окончания ПЦР содержимое пробирок подвергают электрофорезу в агарозном или полиакриламидном геле с последующим окрашиванием — так молекулы ДНК разной длины разделяются пространственно и становятся видны невооруженным глазом [19]. Полиакриламидный гель намного плотнее, поэтому больше подходит для разделения очень коротких фрагментов (несколько десятков пар нуклеотидов), при этом можно увидеть разницу даже в один нуклеотид!
Расплавленный при 65 °С гель заливают в специальную форму (плашку) с установленной в ней гребенкой, формирующей лунки (рис. 9). Когда гель застывает, гребенку вынимают, ставят форму в камеру для электрофореза и заливают специальным буфером. Затем в лунки микропипеткой вносят раствор из ПЦР-пробирок, смешанный с краской — чаще бромфеноловым синим. Чтобы потом определять размеры амплифицированных фрагментов, в отдельную лунку вносят маркер молекулярных масс (ladder), содержащий набор кусочков ДНК известных размеров. Камеру подключают к источнику питания и наблюдают за бегущими от электродов волшебными пузырьками. Десятки минут или несколько часов, зависит от размера фрагментов ДНК, плотности геля и приложенного напряжения (видео 5).
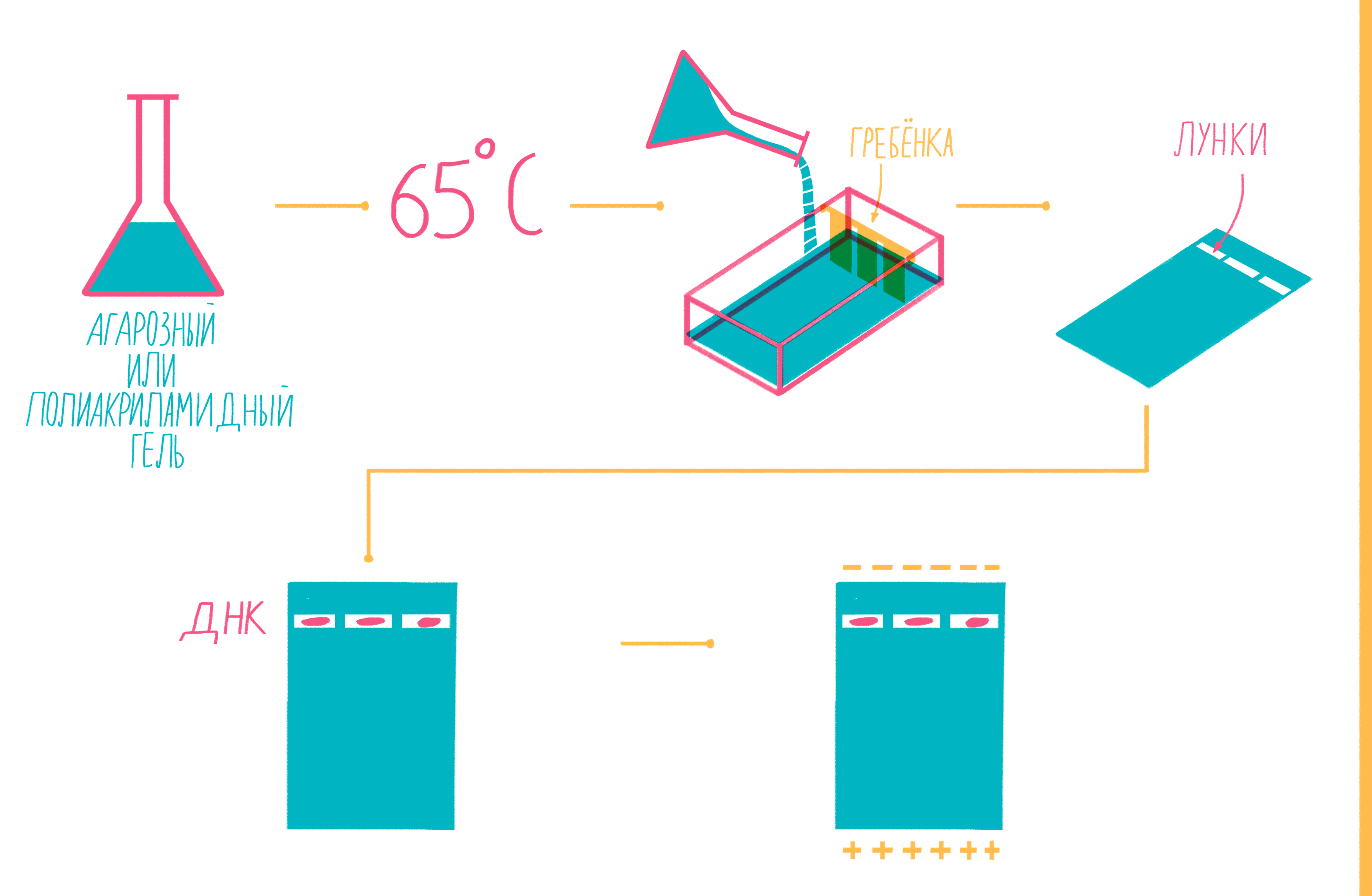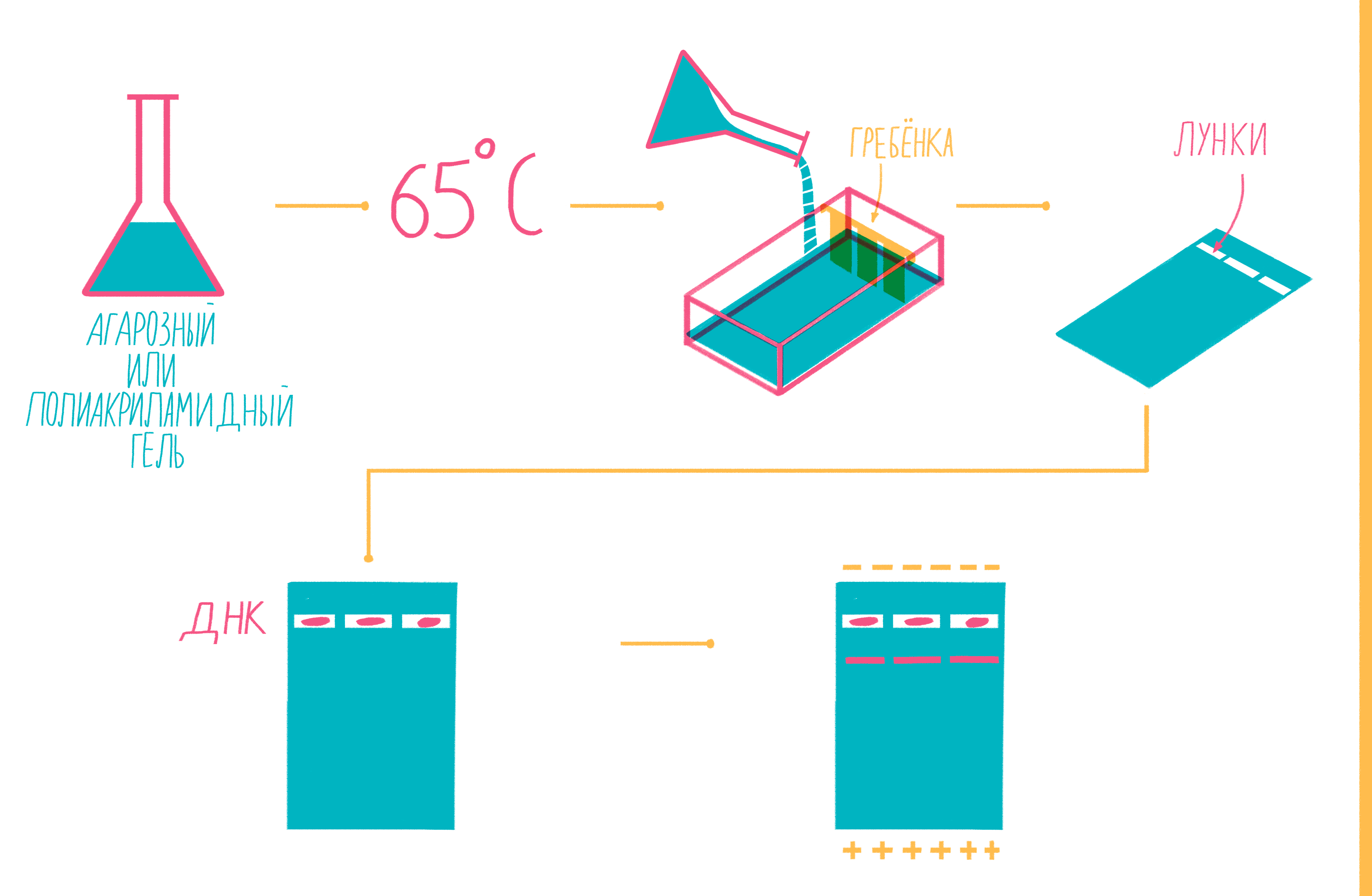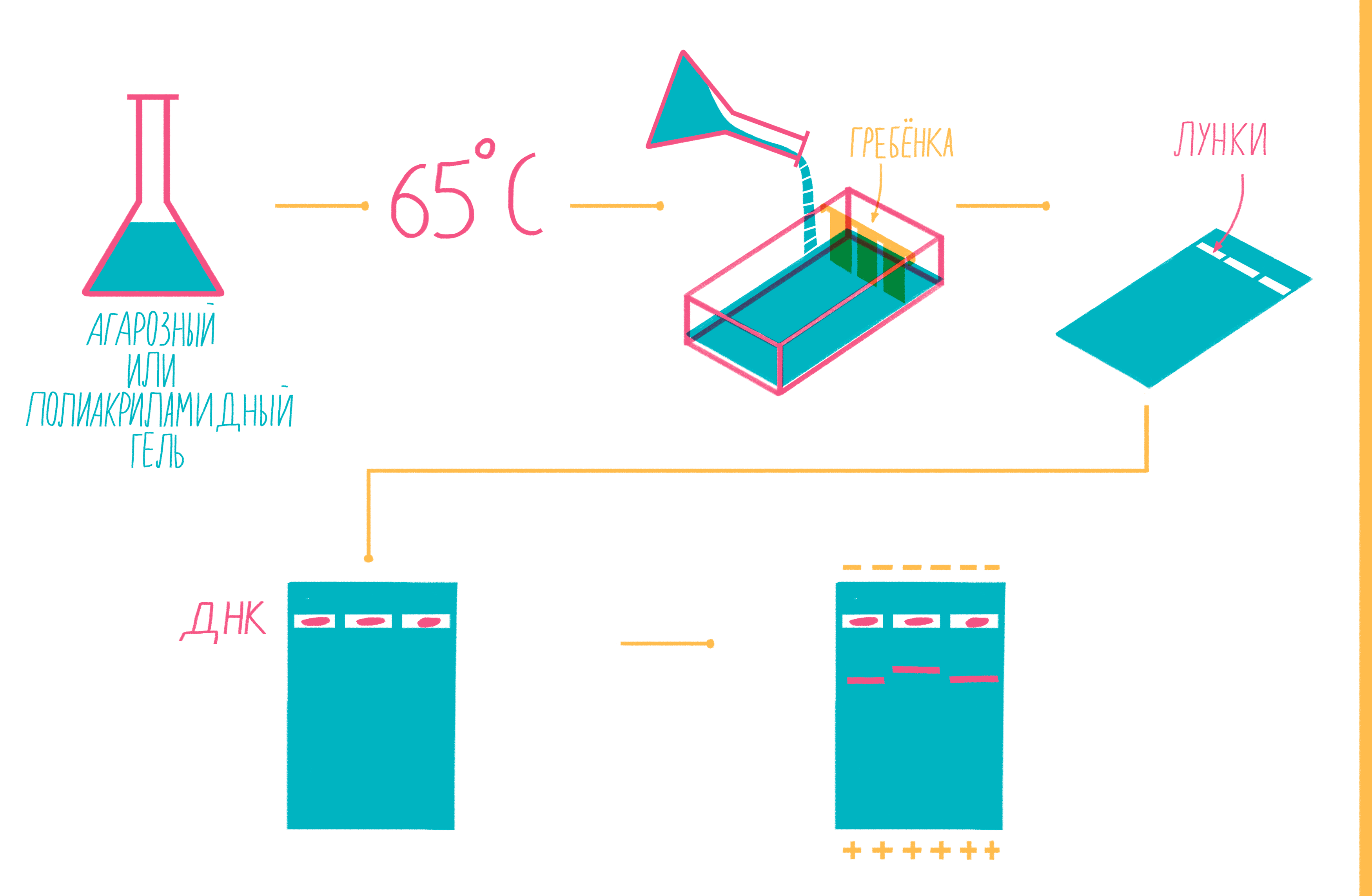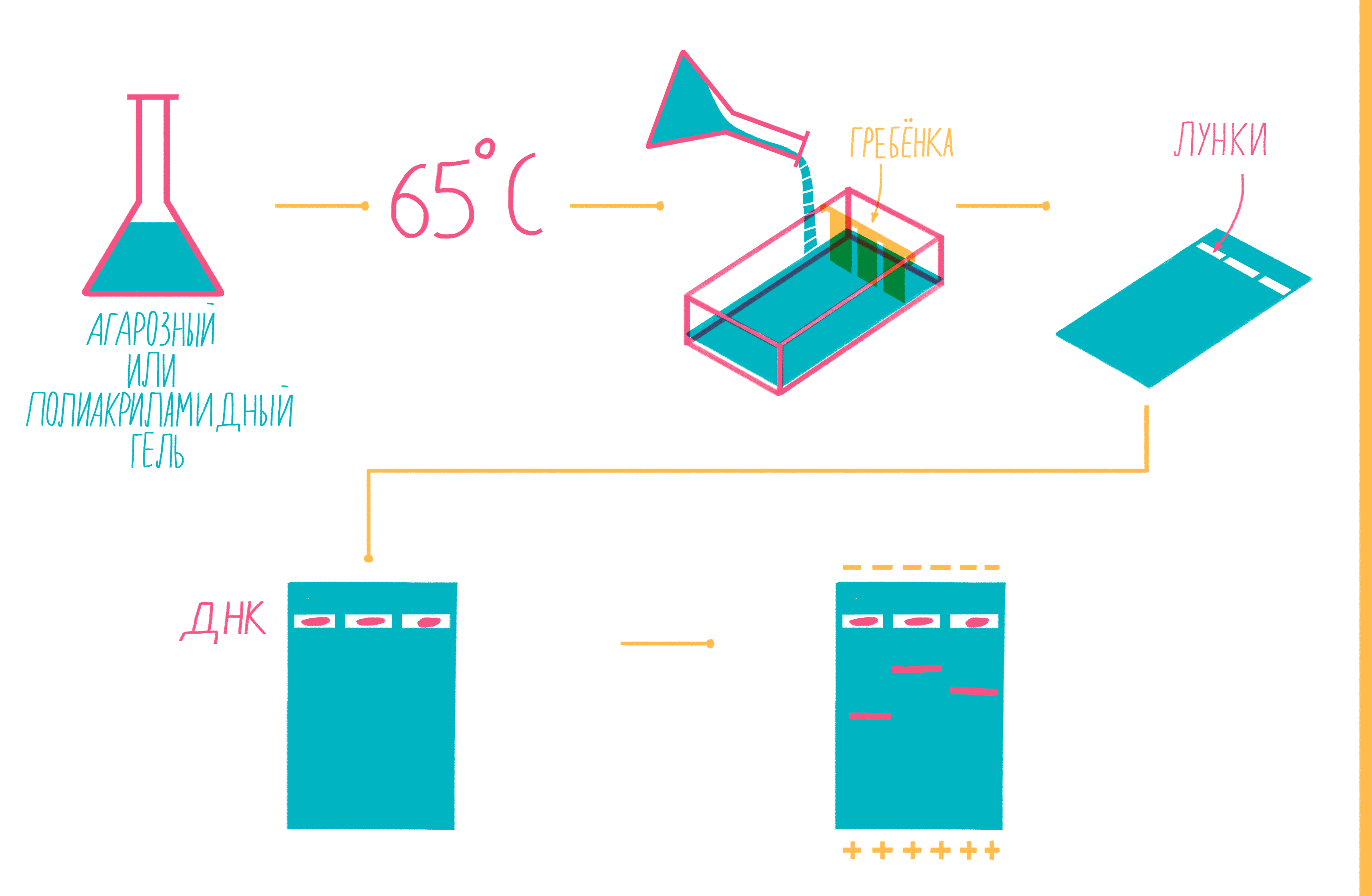
Рисунок 9. Подготовка геля для горизонтального электрофореза.
рисунок Ольги Пташник
Видео 5. Электрофорез в агарозном геле.
Благодаря отрицательно заряженному сахарофосфатному остову ДНК, фрагменты движутся в геле под действием электрического поля от отрицательного катода к положительному аноду. Более короткие молекулы делают это быстрее, чем длинные. Бромфеноловый синий нужен для того, чтобы следить за продвижением фронта проб в геле и не допустить их выхода за его пределы.
После окончания электрофореза гель вынимают из плашки и, чтобы увидеть расположение фрагментов, вымачивают в растворе флюоресцентного красителя, прочно связывающегося с ДНК. Иногда его вводят в гель еще до залития плашки. Если красителем служит бромистый этидий, внедряющийся между нуклеотидами ДНК, визуализацию проводят под ультрафиолетом (рис. 10).
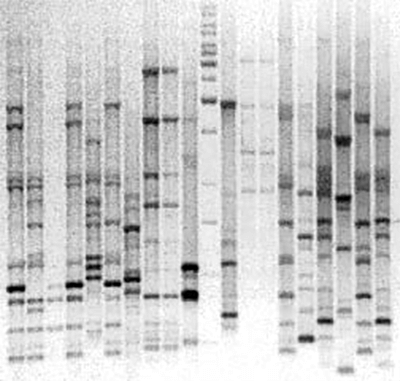
Рисунок 10. Амплифицированные участки ДНК в агарозном геле после электрофореза. Показаны результаты BOX-PCR — варианта rep-PCR (repetitive extragenic palindromic PCR), в котором с праймером BOX-A1R намножаются повторяющиеся внегенные палиндромы (BOX-мотивы) бактерий. Метод BOX-ПЦР — один из видов геномной дактилоскопии. Он обладает высокой разрешающей способностью, позволяя различать бактерии на уровне штаммов: картина распределения амплифицированных фрагментов в геле уникальна для каждого штамма. Вертикальные дорожки — отдельные пробы, соответствующие разным бактериальным штаммам; горизонтальные полоски на каждой дорожке — фрагменты ДНК разной длины.
фото автора статьи
Если экспериментатор преследовал цель просто понять, есть ли нужная последовательность нуклеотидов в ДНК-матрице, то после визуализации гель выбрасывают. Но нужные фрагменты несложно из геля выделить для дальнейшей работы: чтобы резать их на кусочки для сравнения с другими фрагментами, вставлять в плазмиды для дальнейшего изучения, секвенировать и т.д.
Типы ПЦР
Мы описали типичную качественную ПЦР, позволяющую узнать, есть нужная последовательность в анализируемом образце или нет. В качестве матрицы мы всегда упоминали ДНК, но бывают случаи, когда в распоряжении экспериментатора есть лишь РНК (при исследовании РНК-вирусов) или только она может дать необходимую информацию (при определении уровня экспрессии генов). Тогда используют ПЦР с обратной транскрипцией, или ОТ-ПЦР (RT-PCR; не путать с real-time PCR!).
Помимо качественной, существует количественная ПЦР — для определения количества исходной матрицы в образце. Это ПЦР в реальном времени, или real-time PCR. Ее тоже можно проводить в двух вариантах: с ДНК-матрицей и РНК-матрицей.
ПЦР с обратной транскрипцией
Полимеразная цепная реакция может идти исключительно на матрице ДНК, поэтому если у экспериментатора есть мРНК (матричная РНК, на основе которой строятся клеточные белки), то сначала ее надо как-то «переписать» в ДНК. Для этого применяют реакцию обратной транскрипции, в которой фермент обратная транскриптаза по матрице РНК строит комплементарную ДНК (кДНК) [20]. А потом с этой ДНК проводят обычную ПЦР, как описано выше (рис. 11).

Рисунок 11. Схема ОТ-ПЦР. К одноцепочечной РНК-матрице присоединяется праймер, и обратная транскриптаза синтезирует цепь ДНК, которая потом сама уже служит матрицей для синтеза ДНК в процессе обычной ПЦР.
рисунок Ольги Пташник
Впервые ОТ-ПЦР упомянули в 1988 году в журнале Science, в статье о синтезе факторов роста макрофагами [21].
Эту реакцию обычно осуществляют с помощью двух ферментов — обратной транскриптазы и ДНК-полимеразы — либо в двух пробирках (в первой при 37 °С по матрице РНК синтезируют кДНК, затем во второй проводят стандартную ПЦР), либо в одной (все реагенты смешивают вместе, дают отстояться 1 час при 37 °С и помещают в циклер). У каждого из подходов есть свои недостатки и достоинства. В первом случае высока вероятность ошибок при пипетировании и загрязнения образца при переносе во вторую пробирку. Но при этом, однажды проведя обратную транскрипцию, полученную кДНК можно использовать в нескольких экспериментах с разными целями. Во втором случае в ПЦР участвует вся синтезированная кДНК, и повторить реакцию уже невозможно. Однако время проведения эксперимента существенно сокращается. Первый вариант применяют для исследования некоторого набора генов, а второй — при большом количестве образцов, но малом количестве изучаемых генов.
Но ОТ-ПЦР можно проводить и с одним ферментом — Tth-полимеразой. Эту термостабильную полимеразу выделили в 1985 году из бактерии Thermus thermophilus HB-8 [22]. Фермент обладает двойной активностью: в присутствии ионов магния — полимеразной, а в присутствии ионов марганца — обратнотранскриптазной. Причем обе реакции могут идти при 70 °С, что очень важно в случае ГЦ-богатых РНК, которые охотно образуют «шпильки»: высокая температура поддерживает матрицу в денатурированном состоянии, повышает специфичность отжига праймеров и позволяет эффективно копировать сложные молекулы [17].
Однако здесь есть и проблема: в присутствии марганца точность полимеразы сильно снижается, и кДНК содержит множество ошибок. Чтобы этого избежать, после реакции обратной транскрипции в пробирку вносят EDTA, которая образует с марганцем устойчивый комплекс, тем самым выводя его из реакции. Затем добавляют магний и проводят ПЦР [17].
ОТ-ПЦР незаменима при работе с вирусами, геном которых представлен молекулой РНК, в диагностике некоторых видов рака по специфическим транскриптам опухолевых клеток, а также в генной инженерии, если нужно экспрессировать эукариотический ген в бактериальных клетках.
ПЦР (и ОТ-ПЦР) в реальном времени
Этот метод еще называют qPCR (quantitative PCR, или количественная ПЦР), поскольку он позволяет не только обнаружить в пробе целевую нуклеотидную последовательность, но и измерить количество ее копий, а значит, и рассчитать, сколько же было исходной матрицы. Этой матрицей может быть как ДНК (qPCR), так и РНК (RT-qPCR). Понятно, что в последнем случае первой стадией будет обратная транскрипция.
ПЦР в реальном времени разработали в 1992 году в Калифорнии сотрудники Roche Molecular Systems [23]. Они добавили в смесь для ПЦР флуоресцентный краситель бромистый этидий (EtBr) и запустили реакцию под ультрафиолетовым светом, который заставлял EtBr светиться. Весь процесс, а следовательно, и усиление свечения при накоплении копий ДНК, исследователи записали на видеокамеру. Затем они смогли подсчитать исходное число копий целевого фрагмента в реакционной смеси.
Метод real-time PCR не требует визуализации продуктов реакции с помощью гель-электрофореза — их накопление фиксируют в реальном времени оптические датчики, вмонтированные в амплификатор и настроенные на определенную длину волны, испускаемую флуоресцирующими метками (рис. 12). При этом используют два типа меток: интеркалирующие агенты («коллеги» EtBr) и зонды с флуорофорами [24].

Рисунок 12. Амплификатор для qPCR CFX384 Touch™ от Bio-Rad.
сайт www.bio-rad.com
Самый популярный интеркалирующий агент — SYBR Green, флуорофор, резко увеличивающий флуоресценцию (в 1000 раз) после связывания с двухцепочечной ДНК (рис. 13). Таким образом, увеличение флуоресценции будет пропорционально увеличению количества ДНК в каждом цикле ПЦР. К сожалению, интеркалирующие агенты обладают низкой специфичностью: они могут связываться и с «побочными» продуктами реакции, и с димерами праймеров. Однако тщательный подбор праймеров и условий ПЦР минимизируют этот недостаток [24].
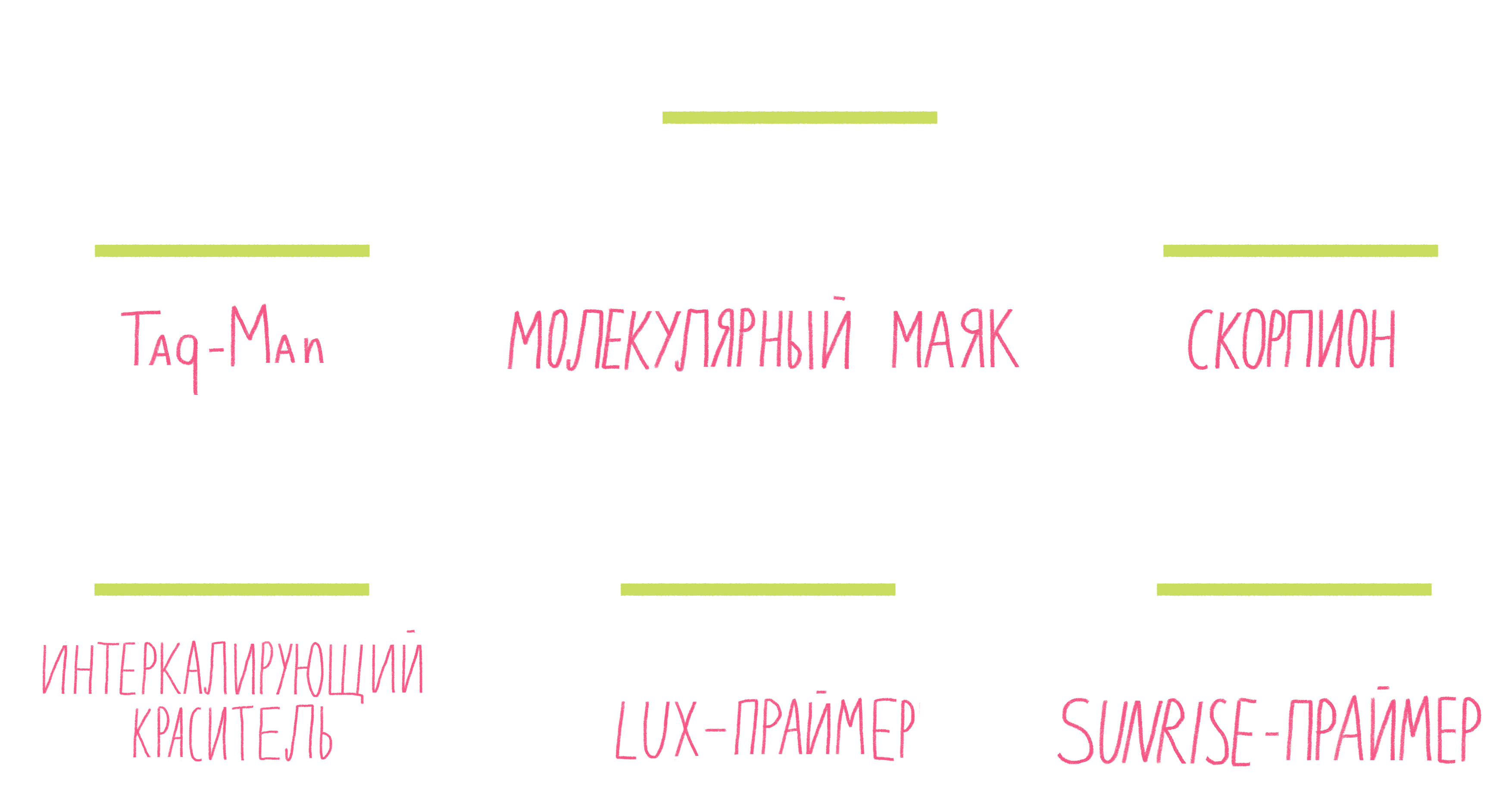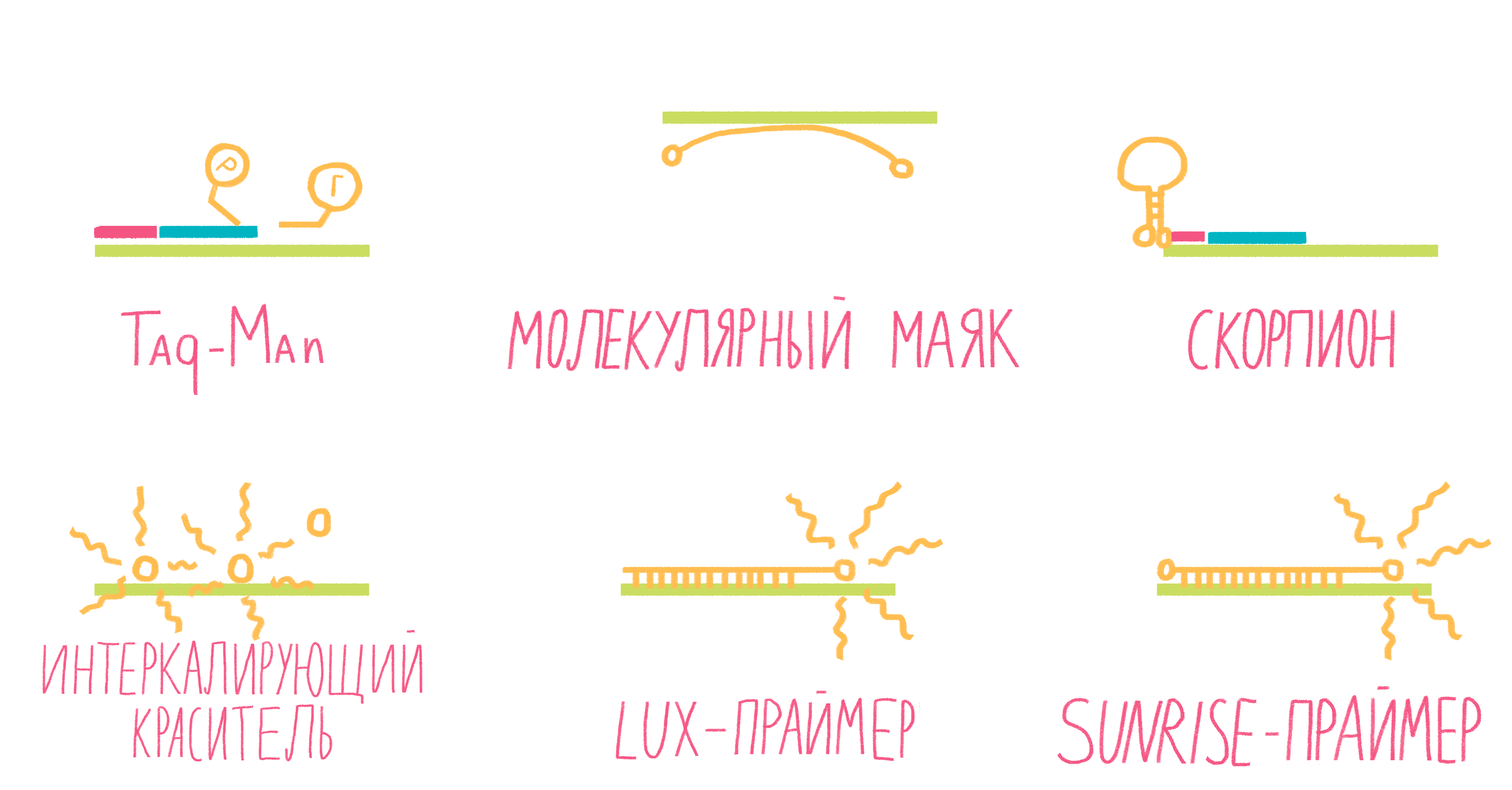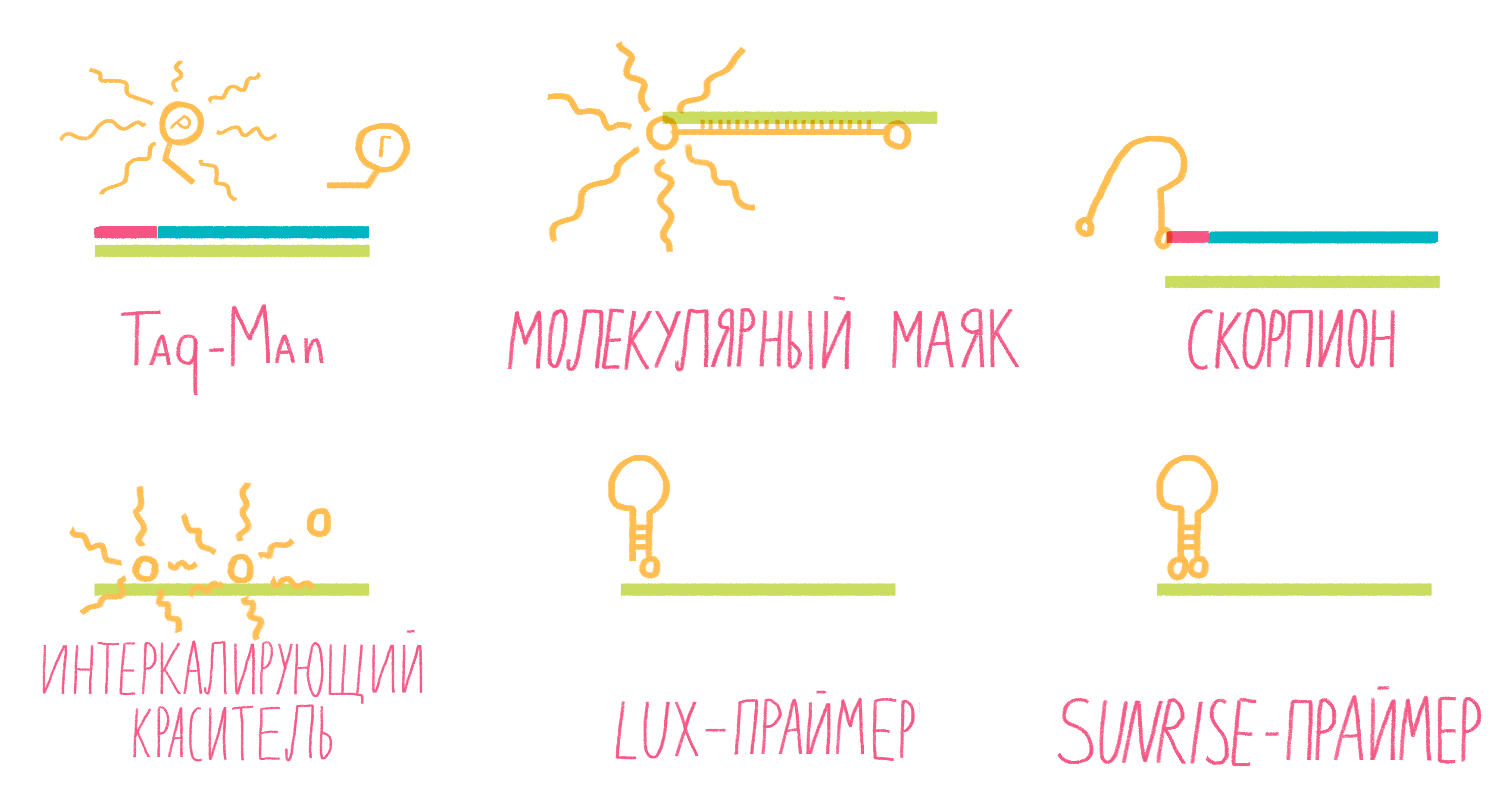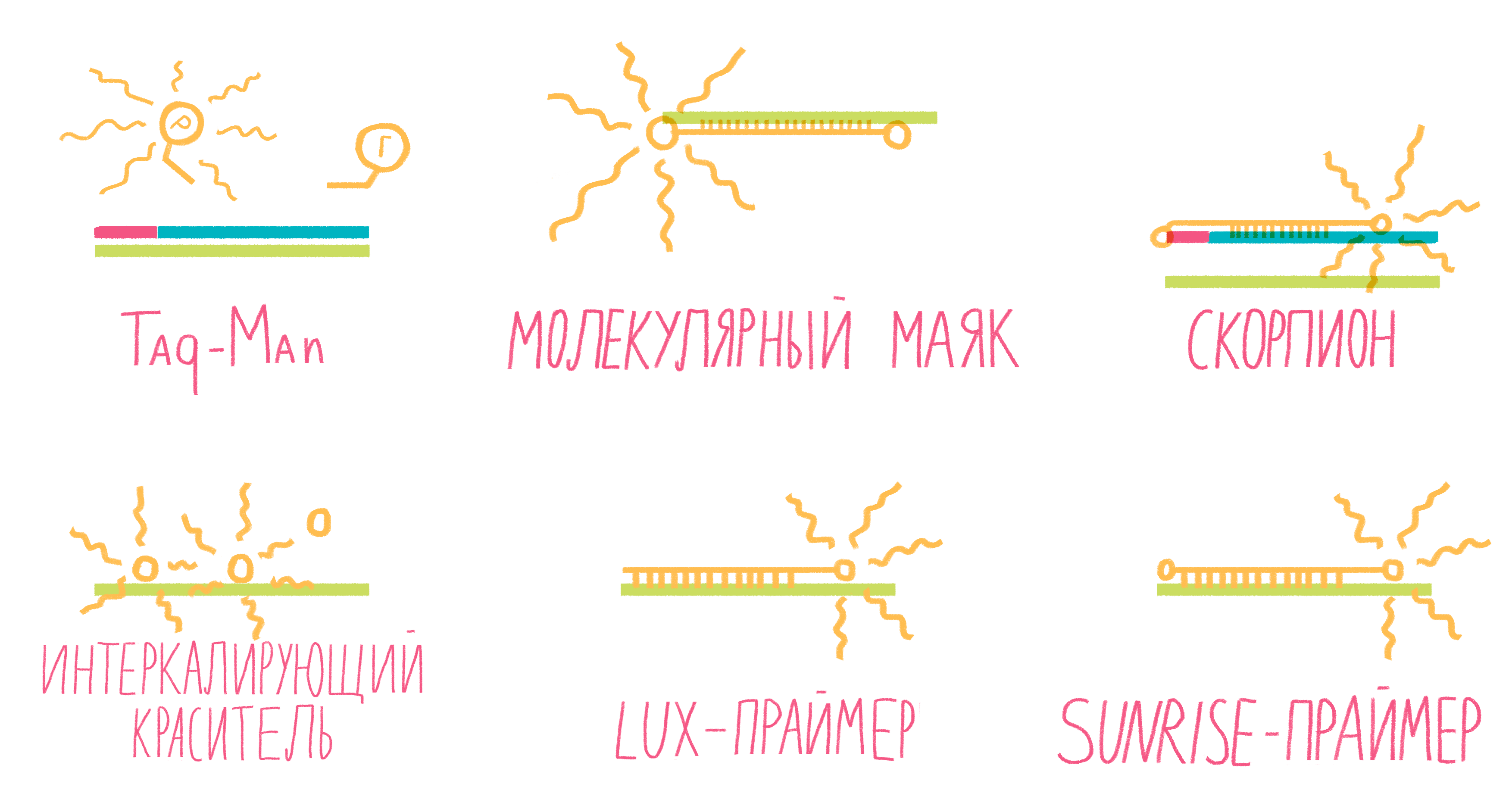
Рисунок 13. Некоторые виды меток для ПЦР в реальном времени.
рисунок Ольги Пташник
Систем зондов с флуорофорами достаточно много (рис. 13). Подробно разберем лишь три самых распространенных.
- TaqMan. Этот небольшой олигонуклеотид, комплементарный внутреннему участку амплифицируемого фрагмента ДНК, содержит два флуорофора: репортер и гаситель. Когда они находятся на одном зонде, то есть близко друг к другу, гаситель поглощает сигнал от репортера. Во время амплификации движущаяся по ДНК полимераза разрушает зонд, репортер и гаситель отдаляются друг от друга, и флуоресценция репортера становится заметной [25].
- Молекулярные маяки. Если в вышеописанном случае детекция свечения происходит на стадии элонгации, то здесь свечение фиксируют на этапе отжига праймеров. Молекулярные маяки — это короткие одноцепочечные фрагменты ДНК, образующие петлю со шпилькой, на концах которой «пришиты» репортер и гаситель (рис. 13). Пока шпилька существует, гаситель находится рядом с репортером, подавляя его свечение. Как только зонд соединяется петлей с комплементарным участком ДНК, репортер и гаситель оказываются достаточно далеко друг от друга, чтобы началась флуоресценция [25].
- Скорпионы. Это структуры, подобные молекулярным маякам, только на 3ˊ-конце после гасителя к ним пришит праймер, с которого и начинается амплификация ДНК. Сигнал от репортера фиксируют в следующем цикле реакции: двухцепочечная ДНК денатурирует, на этапе отжига праймеров раскрывается шпилька зонда, и он, изгибаясь как хвост скорпиона, комплементарно соединяется с цепочкой ДНК, синтезированной как продолжение его праймера (рис. 13). Таким образом репортер с гасителем разносятся в пространстве, и появляется свечение [24].
Применяют ПЦР в реальном времени для анализа экспрессии генов, одиночных нуклеотидных полиморфизмов (SNP) и хромосомных аберраций, для обнаружения конкретных патогенов и, в последние годы, белков (иммуно-ПЦР в реальном времени) [26].
Иммуно-ПЦР в реальном времени

Рисунок 14. Схема иммуно-ПЦР в реальном времени.
рисунок Ольги Пташник
ПЦР служит для качественного и количественного определения в пробе нуклеиновых кислот. Но во многих случаях необходимо детектировать ферменты, гормоны, токсины, антитела и другие молекулы. В большинстве случаев для этого используют метод ИФА (иммуноферментный анализ). Чувствительность метода позволяет определять даже несколько нанограммов антигена в пробе, однако отдельные молекулы ИФА выявить не в состоянии [27].
Чтобы устранить такой недостаток, ученые решили попробовать соединить ИФА и ПЦР. Технологию иммуно-ПЦР (иПЦР, IPCR) разработали сотрудники Калифорнийского университета в Беркли в 1992 году [28]. Сначала для визуализации результатов использовали гель-электрофорез, но сегодня в большинстве случаев применяют ПЦР в реальном времени.
Суть метода заключается в следующем. Пробы, где ищут нужную молекулу (гормон, токсин и т.п.), помещают в специальные пробирки, материал которых обладает высокой антигенсвязывающей способностью и термостойкостью. Далее к пробам добавляют специфические антитела с «пришитыми» к ним ДНК-метками длиной 150–300 п.н. (существует множество вариантов таких систем, но здесь разберем самый простой). Когда антитела присоединятся к искомым молекулам (антигенам), иммобилизованным на стенках пробирок, производят многократную промывку, чтобы удалить непрореагировавшие меченые антитела (рис. 14). Затем в эти же пробирки заливают смесь для qPCR (с интеркалирующим агентом или зондом), помещают их в циклер и проводят реакцию, во время которой амплифицируются ДНК-метки на антителах, связанных с антигенами. Так получают сведения не только о наличии антигенов в пробе, но и об их количестве [29], [30].
Иммуно-ПЦР по разрешающей способности превосходит ИФА на 2–5 порядков и выявляет антиген даже когда невозможно сконцентрировать пробу либо на ранних стадиях бактериальной или вирусной инфекции. Не менее полезно и то, что иПЦР позволяет одновременно обнаруживать много разных антигенов, так как к антителам «пришиты» разные ДНК, амплифицируемые с уникальными праймерами, для которых можно использовать зонды разных конструкций [27], [30].
Применяют иммуно-ПЦР для поиска в пробах вирусных антигенов (например, ВИЧ), опухолеассоциированных антигенов (например, фактора роста эндотелия сосудов), прионов, бактериальных белков, токсинов (в том числе и небелковых, например, диэтилфталата) и других веществ [31].
Варианты проведения реакции
Сегодня существуют десятки вариантов проведения ПЦР для разных целей, для повышения специфичности и эффективности. Разберем лишь несколько наиболее популярных и интересных.
ПЦР с горячим стартом
Известно, что Taq-полимераза может проявлять небольшую активность при комнатной температуре и даже когда пробирка с реакционной смесью находится во льду. Поэтому фермент всегда добавляют в смесь непосредственно перед запуском реакции. Но если, например, проб много, то какие-то из них некоторое время будут стоять уже с полимеразой, пока экспериментатор внесет ее во все пробирки. В этом случае есть вероятность получения неспецифически амплифицированных фрагментов.
Чтобы избежать такой неприятности, используют ПЦР с горячим стартом (hot start PCR), где в смесь добавляют полимеразу в комплексе с антителами, блокирующими ее активность. На первой стадии ПЦР (при 95 °С) антитела денатурируют, полимераза освобождается и только тогда начинает работу.
Ступенчатая ПЦР
При оптимальной температуре отжига праймеры иногда могут связываться и с не идеально комплементарными им участками, а вот если эту температуру немного повысить (например, до 72 °С), то специфичность гибридизации праймеров с матрицей можно существенно увеличить. На этом и основана ступенчатая ПЦР (touchdown PCR): первые циклы проводят при повышенной температуре отжига, постепенно снижая ее до оптимальной в следующих циклах. В результате поначалу вероятность неспецифичной амплификации снижается до минимума, а далее уже размноженные копии нужного фрагмента будут успешно конкурировать за праймеры с не полностью комплементарными им участками ДНК-матрицы.
«Холодная» ПЦР
COLD-PCR (CO-amplification at Lower Denaturation temperature-PCR) используют, когда необходимо выявить, например, однонуклеотидную мутацию гена, но при этом проба содержит ДНК-матрицу как с мутантным геном, так и с геном «дикого типа». Такая смесь типична для биоптатов или образцов крови онкологических больных, и потому этот анализ востребован в медицине для ранней диагностики рака или его рецидивов, а также для назначения индивидуальной терапии на основе молекулярного профилирования. Если делать стандартную ПЦР, то амплифицируются и мутантные, и немутантные аллели интересующего гена, причем последних будет значительно больше, и выявить мутацию будет очень трудно.
Принцип «холодной» ПЦР основан на том, что замена даже одного нуклеотида в одной из цепей ДНК-фрагмента приводит к изменению его температуры плавления, то есть температуры, при которой две цепи ДНК отсоединяются друг от друга. Это изменение составляет обычно 0,2–1,5 °С для фрагментов длиной до 200 п.н. Такая пониженная температура называется критической температурой денатурации (Тс): при ней эффективность ПЦР резко падает из-за малого числа денатурированных матриц [32]. То есть при Тс матрицы «дикого типа» денатурировать уже не будут, но будут те, в которых одна из цепей содержит нуклеотидную замену, отчего их Tm снижается до значения Тс. Для получения таких коротких диагностических матриц интересующий фрагмент вначале выделяют из тотальной ДНК биоматериала с помощью стандартной ПЦР. Мутантные формы среди этих фрагментов ищут уже с помощью COLD-PCR, которая проходит в несколько этапов (рис. 15):
- Стандартная денатурация при 94 °С.
- Гибридизация при 70 °С, во время которой образуются гомодуплексы (обе цепи ДНК «дикого типа» или же обе мутантных, что маловероятно из-за их низкого содержания в исходной пробе) и гетеродуплексы целевых фрагментов (одна цепь «дикого типа», вторая — мутантная). В гетеродуплексе как минимум один нуклеотид (измененный) не может образовать водородных связей со своим «правильным» визави, что выражается в понижении температуры плавления такого дуплекса.
- Критическая денатурация при Тс. Денатурируют лишь гетеродуплексы; гомодуплексы остаются в двухцепочечном состоянии.
- Отжиг праймеров. Может идти только на разошедшихся цепях гетеродуплекса.
- Элонгация.
После окончания всех циклов реакции полученные копии интересующего фрагмента секвенируют, чтобы точно установить место и тип мутации.
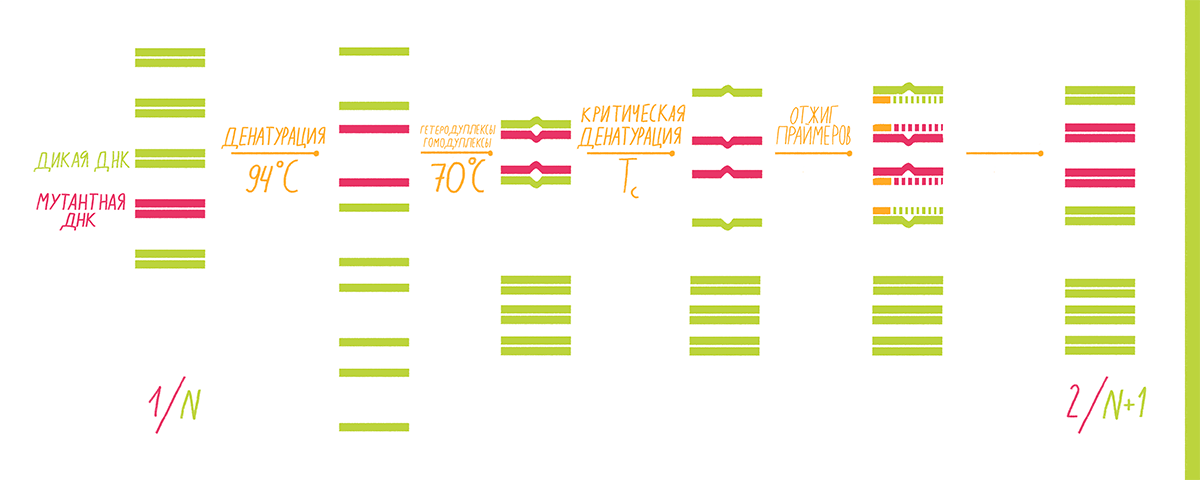
Рисунок 15. Схема «холодной» ПЦР.
Чтобы увидеть рисунок в полном размере, нажмите на него.
рисунок Ольги Пташник
ПЦР длинных фрагментов
Применяют, когда нужно амплифицировать очень длинные фрагменты — более 5 т.п.н. В long-range PCR часто используют две полимеразы: Taq и Pfu. Первая может за один проход синтезировать длинную цепь ДНК, но при этом «застревает», совершив ошибку, потому что не умеет вырезать только что вставленные нуклеотиды. Вторая полимераза менее процессивна, зато способна исправлять ошибки. Так они друг другу и помогают: Taq ошибается, а Pfu исправляет, давая возможность «подружке» закончить синтез.
Мультиплексная ПЦР
Бывают случаи, когда в одной пробе необходимо выявить сразу несколько последовательностей. Например, при инфекции несколькими патогенами, при диагностике комплекса заболеваний или при выявлении мутаций (рис. 16). И чтобы не проводить много реакций, экономят время и реактивы — применяют мультиплексную ПЦР (multiplex PCR), то есть ПЦР со множеством праймеров. Суть ее в том, что в одну пробирку с ДНК-матрицей добавляют целый набор праймеров для одновременной амплификации нескольких интересующих фрагментов [24], [33].
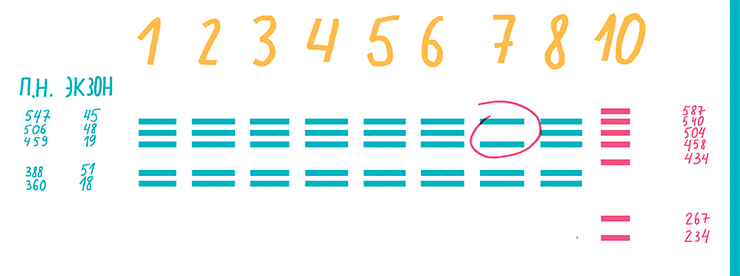
Рисунок 16. Результаты мультиплексной ПЦР ДНК пациента с миодистрофией Дюшенна. К дистрофии приводят различные мутации экзонов гена белка дистрофина. На дорожке 7 нет полосы, соответствующей экзону 48 (длиной 506 п.н.) этого гена.
рисунок Ольги Пташник
Однако при этом надо соблюдать такие условия:
- температуры отжига праймеров не должны сильно разниться (то есть длина каждого праймера должна быть 18–28 нуклеотидов, а ГЦ-состав — 45–60%);
- каждый набор праймеров должен давать фрагмент, отличный от других по размеру, чтобы после электрофореза в геле полосы не совпадали.
RAPD
Это ПЦР случайных полиморфных фрагментов ДНК — Random Amplification of Polymorphic DNA. К RAPD прибегают, когда нужно различить сходные геномы: виды бактерий, сорта растений, породы собак и т.д. Используют небольшой праймер (до 10 н.), который может гибридизоваться со многими случайными участками генома. Если правильно подобрать последовательность праймера и условия реакции, то в геле после электрофореза пробы будут отличаться друг от друга количеством и (или) расположением полос.
При всей схожести метода с rep-PCR, описанной под рисунком 10, последнюю всё-таки считают независимой техникой, отличающейся от RAPD своими характеристиками [34]. Так, RAPD менее специфична и хуже воспроизводится: любая мутация в комплементарном праймеру участке матрицы приводит к тому, что короткий праймер не гибридизуется с ним, и соответствующей полосы в геле не будет. Удлинение праймеров в rep-PCR до 20–22 н. сильно повышает воспроизводимость результатов.
Асимметричная ПЦР
Асимметричную реакцию (asymmetric PCR) проводят при желании получить амплифицированную копию участка только одной из цепей ДНК — например, для последующей гибридизации. В таком случае в реакционной смеси концентрация одного праймера должна быть намного выше, чем другого, и тогда на выходе будут превалировать фрагменты нужной цепи.
Метилспецифичная ПЦР
Геномная ДНК живых организмов, как правило, метилирована: после синтеза ДНК к небольшому проценту цитозинов и аденинов фермент ДНК-метилтрансфераза присоединяет метильную группу. Цели метилирования разнообразны: от регуляции экспрессии отдельных генов до регуляции целых процессов, таких как старение или канцерогенез.
Это свойство геномной ДНК эксплуатирует метилспецифичная ПЦР (methylation-specific PCR). Такой вариант ПЦР применяют, чтобы понять, метилирован ли определенный участок ДНК по цитозину.
Перед постановкой реакции ДНК-матрицу обрабатывают бисульфитом. Он преобразует неметилированные цитозины в урацилы, которые распознаются праймерами и полимеразой как тимины, а метилированные цитозины не трогает. Затем проводят две реакции с разными праймерами: в одну пробирку вносят праймеры, специфичные к последовательности с цитозинами, а в другую — к последовательности с урацилами. Если амплификация прошла в первой пробирке, значит ДНК на этом участке метилирована, если во второй — не метилирована.
ПЦР со вложенной парой праймеров
Она же — вложенная или гнездовая ПЦР (nested PCR). Полезна для уменьшения вероятности амплификации неспецифических фрагментов. Если, например, какие-то из праймеров «сядут» на незапланированные участки, после электрофореза в геле можно получить несколько полос — целевого фрагмента и побочных. Чтобы повысить специфичность реакции, используют два набора праймеров: первый — для амплификации более длинного фрагмента, второй — для амплификации внутреннего участка этого фрагмента (рис. 17). Несколько раундов ПЦР проводят с первым набором, а затем добавляют второй. Чтобы избежать продолжения амплификации с первыми праймерами, оба набора разрабатывают для отжига при разных температурах [24].
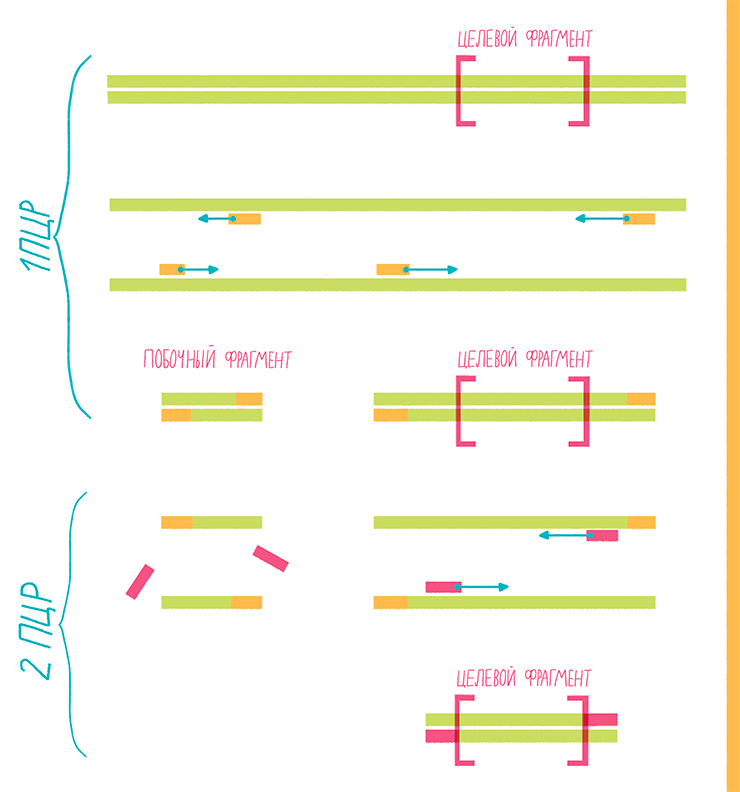
Рисунок 17. Схема вложенной ПЦР.
рисунок Ольги Пташник
Инвертированная ПЦР
Этот вариант используют, когда известна последовательность (сиквенс) какого-то участка ДНК, но нужно амплифицировать вовсе не его, а то неизвестное, что его окружает. Например, необходимо узнать, в какое место генома встроился вирус с известным сиквенсом. Тогда-то и приходит на помощь инвертированная ПЦР (inverse PCR), которая состоит из нескольких этапов (рис. 18):
- ДНК, где есть участок с известной последовательностью, разрезают крупнощепящей рестриктазой. Такие эндонуклеазы распознают сайты длиной 6–8 нуклеотидов, а поскольку таких мест в геноме не может быть слишком много, ДНК режется на крупные куски — фрагменты длиной несколько т.п.н. Важно учесть, чтобы сайта узнавания выбранной рестриктазы не было внутри известного нам участка: он должен войти целиком в один из крупных рестрикционных фрагментов.
- Ферментом лигазой полученные фрагменты закольцовывают.
- Используя праймеры к известной последовательности, но ориентированные кнаружи от нее, амплифицируют ту самую, неизвестную, которую потом можно, например, секвенировать.
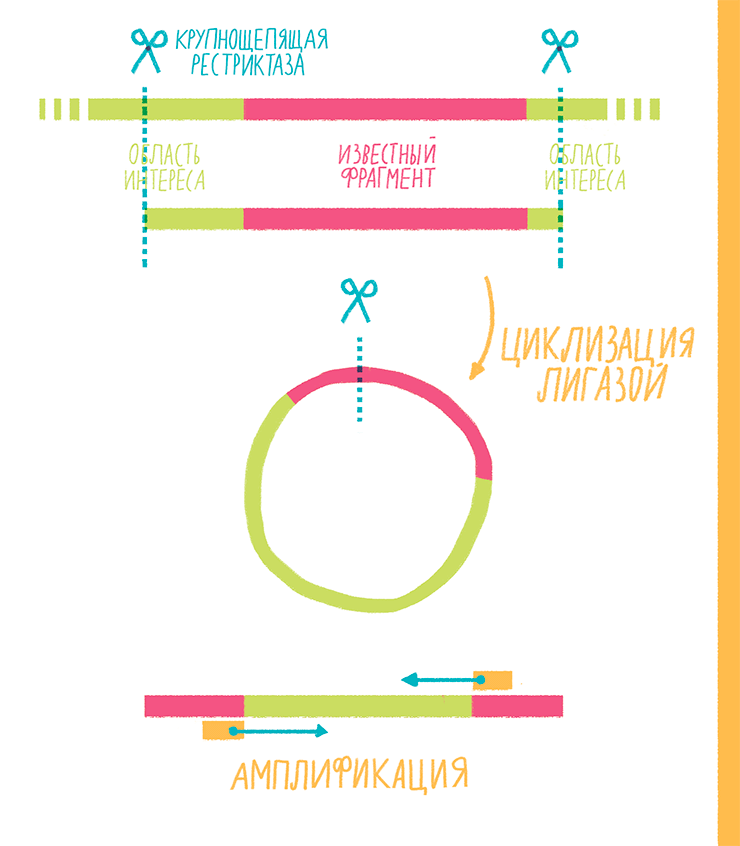
Рисунок 18. Схема инвертированной ПЦР.
рисунок Ольги Пташник
ПЦР с перекрывающимися праймерами
Чаще всего для соединения двух фрагментов двухцепочечной ДНК используют метод рестрикции/лигирования, когда края этих фрагментов разрезают одинаковыми эндонуклеазами с образованием «липких» концов, а потом соединяют их с помощью лигазы. Однако с этой целью можно применять и метод ПЦР.
ПЦР с перекрывающимися праймерами, или продлением перекрывания (overlap extension PCR), выполняется в несколько этапов (рис. 19):
- Для каждого фрагмента ДНК-матрицы конструируют два праймера: один обычный, а второй (гибридизующийся со стороны будущей сшивки фрагментов) на 5ˊ-конце несет небольшую последовательность, соответствующую концу другого фрагмента, того, что будут пришивать.
- Проводят две раздельные ПЦР — для каждого из фрагментов. В результате последовательность, содержащаяся на 5ˊ-концах «стыковочных» праймеров, уже входит в состав новообразованных амплифицированных фрагментов.
- Содержимое обеих пробирок смешивают и используют уже только два праймера — для дальних (внешних) концов. Поскольку оба фрагмента содержат на одном конце последовательности, комплементарные друг другу, во время отжига праймеров фрагменты гибридизуются, а их перекрывающиеся 3ˊ-концы служат праймерами. Таким образом получаются длинные молекулы, состоящие из двух фрагментов.
Этот же вариант ПЦР, но с небольшими модификациями, используют и для внесения мутаций, например, если из длинного фрагмента ДНК надо удалить какой-то участок.
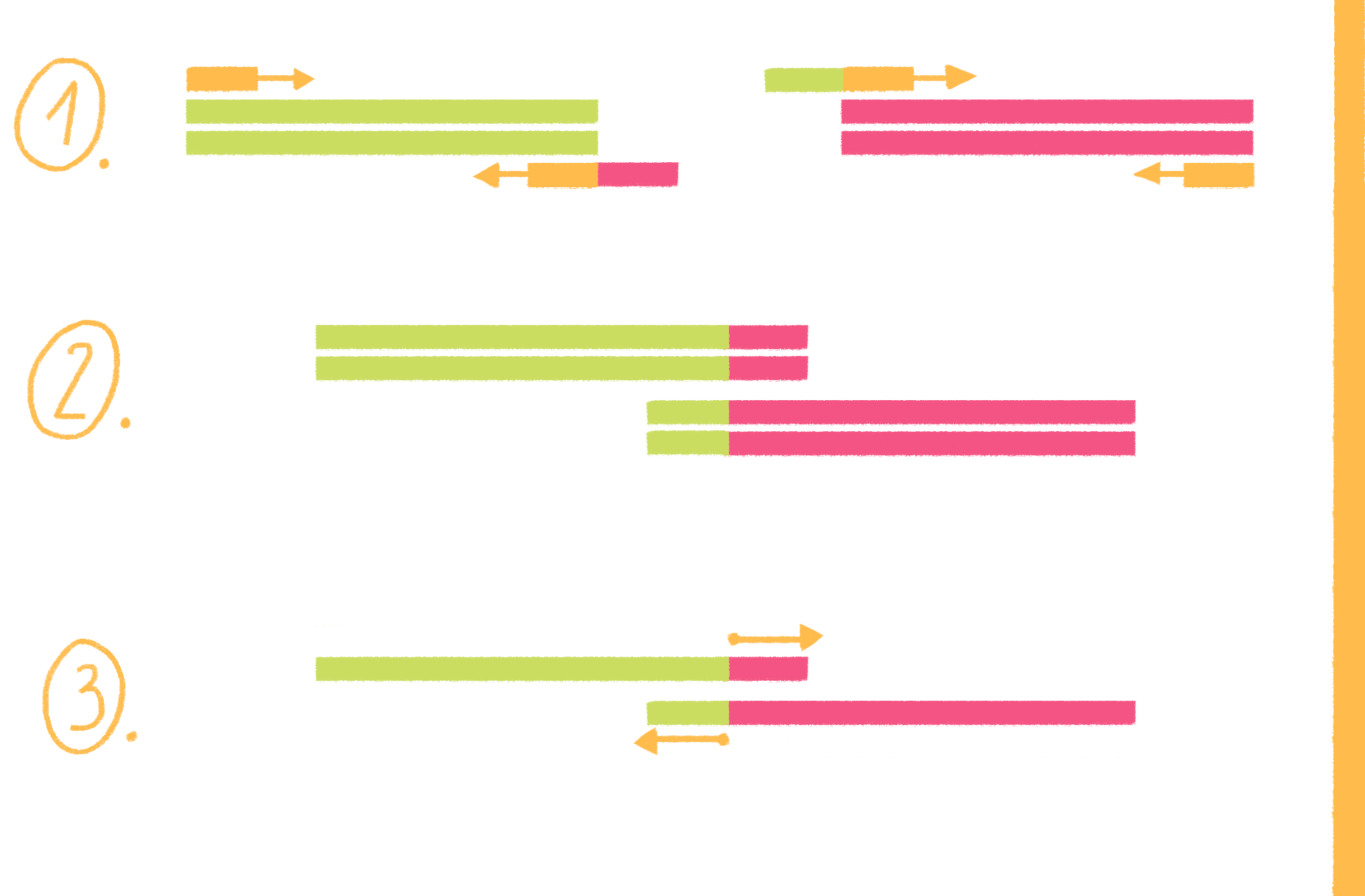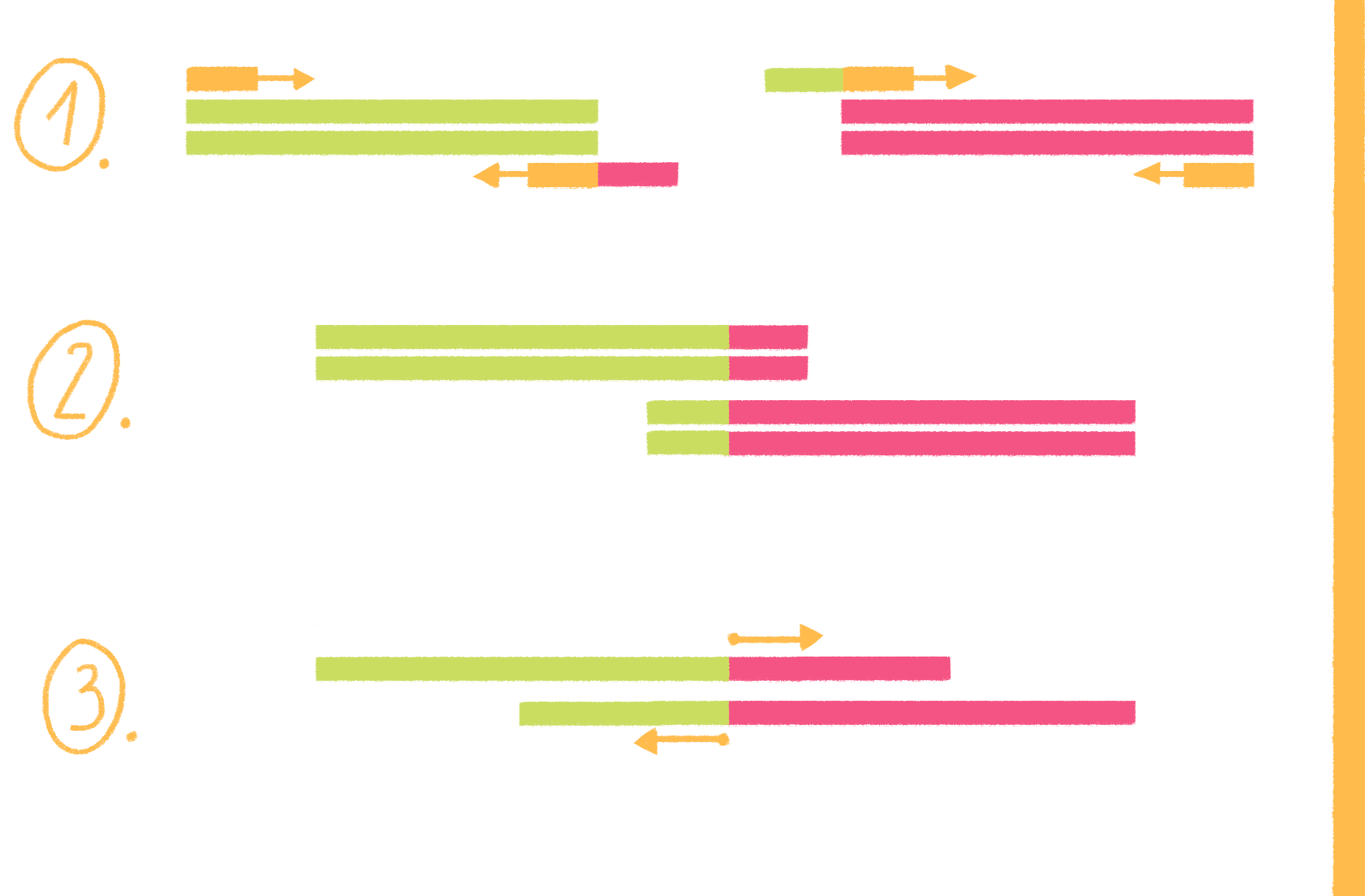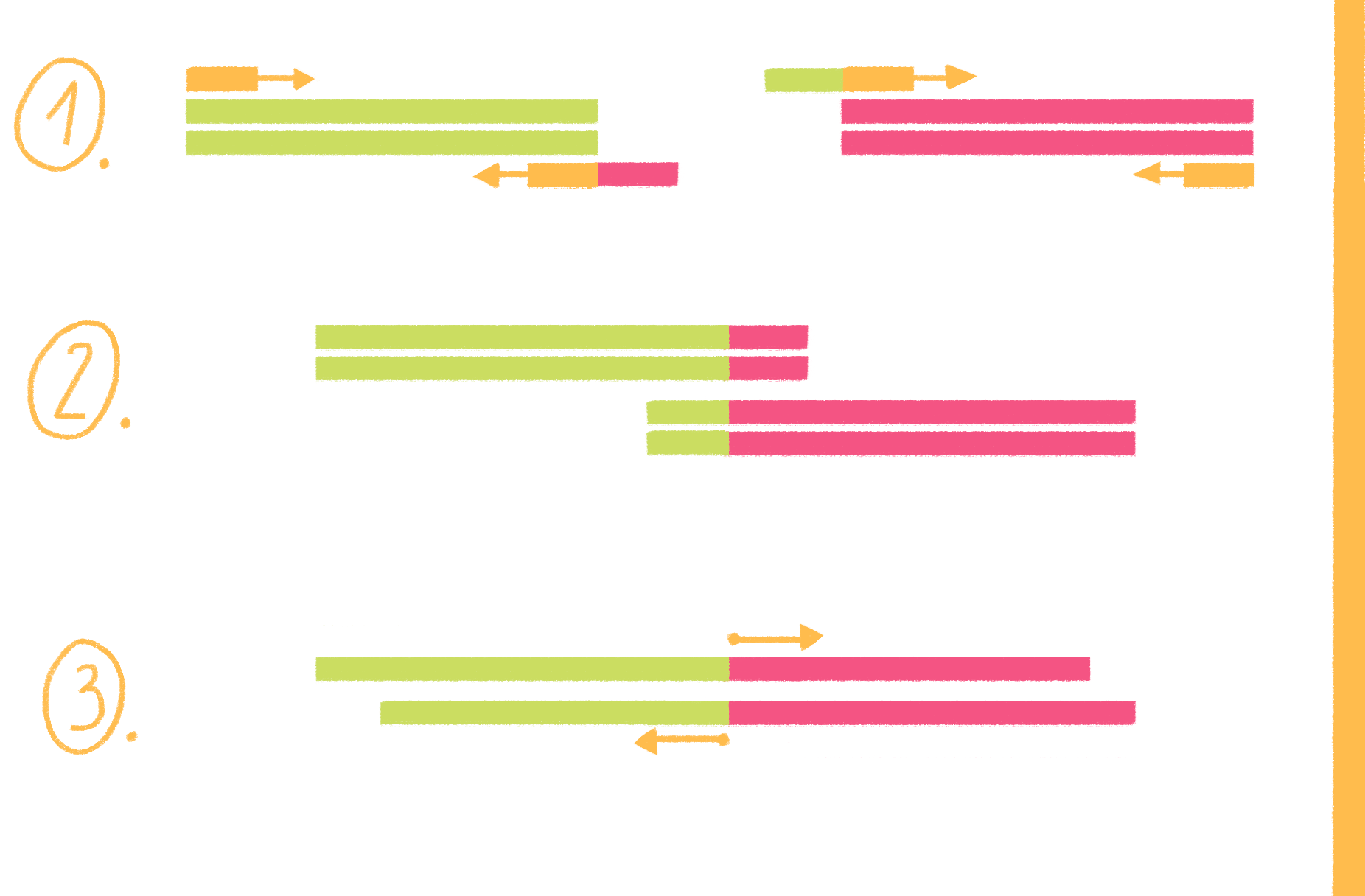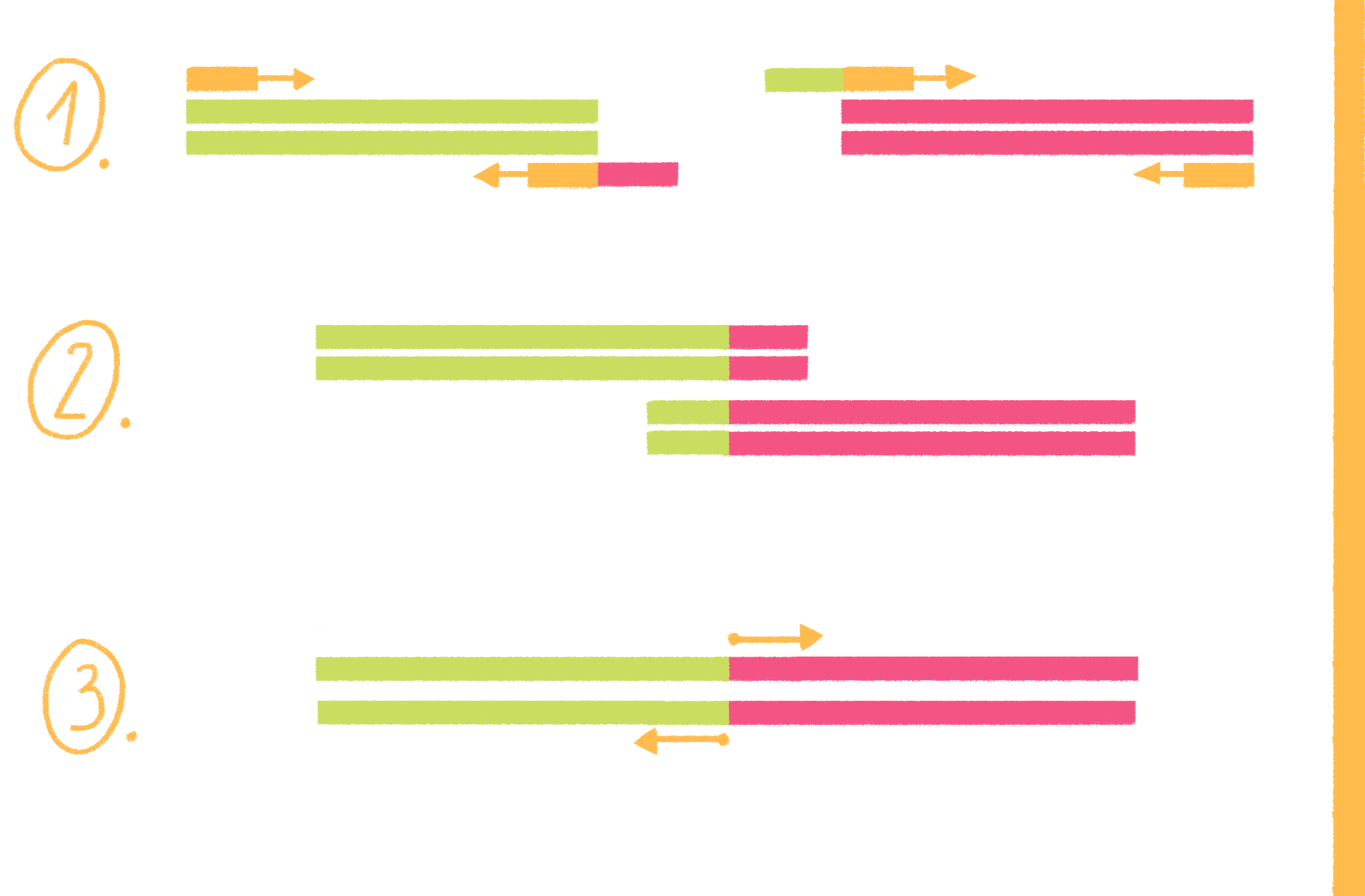
Рисунок 19. Схема ПЦР с перекрывающимися праймерами.
рисунок Ольги Пташник
Сборочная ПЦР
Этот вариант похож на предыдущий. Его используют для сборки синтетических молекул ДНК из отдельных фрагментов, например, чтобы получить синтетические гены или даже целые геномы.
В сборочной ПЦР (assembly PCR) используют одноцепочечные олигонуклеотиды длиной до 50 н., одна часть которых предназначена для образования одной цепи ДНК, а другая — для образования другой (рис. 20). Важно, чтобы эти олигонуклеотиды частично перекрывались концами (примерно на 20 нуклеотидов) с «соседями» на будущей противоположной цепи, поскольку они сами будут работать и праймерами, и матрицей. Во время первых 30 циклов ПЦР концы олигонуклеотидов удлиняются по матрице фрагментов противоположной цепи. К концу процесса каждый олигонуклеотид удлинится настолько, что превратится в отдельную цепочку будущей синтетической ДНК. Тогда в реакцию добавляют пару праймеров, комплементарных концам этой ДНК, и проводят дополнительные 23 цикла, получая на выходе множество копий синтетической ДНК.

Рисунок 20. Схема сборочной ПЦР.
рисунок Ольги Пташник

Рисунок 21. Схема твердофазной ПЦР.
рисунок Ольги Пташник
Твердофазная ПЦР
Ее применяют, например, для получения ДНК-микрочипов или при секвенировании на платформе Illumina. К твердой поверхности 5ˊ-концами пришивают праймеры, добавляют реакционную смесь и проводят твердофазную ПЦР (solid phase PCR, рис. 21). В первом цикле удлиняется некоторое количество праймеров, затем проводят промывку, чтобы удалить свободно плавающие в растворе ДНК-матрицы, добавляют новый раствор (но уже без матриц) и продолжают ПЦР. Одноцепочечные ДНК, торчащие над поверхностью, на этапе отжига изгибаются и гибридизуются с какими-то из соседних праймеров, становясь матрицами для удлинения этих праймеров. На этапе денатурации обе цепи расходятся, но всё равно остаются прикрепленными к поверхности, так как представляют собой продолжения праймеров [35].
In situ ПЦР
Это реакция, которую проводят непосредственно в клетках или тканях, например, для изучения внутриклеточного развития вирусов. Сначала клетки или ткань фиксируют на предметном стекле и обрабатывают протеазой, чтобы расщепить белки и освободить ДНК (или РНК, если собираются проводить ОТ-ПЦР). Затем прямо на стекло добавляют смесь для ПЦР и ставят препарат в амплификатор. Выявляют получившиеся фрагменты либо ДНК-гибридизацией, либо иммунологическими методами.
Капельная цифровая ПЦР
Цифровая ПЦР (digital PCR) — более точный и воспроизводимый метод количественного определения ДНК, чем ПЦР в реальном времени. Стандартная ПЦР проходит во всём объеме образца, а при цифровой пробу делят на большое количество маленьких субъединиц (компартментов) и проводят ПЦР в каждой из них отдельно. Методы разделения на компартменты в различных технологиях цифровой ПЦР отличаются друг от друга (используют масляную эмульсию, капилляры и т.д.), а реакцию проводят в планшетах с микролунками. Результаты визуализируют чаще всего с помощью системы TaqMan, но иногда применяют и интеркалирующие агенты, например, зеленую флуоресцирующую краску EvaGreen.
Метод цПЦР разработали австралийцы Алек Морли и Памела Сайкс в 1992 году, когда исследовали больных лейкемией [36]. В последующие годы разные исследовательские группы разрабатывали свои варианты, в том числе и варианты компартментализации. К сожалению, все они имели существенный недостаток — высокую трудоемкость: пробу надо было разделять на сотни (а то и тысячи) реакций объемом по нескольку микролитров каждая или проводить дополнительные реакции (например, иммобилизацию праймеров на магнитных шариках и гибридизацию продуктов ПЦР с флуоресцентными пробами, как в технологии BEAMing), а затем отдельно анализировать результаты.
От этих проблем экспериментаторов избавила капельная цифровая ПЦР (кцПЦР), или по-английски droplet digital PCR (ddPCR). Общепризнанным лидером в этой области является система цифровой капельной ПЦР — QX200 производства компании Bio-Rad. Эту методику разработала компания QuantaLife, а в 2011 году Bio-Rad приобрела права на технологию [37]. На рынке существует еще несколько систем капельной цифровой ПЦР: QuantStudio 3D и QuantStudio 12K Flex (обе производства Thermo Fisher Scientific), Biomark HD (от Fluidigm), а также RainDrop Plus (от RainDance Technologies), недавно купленный компанией Bio-Rad, но QX200 явно доминирует — за последние несколько лет систему упоминали в более чем 600 публикациях.
В ddPCR из 20 мкл образца, в котором требуется определить количество исследуемой ДНК, создают водно-маслянную эмульсию. Реакционную смесь разделяют на приблизительно 20 000 капель-реакций объемом около 1 нл каждая с помощью автоматического генератора капель (рис. 22). При этом генетический материал распределяется по каплям случайным образом: в них попадают как ДНК-мишени, так и фоновая ДНК. Процесс распределения целевой ДНК по каплям чисто случайный и подчиняется закону распределения малых чисел Пуассона. Перед разделением образца на капли не обязательно разводить его до концентрации, чтобы в каждой капле было либо 0, либо 1 копия ДНК-мишени: при анализе результатов учитываются ситуации, когда в одной капле находится более одной копии мишени.
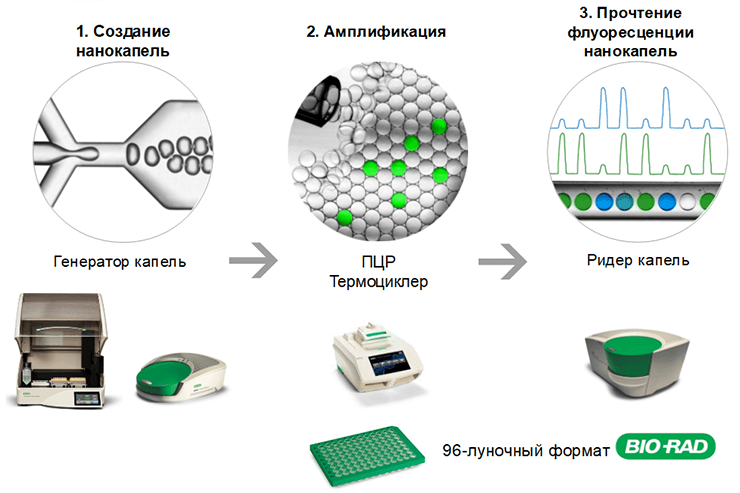
Рисунок 22. Схема цифровой ПЦР с использованием системы QX200™ от Bio-Rad.
сайт www.bio-rad.com
Капли вносят в 96-луночный планшет для ПЦР и помещают в циклер. Реакция проходит независимо в каждой капле (подробное руководство по капельной цифровой ПЦР на английском языке доступно на сайте Bio-Rad: Droplet DigitalTM PCR Applications Guide). В тех каплях, куда попала ДНК-мишень, образуется ПЦР-продукт, что приводит к увеличению уровня флуоресцентного сигнала от флуоресцентной метки: либо TaqMan-зондов, либо интеркалирующего красителя. После ПЦР капли независимо друг от друга проверяют в специальном устройстве (ридере) на наличие или отсутствие в них флуоресцентного сигнала (рис. 23). Количество капель с положительным и отрицательным сигналами подсчитывают для каждого образца, а программное обеспечение выдает концентрацию ДНК-мишени в виде числа копий в микролитре. Анализ продукта проходит в конечной точке после проведения ПЦР [38].

Рисунок 23. Автоматическая шприц-пипетка ридера капель извлекает капли из каждой лунки планшета для ПЦР.
сайт www.bio-rad.com
В кцПЦР определение количества ДНК-мишени проводят не относительно, используя калибровочную кривую, как в случае с ПЦР в реальном времени, а прямым подсчетом капель с наличием или отсутствием в них ДНК-мишени. Это существенно увеличивает стабильность системы и ее устойчивость к ингибиторам ПЦР.
При наличии только двух флуоресцентных каналов (FAM и HEX/VIC) система позволяет запускать 4–5-плексные реакции за счет использования смеси зондов с одной нуклеотидной последовательностью, но меченных красителями FAM и HEX/VIC в различных пропорциях. Возможно также проводить 2–3-плексные реакции с интеркалирующим красителем EvaGreen, используя различную «емкость» разноразмерных ПЦР-продуктов для интеркалятора, что позволяет независимо подсчитать количество этих продуктов по разнице уровня их флуоресценции.
С помощью капельной цифровой ПЦР можно определять:
- количество интересующих молекул ДНК в образце;
- до 0,01% минорного, например мутантного, генома в присутствии генома нормальной ткани;
- вариации числа копий гена (copy namber variation, CNV);
- уровень экспрессии генов и микроРНК, включая детекцию небольших (до 10%) изменений уровня экспрессии;
- различия в геномном контенте и экспрессии между единичными клетками.
Метод активно используют в различных областях, но наиболее часто при:
- проведении онкоисследований, включая анализ свободной циркулирующей ДНК и образцов жидкой биопсии (EGFR, BRAF, KRAS и т.д.);
- детекции нужных вариантов в экспериментах по геномному редактированию (CRISPR/Cas9, ZFN, TALEN);
- изучении геномики единичных клеток (single cell).
Несмотря на сходство применения цифровой ПЦР и ПЦР в реальном времени, скорее всего, в будущем ddPCR будет постепенно вытеснять real-time PCR как основной ПЦР-метод количественного определения ДНК.
Альтернативы ПЦР
В этой главе опишем наиболее интересные и перспективные методы изотермической амплификации, которые могут составить конкуренцию стандартной ПЦР, если нужно узнать, есть ли в пробе та или иная последовательность. Эти методы особенно полезны для медицины и сельского хозяйства в полевых условиях, когда нужно обнаружить патогены в образцах растительного или животного происхождения. Если же перед экспериментатором стоят генно-инженерные цели, например, клонирование генов, то здесь, конечно, ПЦР вне конкуренции.
Опосредованная образованием петель изотермическая амплификация
Метод описали японские ученые в 2000 году [39]. LAMP (loop-mediated isothermal amplification) использует Bst-полимеразу из Geobacillus stearothermophilus, совмещающую полимеразную и хеликазную активности, что дает возможность исключить фазу денатурации и проводить реакцию при 60–65 °С. Подробно о методе написано в статье «Поиск иголки в стоге сена за 10 минут — подсвети себе LAMPой» [40].
LAMP, в отличие от обычной ПЦР, использует не два, а четыре или шесть праймеров, что увеличивает специфичность реакции, но в то же время повышает вероятность артефактов и эффекта множественности полос в электрофорезном геле. Однако это, по сути, не недостаток, так как метод рассчитан на детектирование результата невооруженным глазом («позитив»/«негатив») без электрофореза.
Хеликазозависимая амплификация
В 2004 году сотрудники компании New England Biolabs (NEB) предложили свою альтернативу полимеразной цепной реакции — хеликазозависимую амплификацию (helicase-dependent amplification, HDA). В реакции авторы предложили использовать хеликазу UvrD Escherichia coli, SSB-белки gp32 фага Т4 и фрагмент Клёнова exo− (рис. 24). Хеликаза раскручивает ДНК, SSB-белки стабилизируют цепи в разделенном состоянии, а фрагмент Клёнова синтезирует новую ДНК от 3ˊ-конца праймеров. Реакция проходит при 37 °С. Однако если перед ее постановкой провести денатурацию ДНК при 95 °С, эффективность повышается в 1,5–2 раза [41], [42]. Коммерческие наборы, выпускаемые сейчас NEB, вместо фрагмента Клёнова содержат Bst-полимеразу, а вместо UvrD E. coli — термостабильную хеликазу UvrD Thermoanaerobacter tengcongensis (Tte-UvrD), что позволяет проводить реакцию при 65 °С [43].
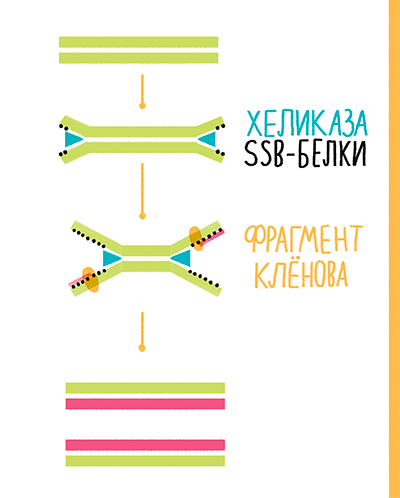
Рисунок 24. Схема хеликазозависимой амплификации.
рисунок Ольги Пташник
К сожалению, у этого метода есть и недостатки. Для получения результатов, сравнимых с результатами стандартной ПЦР, всё-таки необходимо провести предварительную денатурацию образца. К тому же максимальный размер амплифицируемого фрагмента не превышает 120 п.н. Однако по некоторым данным, HDA улавливает в пробе меньшую концентрацию целевой ДНК, чем LAMP [44]. А если использовать хелимеразу (особым образом сшитые Tte-UvrD и Bst-полимеразу), созданную компанией BioHelix, то размер амплифицируемого фрагмента увеличивается до 1,5 т.п.н. [45].
Рекомбиназная полимеразная амплификация
В 2006 году британские ученые из биотехнологической компании TwistDX LTD разработали метод изотермической рекомбиназной полимеразной амплификации (РПА), который может составить сильную конкуренцию как обычной ПЦР, так и ПЦР в реальном времени [46].
Главное отличие РПА от обычной ПЦР в том, что реакция может проходить при комнатной температуре (25 °С), а оптимально — при 37–40 °С. В РПА используют большой фрагмент Bsu-полимеразы, SSB-белки gp32 фага Т4 и его же рекомбиназу uvsX. Часть молекул рекомбиназы связывают с одним праймером, часть — с другим. После внесения в реакционную смесь такие молекулы сканируют ДНК в поисках комплементарных праймеру участков и, найдя их, инициируют процесс расплетения двухцепочечной молекулы и присоединения праймера (рис. 25). Расплетенную ДНК стабилизируют SSB-белки, а полимераза начинает синтез. Процесс АТФ-зависимый, поскольку энергия нужна для освобождения 3ˊ-конца праймера от молекул рекомбиназы. Размер амплифицируемого фрагмента ограничивается тысячей пар нуклеотидов, а минимальная длина праймера должна быть 30 н. для повышения его специфичности [46].

Рисунок 25. Схема рекомбиназной полимеразной амплификации.
рисунок Ольги Пташник
Процесс может длиться 15–30 минут, включая экстракцию образцов, что намного быстрее стандартной ПЦР [47]. Этот метод амплификации лишен значительных недостатков (кроме требования строгого соблюдения условий реакции) и может легко модифицироваться в РПА с обратной транскрипцией и РПА в реальном времени — как с интеркалирующими агентами, так и с флуоресцентными зондами.
Весной 2017 года американские ученые предложили на основе рекомбиназной полимеразной амплификации в сочетании с технологией CRISPR-Cas новый метод диагностики — SHERLOCK. О нем «Биомолекула» уже рассказывала в статье «SHERLOCK — молекулярный сыщик спешит на помощь!» [48].
Применение ПЦР
1. Клиническая медицина
- Анализ клинических образцов на наличие инфекционных агентов бактериальной и вирусной природы: ВИЧ, вирусов гепатита и герпеса, хламидий, хеликобактера, туберкулезных микобактерий и т.д.
- Диагностика лейкемии, лимфомы и других видов неоплазий, которые можно определить по мутациям в определенных генах. Мониторинг опухолевых заболеваний после терапии.
- Диагностика наследственных заболеваний, причина которых — мутации отдельных генов: серповидноклеточной анемии, бокового амиотрофического склероза, фенилкетонурии, муковисцидоза, мышечной дистрофии и т.п.
- Персонализированная медицина. Далеко не все лекарства одинаково действуют на всех людей. Одно и то же вещество может помогать одному пациенту и быть токсичным или аллергенным для другого из-за особенностей метаболических процессов у разных людей. Поэтому наиболее эффективны индивидуально подобранные дозировки индивидуально подобранных препаратов. Различия метаболизма обычно обусловлены генетически. Сделав пациенту своеобразный «генетический паспорт» таких особенностей, можно на его основе подбирать правильное лечение [49].
- Тканевая типизация перед трансплантацией органов.
- Обнаружение хромосомных кроссинговеров, делеций, инсерций, транслокаций и инверсий в отдельных сперматозоидах до оплодотворения.
2. Криминалистика и судебная медицина
- Установление личности преступников и жертв по ДНК из капель крови, волос и спермы с места преступления.
- Установление отцовства и иного родства.
- Расследование причин необъяснимой смерти («молекулярная аутопсия») — в комплексе с морфологическими и физиологическими данными [24].
3. Генная инженерия
- Клонирование ДНК для исследования функций генов, их взаимодействия, создания синтетических ДНК, генетически модифицированных организмов и пр. [50].
- Секвенирование ДНК [8], основанное на ПЦР, в ходе которой в синтезируемую цепь включается меченный флуоресцентной меткой или радиоактивным изотопом дидезоксинуклеотид, что приводит к терминации синтеза и позволяет определить положение конкретных нуклеотидов после разделения в геле (то есть пошагово прочитать их последовательность).
- Мутагенез, основанный на внесении изменений в ДНК посредством ПЦР.
- Создание гибридизационных зондов для различных видов блоттинга.
- Анализ экспрессии генов в тканях и отдельных клетках в разных условиях.
4. Антропология, палеонтология
- Изучение взаимосвязей между видами в эволюционной биологии.
- Исследование миграции людей и связи различных этносов, национальностей и рас.
- Изучение вымерших животных и предков человека.
- Исследование ДНК исторических личностей, например, Николая II [51], английского короля Ричарда III [52], египетских фараонов [53] и т.д.
5. Сельское хозяйство
- Обнаружение патогенов (бактерий, грибов, вирусов) у растений и животных.
- Диагностика наследственных заболеваний домашних и сельскохозяйственных животных.
- Анализ пищевых продуктов на содержание генетических модификаций.
- Обнаружение Х-хромосомы у животных, пол которых трудно определить невооруженным глазом: например, рыб, рептилий, попугаев.
Полимеразная цепная реакция — один из самых мощных и дешевых лабораторных методов. Его появление привело к воистину революционным изменениям в науке и медицине. И если сейчас для генетических экспресс-анализов появляются альтернативные техники, не требующие сложной аппаратуры, то в генетической инженерии ПЦР по-прежнему просто незаменима. Пожалуй, самые ценные свойства этого метода — совместимость с другими техниками и невероятная пластичность: они позволяют биологам и врачам с минимальными усилиями решать совершенно разные задачи. А главное, метод пока не исчерпал свой потенциал: до сих пор появляются всё новые его варианты, и, возможно, нас еще не раз удивят остроумные и неожиданные протоколы экспериментов, основанных на ПЦР.
Календарь
На основе статей спецпроекта мы решили сделать календарь «12 методов биологии» на 2019 год. Эта статья представляет февраль.
Литература
- Артур Корнберг (1918–2007);
- Peter M. Burgers. (2007). Arthur Kornberg (1918–2007). Molecular Cell. 28, 530-532;
- Lehman I.R., Bessman M.J., Simms E.S., Kornberg A. (1958). Enzymatic synthesis of deoxyribonucleic acid. I. Preparation of substrates and partial purification of an enzyme from Escherichia coli. J. Biol. Chem. 233, 163–170;
- K. Kleppe, E. Ohtsuka, R. Kleppe, I. Molineux, H.G. Khorana. (1971). Studies on polynucleotides. Journal of Molecular Biology. 56, 341-361;
- Chien A., Edgar D.B., Trela J.M. (1976). Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus. J. Bacteriol. 127, 1550–1557;
- The Nobel Prize in Chemistry 1958. (2014). Nobelprize.org;
- Sanger F., Nicklen S., Coulson A.R. (1977). DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. USA. 74, 5463–5467;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- The Nobel Prize in Chemistry 1980. (2014). Nobelprize.org;
- Fore J.Jr., Wiechers I.R., Cook-Deegan R. (2006). The effects of business practices, licensing, and intellectual property on development and dissemination of the polymerase chain reaction: case study. J. Biomed. Discov. Collab. 1, 7;
- Mullis K.B. (1990). The unusual origin of the polymerase chain reaction. Scientific American. 4, 56–61;
- R. Saiki, S Scharf, F Faloona, K. Mullis, G. Horn, et. al.. (1985). Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science. 230, 1350-1354;
- Bartlett J.M. and Stirling D. (2003). A short history of the polymerase chain reaction. Methods Mol. Biol. 226, 3–6;
- Википедия: Kary Mullis (англ.);
- R. Saiki, D. Gelfand, S Stoffel, S. Scharf, R Higuchi, et. al.. (1988). Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science. 239, 487-491;
- Википедия: History of polymerase chain reaction (англ.);
- M. J. McPherson and S. G. Moller. PCR (The dasics). 2nd edition. NY: Taylor & Francis Group, 2006. — 292 p.;
- Vondrejs V. a Storchová Z. Genové inženýrsrví I. Praha: Nakladatelství University Karlovy, 1997;
- 12 методов в картинках: очистка молекул и разделение смесей;
- Мода на ретро. Где встречается обратная транскрипция, и как она эволюционировала;
- D. Rappolee, D Mark, M. Banda, Z Werb. (1988). Wound macrophages express TGF-alpha and other growth factors in vivo: analysis by mRNA phenotyping. Science. 241, 708-712;
- Carmen RUTTIMANN, Milena COTORAS, Josefina ZALDIA, Rafael VICUNA. (1985). DNA polymerases from the extremely thermophilic bacterium Thermus thermophilus HB-8. Eur J Biochem. 149, 41-46;
- Russell Higuchi, Gavin Dollinger, P. Sean Walsh, Robert Griffith. (1992). Simultaneous Amplification and Detection of Specific DNA Sequences. Nat Biotechnol. 10, 413-417;
- Hernandez-Rodriguez P. and Ramirez A.G. Polymerase chain reaction: types, utilities and limitations. In: Polymerase chain reaction / ed. by Hernandez-Rodriguez P. Rijeka, Croatia: InTech, 2012. — 566 p.;
- Seifi M., Ghasemi A., Heidarzadeh S., Khosravi M., Namipashaki A., Soofiany V.M. et al. Overview of real-time PCR principles. In: Polymerase chain reaction / ed. by Hernandez-Rodriguez P. Rijeka, Croatia: InTech, 2012. — 566 p.;
- Mikael Kubista, José Manuel Andrade, Martin Bengtsson, Amin Forootan, Jiri Jonák, et. al.. (2006). The real-time polymerase chain reaction. Molecular Aspects of Medicine. 27, 95-125;
- Маерле А.В., Сергеев И.В., Алексеев Л.П. (2014). Метод иммуно-ПЦР: перспективы использования. Иммунология. 1, 44–48;
- T Sano, C. Smith, C. Cantor. (1992). Immuno-PCR: very sensitive antigen detection by means of specific antibody-DNA conjugates. Science. 258, 120-122;
- Рязанцев Д.Ю., Воронина Д.В., Завриев С.К. (2016). Иммуно-ПЦР: достижения и перспективы. Успехи биологической химии. 56, 377–410;
- Michael Adler, Ron Wacker, Christof M. Niemeyer. (2003). A real-time immuno-PCR assay for routine ultrasensitive quantification of proteins. Biochemical and Biophysical Research Communications. 308, 240-250;
- Christof M. Niemeyer, Michael Adler, Ron Wacker. (2005). Immuno-PCR: high sensitivity detection of proteins by nucleic acid amplification. Trends in Biotechnology. 23, 208-216;
- Coren A Milbury, Jin Li, Pingfang Liu, G Mike Makrigiorgos. (2011). COLD-PCR: improving the sensitivity of molecular diagnostics assays. Expert Review of Molecular Diagnostics. 11, 159-169;
- P. Markoulatos, N. Siafakas, M. Moncany. (2002). Multiplex polymerase chain reaction: A practical approach. J. Clin. Lab. Anal.. 16, 47-51;
- Baldy-Chudzik K. (2001). Rep-PCR — a variant to RAPD or an independent technique of bacteria genotyping? A comparison of the typing properties of rep-PCR with other recognised methods of genotyping of microorganisms. Acta Microbiol. Pol. 50, 189–204;
- Jean-Francois Mercier, Gary W. Slater, Pascal Mayer. (2003). Solid Phase DNA Amplification: A Simple Monte Carlo Lattice Model. Biophysical Journal. 85, 2075-2086;
- Sykes P.J., Neoh S.H., Brisco M.J., Hughes E., Condon J., Morley A.A. (1992). Quantitation of targets for PCR by use of limiting dilution. Biotechniques. 13, 444–449;
- Benjamin J. Hindson, Kevin D. Ness, Donald A. Masquelier, Phillip Belgrader, Nicholas J. Heredia, et. al.. (2011). High-Throughput Droplet Digital PCR System for Absolute Quantitation of DNA Copy Number. Anal. Chem.. 83, 8604-8610;
- Киселева Я.Ю., Птицин К.Г., Радько С.П., Згода В.Г., Арчаков А.И. (2016). Цифровая капельная ПЦР — перспективный технологический подход к количественному профилированию микроРНК. Биомедицинская химия. 4, 403–410;
- Notomi T., Okayama H., Masubuchi H., Yonekawa T., Watanabe K., Amino N., Hase T. (2000). Loop-mediated isothermal amplification of DNA. Nucleic Acids Res. 28, e63;
- Поиск иголки в стоге сена за 10 минут — подсвети себе LAMPой;
- Myriam Vincent, Yan Xu, Huimin Kong. (2004). Helicase-dependent isothermal DNA amplification. EMBO Rep. 5, 795-800;
- Lei Yan, Jie Zhou, Yue Zheng, Adam S. Gamson, Benjamin T. Roembke, et. al.. (2014). Isothermal amplified detection of DNA and RNA. Mol. BioSyst.. 10, 970;
- Fei Ma, Meng Liu, Bo Tang, Chun-yang Zhang. (2017). Sensitive Quantification of MicroRNAs by Isothermal Helicase-Dependent Amplification. Anal. Chem.. 89, 6182-6187;
- Celine Zahradnik, Claudia Kolm, Roland Martzy, Robert L. Mach, Rudolf Krska, et. al.. (2014). Detection of the 35S promoter in transgenic maize via various isothermal amplification techniques: a practical approach. Anal Bioanal Chem. 406, 6835-6842;
- Aurélie Motré, Ying Li, Huimin Kong. (2008). Enhancing helicase-dependent amplification by fusing the helicase with the DNA polymerase. Gene. 420, 17-22;
- Olaf Piepenburg, Colin H Williams, Derek L Stemple, Niall A Armes. (2006). DNA Detection Using Recombination Proteins. PLoS Biol. 4, e204;
- Binoy Babu, Brian K. Washburn, Tülin Sarigül Ertek, Steven H. Miller, Charles B. Riddle, et. al.. (2017). A field based detection method for Rose rosette virus using isothermal probe-based Reverse transcription-recombinase polymerase amplification assay. Journal of Virological Methods. 247, 81-90;
- SHERLOCK — молекулярный сыщик спешит на помощь!;
- От медицины для всех — к медицине для каждого!;
- Молекулярное клонирование, или как засунуть в клетку чужеродный генетический материал;
- Фишман Р. (2016). Как идентифицировали останки Николая II и его семьи по ДНК. «Популярная механика»;
- Антонов Е. (2014). «Дело» короля Ричарда III «закрыли» спустя 529 лет. «Наука и жизнь»;
- Власова Е. (2017). Палеогенетики секвенировали ДНК древних египтян. N+1.