Как молекулы РНК общаются с хроматином
24 июля 2020
Как молекулы РНК общаются с хроматином
- 996
- 0
- 13
Исследование контактов РНК с геномной ДНК позволило выявить не только новые функциональные группы прежде неизвестных РНК, но и разобраться в некоторых фундаментальных вопросах молекулярной биологии
-
Автор
-
Редактор
Несмотря на то, что лишь несколько процентов генов эукариот кодируют белки, транскрипция затрагивает почти все участки генома. В результате этого процесса образуется огромное количество всевозможных некодирующих РНК, причем функции большинства из них неизвестны. Тем не менее установлено, что некоторые некодирующие РНК участвуют в поддержании и регуляции пространственной организации генома. Исследователи из Института биологии гена Российской академии наук и с факультета биоинженерии и биоинформатики МГУ им. М.В. Ломоносова разработали новый метод, позволяющий изучать контакты молекул РНК с геномом, и с его помощью идентифицировали сотни видов РНК, ассоциированных с активным или неактивным хроматином. С помощью нового метода ученым даже удалось детально изучить кинетику образования мРНК и подтвердить гипотезу о котранскрипционном сплайсинге интронов.
В геномах эукариотических организмов закодировано огромное количество разнообразных некодирующих РНК. Хотя эти РНК, за рядом любопытных исключений , не служат матрицей для синтеза полипептидов, они задействованы во множестве важнейших биологических процессов, от регуляции активности ферментов до образования ядерных телец, однако функции большинства из них остаются неизученными.
О длинных некодирующих РНК, которые содержат транслируемые открытые рамки считывания, читайте в статье «Кодирующие некодирующие РНК» [1].
Многочисленные эксперименты показали, что некодирующие РНК участвуют в регуляции пространственной организации генома. Самой известной некодирующей РНК, напрямую влияющей на структуру хроматина, является РНК под названием XIST (от анг. X-inactive specific transcript) , которая обеспечивает гетерохроматинизацию одной из двух X-хромосом в клетках самок млекопитающих. Кроме того, особые энхансерные РНК способствуют сближению энхансера и соответствующего ему промотора, что играет важную роль в регуляции экспрессии генов. Любопытно, что архитектурный белок хроматина CTCF, который является важным регулятором пространственной организации генома у млекопитающих, взаимодействует с множеством некодирующих РНК. При этом стоит иметь в виду, что, поскольку в живой клетке геномная ДНК плотно покрыта белками, РНК, как правило, взаимодействуют с ДНК не напрямую, а опосредованно, через белки. Как же разобраться, с какими участками генома какие именно РНК взаимодействуют?
Об инактивации X-хромосомы некодирующей РНК XIST читайте в статье «Загадочное путешествие некодирующей РНК Xist по X-хромосоме» [2]. — Ред.
Red-C: новый метод для идентификации контактов РНК и геномной ДНК
Ученые из Института биологии гена РАН и с факультета биоинженерии и биоинформатики МГУ им. М.В. Ломоносова разработали новый метод, предназначенный для картирования контактов РНК с геномом [3]. Новый метод получил название Red-C от англ. RNA ends on DNA capture (дословно — «захват концов РНК на ДНК»). Сначала клетки, в которых исследуют контакты РНК с геномом, фиксируют формальдегидом, из-за чего формируются комплексы ДНК—белок—РНК, компоненты которых ковалентно сшиты друг с другом. Далее ДНК фрагментируют рестриктазой, а к молекулам РНК пришивают на 3′-концы особую нуклеотидную последовательность — адаптер (с одного конца одноцепочечный для сшивания с РНК, с другого — двухцепочечный), в состав которого входит биотинилированный нуклеотид (рис. 1). Затем проводят лигирование, и, если вблизи адаптера у конца молекулы РНК находится конец молекулы ДНК, то двухцепочечный конец адаптера и ДНК будут сшиты лигазой. Теперь нужно разрушить белок, который когда-то был посредником между контактирующими РНК и геномной ДНК, и выделить получившиеся химерные молекулы, включающие взаимодействующие РНК и ДНК, разделенные адаптером. ДНК далее дополнительно обрезают с помощью весьма хитрой рестриктазы MmeI, сайт узнавания которой специально включают в последовательность адаптера — с того конца, который соединяется с ДНК. В отличие от наиболее часто используемых рестриктаз, MmeI вносит разрыв не в ту же последовательность, которую распознает, а на расстоянии 20 нуклеотидов выше нее. Благодаря обработке MmeI получаются гибридные молекулы, на одном конце которых находится РНК, в середине — адаптер с биотинилированным нуклеотидом, а после него идет небольшой фрагмент геномной ДНК (около 20 нуклеотидов), который с этой РНК и взаимодействует в живой клетке. Поскольку в состав адаптера входит биотинилированный нуклеотид, такие химерные молекулы можно «вытянуть» из раствора за него, используя стрептавидин, обладающий высоким сродством к биотину. После дополнительной процедуры, включающей обратную транскрипцию (достраивание комплекса путем синтеза цепочки ДНК по матрице РНК), химеры амплифицируют и секвенируют, что позволяет определить последовательности РНК и ДНК, взаимодействующие в живой клетке.
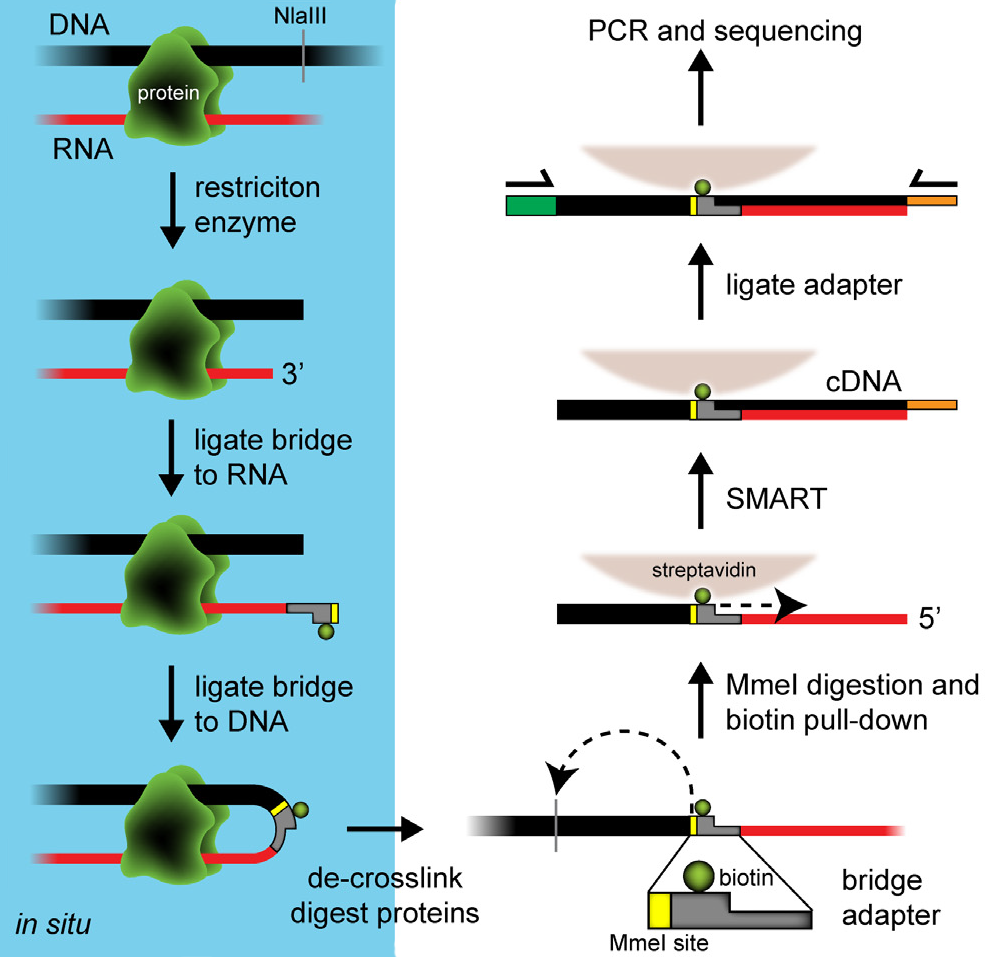
Рисунок 1. Схема протокола Red-C
Интерактом РНК и геномной ДНК
Используя Red-C, ученые изучили интерактом (совокупность всех взаимодействий) РНК и геномной ДНК в культуре клеток эритролейкемии человека (линия клеток K562). Оказалось, что наиболее интенсивно с геномной ДНК контактируют мРНК, а также длинные и очень длинные межгенные некодирующие РНК. Кроме того, с геномом взаимодействуют антисмысловые РНК, малые ядерные и ядрышковые РНК, микроРНК, пиРНК и некоторые другие виды некодирующих РНК . Существенную часть выявленных контактов не удалось отнести к какой-либо из ранее аннотированных РНК. Во многих случаях фрагменты таких неопознанных РНК в действительности принадлежали одному неизвестному транскрипту. Исследователи предложили называть «темную материю» неизвестных РНК, взаимодействующих с геномом, общим термином «X-РНК».
Об огромном разнообразии РНК рассказывает статья «Обо всех РНК на свете, больших и малых» [4]. — Ред.
Чтобы выяснить, насколько хорош метод Red-C для определения положения РНК в пространстве ядра, исследователи проанализировали, с какими областями генома наиболее активно взаимодействуют РНК того или иного вида. Выяснилось, что бóльшая часть мРНК локализуется неподалеку от гена, с которого они были считаны, то есть в месте своего синтеза. В самом деле, фиксация формальдегидом пришивает созревающий транскрипт к ДНК, на матрице которой он синтезируется. А вот некодирующая РНК MALAT1, которая локализуется в ядерных спеклах (ядерных тельцах, в которых проходит сплайсинг) и участвует в процессинге предшественников мРНК, располагается далеко от гена, с которого она была считана. Как и следовало ожидать, метод Red-C подтвердил, что РНК XIST покрывает всю X-хромосому, с которой она и считывается (разумеется, это делает не одна-единственная молекула РНК XIST, а множество ее молекул; у человека длина XIST составляет 17 т.п.о. [5]).
Но MALAT1 и XIST — редкие исключения среди некодирующих РНК, которые подробно изучены, в том числе и экспериментально, и их биология хорошо известна. А что насчет всего остального океана безымянных РНК, которым в плане изученности повезло меньше? Авторы работы провели систематический анализ предпочтений каждой РНК в плане взаимодействий с хромосомами, причем как на близком расстоянии, так и на большей дистанции. Для каждой РНК рассчитали плотность контактов с той хромосомой, с которой она транскрибируется, на определенных интервалах: сам ген, который кодирует РНК (интервал G), на расстоянии от 0 до 50 тысяч пар оснований (т.п.о.) от границ гена в обе стороны (интервал S), на расстоянии от 50 до 500 т.п.о. от границ гена (интервал M), на расстоянии от 500 тысяч до 5 миллионов п.о. от границ гена (интервал L) и на расстоянии свыше 5 миллионов п.о. от границ гена (интервал R) (рис. 2).
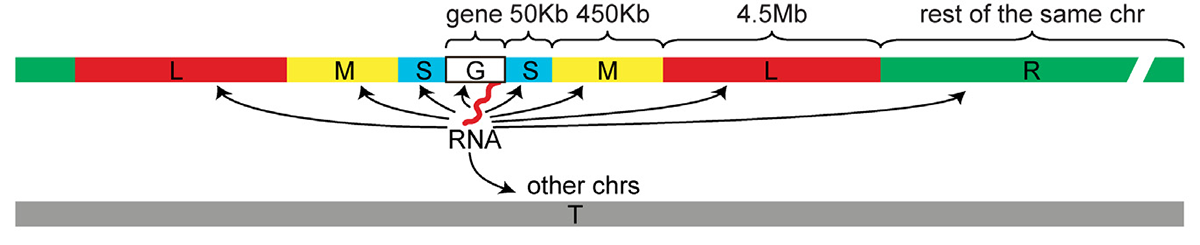
Рисунок 2. Интервалы, на которых рассчитывалась частота контактов для каждой РНК
Далее для каждого интервала рассчитывали относительную частоту попадания в него конкретного контакта путем деления общего числа контактов в интервале на количество контактов этой конкретной РНК с другими хромосомами. Оказалось, что почти все РНК наиболее часто взаимодействуют с тем геном, с которого они были считаны, однако количество контактов сильно различалось у разных видов РНК. Так, малые ядерные и ядрышковые РНК взаимодействуют со «своими» генами и прилежащими областями реже, чем РНК других видов. XIST тоже ведет себя нестандартно: вблизи своего гена плотность контактов у нее относительно невелика по сравнению с другими РНК, зато она активно взаимодействует с дальними участками X-хромосомы, с которой транскрибируется.
Информация о том, как часто РНК взаимодействует с разными участками генома, может многое рассказать о ее биологических функциях. Верно и обратное: биологические функции во многом определяют пространственную локализацию РНК. Например, РНК, участвующие в сплайсинге, активно контактируют со всеми хромосомами (оно и понятно: сплайсинг нужен генам на всех хромосомах), а уже не раз упомянутая XIST локализуется почти исключительно вблизи неактивной X-хромосомы, потому что она обеспечивает ее инактивацию. Для идентификации РНК, преимущественно взаимодействующих с определенными участками генома, ученые разработали алгоритм, распределяющий их на группы в зависимости от частот контактов РНК с геномом в разных интервалах: S+M и L, L и R, R и хромосомы, отличные от той, с которой была считана РНК (T на рисунке 1). С помощью этого алгоритма ученые идентифицировали 313 РНК, которые преимущественно взаимодействуют с участками генома вокруг «родного» гена, 30 XIST-подобных РНК, которые полностью «облепляют» хромосому, на которой они закодированы, 224 РНК, взаимодействующих буквально со всем геномом, а всего выявили 10 367 РНК, образующих 500 и более контактов с геномом.
Взаимодействие различных РНК с активным и неактивным хроматином
Еще один аспект исследования касался того, какие РНК предпочитают взаимодействовать с активным хроматином, а какие — с репрессированным. Для изучения этого вопроса исследователи воспользовались готовой аннотацией участков активного и неактивного хроматина в клетках K562. Авторы работы проанализировали, с какими участками генома наиболее активно взаимодействуют энхансерные РНК, то есть некодирующие РНК, которые считываются с участков генома, функционирующих как энхансеры. Оказалось, что РНК, считанные с сильных энхансеров, предпочтительнее взаимодействуют с сильными энхансерами на той же хромосоме, что, возможно, указывает на образование кластеров энхансеров в пространстве клеточного ядра. С активным хроматином наиболее интенсивно взаимодействуют сплайсосомные РНК, напрямую участвующие в реакциях сплайсинга, а очень длинные некодирующие РНК и X-РНК, напротив, тяготеют к репрессированному хроматину, расположенному на расстоянии до 10 миллионов пар оснований от границ генов, с которых эти РНК считываются. Авторы работы предположили, что очень длинные некодирующие РНК и X-РНК участвуют в сайленсинге (подавлении работы) соседних с ними генов.
Особенно интересна оказалась локализация микроРНК MIR3648 и MIR3687, которые вырезаются из транскрипта-предшественника рРНК. Эти микроРНК взаимодействуют с неактивным хроматином по всему геному и являются одними из самых многочисленных РНК репрессированного хроматина. Авторы исследования показали, что они ассоциированы с областями поздней репликации, практически не взаимодействуют с активно транскрибируемыми генами, а также весьма многочисленны в «генных пустынях» — областях генома, почти лишенных генов, — и бедной генами хромосоме 18. Похожую локализацию имеют и некоторые другие РНК, происходящие из длинного предшественника рРНК, хотя зрелые рРНК взаимодействуют и с активным, и с неактивным хроматином с одинаковой частотой. Возможно, это отчасти объясняет наличие гетерохроматинового «обрамления» вокруг ядрышка, ведь именно там накапливаются предшественники рРНК.
Red-C и модель сплайсинга
Исходя из данных, полученных с помощью Red-C, ученые оценили, как экзоны и интроны внутри мРНК взаимодействуют с хромосомой, с которой она была синтезирована, и с другими хромосомами. Оказалось, что мРНК не «сидит» на родном гене бóльшую часть времени, а активно взаимодействует с другими участками хромосомы на этапе ухода зрелого транскрипта от гена, с которого он был синтезирован, причем длинные мРНК более активно взаимодействуют со «своей» хромосомой, чем с другими — видимо, эти контакты устанавливаются во время долгой транскрипции. Любопытно, что мРНК более интенсивно взаимодействует с участками, локализованными ближе к концу, нежели началу гена. Вероятно, это связано с тем, что по ходу транскрипции РНК-полимераза «тащит» транскрипт за собой вдоль по ДНК.
Примечательно, что, хотя суммарное число контактов для интронов больше, чем для экзонов (вероятно, в силу того, что интроны, как правило, длиннее экзонов), на единицу длины экзоны устанавливают в два раза больше контактов, чем интроны. Первый экзон транскрипта обычно очень активно взаимодействует со всем «родным» геном, и после точки завершения транскрипции частота контактов резко падает. В случае первого интрона частота контактов снижается по мере приближения к концу гена, и резкого падения частоты контактов после терминатора транскрипции, как у первого экзона, не наблюдается. Вероятно, первый интрон удаляется еще во время транскрипции, а первый экзон «тащится» вслед за РНК-полимеразой вдоль всего гена до самого конца транскрипции. В то же время последний экзон и последний интрон демонстрируют сравнительно невысокие частоты контактов с участками собственного гена, что свидетельствуют о быстром высвобождении мРНК после завершения траснкрипции последних интрона и экзона.
Таким образом, полученные с помощью Red-C данные подтверждают представление о котранскрипционном сплайсинге интронов. Примечательно, что экзонные участки мРНК показывают повышенную частоту взаимодействия с участками ДНК, расположенными сразу за геном. Вероятно, РНК-полимераза II «волочит» новосинтезированную мРНК за собой уже после завершения транскрипции до того момента, пока в мРНК не будет внесен разрыв в сайт полиаденилирования.
В то же время популярная гипотеза образования петли из гена, когда промотор и терминатор пространственно сближаются, что могло бы облегчать повторную инициацию транскрипции, не нашла своего подтверждения в данных Red-C. Если бы промотор и терминатор гена действительно взаимодействовали друг с другом, а расположенная между ними часть гена сворачивалась в петлю, то первые интрон и экзон мРНК наиболее интенсивно взаимодействовали бы с концом гена, а последние интрон и экзон — наоборот, с началом гена. Однако ни одно, ни второе с помощью Red-C не подтвердилось.
Что дальше?
Уже первое применение Red-C на всего лишь одной линии клеток позволило не только выделить несколько интересных с функциональной точки зрения групп ранее неизвестных РНК, но и разобраться в таких фундаментальных вопросах молекулярной биологии, как сплайсинг и инициация транскрипции.
Первый автор статьи в Nucleic Acids Research Алексей Гаврилов комментирует результаты работы следующим образом:
В настоящее время мы ведем разработку нового метода, сочетающего технику Red-C с техникой иммунопреципитации хроматина. В основе метода лежит преципитация РНК-ДНК-белковых комплексов с использованием антител к интересующему белку. В первую очередь, мы проанализируем РНК-ДНК-взаимодействия, опосредованные белком CTCF. Мы надеемся, что полученные данные позволят идентифицировать спектры некодирующих РНК, принимающих участие в организации СTCF-зависимых петель хроматина, что поможет раскрыть механизмы формирования этих петель. Мы также предполагаем разработать метод, комбинирующий техники Red-C и DamID, и с его помощью идентифицировать РНК, контактирующие с участками ДНК, локализованными на периферии ядра. Эта работа может пролить свет на механизмы репрессии генов вблизи ядерной ламины.
Остается ожидать новых впечатляющих результатов, которые будут получены благодаря этим новым методам.
Литература
- Кодирующие некодирующие РНК;
- Загадочное путешествие некодирующей РНК Xist по X-хромосоме;
- Alexey A Gavrilov, Anastasiya A Zharikova, Aleksandra A Galitsyna, Artem V Luzhin, Natalia M Rubanova, et. al.. (2020). Studying RNA–DNA interactome by Red-C identifies noncoding RNAs associated with various chromatin types and reveals transcription dynamics. Nucleic Acids Research. 48, 6699-6714;
- Обо всех РНК на свете, больших и малых;
- Carolyn J. Brown, Brian D. Hendrich, Jim L. Rupert, Ronald G. Lafrenière, Yigong Xing, et. al.. (1992). The human XIST gene: Analysis of a 17 kb inactive X-specific RNA that contains conserved repeats and is highly localized within the nucleus. Cell. 71, 527-542.