
Коронавирус из палеолита
12 июля 2021
Коронавирус из палеолита
- 1717
- 0
- 4
«Захоронение охотников на мамонтов». Работа кисти чешского палеоиллюстратора Зденека Буриана.
-
Автор
-
Редактор
Коронавирусы человека — относительно молодое для науки явление. Первый из них был описан в середине 60-х годов XX века, седьмой стал общемировой проблемой лишь в XXI-м. А что, если от коронавирусной инфекции люди страдали еще в те далекие времена, когда никто не знал слов «локдаун» и «самоизоляция»? Коллективу ученых из США и Австралии удалось заглянуть в доисторическую эпоху верхнего палеолита и с помощью методов эволюционной генетики найти следы масштабной коронавирусной эпидемии, поразившей группы охотников-собирателей более 20 тыс. лет назад на территории Восточной Азии.
Пандемия новой коронавирусной инфекции, которую мы переживаем в начале третьего десятилетия XXI века, подчеркнула уязвимые места систем общественного здравоохранения во всех, без исключения, странах мира. Несмотря на достаточно широкий арсенал биомедицинских и эпидемиологических методов исследования, человечество оказалось уязвимым к атакам вирусных инфекций. Всемирная организация здравоохранения (ВОЗ) активно призывает нас извлекать уроки из сложившейся ситуации и быть готовыми к тому, что нынешняя эпидемия будет, к сожалению, не последней. В мае 2021 года Питер Хотез — профессор медицинского колледжа Бейлора (Техас, США) в интервью NBC отмечал важность мультидисциплинарного изучения природы происхождения вируса COVID-19, «иначе нас ждет и COVID-26, и COVID-32 в дальнейшем». Действительно, эта проблема уже давно перестала быть предметом внимания исключительно вирусологов, эпидемиологов и специалистов по санитарной медицине.
Палеогенетики также не остались в стороне от изучения причин распространения коронавирусной эпидемии. Изучение древних эпидемий не является принципиально новым направлением в науке [1], однако современные методы исследования позволяют ученым открыть много нового. Сейчас палеопатология в целом и палеогенетика в частности — перспективные быстроразвивающиеся отрасли научного знания. Еще совсем недавно мы могли судить лишь о тех заболеваниях, что способны так или иначе отражаться на окаменевающих останках [2]. В 2019 году секвенирование древней ДНК позволило международной исследовательской группе сделать вывод о том, что эпидемия чумы, произошедшая в Европе примерно 5000 лет назад, привела к неолитическому упадку — резкому сокращению численности людей, населявших западную Евразию. Авторы полагают, что выделенный из человеческих останков штамм чумной палочки содержал ген активатора плазминогена, необходимого для развития легочной чумы — чрезвычайно смертельной формы этой болезни, способной передаваться по воздуху [3].
Объединенному австрало-американскому коллективу авторов удалось заглянуть еще глубже в прошлое. В конце июня 2021 г. в журнале Current Biology были опубликованы результаты исследования южно-азиатских популяций людей, проживавших на территории современных Китая, Вьетнама и Японии более 20 тыс. лет назад. Авторам удалось найти генетические следы древней коронавирусной эпидемии, случившейся в эпоху позднего палеолита — суровое время, когда нашу планету терзала череда цикличных оледенений, а по Земле ходили мамонты [4].
Исследовательская группа применила эволюционный анализ к наборам геномных данных человека для реконструкции событий естественного отбора, отразившегося на десятках человеческих генов, кодирующих иммунные белки, которые взаимодействовали с древними и, вероятно, взаимодействуют с современными коронавирусами (включая SARS-CoV-2). На протяжении всей истории эволюции нашего вида отбор был нацелен на белки, напрямую взаимодействующие с вирусами — например, те, что участвуют в иммунном ответе или используются вирусами для проникновения в клетки хозяина. За миллионы лет эволюции человека отбор привел к скорости фиксации вариантов генов белков, взаимодействующих с вирусами (virus interacting proteins, VIP), в три раза большей, чем других классов генов.
Авторы исследования проанализировали сигналы отбора в 420 VIP, взаимодействовавших с коронавирусом (CoV-VIP), на базе 26 популяций по всему миру (по данным проекта «1000 геномов»). Они выявили, что в восьми парах хромосом жителей Восточной Азии присутствует большое количество адаптационных сигналов CoV-VIP, и их уровень в верхнепалеолитический период в три раза превышает ожидаемый, в то время как в популяциях на других континентах генетические сигналы COV-VIP не встречается вовсе (в том числе в соседнем южно-азиатском регионе) (рис. 1).

Рисунок 1. Гены восточно-азиатской популяции, кодирующие белки взаимодействия с древним коронавирусом, были обнаружены в восьми парах хромосом, содержащих информацию об адаптации к инфекции. Обнаруженное количество генов, кодирующих адаптивные к коронавирусной инфекции признаки, в восточно-азиатской популяции превышало ожидаемый для верхнего палеолита уровень в 3 раза (значение справедливо для людей, живших 900–1000 поколений назад, приблизительно 20–25 тыс. лет назад).
[4], рисунок адаптирован
Примечательно, что 42 гена, содержащие информацию об устойчивости к древней коронавирусной инфекции, кодируют те же противовирусные адаптации, которые снижают процент госпитализаций в тяжелом состоянии при ковиде у современных британцев.
Авторы также отмечают крайне низкую вероятность того, что резкое обогащение генома сигналами CoV-VIP может быть вызвана каким-либо другим вирусом, имеющим сходные взаимодействующие белки. Результаты масс-спектрометрии (определение коэффициента коалесценции и возраста аллелей) продемонстрировали высокую для подобных исследований (73%) степень взаимодействия белков CoV-VIP с вирусом SARS-CoV-2. Экологическое давление абиотических факторов тоже представляется маловероятным фактором возникновения подобных изменений в генотипе. 21 из 42 (50%) CoV-VIP, прошедших отбор 900 поколений назад, обладают способностью формировать иммунный ответ к коронавирусной инфекции с высокой степенью достоверности.
Все это позволяет говорить о том, что в эпоху позднего палеолита на территории Восточной Азии свирепствовала настоящая эпидемия древнего коронавируса. На именно внезапный, свойственный вирусным эпидемическим заболеваниям характер распространения также указывают данные, полученные на основе анализа графика, показывающего время появления на хромосомах специфических локусов для генов, ответственных за белки, взаимодействовавшие с коронавирусами. Установлено, что наследственная рекомбинация привела к появлению новой комбинации генов, возникнувших стихийно примерно 900 поколений назад. Количество мутаций в генах CoV-VIP превышало общее количество всех мутаций за исследуемый период в три раза. Период возникновения наиболее значительного количества сигналов CoV-VIP приходится именно на людей, живших 870±100 поколений назад (примерно 25 тыс. лет назад, если в среднем давать 28 лет на поколение с учетом средней продолжительности жизни охотников-собирателей в позднем палеолите) (рис. 2). События, начавшиеся 900 поколений назад, явно предшествовали предполагаемому разделению различных популяций Восточной Азии, включенных в проект «1000 геномов», от их общей предковой популяции, что также указывает на эпидемических характер появления выявленного в результате исследования резкого всплеска мутационных изменений.
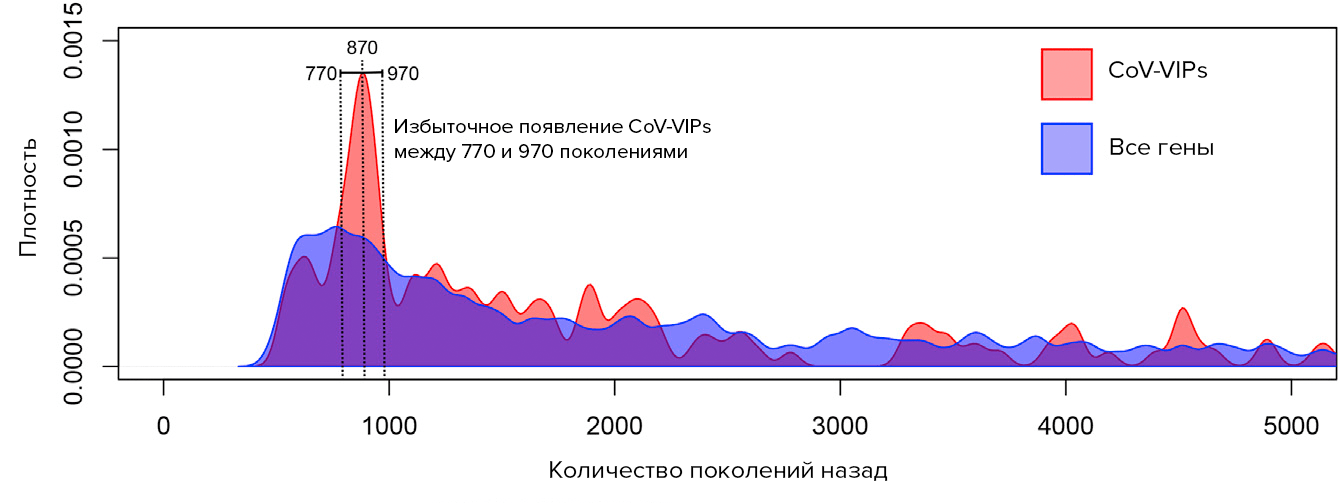
Рисунок 2. Динамика плотности мутаций, встречающихся в прошлых поколениях. Красным маркером отмечены мутационные сигналы в генах, ответственных за CoV-VIP, синим — общее количество во всех генах.
[4], рисунок адаптирован
Как мы видим, современные человеческие геномы содержат информацию о нашей эволюционной борьбе с болезнями, поразившими предков современных жителей Восточной Азии более 20 тыс. лет назад. Такое знание несомненно будет полезно в определении того, какие вирусы будут иметь пандемический потенциал в будущем. Эволюционная информация может в конечном итоге помочь в разработке новых лекарств и методов лечения, дополняя информацию, полученную в результате более традиционных эпидемиологических и биомедицинских исследований. Ученые полагают, что CoV-VIP, ставшие мишенью древней коронавирусной эпидемии, могут играть свою роль в этиологии SARS-CoV-2 у современных популяций людей. Из 42 появившихся еще у древних людей генов CoV-VIP, хранящих информацию об адаптационных сигналах, только четыре (SMAD3, IMPDH2, PPIB и GPX1) сейчас изучаются в целях разработки 11 противосимптомных препаратов, на данный момент проходящих клинические испытания, остальные еще ждут своего анализа в рамках антикоронавирусной терапии [4], [6].
В заключение, конечно, нужно отметить, что адаптация к древним вирусным эпидемиям в конкретных популяциях совсем не обязательно подразумевает какую-либо разницу в генетической восприимчивости к современному коронавирусу между разными популяциями людей. Актуальные научные данные указывают на подавляющее влияние социально-экономических факторов (плотность населения, благоустройство населенных мест, материальное благосостояние населения, санитарная культура, состояние здравоохранения и др.) в эпидемиологии новой коронавирусной инфекции COVID-19.
Литература
- Секретное оружие конкисты;
- Пилипенко А.С. и Молодин В.И. (2010). Палеогенетический анализ в археологических исследованиях. «Информ. вестн. ВОГиС». 2, 280–311;
- Nicolás Rascovan, Karl-Göran Sjögren, Kristian Kristiansen, Rasmus Nielsen, Eske Willerslev, et. al.. (2019). Emergence and Spread of Basal Lineages of Yersinia pestis during the Neolithic Decline. Cell. 176, 295-305.e10;
- Yassine Souilmi, M. Elise Lauterbur, Ray Tobler, Christian D. Huber, Angad S. Johar, et. al.. (2021). An ancient viral epidemic involving host coronavirus interacting genes more than 20,000 years ago in East Asia. Current Biology;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Chris Finan, Anna Gaulton, Felix A. Kruger, R. Thomas Lumbers, Tina Shah, et. al.. (2017). The druggable genome and support for target identification and validation in drug development. Sci. Transl. Med.. 9, eaag1166.





