мРНКаааауу
30 июня 2009
мРНКаааауу
- 3471
- 0
- 5
Структура каждой матричной РНК уникальна, однако некоторые общие черты, такие как, например, поли(А)-хвост, у них всё-таки есть. Но и это не абсолютно: недавно обнаружилось, даже хвост у них может варьировать.
-
Автор
-
Редакторы
В классических учебниках молекулярной биологии структура мРНК эукариот охарактеризована весьма чётко — метилированный гуанин, связанный через трифосфатную группу с 5′-концом РНК (5′-кэп), 5′-нетранслируеамая область (НТО) с сайтом «посадки» рибосомы, кодирующая белóк последовательность, 3′-НТО и поли(А)-хвост. Наличие последнего считается критическим фактором — отщепление поли(А)-хвоста, или деаденилирование, вызывает деградацию мРНК. Однако последние работы показали, что поли(А) является не единственно возможной 3′-терминальной последовательностью — к этому «хвосту» может быть дополнительно присоединён поли(У)-«хвостик» различной длины.
Известно, что матричная РНК (мРНК) является главным посредником между ДНК и белок-синтезирующим аппаратом. У прокариотических организмов только что транскрибированная мРНК функционально активна и начинает транслироваться немедленно. У эукариот же после транскрипции пре-мРНК подвергается созреванию. Только правильно созревшая мРНК транспортируется из ядра в цитоплазму и служит матрицей для синтеза белка.
Поскольку количество вновь синтезированного белка прямо пропорционально количеству содержащейся в цитоплазме соответствующей мРНК, регуляция времени жизни последней является критическим фактором, регулирующим скорость синтеза белкá. Нарушения этой регуляции ведут к катастрофическим процессам, — в частности, потере контроля над основными процессами в клетке и трансформации клетки в раковую. Поэтому любая мРНК имеет схожую структуру — кодирующая область в центре и регуляторные фрагменты по краям (рис. 1).
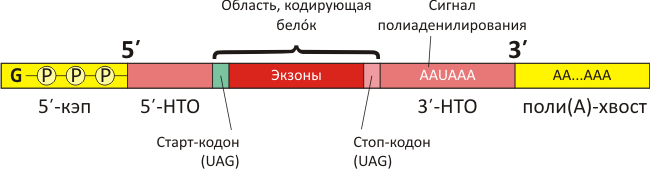
Рисунок 1. Строение мРНК эукариот. Любая мРНК начинается с кэпа (от англ. cap — шапка, кепка) — метилированного остатка гуанина, который присоединён нестандартной связью (через три фосфатных группы). Наличие кэпа критично для функционирования мРНК в клетке. За ним следует 5′-нетранслируемая область (НТО), которая не кодирует белóк: её функция — «узнавание» рибосомы и регулирование трансляции. Сама кодирующая область начинается после «стартового» нуклеотидного триплета АUG и заканчивается стоп-кодоном (UGA или другими). За стоп-кодоном следует 3′-НТО, которая также важна для регуляции синтеза белка. Заканчивается вся молекула поли(А)-хвостом, который, как оказалось, всё-таки может состоять не только из аденозинов, но также включать остатки уридина (см. текст).
Основной механизм регуляции известен: на 3′-конце каждой мРНК находится регуляторная последовательность, состоящая практически полностью из одного нуклеотида — аденозина (А), из-за чего она и получила название поли(А)-хвост. Её длина регулируется рядом ферментов — она может и удлиняться, и обрезаться. Известно, что почти всегда длина поли(А)-хвоста определяет время жизни (точнее, полураспада) всей молекулы. Когда поли(А)-хвост укорачивается настолько, что не может более выполнять свои функции (связывать регуляторный комплекс белков), происходит отрезание кэпа ферментом Dcp1. Стандартная схема распада мРНК начинается с постепенного деаденилирования 3′-конца экзонуклеазой PARN. После этого мРНК уже не может участвовать в инициации транскрипции, и расщепляется экзонуклеазами.
В общем, наличие и функции поли(А)-хвоста неоспоримо важны. Но недавно оказалось, что не только поли(А) выполняет роль такого регулятора. Было открыто явление уридинилирования 3′-конца мРНК, т. е. модификация поли(А)-хвоста поли-уридином (поли(У)) [2]. Обнаружилось, что поли(А)-хвост может модифицироваться одним или многими остатками уридина, что, по-видимому, ведёт к инициации расщепления мРНК. Такой механизм уже обнаружен у ряда организмов (одноклеточных водорослей Chlamydomonas reinhardtii, дрожжей Schizosaccharomyces pombe, лягушки Xenopus laevis, цветкового растения Arabidopsis thaliana) и, вероятно, будет обнаружен и у многих других.
Такая распространённость заставляет взглянуть на поли-уридинилирование мРНК как на универсальный второй путь регуляции «жизни» мРНК в клетке. Сведений пока ещё очень мало, но уже открыта белковая машинерия, обеспечивающая «пришивку» уридина к мРНК, — это семейство терминальных уридиниловых терминаз (TUTases, PUPases) [3]. Ранее было известно, что они модифицируют некоторые малые ядерные РНК (например U6) и таким образом стабилизируют их структуру и время жизни. Однако в случае мРНК, модификация её «поверх» поли(А) уридином (достаточно всего нескольких нуклеотидов) вызывает узнавание мРНК специальным комплексом вызывающих деградацию ферментов — Lsm1–7, — которые запускают процесс деградации мРНК по стандартной схеме: деаденилирование, декэпирование и экзонуклеазное расщепление. И хотя наличие нескольких уридинов на 3′-конце может показаться не очень важной деталью, всё-таки это не так. Для этого сделаем короткий экскурс в методы современной молекулярной биологии.
Мониторинг мРНК в клетках (как тотальной, так и отдельных типов) на сегодня является одним из самых точных методов для описания состояния клетки и, соответственно, — целых органов. Дело в том, что ДНК является стандартной «базой данных», одинаковой во всех тканях организма (с некоторыми оговорками, на которых мы тут останавливаться не будем). Однако намного более важно узнать, какие гены и с какой интенсивностью активируются в каждой ткани или органе. Самая ценная информация — это уровень экспрессии индивидуальных генов в определённый отрезок времени. Анализ профиля синтеза белков по регистрации присутствия самих белковых молекул может быть недостаточно точен, поскольку многие белки довольно стабильны и могут сохраняться достаточно долгое время. Кроме того, основной метод их регистрации — при помощи антител (вестерн-блоттинг) — довольно капризен, и не во всех случаях может помочь. По этой причине анализ присутствия самих белков не во всех случаях может дать ответ на многие вопросы, которые можно получить, анализируя содержание мРНК и профили её экспрессии. Анализ «спектра» мРНК даёт намного более ценную информацию об активности генов — за счёт короткого времени жизни РНК в цитоплазме мы всегда узнаём самые «свежие» новости, а сам метод намного более чувствителен (за счет ПЦР-амплификации исходных образцов).
Основной подход к исследованию мРНК базируется на методе обратной транскрипции (рис. 2), происходящем из открытия Д. Балтимором, Р. Дульбекко и Г. Теминым явления обратной транскрипции у вирусов (Нобелевская премия по физиологии и медицине, 1975). Ими был описан фермент обратная транскриптаза, который, используя молекулу РНК в качестве матрицы, строит вторую, комплементарную, цепь, но уже ДНК. Такая одноцепочечная комплементарная ДНК получила название кДНК (cDNA). Лабораторными методами эту кДНК можно «умножить» в миллионы раз и сохранить для последующего анализа. На методе обратной транскрипции базируются многие современные методы исследования для анализа клеточной системы транскрипции-трансляции.
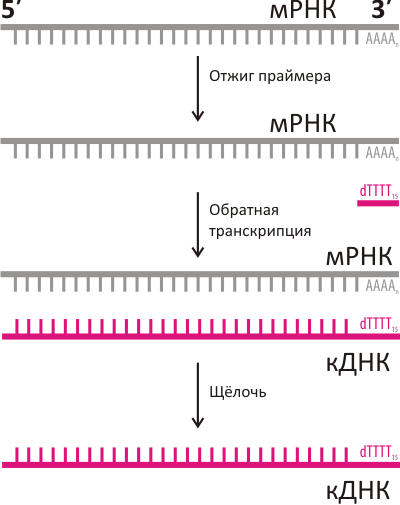
Рисунок 2. Схема обратной транскрипции
Однако тут есть один нюанс — чтобы обратная транскрипция началась, нужна короткая стартовая «затравка» (праймер), которая была бы комплементарна 3′-концу РНК (рис. 2). До сегодняшнего дня в качестве универсального праймера в основном использовался олигонуклеотид поли(Т), комплементарный поли(А)-хвосту. Теперь, когда оказалось, что поли(А)-хвост не столь уж инвариантен, возникает вопрос: является ли «универсальный» поли(Т)-праймер настолько уж универсальным? В свете описанного открытия, возможно, он может не связываться и не выявлять целые пулы мРНК с уридинами на 3′-конце, которые оказываются фактически «невидимыми» для традиционных алгоритмов в молекулярной биологии. А если учесть, что метод обратной транскрипции на основе поли(Т) и последующая ПЦР-диагностика являются сейчас основными инструментами для определения экспрессии генов, открытие альтернативных 3′-концов может вызвать необходимость пересмотреть истинность целых массивов данных, которые сегодня считаются исходной базой для молекулярных исследований.
Литература
- S. Ito-Harashima, K. Kuroha, T. Tatematsu, T. Inada. (2007). Translation of the poly(A) tail plays crucial roles in nonstop mRNA surveillance via translation repression and protein destabilization by proteasome in yeast. Genes & Development. 21, 519-524;
- Olivia S Rissland, Chris J Norbury. (2009). Decapping is preceded by 3′ uridylation in a novel pathway of bulk mRNA turnover. Nat Struct Mol Biol. 16, 616-623;
- William Marzluff. (2009). A new way to initiate mRNA degradation. Nat Struct Mol Biol. 16, 613-614.