
Мультиомики в сельском хозяйстве: когда ДНК встречается с трактором
26 января 2024
Мультиомики в сельском хозяйстве: когда ДНК встречается с трактором
- 876
- 0
- 6
Вместе с приходом мультиомиксных технологий получать новые породы животных и сорта растений стало еще проще. Ускорилось и производство полезных компонентов для пищевой и химической индустрии.
Рисунок в оригинальном разрешении.
иллюстрация Карины Илиопуло
-
Автор
-
Редакторы
-
Иллюстратор
В новой статье спецпроекта «Мультиомиксные технологии» мы поговорим о том, какие технологические тренды сегодня в моде у специалистов, занятых сельским хозяйством. И здесь не будет долгих рассуждений о лучших сортах редиса — только самые амбициозные цели и по-настоящему большие данные. Вместе мы убедимся, что картинка, которую большинство из нас рисует себе, когда слышит слово «агроном», давно не соответствует действительности. Работники современного сельского хозяйства — это не пахари с сохой наперевес. Зачастую всё самое интересное в этой приземленной отрасли происходит вообще не в поле, а гораздо раньше — в НИИ и лабораториях, где люди учатся предсказывать природные процессы и изменять их, оперируя поистине огромными массивами информации.
Мультиомиксные технологии
На «Биомолекуле» уже разбирались методы и технологии молекулярной биологии, в том числе протеомные и геномные. В связи с технологическим прогрессом всё чаще применяют их комбинацию, причем к ним добавляются и другие «омики». В спецпроекте «Мультиомиксные технологии» мы расскажем не столько о самих методах, сколько об их применении в самых разных областях — фундаментальной науке, медицине, экологии, сельском хозяйстве. Мы рассмотрим, как калейдоскоп множества методов позволяет лучше увидеть общую картину, ответить на интересующие ученых вопросы и продвинуться в решении практических задач.
Партнер спецпроекта — аффилированные компании «Филип Моррис Интернэшнл» в России, ФМИ (PMI Affiliates in Russia).
С момента появления сельского хозяйства, а произошло это больше 10 тысяч лет назад, люди неуклонно прикладывают силы, чтобы эта отрасль процветала и продолжала бесперебойно снабжать нас необходимым питанием. Наращивание объемов выпускаемой продукции, захват новых территорий под пахотные земли, неуклонный рост животноводческой отрасли даже с учетом появления альтернативных источников пищевого белка — всё это необходимо для того, чтобы прокормить растущее население планеты. И население это не маленькое. В 2011 году количество людей на Земле превысило 7 млрд [1], в 2022 была преодолена планка 8 млрд, а 2055 год и вовсе обещает стать знаменательным — к этому времени общее число людей должно достигнуть круглых 10 млрд.
Разумеется, столь быстрый рост населения — это не только повод для нашей гордости как вида, но и серьезная проблема, если вы занимаетесь обеспечением людей продовольствием. И если раньше достаточно было просто сеять больше пшеницы и выращивать больше коров, то теперь такой подход не работает. Времена изменились, и нам приходится думать об экологии и глобальном потеплении, а значит, экстенсивный путь развития аграрного сектора должен заменяться интенсивным, где ставка делается на увеличение эффективности уже существующих процессов. Иными словами, нам необходимо постоянно увеличивать урожайность растений, которые мы едим, и продуктивность животных, которых разводим. И тут на помощь человеку приходят современные технологии.
Мультиомиксные технологии и растениеводство
Омики и их комбинации с успехом используются в работе с растениями. И речь здесь идет не только о геномике, транскриптомике и протеомике, благодарить за которые нам следует новые методы секвенирования нуклеиновых кислот (NGS) , но и о менее очевидных совокупностях данных: метаболомике, иономике (это одновременная оценка количественного состава минеральных элементов в тканях и органах растений) и феномике — исследовании черт организма (рис. 1) [3]. Например, соединение геномики с феномикой и другими омиками делает видимой взаимосвязь между геномом и фенотипом культуры в конкретных условиях их роста.
Подробнее о секвенировании читайте в статье «12 методов в картинках: секвенирование нуклеиновых кислот» [2].
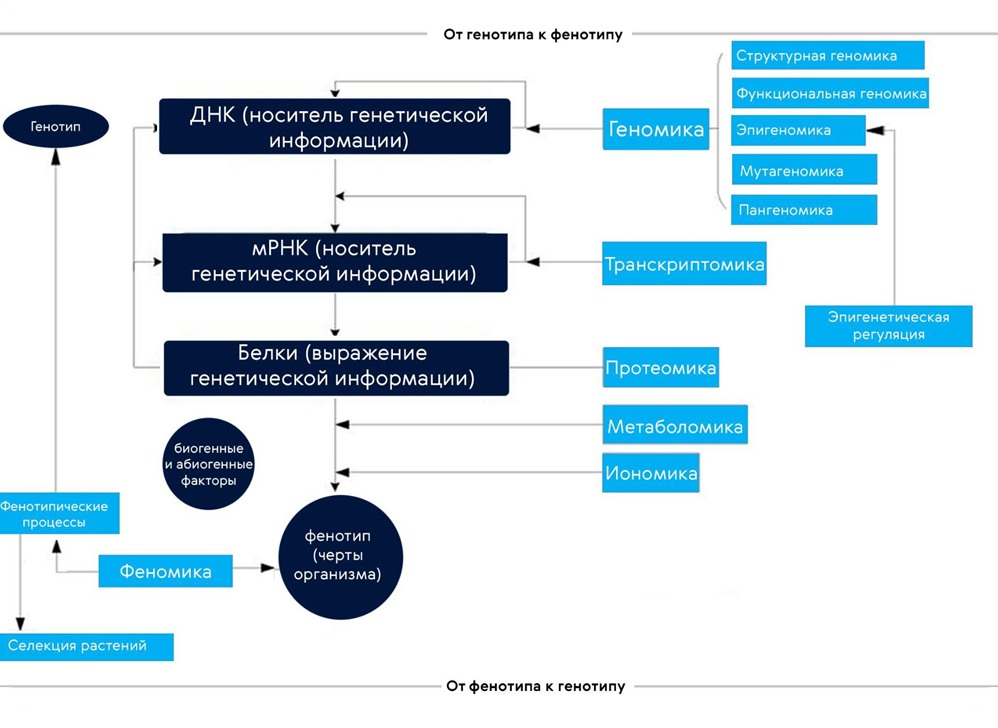
Рисунок 1. Что изучают омики, которые часто используются в сельском хозяйстве. Все виды геномики сконцентрированы на изучении ДНК. Это может быть ее структура, функции, эпигенетические модификации, мутации, влияющие на организм, или даже совокупный геном близкородственных микроорганизмов. Транскриптомика исследует мРНК, протеомика — совокупность экспрессируемых клеткой белков, а метаболомика и иономика ищут связь между белками и имеющимся фенотипом (свойствами), за который, в свою очередь, ответственна феномика. Оперируя большими данными, ученые изучают фенотип, опираясь на знания о геноме, и наоборот. Примечательно, что на данные всех этих омик оказывает влияние множество биотических и абиотических факторов.
иллюстрация Карины Илиопуло на основе [3]
Мультиомики уже зарекомендовали себя в работе с самыми разными культурами: пшеницей, соей, томатами, ячменем, просом, рисом, хлопком [3–5]. Они отлично подходят для таких задач, как выяснение механизмов роста и старения растений, а также предсказание их урожайности и реакций на биотический и абиотический стресс, что в конечном счете приводит нас к прогнозной селекции [6–11]. Сегодня это крайне перспективное направление. Климат в ближайшем будущем будет меняться, оказывая влияние в том числе на распространение бактериальных и грибных фитопатогенов и вредителей, и человеку нужно уже сейчас думать над тем, какие растения он будет выращивать через 50 лет. Кроме того, эти подходы могут пригодиться для разработки методов одомашнивания растений de novo с целью получения культур, максимально приспособленных к жизни в существующих условиях [12].
Другая важная цель применения современных подходов к селекции растений, которая актуальна и для животноводства, — создать такую индустрию, которая будет не только обеспечивать достаточное предложение продукта, но и минимально загрязнять окружающую среду. Для растениеводства главным загрязняющим элементом со времен зеленой революции 1960-х годов остаются пестициды (на «Биомолекуле» есть небольшой спецпроект, посвященный этим химикатам). Объемы их потребления необходимо снижать уже сейчас, пока популяции опылителей и микробные сообщества способны самовосстанавливаться после наносимого им ущерба. Не стоит также забывать про вырубку лесов для освобождения новых посадочных площадей. Этот вопрос стоит крайне остро, например, для кофейной индустрии. Ее особенность состоит в том, что плантации кофе планомерно движутся в сторону тропических лесов, и как остановить этот процесс, сохранив кофейный рынок, непонятно. В то же время, фермеры вынуждены подчиняться новым «зеленым» законам, запрещающим дальнейшую вырубку, если не хотят потерять европейскую часть рынка.
Как омики помогают ученым работать с растениями
Решать такие глобальные задачи, как обеспечение населения Земли едой в условиях меняющегося климата проще, если под рукой у тебя есть современные инструменты. И сегодня самыми навороченными из них считаются именно подходы различных «омик» . Самый шик — использовать сразу несколько.
О том, что такое эти «омики», речь шла в первой статье нашего спецпроекта — «Факты и легенды о мультиомиксных технологиях» [13].
Сегодня уже никого не удивишь эталонными геномами для самых разных видов растений — их накопились даже не сотни, а тысячи. А для некоторых видов есть данные и о пангеноме — совокупном геноме растений одного вида, но, например, разных сортов. Для других уровней информации о живых организмах существуют другие «омики» (рис. 1). Они позволяют узнать много нового о работе генома и о функциях экспрессируемых клетками белков. Прибавьте сюда сбор данных в различных условиях среды обитания подопытных, и вы примерно поймете, о каких колоссальных объемах информации идет речь (рис. 2) [14]. И эта информация из года в год становится всё доступнее. Если еще недавно полногеномное секвенирование мог позволить себе далеко не каждый, то сейчас для биологов оно стало рутинным.
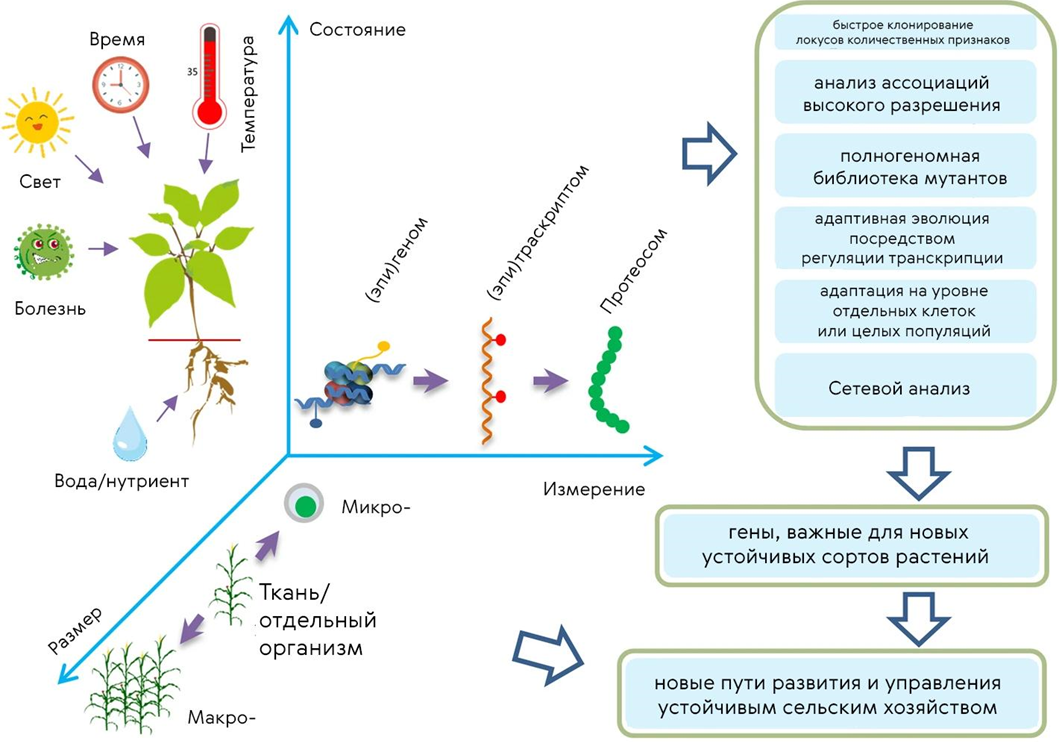
Рисунок 2. Данные, которые ученые собирают с целью развития отрасли сельского хозяйства. Омиксные технологии, оперирующие генетической и фенотипической информацией, позволяют находить гены, важные для создания новых устойчивых к стрессам и болезням сортов растений и искать новые пути управления сельским хозяйством.
Здесь назревает два вопроса: как все эти данные обрабатывать и что нового можно узнать и сделать, разжившись самой разной информацией о растениях?
Ответом на первый, конечно, будет биоинформатика. Эта область науки применяет вычислительные технологии, чтобы обрабатывать, интерпретировать и хранить данные о биологических объектах. Именно благодаря ей биологам доступно моделирование макромолекул и симуляции процессов, происходящих в клетках, что значительно упрощает поисковые исследования. Свои базы данных, созданные с помощью методов биоинформатики, существуют для самых разных областей знаний, не исключая и растениеводство. Многие из таких баз объединяют в себе информацию о геноме, транскриптоме, протеоме и метаболоме растений и играют ключевую роль в решении задач по селекции.
Отвечая на второй вопрос, первым делом снова возвращаемся к геномике. Она позволяет быстрее искать функциональные гены, которыми затем можно будет манипулировать для создания новых, улучшенных сортов (о том, как происходит современная селекция растений, мы рассказывали в обзоре «От ГМО к растениям будущего» [15]). Например, с развитием омиксных подходов заметно ускорилось картирование локусов количественных признаков (тех, за наследование которых отвечает два или более генов [16]). Это поиск и определение локализации участков ДНК, в которых содержатся гены, влияющие на сложные фенотипические признаки, такие как урожайность зерна, качество молока у коров или размер яиц у птицы.
Кроме того, большие геномные данные, такие как повторное секвенирование панели разных линий, помогают проводить эффективный анализ ассоциаций генов, влияющих на фенотип растений, и лучше их картировать [19]. Наконец, использование библиотек полногеномных мутаций в сочетании с полногеномным или целевым секвенированием позволяет быстро находить важные гены, которые ранее не использовались с целью селекции [20].
С другой стороны, сравнительный транскриптомный анализ в различных условиях окружающей среды хорош для идентификации генов, важных для адаптации растений к изменяющемуся климату [21], а использование омик в масштабе одной клетки — для выявления типов клеток, необходимых для такой адаптации [22].
В будущем вполне возможно объединение данных всех применимых для целей сельского хозяйства омик в одну сеть, что сделает поиск самых значимых генов и метаболических путей у растений в разы быстрее и точнее.
Повторное одомашнивание и новые культуры растений
Сегодня особенно актуальным становится поиск новых сортов и культур с высоким коэффициентом выживаемости в условиях изменяющегося климата. При этом в процессе селекции человек раз за разом выбирал выращивать одни варианты растений, упуская из виду другие, которые в настоящее время могли бы пригодиться. Если бы мы могли вернуться к геномам диких предков тех растений, с которыми работаем сейчас, мы бы могли обнаружить более удачные генетические вариации с точки зрения устойчивости сортов к экстремальным температурам или засухам. И повторное одомашнивание растений действительно возможно с использованием современных технологий и при наличии знаний о доместикации [23]. В процессе такой работы можно целенаправленно включать те или иные гены у растений, преуспевающих в адаптации, регулируя их урожайность и другие свойства для создания новых сортов, обладающих преимуществами адаптации к окружающей среде и в то же время высоким потенциалом урожайности.
Помимо разработки устойчивых к климату сортов с использованием диких версий культурных растений, омиксные подходы подходят и для разработки растений с совершенно новыми характеристиками. Так, с растущей популярностью вертикальных ферм востребованными стали изменения в архитектуре растений, которые бы улучшили их эффективность при выращивании не в открытом грунте, а в ограниченном пространстве [24]. В этом случае урожайность одного растения будет небольшой, но за счет компактности и сокращенного цикла роста выращивать такой сорт будет выгодно.
Преимущества объединения омиков
Чтобы лучше понять, как работают клетки и, тем более, сложные организмы, подходы омик удобно объединять между собой. Сочетание метаболомики с геномикой, транскриптомикой и протеомикой дает портрет функций, охватывающий геном и фенотипические взаимодействия растений с окружающей средой [25]. Так, использование мультиомиков помогло определить роль регуляции биосинтеза липидов при формировании хлопкового волокна [26], а исследование ассоциаций между геномными вариантами и фенотипом растений в совокупности с изучением метаболома оказалось мощным методом анализа биохимических и генетических процессов нескольких модельных видов сельскохозяйственных культур, включая рис, кукурузу и томаты [27], [28]. Дальше — больше. Объединение омик оказывается полезно и при создании новых сортов растений, устойчивых к стрессу [29], и при идентификации интересных мутантных форм с перспективными свойствами [30], и даже при поиске наиболее эффективных агрономических техник [31].
В целом, геномная информация в сочетании с подходами, которые концентрируются на признаках, дает лучшее понимание процессов их формирования у растений. Используемые вместе, подходы омики дополняют друг друга и позволяют проводить более глубокий анализ жизненных процессов самых разных объектов, открывая нам функциональные механизмы генов.
Зачем мультиомиксные технологии нужны при изучении сельскохозяйственных почв?
Размышляя подобным образом, для начала стоит подумать о том, что такое почва в понимании биолога, и взглянуть на нее как на место обитания миллионов живых микро- и макроорганизмов. Интересовать нас будут первые. Бактерии и грибы — это неотъемлемая часть почвы, они влияют на ее свойства и вступают во взаимоотношения с растениями так же, как микроорганизмы, обитающие на поверхности и внутри нашего собственного тела, вступают в контакт с нами. В какой-то степени почва — это «кишечник» нашей планеты, и у этого кишечника есть свой неповторимый микробиом.
Растения, как и людей с животными, ошибочно рассматривать исключительно в качестве самостоятельных генетических объектов, так как они постоянно сотрудничают с почвенными микробами. От этого сотрудничества зависит очень многое, начиная с эффективности извлечения растением из земли питательных веществ и заканчивая его способностью синтезировать те или иные соединения. Получается, что фенотип растений зависит не только от их генома и факторов внешней среды, но и от того, с какими микробами они вступают в симбиоз. Для выделения этой значимой связи между макро- и микроорганизмами появился даже специальный термин — холобионт. Так стали называть совокупность из организма-хозяина и всех его микроскопических симбионтов.
Так вот, для изучения растений с этой точки зрения мультиомиксные технологии просто необходимы. На рисунке 4 схематично показано, чем интересуются хологеномика и холо-омиксные технологии и какую информацию помогают собрать.
Так вот, для изучения растений с этой точки зрения мультиомиксные технологии просто необходимы. На рисунке 4 схематично показано, чем интересуются хологеномика и холо-омиксные технологии и какую информацию помогают собрать [32].
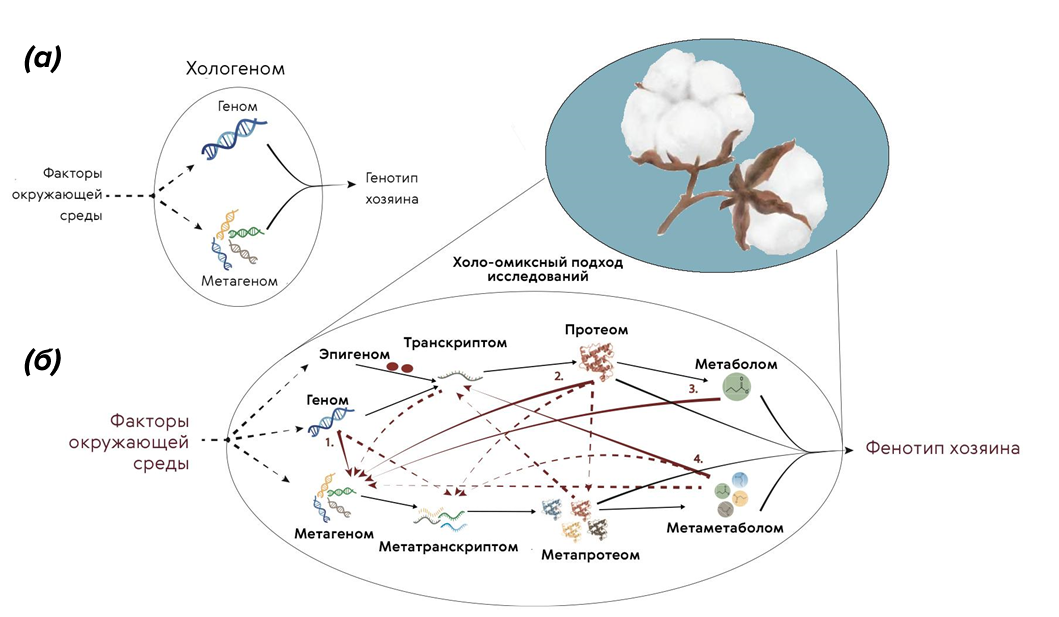
Рисунок 4. Что изучает хологеномика и холо-омика. Отметим, что холо-омика объединяет не только разные уровни информации о работе какого-то организма, но и информацию о разных генетических объектах. Она работает с мета-транскриптомами, мета-протеомами и мета-метаболомами.
иллюстрация Карины Илиопуло на основе [32]
Кроме того, что мультиомики активно используются для поиска биохимических связей между растениями и почвенным микробиомом (который везде разный), они также охватывают эпигеном и поддерживают экспосомный подход, то есть стремятся учесть все воздействия на организм извне, а значит, в перспективе могут много рассказать и о взаимодействиях холобионтов с окружающей средой. Разумеется, главной целью для исследователей здесь будет оставаться понимание, как все эти сложные взаимодействия отражаются на фенотипе главных объектов исследований — растений.
Конечно, пока холо-омика — даже не молодое, а только зарождающееся направление. У него всё еще много ограничений, включая дороговизну исследований и сложность интерпретации и воспроизводимости результатов. Тем не менее, уже сейчас его прикладное значение для отрасли очевидно, а в изучении почвенных микробных сообществ и их функциональных свойств ученые уже неплохо продвинулись (рис. 5) [33], [34].
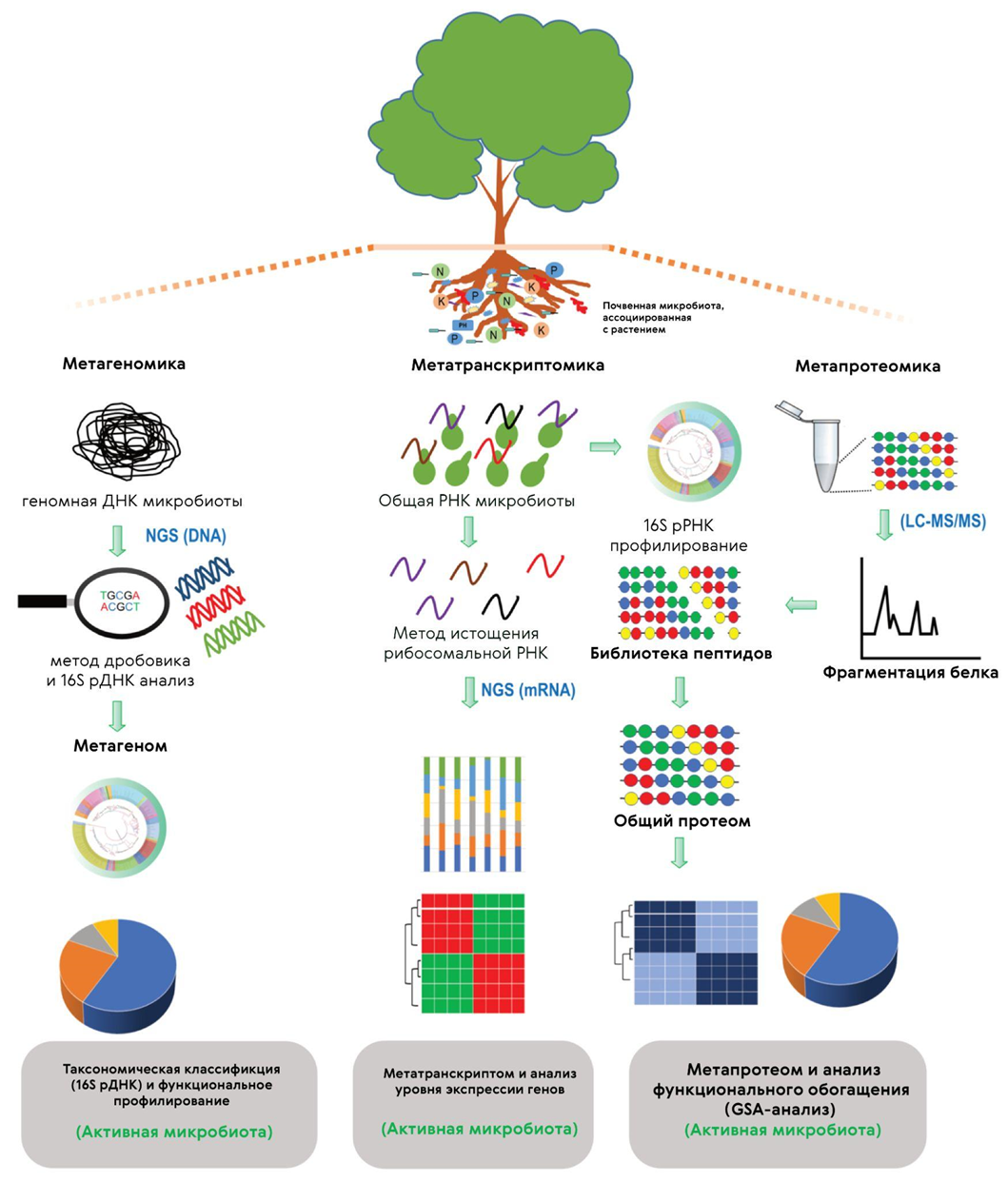
Рисунок 5. Мультиомиксные технологии успешно используются для исследований микробно-растительных взаимодействий. Разнообразные геномные (метагеномика) и постгеномные (метатранскриптомика и метапротеомика) подходы используются для лучшего понимания взаимодействий почвенной микробиоты и растений.
LC-MS/MS — жидкостная хроматография с масс-спектрометрией или масс-спектрометрия.
Метагеномный анализ почв показывает, что сегодня культивировать в лаборатории ученым удается лишь порядка 5% всех присутствующих в земле бактерий, а значит, многие почвенные микробы нам до сих пор неизвестны, а их функции не раскрыты [35]. С другой стороны, с помощью мультиомиксных технологий можно выявлять наиболее благоприятных микробных соседей для возделываемых культур, а после — обеспечивать растениям такое соседство, улучшая им жизнь и сокращая использование пестицидов, ведь почвенный микробиом защищает растения от патогенов и уберегает их от стресса (симбиотические микоризные грибы и бактерии в ходе эволюции приобрели устойчивость к засухе, повышенному содержанию в почве тяжелых металлов и засолению почв, а значит, могут поддерживать здоровье растений даже в стрессовых для них условиях [36]). Главный коммерческий продукт этого направления исследований — биоудобрения (рис. 6) [37].
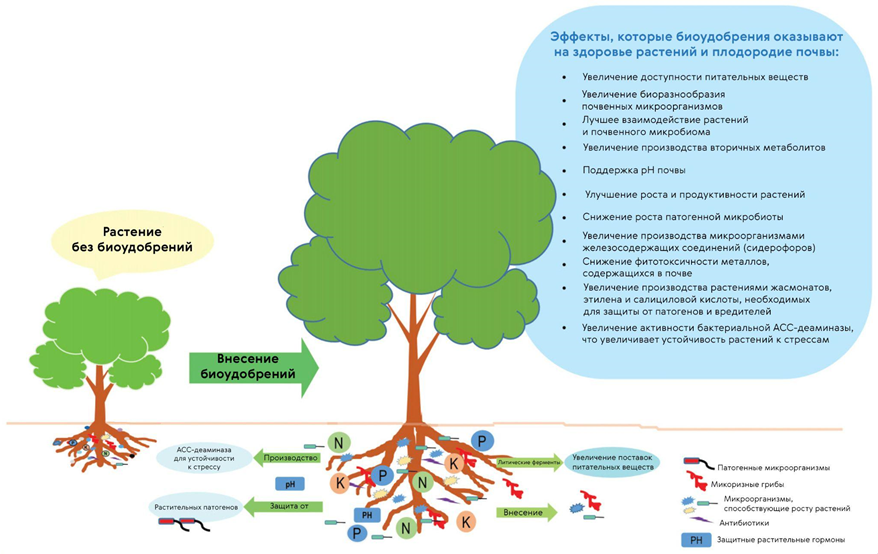
Рисунок 6. Как биоудобрения влияют на здоровье растений. Биоудобрения на базе микоризных грибов и микроорганизмов, влияющих на рост растений, способствуют лучшему снабжению и усвоению растениями питательных веществ, защищают растения от патогенов, повышают их устойчивость к стрессу.
N — азот, P — фосфор, K — калий.
Как выясняется, они, среди прочего, способны осуществлять биоремедиацию почв, что в настоящее время крайне актуально [38].
Использовать мультиомиксные данные о почвенных микробиомах для создания новых сортов растений гораздо сложнее, для этого их нужно объединять в многомерные биологические сети. Тем не менее, уже сейчас омиксные исследования помогают раскрывать состав микробных сообществ и определять роль различных генов, метаболитов, белков и нуклеиновых кислот, участвующих в растительно-микробных взаимодействиях. Эта информация в дальнейшем может использоваться для корректировки или усиления этих взаимодействий [39]. Например, секвенирование генома некоторых эндофитных бактерий (это бактерии, которые колонизируют растительные ткани) помогло узнать много нового об их свойствах, включая способность фиксировать азот, поглощать железо и образовывать биопленки. Результаты работ в этом направлении говорят о том, что симбиотический образ жизни этих микроорганизмов сделал их крайне приспособленными к существованию в среде с ограниченным количеством железа и даже наделил способностью изолировать этот металл от растительных патогенов [40].
Метаболомика также облегчает понимание того, как растения и микроорганизмы меняют свой метаболический профиль, реагируя друг на друга. Причем эти изменения затрагивают как вторичные метаболиты — например, фитоалексины и антимикробные вещества растений, — так и первичные — сахара, аминокислоты и органические кислоты. Идентификация метаболитов, которыми обмениваются корни растений и микоризные грибы, может помочь воздействовать на микробиомы извне и увеличивать устойчивость растений к различного рода стрессам [41], [42].
На модели арабидопсиса было показано, что важную роль для почвенных микробов играют корнеспецифичные терпеноиды, которые могут накапливаться в тканях растений в виде тритерпеновых гликозидов [43] и поддерживать формирование микробиоты, регулируя рост специфических корневых бактерий [44]. Кроме того, некоторые метаболиты (флавоноиды и молекулы, чувствительные к кворуму, то есть те, выработка которых изменяется в зависимости от плотности их микроскопического окружения) участвуют в регуляции симбиоза между растениями и отдельными микробами — ризобиями, арбускулярной микоризой и эктомикоризой.
Для идентификации, скрининга и количественного определения метаболитов существуют разные технологии и подходы, каждый из которых имеет свои преимущества и недостатки. Например, спектрофотометрия отличается быстротой и низкой стоимостью, а инфракрасная спектроскопия с Фурье-преобразованием (FTIR) позволяет ежедневно анализировать тысячи образцов различных метаболитов, не причиняя образцам никакого вреда [45], [46]. Аналогичным образом, анализ генома диазотрофной бактерии Klebsiella pneumonia показал ее способность бороться с вредными для растений активными формами кислорода путем экспрессии нескольких защитных ферментов, среди которых — супероксиддисмутаза, каталазы, пероксидазы и редуктазы [47], [48]. В 2020 году для лучшего понимания салицилового пути защиты растений был предложен мультиомиксный подход, включающий интеграцию метаболомных, геномных и волатиломных (относящихся ко всем выделяемым организмом летучим веществам) данных для фиксирования состояния стресса у растений [49]. Очевидно, более глубокое понимание взаимодействий между растениями и эндофитами может быть достигнуто путем связывания геномной информации с данными о фенотипе растений. В этом направлении активно начинают применяться метагеномный анализ [41] и постгеномные подходы — метатранскриптомика (анализ экспрессии мРНК), метапротеомика и метапротеогеномика. Так, метапротеогеномное исследование микробных сообществ филлосферы и ризосферы риса показало, что, несмотря на присутствие генов nifH в обеих микросредах, динитрогеназоредуктаза идентифицируется исключительно в ризосфере [50]. Если бы такие подходы использовались для множества возделываемых культур в рутинном порядке и затем применялись для инженерии почвенных микробиомов с помощью методов синтетической биологии, биоудобрения из года в год становились бы все эффективнее, конкурируя с агрохимией. А это бы означало кардинальные изменения в хозяйствовании.
В целом, стратегии мультиомики предлагают мощный арсенал для понимания сложных связей между растениями, их геномами и окружающей средой. Использование этих знаний в будущем поможет нам разрабатывать и внедрять новые устойчивые методы ведения сельского хозяйства, которые не только повысят производительность и эффективность использования природных ресурсов, но и будут минимально влиять на окружающую среду.
Мультиомики в животноводстве
Современные подходы к разведению домашнего скота разительно отличаются от тех, что использовались еще 50 лет назад. Если раньше методы отбора и разведения животных основывались на их фенотипических признаках (тогда генетические изменения накапливались крайне медленно, а оценить результат работы, например, качество туши, можно было только через длительный промежуток времени), то сегодня мы уже не можем представить себе селекцию без методов молекулярной биологии. При этом данных только о геноме животных порой становится недостаточно [54]. Мультиомиксные технологии, позволяющие искать закономерности в сложных системах, крайне перспективны как инструмент умной селекции животных [55]. Примером тут может служить выведение пород, устойчивых к различного вида стрессам [56]. В отрасли также очень остро стоят вопросы повышения производительности пород и улучшения их здоровья [57].
С появлением высокопроизводительных методов и сложных аналитических систем оценивать генетический потенциал животных стало гораздо проще. Большие данные геномики, транскриптомики, протеомики, метаболомики и феномики, а также характеристика самих животных (продолжительность их жизни, поведение и так далее) вместе дают возможность лучше понять механизмы, влияющие на эффективность производственных процессов.
Вот один из примеров. Несмотря на то, что идентифицированных локусов количественных признаков становится всё больше, нам известны далеко не все гены и мутации, вызывающие фенотипические изменения. Для выявления генов, ответственных за сложные признаки, такие как качество мяса, активно используется транскриптомика. Объединив транскриптомные профили нескольких тканей, можно идентифицировать предполагаемые механизмы, лежащие в основе дифференциальной регуляции тканей у эмбрионов крупного рогатого скота [58]. Данные эпигеномики, протеомики и метаболомики, в свою очередь, тоже находят свое применение. Так, мультиомиксный подход, включающий генотипирование, определение транскриптома и протеома, позволил составить молекулярный профиль для бесплодных и фертильных коров, что углубило знания природы фертильности млекопитающих [59]. В конечном счете, омиксные технологии помогают более точно оценивать племенную ценность, в том числе за счет обнаружения малых фенотипических изменений, таких как реакция на определенный корм, и проводить селекцию на ранней стадии жизни животных, ускоряя процесс получения пород с лучшими качествами (рис. 9) [60].
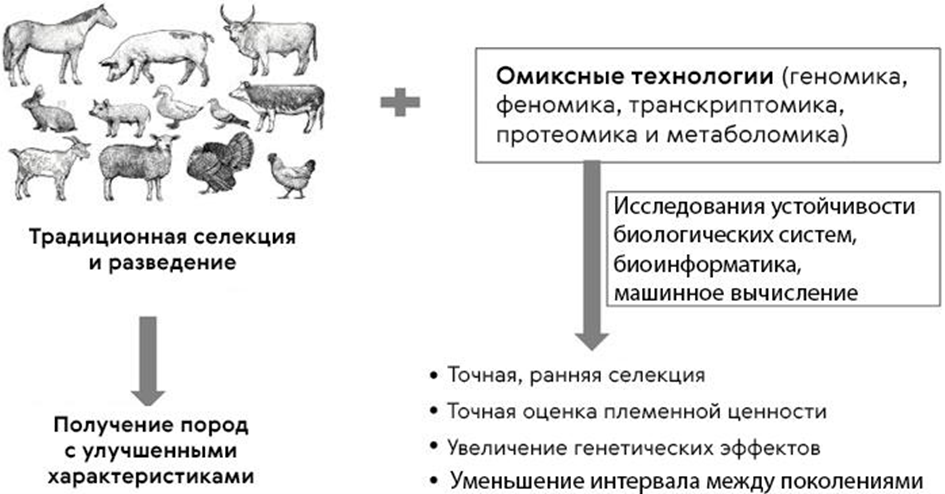
Рисунок 9. Как омиксные технологии помогают селекции животных.
Конечно, сложный анализ предполагает использование сложных методов биоинформатики и объемную статистическую работу (рис. 10), поэтому в настоящее время основная масса исследований ограничивается какой-то одной омикой. Специфика отрасли — большой размер популяций животных и огрехи в записи их фенотипических особенностей, что также тормозит выработку новых стратегий отбора качественного генетического материала.
Холо-омика, о которой мы уже говорили, в животноводстве востребована даже больше, чем в растениеводстве. Давно известно, что взаимосвязь между геномом животного-хозяина и микробиотой его кишечника играет ключевую роль в показателях продуктивности особи. Современные корма для животных и кормовые добавки (пробиотики, пребиотики, синбиотики) рутинно используются для модуляции микробиоты кишечника животных и улучшения их роста и общего здоровья. Кроме того, постоянно разрабатываются более экологичные формулы кормов, позволяющие снижать нагрузку отрасли на биосферу. Например, использование морских водорослей в кормах для коров помогает снижать выбросы метана [66]. Также производители кормов всё время находятся в поиске более эффективных ферментных добавок, призванных улучшать перевариваемость корма, а для их производства нужны микробные штаммы-продуценты — либо природные, либо рекомбинантные. Внедрению в промышленность и тех, и других обычно сопутствуют продолжительные исследования, время на которые может быть значительно сокращено с использованием больших данных о геноме, транскриптоме и протеоме.
Новые подходы к производству питания
Сфера питания сегодня развивается, следуя за трендами здорового образа жизни и устойчивого хозяйствования. Люди стремятся есть полезную еду, знать, из чего она сделана, и платить за продукты, производители которых заботятся об окружающей среде. Из года в год растет рынок эко-продуктов , разнообразнее становится ассортимент диетического питания, на полках супермаркетов всё чаще можно встретить вегетарианские и веганские альтернативы молока и мяса.
На «Биомолекуле» выходила статья о пестицидах, в которой речь шла и о том, как работает рынок биопродуктов (глава «Органические продукты для лучшей жизни») [67].
Поиск новых источников пищевого белка — это одно из крайне востребованных прикладных направлений биологических исследований. Основные источники такого белка сегодня — это растения (например, горох и соя), насекомые и одноклеточные организмы [68]. Да-да, белок микроскопических грибов и водорослей — ценный источник энергии и аминокислот, и его использование имеет свои преимущества. Во-первых, микроорганизмы можно выращивать на вторичном сырье, которое в другом случае нужно было бы утилизировать. Во-вторых, производство такого белка меньше влияет на биосферу, чем традиционное животноводство. Наконец, состав микробного белка можно регулировать, находя или создавая новые промышленные штаммы микроорганизмов-продуцентов. Доказательством того, что игра стоит свеч, выступают успешные стартапы — молоко PerfectDay из генетически модифицированных дрожжей [69] или грибное «мясо» от брендов Meati, MyBacon, Nature’s Fynd и Quorn.
Разумеется, работа с перспективными продуцентами подразумевает исследование их свойств, а иногда — вмешательство в их геном. И здесь нам на помощь снова приходят омики.
Еще один интересный сегмент пищевой промышленности — производство персонализированного питания и продукции для отдельных групп населения, например, диабетиков или спортсменов. Казалось бы, ничего особенного. Но что, если питание может быть действительно «персонализированным», то есть подобранным для каждого отдельного человека? Что вообще для этого нужно и как это может выглядеть? Фантазии есть куда разыграться. Это может быть и персональный пищевой компьютер, который позволит выращивать еду с заданными свойствами, регулируя условия произрастания пищевых культур, и специальный сканер для еды, с помощью которого можно будет определять состав и «полезность» тех или иных продуктов или блюд по фотографии. Ну и конечно, какая персонализация без согласования меню с твоей собственной ДНК? И в этом случае это не просто слова, такая инициатива уже есть! Британская компания DnaNudge предлагает клиентам браслеты, где будет храниться информация об их геноме, которая в дальнейшем поможет специальной программе давать персональные рекомендации по питанию, уходу за кожей и спорту (рис. 11). В основе технологии лежит анализ SNP, связанных с предрасположенностью к ряду заболеваний.
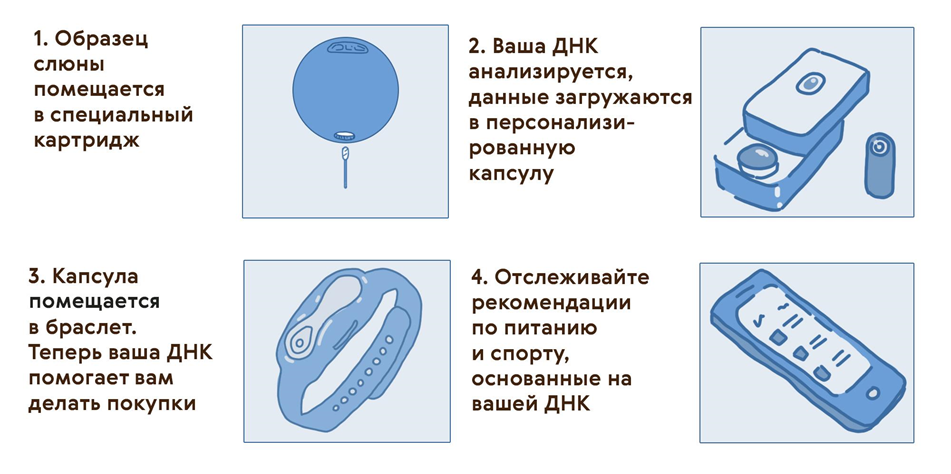
Рисунок 11. Как работают «покупки с ДНК» от компании DnaNudge. Сперва образец слюны с ДНК помещается в NudgeBox — портативный ПЦР-аппарат. Затем с помощью микрочипа с 72 ДНК-маркерами геном человека анализируется, после чего вся информация загружается в браслет.
иллюстрация Карины Илиопуло в соответствии с данными, публикуемыми производителем
Другое направление использования омиксных технологий — изучение пищевых аллергий, от которых сегодня страдает около 8% детей и 5% взрослых [70]. Мультиомиксный подход способен углубить знания о факторах риска аллергии, различных иммунных реакциях и метаболических изменениях, а также роли микробиома в развитии аутоиммунных заболеваний. В конечном счете, системные данные должны помочь вывести диагностику и лечение аллергий на качественно новый уровень.
Если говорить об исследовании ДНК, то GWAS сегодня используется для изучения генетических вариаций пищевых аллергий путем поиска локусов, ассоциированных с заболеванием. Многое о функционировании генома в разрезе аллергических реакций становится понятно благодаря транскриптомным исследованиям [71]. Эпигеномные исследования, с другой стороны, дают нам ценную информацию о том, как гены взаимодействуют с окружающей средой [72].
Так как причины развития аллергии довольно сложны, для их анализа нередко используют комбинации разных подходов: транскриптомики и эпигеномики [73], транскриптомики и метаболомики и так далее [74]. О том, как проходят омиксные исследования, которые применяют при изучении пищевых аллергий, показано на рисунке 12 [75].
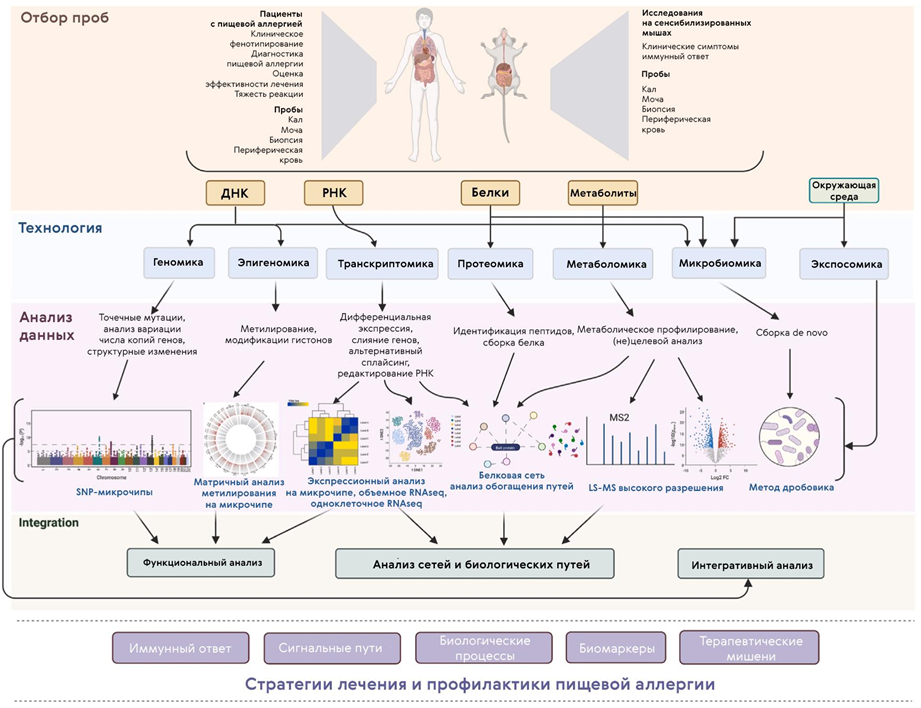
Рисунок 12. Омиксный анализ, применяемый для изучения пищевой аллергии. Эта комплексная работа включает сбор образцов и анализ данных, объединяя несколько омиксных подходов, включая геномику, эпигеномику, транскриптомику, протеомику, метаболомику, микробиомику и экспосомику (изучение факторов окружающей среды, воздействующих на регуляцию генов и развитие организмов).
SNP — однонуклеотидный полиморфизм, WES/WGS — полное секвенирование экзома или генома, ChIP-chip — иммунопреципитация хроматина в сочетании с микроматрицей ДНК, ChIP-seq — иммунопреципитация хроматина в сочетании с секвенированием, RNA-seq — РНК-секвенирование, LC-MS — жидкостная хроматография с масс-спектрометрией, GC-MS — газовая хроматография с масс-спектрометрией.
Используются мультиомики и в более приземленных сферах. Везде в пищевой промышленности, где речь идет о ферментации и микробных сообществах, есть место геномным и постгеномным исследованиям. Например, в виноделии микробы, живущие на виноградной лозе, на предприятиях и те, что вводятся искусственно перед началом стадии брожения, играют решающую роль в формировании аромата и вкуса готового напитка (главные микробы вина — это Saccharomyces cerevisiae и Oenococcus oeni). Изучение биоразнообразия диких спиртовых дрожжей и микробных популяций, участвующих в сбраживании сусла, несомненно, в будущем поспособствует лучшему управлению процессами создания вина. С помощью методов метагеномики и метабаркодирования ученые постоянно узнают о микробном мире что-то новое [76], [77]. Так, недавно было обнаружено, что грибковые сообщества, переносимые дрозофилами, на разных винодельнях имеют разный состав, а значит, микробиом виноделен отчасти формируется с участием плодовых мушек [78]. Изучение транскриптома, протеома и метаболома виноматериалов или других ферментируемых продуктов (сыров, комбучи, пива) на разных стадиях производства тоже может быть крайне полезно. В частности, метапротеомика может как помогать оценивать функциональность генов, которые ответственны за экспрессию отдельных белков, так и исследовать белок-белковые взаимодействия в субстратах. Кроме того, наряду с метаболомикой [79] она может использоваться для исследования вкусоактивных веществ в продуктах питания [80].
Комбинирование нескольких омик в проектах для пищевой промышленности также встречается всё чаще. Метагеномные и метаболомные данные объединяют, чтобы найти микроорганизмы, влияющие на вкус чая Пуэр, который он приобретает в ходе ферментации [81], или колбасы, проходящей через процесс созревания [82]. А дополнив данные о геноме транскриптомом, можно узнать больше о микробном сообществе, формирующемся при созревании сыра [83].
«Зеленая» экономика и ее причуды: растения и животные как продуценты
Тренд устойчивого развития в последние годы сказался на развитии многих рынков, включая химическую промышленность. То, что раньше получали с помощью химического синтеза, сегодня предпочтительно производить, используя «зеленые» технологии. Отсюда растущее количество проектов, где продуцентами самых разных веществ выступают растения и животные (пусть и в меньшей степени). Фармакологическая отрасль также активно использует «живые фабрики» для производства сложных молекул.
Для того, чтобы создать собственную «биофабрику», достаточно получить генетически модифицированное растение, которое будет производить целевые рекомбинантные продукты прямо у себя в листьях, плодах или корнях. Диапазон используемых видов тут не маленький — это и табак, и зерновые, и бобовые, и овощи.
Создание трансгенных растений-фабрик может происходить посредством разных методов [84]. И даже с учетом ограничений (сложностей с одобрением новых фармпрепаратов, различий в посттрансляционном гликозилировании растительных и человеческих белков, низких темпов производства) такое «молекулярное сельское хозяйство» вполне может стать рутиной для производителей вакцин, антител, терапевтических белков [85] и множества других веществ, включая биопластики, в ближайшем будущем.
В биореакторах можно выращивать клетки растений, мхи и ряску. Из растительных клеток, например, получают противораковый препарат паклитаксел и терапевтические ферменты [90].
Крупнотоннажное биотех-производство также с успехом использует водоросли. Они уже стали классикой в сфере получения удобрений и растительных подкормок, средств для очистки сточных вод, бета-каротина и полиненасыщенных жирных кислот. Самая популярная промышленная микроводоросль — это Chlorella.
Генетически модифицированные млекопитающие — задачка посложнее. Тем не менее, они всерьез рассматриваются как источники терапевтических белков, которые будут накапливаться в биологических жидкостях (крови, молоке). Так, в 2014 году FDA одобрило рекомбинантный белок Ruconest, полученный из трансгенного кроличьего молока. Его выделяют с целью применения в терапии наследственного ангионевротического отека.
Мультиомиксные подходы в разрезе создания новых экспрессионных систем, как растительных, так и животных, позволяют больше узнавать об их метаболических путях и корректировать их с целью более эффективной наработки ценных продуктов. Они могут быть также полезны при выборе тех или иных сортов и пород [87] под конкретные производственные задачи. Кроме того, изучение генома наряду с транскриптомом и протеомом может помочь при подборе оптимальных условий культивирования растений-продуцентов [91].
Заключение
Мультиомиксный подход, который объединяет в себе геномные данные разных уровней, а также информацию о признаках живых организмов, прямо сейчас меняет сельское хозяйство и связанные с ним производства, делая их беспрецедентно более эффективными, быстрыми и точными. Новые знания, которые удается вычленить из потоков обрушивающейся на биологов информации, будь то данные о геноме целой популяции почвенных микроорганизмов или связь множества генов в разных участках ДНК со сложными признаками у животных и растений, позволяют предсказывать результаты селекционной работы и гораздо шире смотреть на объекты исследований. Мы уже не удивляемся мысли о том, что растение неотделимо от почвенного микробиома, а человек — от кишечного, более того, мы активно вмешиваемся в эти взаимосвязи, извлекая из них пользу, а растения и микроорганизмы все чаще воспринимаются биотехнологами как универсальные фабрики для производства самых разных молекул. И это только начало пути. Когда ДНК встречается с трактором, рождается новая реальность, где человек берет в свои руки саму суть живого и управляет ею по своему усмотрению, чтобы накормить весь мир, сохранив его для будущих поколений.
Литература
- 7 000 000 000;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Yaodong Yang, Mumtaz Ali Saand, Liyun Huang, Walid Badawy Abdelaal, Jun Zhang, et. al.. (2021). Applications of Multi-Omics Technologies for Crop Improvement. Front. Plant Sci.. 12;
- Sidra Aslam, Muhammad Aamer Mehmood, Mehboob-ur Rahman, Fatima Noor, Niaz Ahmad. (2022). Bioinformatics-assisted multiomics approaches to improve the agronomic traits in cotton. Bioinformatics in Agriculture. 233-251;
- Manas Ranjan Sahoo, Ponnam Naresh, Meenu Kumari, Gobinda Chandra Acharya. (2022). Omics in leafy vegetables: Genomics, transcriptomics, proteomics, metabolomics, and multiomics approaches. Omics in Horticultural Crops. 281-302;
- Federico Scossa, Saleh Alseekh, Alisdair R. Fernie. (2021). Integrating multi-omics data for crop improvement. Journal of Plant Physiology. 257, 153352;
- Minxuan Li, Sen Cai, Shijun You, Yuanyuan Liu. (2023). Multiomics techniques for plant secondary metabolism engineering: Pathways to shape the bioeconomy. Genomics and the Global Bioeconomy. 205-252;
- Yunbi Xu, Xingping Zhang, Huihui Li, Hongjian Zheng, Jianan Zhang, et. al.. (2022). Smart breeding driven by big data, artificial intelligence, and integrated genomic-enviromic prediction. Molecular Plant. 15, 1664-1695;
- Dinesh Chandra, Pankaj Bhatt. (2023). Multiomics strategies for alleviation of abiotic stresses in plants. Unravelling Plant-Microbe Synergy. 1-35;
- Hong-Kyu Choi. (2019). Translational genomics and multi-omics integrated approaches as a useful strategy for crop breeding. Genes Genom. 41, 133-146;
- Mehanathan Muthamilarasan, Nagendra Kumar Singh, Manoj Prasad. (2019). Multi-omics approaches for strategic improvement of stress tolerance in underutilized crop species: A climate change perspective. Advances in Genetics. 1-38;
- Liumei Jian, Jianbing Yan, Jie Liu. (2022). De Novo Domestication in the Multi-Omics Era. Plant and Cell Physiology. 63, 1592-1606;
- Факты и легенды о мультиомиксных технологиях;
- Qing Li, Jianbing Yan. (2020). Sustainable agriculture in the era of omics: knowledge-driven crop breeding. Genome Biol. 21;
- От ГМО к растениям будущего. Всё самое интересное о сложной работе современного селекционера;
- Hongwei Zhang, Xi Wang, Qingchun Pan, Pei Li, Yunjun Liu, et. al.. (2019). QTG-Seq Accelerates QTL Fine Mapping through QTL Partitioning and Whole-Genome Sequencing of Bulked Segregant Samples. Molecular Plant. 12, 426-437;
- Shan Li, Yonghang Tian, Kun Wu, Yafeng Ye, Jianping Yu, et. al.. (2018). Modulating plant growth–metabolism coordination for sustainable agriculture. Nature. 560, 595-600;
- Kun Wu, Shuansuo Wang, Wenzhen Song, Jianqing Zhang, Yun Wang, et. al.. (2020). Enhanced sustainable green revolution yield via nitrogen-responsive chromatin modulation in rice. Science. 367;
- Ning Yang, Jie Liu, Qiang Gao, Songtao Gui, Lu Chen, et. al.. (2019). Genome assembly of a tropical maize inbred line provides insights into structural variation and crop improvement. Nat Genet. 51, 1052-1059;
- Hai-Jun Liu, Liumei Jian, Jieting Xu, Qinghua Zhang, Maolin Zhang, et. al.. (2020). High-Throughput CRISPR/Cas9 Mutagenesis Streamlines Trait Gene Identification in Maize. The Plant Cell. 32, 1397-1413;
- Simon C. Groen, Irina Ćalić, Zoé Joly-Lopez, Adrian E. Platts, Jae Young Choi, et. al.. (2020). The strength and pattern of natural selection on gene expression in rice. Nature. 578, 572-576;
- Cheng Luo, Alisdair R. Fernie, Jianbing Yan. (2020). Single-Cell Genomics and Epigenomics: Technologies and Applications in Plants. Trends in Plant Science. 25, 1030-1040;
- Alisdair R. Fernie, Jianbing Yan. (2019). De Novo Domestication: An Alternative Route toward New Crops for the Future. Molecular Plant. 12, 615-631;
- Choon-Tak Kwon, Jung Heo, Zachary H. Lemmon, Yossi Capua, Samuel F. Hutton, et. al.. (2020). Rapid customization of Solanaceae fruit crops for urban agriculture. Nat Biotechnol. 38, 182-188;
- Wolfram Weckwerth. (2011). Green systems biology — From single genomes, proteomes and metabolomes to ecosystems research and biotechnology. Journal of Proteomics. 75, 284-305;
- Maojun Wang, Pengcheng Wang, Lili Tu, Sitao Zhu, Lin Zhang, et. al.. (2016). Multi-omics maps of cotton fibre reveal epigenetic basis for staged single-cell differentiation. Nucleic Acids Res. 44, 4067-4079;
- Jie Luo. (2015). Metabolite-based genome-wide association studies in plants. Current Opinion in Plant Biology. 24, 31-38;
- Fumio Matsuda, Ryo Nakabayashi, Zhigang Yang, Yozo Okazaki, Jun‐ichi Yonemaru, et. al.. (2015). Metabolome‐genome‐wide association study dissects genetic architecture for generating natural variation in rice secondary metabolism. The Plant Journal. 81, 13-23;
- Sudisha Jogaiah, Sharathchandra Ramsandra Govind, Lam-Son Phan Tran. (2013). Systems biology-based approaches toward understanding drought tolerance in food crops. Critical Reviews in Biotechnology. 33, 23-39;
- Dibyendu Talukdar, Andrey Sinjushin. (2015). Cytogenomics and Mutagenomics in Plant Functional Biology and Breeding. PlantOmics: The Omics of Plant Science. 113-156;
- Wanneng Yang, Zilong Guo, Chenglong Huang, Lingfeng Duan, Guoxing Chen, et. al.. (2014). Combining high-throughput phenotyping and genome-wide association studies to reveal natural genetic variation in rice. Nat Commun. 5;
- Lasse Nyholm, Adam Koziol, Sofia Marcos, Amanda Bolt Botnen, Ostaizka Aizpurua, et. al.. (2020). Holo-Omics: Integrated Host-Microbiota Multi-omics for Basic and Applied Biological Research. iScience. 23, 101414;
- Muzaffer Arıkan, Thilo Muth. (2023). Integrated multi-omics analyses of microbial communities: a review of the current state and future directions. Mol. Omics. 19, 607-623;
- Umesh Kumar, Subhisha Raj, Arathi Sreenikethanam, Rahul Maddheshiya, Seema Kumari, et. al.. (2023). Multi-Omics Approaches in Plant–Microbe Interactions Hold Enormous Promise for Sustainable Agriculture. Agronomy. 13, 1804;
- Raíssa Mesquita Braga, Manuella Nóbrega Dourado, Welington Luiz Araújo. (2016). Microbial interactions: ecology in a molecular perspective. Brazilian Journal of Microbiology. 47, 86-98;
- Shobhit Raj VIMAL, Jay Shankar SINGH, Naveen Kumar ARORA, Surendra SINGH. (2017). Soil-Plant-Microbe Interactions in Stressed Agriculture Management: A Review. Pedosphere. 27, 177-192;
- Bernard R. Glick Beneficial Plant-Bacterial Interactions — Springer International Publishing, 2015;
- Muneer A. Malla, Anamika Dubey, Shweta Yadav, Ashwani Kumar, Abeer Hashem, Elsayed Fathi Abd Allah. (2018). Understanding and Designing the Strategies for the Microbe-Mediated Remediation of Environmental Contaminants Using Omics Approaches. Front. Microbiol.. 9;
- Nicholas O. Igiehon, Olubukola O. Babalola. (2018). Below-ground-above-ground Plant-microbial Interactions: Focusing on Soybean, Rhizobacteria and Mycorrhizal Fungi. TOMICROJ. 12, 261-279;
- A. Sessitsch, P. Hardoim, J. Döring, A. Weilharter, A. Krause, et. al.. (2012). Functional Characteristics of an Endophyte Community Colonizing Rice Roots as Revealed by Metagenomic Analysis. MPMI. 25, 28-36;
- Shawn M. Higdon, Tania Pozzo, Nguyet Kong, Bihua C. Huang, Mai Lee Yang, et. al.. (2020). Genomic characterization of a diazotrophic microbiota associated with maize aerial root mucilage. PLoS ONE. 15, e0239677;
- N SCHAUER, A FERNIE. (2006). Plant metabolomics: towards biological function and mechanism. Trends in Plant Science. 11, 508-516;
- Alberto Pascale, Silvia Proietti, Iakovos S. Pantelides, Ioannis A. Stringlis. (2020). Modulation of the Root Microbiome by Plant Molecules: The Basis for Targeted Disease Suppression and Plant Growth Promotion. Front. Plant Sci.. 10;
- Pengchao Wang, Ben Niu. (2019). Plant specialized metabolites modulate root microbiomes. Sci. China Life Sci.. 62, 1111-1113;
- Nozomu Sakurai. (2022). Recent applications of metabolomics in plant breeding. Breed. Sci.. 72, 56-65;
- David I. Ellis, Royston Goodacre. (2006). Metabolic fingerprinting in disease diagnosis: biomedical applications of infrared and Raman spectroscopy. Analyst. 131, 875;
- Claudia Knief, Nathanaël Delmotte, Samuel Chaffron, Manuel Stark, Gerd Innerebner, et. al.. (2012). Metaproteogenomic analysis of microbial communities in the phyllosphere and rhizosphere of rice. ISME J. 6, 1378-1390;
- Luz K. Medina-Cordoba, Aroon T. Chande, Lavanya Rishishwar, Leonard W. Mayer, Lina C. Valderrama-Aguirre, et. al.. (2021). Genomic characterization and computational phenotyping of nitrogen-fixing bacteria isolated from Colombian sugarcane fields. Sci Rep. 11;
- Sharifa G. Crandall, Kaitlin M. Gold, María del Mar Jiménez-Gasco, Camila C. Filgueiras, Denis S. Willett. (2020). A multi-omics approach to solving problems in plant disease ecology. PLoS ONE. 15, e0237975;
- Jing Ke, Bing Wang, Yasuo Yoshikuni. (2021). Microbiome Engineering: Synthetic Biology of Plant-Associated Microbiomes in Sustainable Agriculture. Trends in Biotechnology. 39, 244-261;
- James J. Stapleton. (2000). Soil solarization in various agricultural production systems. Crop Protection. 19, 837-841;
- Gamliel A., Katan J. Soil Solarization: Theory and Practice. Amer Phytopathological Society, 2017. — 266 p.;
- Yasunori Ichihashi, Yasuhiro Date, Amiu Shino, Tomoko Shimizu, Arisa Shibata, et. al.. (2020). Multi-omics analysis on an agroecosystem reveals the significant role of organic nitrogen to increase agricultural crop yield. Proc. Natl. Acad. Sci. U.S.A.. 117, 14552-14560;
- Ya-lan YANG, Rong ZHOU, Kui LI. (2017). Future livestock breeding: Precision breeding based on multi-omics information and population personalization. Journal of Integrative Agriculture. 16, 2784-2791;
- Lucas Lima Verardo, Luiz F. Brito, Nuno Carolino, Ana Fabrícia Braga Magalhães. (2023). Editorial: Omics applied to livestock genetics. Front. Genet.. 14;
- Claudia Kasper, David Ribeiro, André M. de Almeida, Catherine Larzul, Laurence Liaubet, Eduard Murani. (2020). Omics Application in Animal Science—A Special Emphasis on Stress Response and Damaging Behaviour in Pigs. Genes. 11, 920;
- Dinesh Bhattarai, Xing Chen, Zia ur Rehman, Xingjie Hao, Farman Ullah, et. al.. (2017). Association of MAP4K4 gene single nucleotide polymorphism with mastitis and milk traits in Chinese Holstein cattle. Journal of Dairy Research. 84, 76-79;
- Wellison Jarles Da Silva Diniz, Alison K Ward. (2021). 282 Multi-omics Approaches to Improve Animal Production. Journal of Animal Science. 99, 20-21;
- Mackenzie A. Marrella, Fernando H. Biase. (2023). A multi-omics analysis identifies molecular features associated with fertility in heifers (Bos taurus). Sci Rep. 13;
- Dibyendu Chakraborty, Neelesh Sharma, Savleen Kour, Simrinder Singh Sodhi, Mukesh Kumar Gupta, et. al.. (2022). Applications of Omics Technology for Livestock Selection and Improvement. Front. Genet.. 13;
- Lakshmi K. Matukumalli, Cynthia T. Lawley, Robert D. Schnabel, Jeremy F. Taylor, Mark F. Allan, et. al.. (2009). Development and Characterization of a High Density SNP Genotyping Assay for Cattle. PLoS ONE. 4, e5350;
- M. Muñoz, R. Bozzi, J. García-Casco, Y. Núñez, A. Ribani, et. al.. (2019). Genomic diversity, linkage disequilibrium and selection signatures in European local pig breeds assessed with a high density SNP chip. Sci Rep. 9;
- The Bovine Genome Sequencing and Analysis Consortium, Christine G. Elsik, Ross L. Tellam, Kim C. Worley, Richard A. Gibbs, et. al.. (2009). The Genome Sequence of Taurine Cattle: A Window to Ruminant Biology and Evolution. Science. 324, 522-528;
- Ling-Yun Chang, Sajjad Toghiani, Samuel E. Aggrey, Romdhane Rekaya. (2019). Increasing accuracy of genomic selection in presence of high density marker panels through the prioritization of relevant polymorphisms. BMC Genet. 20;
- Pattarapol Sumreddee, El Hamidi Hay, Sajjad Toghiani, Andrew Roberts, Samuel E. Aggrey, Romdhane Rekaya. (2021). Grid search approach to discriminate between old and recent inbreeding using phenotypic, pedigree and genomic information. BMC Genomics. 22;
- Lorenna Machado, Marie Magnusson, Nicholas A. Paul, Rocky de Nys, Nigel Tomkins. (2014). Effects of Marine and Freshwater Macroalgae on In Vitro Total Gas and Methane Production. PLoS ONE. 9, e85289;
- Контроль за пестицидами. Полиция в белых халатах;
- Протеиновые инвестиции;
- Intel индустрии питания. Как двое вчерашних студентов меняют наше представление о еде с помощью ГМО-дрожжей и молока без коров;
- Scott H. Sicherer, Hugh A. Sampson. (2018). Food allergy: A review and update on epidemiology, pathogenesis, diagnosis, prevention, and management. Journal of Allergy and Clinical Immunology. 141, 41-58;
- C. T. Watson, A. T. Cohain, R. S. Griffin, Y. Chun, A. Grishin, et. al.. (2017). Integrative transcriptomic analysis reveals key drivers of acute peanut allergic reactions. Nat Commun. 8;
- Xiumei Hong, Christine Ladd-Acosta, Ke Hao, Ben Sherwood, Hongkai Ji, et. al.. (2016). Epigenome-wide association study links site-specific DNA methylation changes with cow's milk allergy. Journal of Allergy and Clinical Immunology. 138, 908-911.e9;
- Samira Imran, Melanie R Neeland, Jennifer Koplin, Shyamali Dharmage, Mimi LK Tang, et. al.. (2021). Epigenetic programming underpins B‐cell dysfunction in peanut and multi‐food allergy. Clin & Trans Imm. 10;
- Elena Goleva, Agustin Calatroni, Petra LeBeau, Evgeny Berdyshev, Patricia Taylor, et. al.. (2020). Skin tape proteomics identifies pathways associated with transepidermal water loss and allergen polysensitization in atopic dermatitis. Journal of Allergy and Clinical Immunology. 146, 1367-1378;
- Qiaozhi Zhang, Yanbo Wang, Linglin Fu. (2022). Application of (multi-)omics approaches for advancing food allergy: an updated review. Current Opinion in Food Science. 46, 100854;
- Peter R. Sternes, Danna Lee, Dariusz R. Kutyna, Anthony R. Borneman. (2017). A combined meta-barcoding and shotgun metagenomic analysis of spontaneous wine fermentation. GigaScience. 6;
- Marie Lisandra Zepeda-Mendoza, Nathalia Kruse Edwards, Mikkel Gulmann Madsen, Martin Abel-Kistrup, Lara Puetz, et. al. Influence ofOenococcus oeniandBrettanomyces bruxellensison Aged Wine Microbial Taxonomic and Functional Profiles — Cold Spring Harbor Laboratory;
- Allison S. Quan, Michael B. Eisen. (2018). The ecology of the Drosophila-yeast mutualism in wineries. PLoS ONE. 13, e0196440;
- D. Cozzolino. (2016). Metabolomics in Grape and Wine: Definition, Current Status and Future Prospects. Food Anal. Methods. 9, 2986-2997;
- Karin Sebald, Andreas Dunkel, Johannes Schäfer, Jörg Hinrichs, Thomas Hofmann. (2018). Sensoproteomics: A New Approach for the Identification of Taste-Active Peptides in Fermented Foods. J. Agric. Food Chem.. 66, 11092-11104;
- Zhongyuan Li, Cuixia Feng, Xuegang Luo, Huiling Yao, Dechun Zhang, Tongcun Zhang. (2018). Revealing the influence of microbiota on the quality of Pu-erh tea during fermentation process by shotgun metagenomic and metabolomic analysis. Food Microbiology. 76, 405-415;
- Ilario Ferrocino, Alberto Bellio, Manuela Giordano, Guerrino Macori, Angelo Romano, et. al.. (2018). Shotgun Metagenomics and Volatilome Profile of the Microbiota of Fermented Sausages. Appl Environ Microbiol. 84;
- Eric Dugat-Bony, Cécile Straub, Aurélie Teissandier, Djamila Onésime, Valentin Loux, et. al.. (2015). Overview of a Surface-Ripened Cheese Community Functioning by Meta-Omics Analyses. PLoS ONE. 10, e0124360;
- Juho Lee, Seon-Kyeong Lee, Jong-Sug Park, Kyeong-Ryeol Lee. (2023). Plant-made pharmaceuticals: exploring studies for the production of recombinant protein in plants and assessing challenges ahead. Plant Biotechnol Rep;
- Melina Laguia-Becher, Zurima Zaldúa, Weijie Xu, Patricia Laura Marconi, William Velander, María Alejandra Alvarez. (2019). Co-expressing Turnip Crinkle Virus-coat protein with the serine protease α-thrombin precursor (pFIIa) in Nicotiana benthamiana Domin. In Vitro Cell.Dev.Biol.-Plant. 55, 88-98;
- Oscar K. K. Bedzo, Lalitha D. Gottumukkala, Giuseppe Lo Sasso, Kacper Kaminski, Walter Schlage, et. al.. (2023). Process development for efficient pectin extraction from tobacco residues and its characterisation. Biomass Conv. Bioref.;
- Csaba Laszlo, Kacper Kaminski, Haifeng Guan, Maria Fatarova, Jianbing Wei, et. al.. (2022). Fractionation and Extraction Optimization of Potentially Valuable Compounds and Their Profiling in Six Varieties of Two Nicotiana Species. Molecules. 27, 8105;
- Kacper Piotr Kaminski, Lucien Bovet, Helene Laparra, Gerhard Lang, Damien De Palo, et. al.. (2020). Alkaloid chemophenetics and transcriptomics of the Nicotiana genus. Phytochemistry. 177, 112424;
- Hartmut Foerster, James N D Battey, Nicolas Sierro, Nikolai V Ivanov, Lukas A Mueller. (2020). Metabolic networks of the Nicotiana genus in the spotlight: content, progress and outlook. Briefings in Bioinformatics;
- Jeffrey L Fox. (2012). First plant-made biologic approved. Nat Biotechnol. 30, 472-472;
- Michela Cecchin, Jovan Simicevic, Louise Chaput, Manuel Hernandez Gil, Laura Girolomoni, et. al.. (2023). Acclimation strategies of the green alga Chlorella vulgaris to different light regimes revealed by physiological and comparative proteomic analyses. Journal of Experimental Botany. 74, 4540-4558.






