
Факты и легенды о мультиомиксных технологиях
01 сентября 2023
Факты и легенды о мультиомиксных технологиях
- 1951
- 0
- 11
Мультиомиксные технологии похожи на котел, в котором варится всё: большие данные, самые современные методы исследований и различные области науки. В результате становится возможным сделать новые открытия и лучше понять, что же происходит в живых организмах и вокруг них.
Рисунок в оригинальном разрешении.
иллюстрация Карины Илиопуло
-
Автор
-
Редакторы
-
Рецензент
-
Иллюстратор
Мультиомиксные (МО) технологии оперируют огромными массивами экспериментальных и биоинформатических данных с целью найти скрытые взаимосвязи между биологическими процессами на самых разных уровнях. Например, оценить влияние эпигенетических модификаций на клинические симптомы болезней или даже проследить пути развития нервной системы в живом организме. В первой статье спецпроекта по мультиомиксным технологиям мы разбираемся, что же это такое, и почему их всё чаще упоминают в обзорах и статьях. Чем мультиомиксный подход отличается от «обычного» омиксного? И вытесняют ли МО-технологии классические методы и подходы молекулярной биологии или просто их дополняют?
Мультиомиксные технологии
На «Биомолекуле» уже разбирались методы и технологии молекулярной биологии, в том числе протеомные и геномные. В связи с технологическим прогрессом всё чаще применяют их комбинацию, причем к ним добавляются и другие «омики». В спецпроекте «Мультиомиксные технологии» мы расскажем не столько о самих методах, сколько об их применении в самых разных областях — фундаментальной науке, медицине, экологии, сельском хозяйстве. Мы рассмотрим, как калейдоскоп множества методов позволяет лучше увидеть общую картину, ответить на интересующие ученых вопросы и продвинуться в решении практических задач.
Партнер спецпроекта — аффилированные компании «Филип Моррис Интернэшнл» в России, ФМИ (PMI Affiliates in Russia).
Жизнь современных ученых уже не та, что вели их предшественники 100 и более лет назад. Приходится признать, что всё, что «лежало на поверхности» и могло быть обнаружено относительно простыми методами, уже открыто. Яблоко уже упало на голову Ньютону, антибиотики найдены Флемингом на заросшей чашке Петри, а структуру ДНК открыли Крик и Уотсон.
Конечно, наука не стоит на месте: с момента открытия структуры ДНК прошло 70 лет, и сегодня генетический тест доступен почти каждому; мы научились редактировать геномы, разрабатывать новые лекарства, ориентируясь на молекулярную мишень, анализировать огромные данные для персонализации медицины. Но подобные достижения требуют других методов работы: наука стала индустрией, а не увлечением избранных визионеров, в ней работает много людей, выделяются бурные потоки денег, а результаты научных исследований оцениваются в том числе через «крутизну» публикаций и призму практического применения.
Исследования в области естественных наук сейчас — это во многом работа с так называемыми «большими данными», когда полученные результаты совмещаются с уже известными базами данных с целью поиска новых закономерностей. Почти все современные проекты затрагивают несколько областей: биологи, химики и медики работают вместе с математиками, биоинформатиками и физиками (рис. 1) [1].
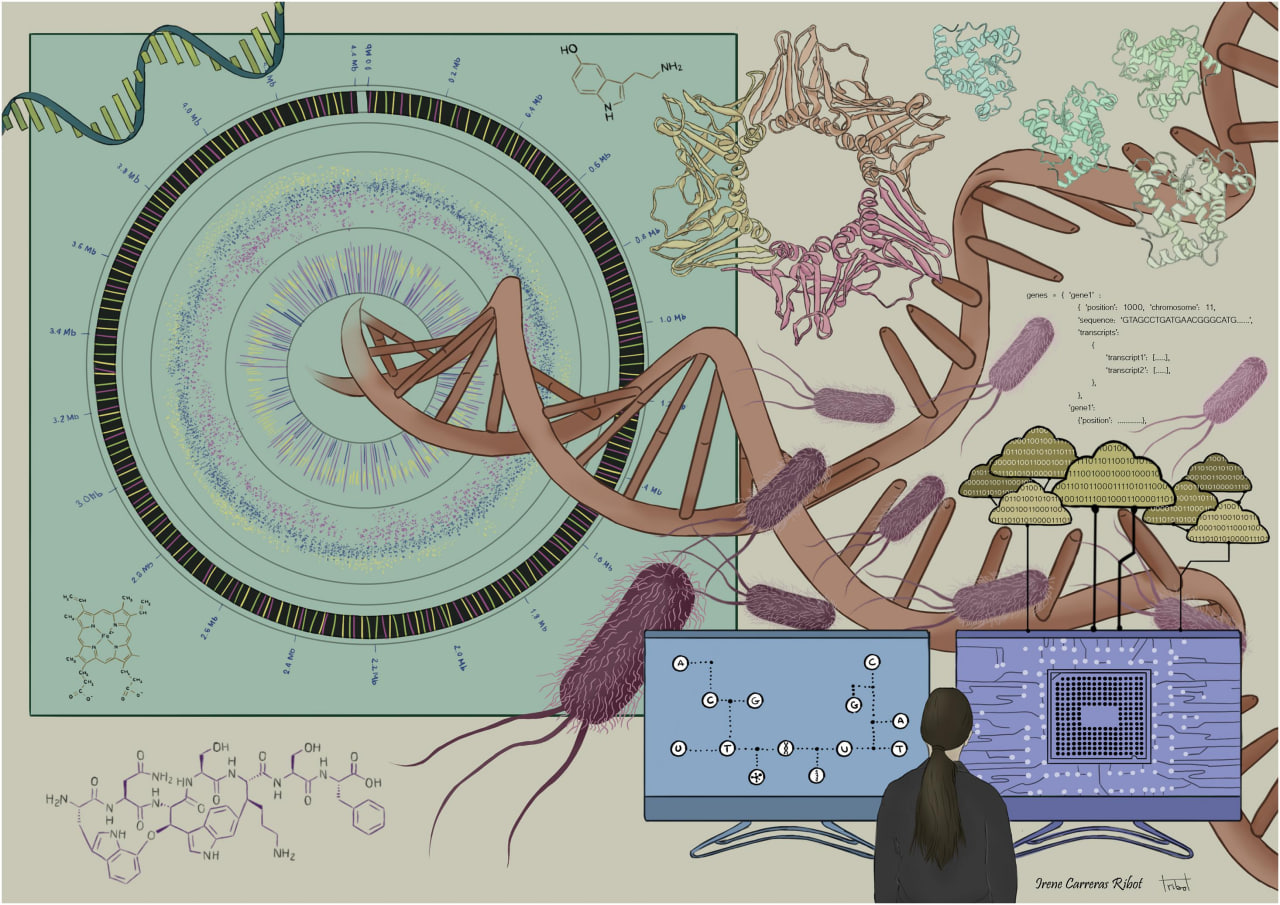
Рисунок 1. Метафора современного подхода к исследованиям как к комбинации разных наук и подходов: биология, химия, физика и медицина замешиваются на высокопроизводительных экспериментальных методах и приправляются хитроумными вычислительными моделями.
Омиксные и мультиомиксные технологии, о которых пойдет речь в этом спецпроекте, — это не конкретные методы. Это скорее современная парадигма экспериментальной работы и научных открытий, основанная на получении большого количества данных для последующего поиска взаимосвязей. Развитие мультиомики напрямую связано с технологиями: высокопроизводительными и автоматизированными биохимическими методами анализа (секвенирование, масс-спектрометрия, цитометрия и другие), программным обеспечением, базами данных и искусственным интеллектом.
Давайте разбираться с этим подробнее.
Что же такое «омики» и зачем их объединять?
Любой, даже не связанный с биологией читатель, слышал про генóм, а может быть, даже задумывался о том, чтобы сделать генетический тест. Генóм, транскриптóм, протеóм, микробиóм — все эти термины объединяет греческий суффикс «-ом», который означает «имеющий природу» и используется для обозначения совокупности чего-либо. Например, геном человека — это совокупность всего генетического материала (то есть молекул ДНК) организма. Транскриптом — совокупность всех молекул РНК, которые синтезируются в клетке, органе или ткани. Микробиом — совокупность всех микробов, населяющих организм человека.
По этой аналогии, -омика — это наука, которая изучает совокупности чего-либо (и уже в силу этого оперирует большими массивами данных). Например, геномика изучает совокупность генов организма, их структуру и функции, переходя от последовательного изучения конкретных генов к изучению их полного (или почти полного) массива. В каком-то смысле геномику можно считать «генетикой наоборот». Подход генетики — выбрать интересующий признак, после чего искать кодирующий его ген (или гены). Подход геномики — секвенировать геном или его фрагменты, а уже потом разбираться, что они кодируют. Проект «Геном человека» [2], успешно завершенный в 2000 году и ставший первым предвестником широкой доступности генетических тестов, прекрасно иллюстрирует этот подход: несмотря на то, что геном расшифрован более 20 лет назад, функции многих конкретных генов до сих пор известны не до конца. И это не уникальная ситуация: в любой омике поиск закономерностей может продолжаться сильно дольше экспериментальной работы, требуемой для получения данных.
Первая статья об омиксных технологиях — «Омики — эпоха большой биологии» [3] — вышла на «Биомолекуле» 10 лет назад и до сих пор не теряет своей актуальности.
Целью «омиксного» исследования становится изучение не отдельных объектов, как это было раньше, а совокупности всех объектов этого класса. Современное высокопроизводительное оборудование позволяет быстро делать огромное количество однотипных измерений (например, современный анализатор крови работает с десятками образцов, а автоматическая станция может подготовить почти сотню проб без участия человека), получая данные как бы «про запас», чтобы потом заняться их изучением и сопоставлением с уже известными базами данных, поиском закономерностей и взаимосвязей.
Подробнее о методах, применяемых в геномике и протеомике, а также успехах в их автоматизации мы говорили в спецпроекте «12 биологических методов в картинках» и в статье «Автоматизация омиксных технологий» [4].
Вполне логичным — в рамках этого общего тренда и на фоне стремительного развития вычислительных методов — выглядит желание объединять несколько омик. Например, мы могли бы получить больше интересных результатов, если бы искали взаимосвязи изменений в геноме, транскриптоме и протеоме; и не одного организма, а, например, в сотне проб от разных пациентов. Именно комбинация нескольких омик лежит в основе появления такого термина, как мультиомика. Но перед тем, как обсуждать плюсы и минусы мультиомиксных исследований, будет не лишним разобраться, какие бывают омики — чтобы лучше понимать, что именно мы можем комбинировать для получения новых данных.
Какие бывают «омики» и что они изучают?
В таблице 1 мы постарались собрать наиболее известные омики со страниц современных научных журналов. Начиная от (эпи)геномики, транскриптомики и протеомики, которые у всех (ну, как минимум, у читателей «Биомолекулы») на слуху уже с десяток лет, и заканчивая более редкими — такими, как коннектомика или экспосомика.
Коннектомика картирует и анализирует связи между нервными клетками организма — возможно, чтобы понять в итоге, как же работают нервная система и мозг. Построить детальный атлас такого предельно сложного объекта, как человеческий мозг, пока не получается, но наука движется к этому — начиная, как водится, с модельного объекта: горячо любимого биологами за свою простоту червя Caenorhabditis elegans [5].
В 2021 году при помощи электронной микроскопии была сделана уже не первая детальная реконструкция мозга нескольких особей этого червя [6], породившая модель созревания мозга — с учетом перестраивания сенсорных и моторных путей (рис. 2 и видео 1 и 2). Первые шаги в этом направлении были сделаны еще в 1980 году [7] — кстати, тогда же и появился термин коннектом. Столь горячий интерес ученых к изучению мозга червя объясняется крайне скромным числом нейронов: у самца червя их всего 385, а поэтому возникающую в них коннектомику изучить сравнительно несложно. Кроме того, «функционал» червя также весьма ограничен, а потому полученные данные реально перенести в червя-робота — этим занимался проект OpenWorm.
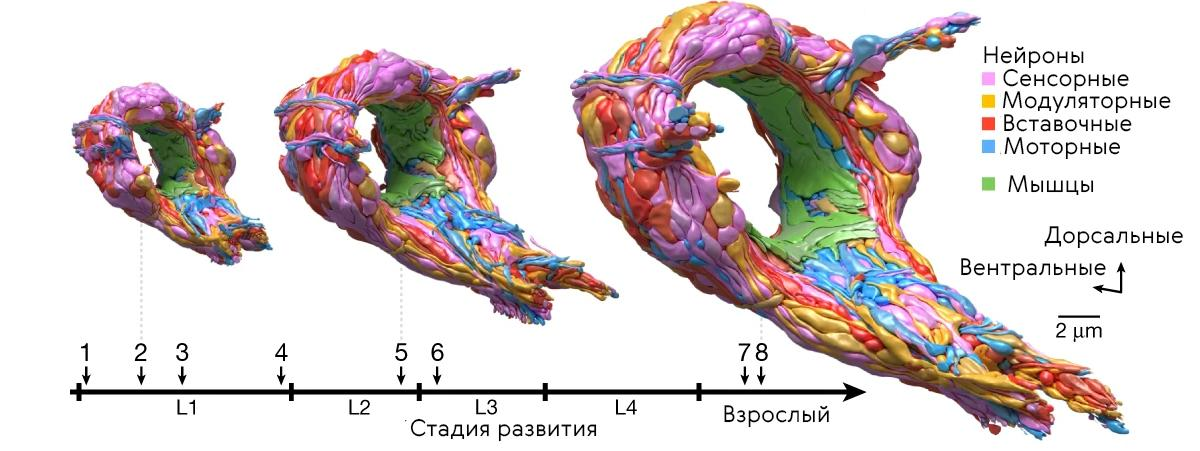
Рисунок 2. Нервная система червя C. elegans меняется по мере развития и созревания его поведения. На рисунке изображена 3D-модель нервной системы, изготовленная по электронной микроскопии серийных срезов мозга (см. подробнее на видео 1) восьми особей Caenorhabditis elegans на постнатальных стадиях (стадии взросления L1—L4 и полностью взрослая особь). Общая геометрия мозга сохраняется от рождения до зрелого возраста, но на этом постоянном каркасе возникают существенные изменения в синаптических связях. Сравнение коннектомов в процессе созревания демонстрирует перестройку сенсорных и моторных путей, а также повышение модульности мозга. Для визуализации и построения коннектома в каждой точке развития исследователи измеряли длину, форму и положение каждого нервного и мышечного волокна, а также картировали каждый физический контакт между всеми нейронами и мышцами и каждый синапс между нейронами, мышцами и глией.
Видео 1. Построение модели мозга взрослого червя C. elegans с последующим выделением отдельных типов нейронов и синапсов (реконструкция сделана с использованием данных электронной микроскопии). Различия в расположении нейронов по мере созревания мозга представлены на видео 2.
видео из статьи Nature
Видео 2. Расположение отдельных классов нейронов. На видео показано развитие некоторых нейронов по ходу взросления — меняется их размер и, в некоторых случаях, форма.
видео из статьи Nature
Разумеется, конечная цель коннектомики — изучить работу мозга человека. Подробно прокартировав все его соединения, можно было бы понять, как именно связи и сигналы влияют на когнитивные и эмоциональные способности и что идет не так при болезнях. Но связи нейронов между собой столь многочисленны, что уже при попытке в 2019 году составить коннектом малька рыбки данио-рерио (всего-то 2000 нейронов) исследователи были вынуждены разделить его на кластеры и изучать их по частям [8]. Мозг человека же несравненно сложнее: хотя точное число синапсов неизвестно, оно может превышать сто триллионов (1014) — для сравнения, в средней галактике типа нашего Млечного пути число звезд, по разным оценкам, составляет 109–1012 (но во всей Вселенной, конечно, много больше: 1022–1024, хотя точно это неизвестно, как и число синапсов). Ввиду такого гигантского количества синаптических связей, изучать, видимо, возможно только отдельные фрагменты мозга, а не весь его целиком. По крайней мере, с учетом современного развития технологий.
Например, существует проект по коннектому сетчатки глаза, принять участие в котором может любой желающий: он реализован в форме игры, запущенной в результате кооперации между Массачусетским технологическом институтом (MIT) и Институтом медицинских исследований Общества Макса Планка. И всё же, говоря о таких частичных моделях, важно помнить, что коннектомика стремится описать не столько отдельную функциональную зону (сетчатку или моторную кору), сколько взаимосвязи между ними.
Приведем пример еще одной диковинки, выходящей за рамки молекулярной биологии: экспосомика изучает воздействия окружающей среды на здоровье и болезни. Изучаемые параметры могут включать аспекты естественной среды (например, качество воздуха) и «техногенных» особенностей, таких как тип жилья или оснащенность рабочего места. Факторы такого рода могут влиять на биологические реакции организма — например, повышать уровень стресса или влиять на экспрессию генов, что, в свою очередь, повлечет изменение микробиома или эпигенома. Экспосомику обычно связывают с другими омиками (например, метаболомикой), чтобы отследить взаимосвязь внешних условий с функциональными изменениями организма — скажем, с повышением вероятности развития хронического заболевания [9].
| Направление | Задачи | Наиболее часто применяемые технологии |
|---|---|---|
| Геномика | Изучение информации, закодированной в геноме организма, включая структуру и функции ДНК, ее картирование и редактирование. | |
| Эпигеномика | Исследование механизмов контроля генов клеткой с помощью негенетических модификаций генома: метилирования ДНК, модификаций гистонов и др. | |
| Транскриптомика | Изучение транскриптома (полного набора РНК-транскриптов) и его реакций в ответ на разные факторы. | NGS: секвенирование полного транскриптома и мРНК, направленное секвенирование РНК. |
| Протеомика [14] | Анализ белков в масштабе их совокупности (протеома) для инвентаризации и поиска изменений под воздействием различных стимулов. |
|
| Метаболомика | Описание совокупности небольших молекул-метаболитов, которые можно найти в клетке, ткани или целом организме. Они важны для понимания физиологии организма, диагностики и лечения заболеваний (могут быть их маркерами). | |
| Липидомика | Изучает липиды, их классификацию и идентификацию, а также взаимодействия между ними. | Масс-спектрометрия, в том числе трехквадрупольная, ВЭЖХ—МС, масс-спектрометрическая визуализация. |
| Коннектомика | Полное описание и картирование структуры связей в нервной системе организма. |
|
| Микробиомика | Изучает микробиом — совокупность всех микробов, населяющих организм человека, а также взаимодействия между микроорганизмами и организмом. |
|
| Феномика | Изучает феном — описание фенотипа, всех его физических и поведенческих характеристик. Ключевой целью является связь различий в фенотипе с изменениями в геноме. | Математические методы анализа, комбинация геномики и данных о фенотипах (например, пациентские реестры в случае медицины или данные компьютерного зрения в сельском хозяйстве). |
| Интерактомика | Стык биоинформатики и биохимии, изучающий белок-белковые взаимодействия и их последствия для организма. |
|
| Токсомика | Изучает процессы, связанные с проявлением токсичности различных веществ для человека. | Компьютерный и статистический анализ проведенных доклинических исследований, фармакокинетики, исследований токсичности и токсикокинетики, в сочетании с данными метаболомных и транскриптомных анализов. |
| Интегромика | Термин «интегром» почти не используется сейчас, но в 2012 году был придуман для объединения информации по всем -омам организма — своеобразный прототип мультиомиксного подхода. | Совокупный компьютерный анализ всех омиксных данных, известных для организма. |
| Когнитивная (нейративная) геномика | Исследует гены и некодирующие последовательности, необходимые для развития нервной системы и функционирования головного мозга. |
|
| Фармакогеномика [17] | Описывает влияние генома конкретного человека на эффект принимаемых этим человеком лекарств. |
|
| Виромика [9] | Изучение вирусных таксонов — сложная задача, учитывая небольшой геном и субмикроскопические размеры самих вирусов, а также крайне высокую изменчивость генома. | NGS: метагеномное и метатранскриптомное. |
| Экспосомика [9] | Влияние окружающей среды на здоровье человека и других животных. | Методы анализа воздуха, воды, почв, продуктов питания, освещенности в комбинации с метаболомикой и клиническими симптомами. |
| Киномика (фосфопротеомика) | Изучение всей совокупности киназ и фосфатаз в организме, их субстратов и сигналинга, в первую очередь, с точки зрения поиска новых мишеней [18]. | Методы геномики и протеомики, данные об активности модуляторов киназ in vitro и in vivo. |
Об этих омиках и соответствующих им -омах мы писали подробно в статье «Омики — эпоха большой биологии» [3].
Некоторые омиксные технологии более развиты и автоматизированы, чем другие. Например, методы, используемые в протеомике [14] — высокопроизводительном анализе белков, — по сравнению с геномными исследованиями [10] находятся на «предыдущей ступени». Это связано как с более разнообразным составом белков (более 20 аминокислот с различными модификациями вместо четырех нуклеотидов в молекуле ДНК или РНК), так и с более низкой чувствительностью методов из-за невозможности амплификации . В основном, в протеомике используются такие методы, как масс-спектрометрия, масс-цитометрия и, в последнее время, основанное на NGS определение белков, например, объемное секвенирование эпитопов и нуклеиновых кислот.
Подробнее о методах, применяемых в геномике и протеомике, а также успехах в их автоматизации, мы говорили в спецпроекте «12 биологических методов в картинках» и в статье «Автоматизация омиксных технологий» [4].
Геномные исследования базируются на секвенировании нового поколения (NGS) и его модификациях. В геномике чаще всего используются полногеномное и экзомное секвенирование генома, а также секвенирование на микрочипах и нанопоровое секвенирование [11]. Эпигеномные исследования (читайте наш спецпроект «Эпигенетика») уже невозможно представить без бисульфитного и хроматин-иммунопреципитационногое секвенирования, ATAC-seq (полногеномная оценка степени открытости хроматина), HiC/3C (определение конформации хромосом), а также секвенирования на микрочипах для исследования метилирования [12]. Транскриптомика использует NGS для секвенирования полного транскриптома и мРНК, а также направленного секвенирования РНК. Без секвенирования не было бы и некоторых других омик. Например, вирусная метагеномика обычно опирается на данные метагеномного или метатранскриптомного секвенирования.
Развитие омиксных исследований «упирается» не только в квалификацию исследователей, но и в развитие технологий, увеличение скорости и снижение стоимости анализа, а также в вычислительные мощности для обработки и хранения данных.
Результаты омкиксных исследований часто публикуются в виде открытых баз данных. Существует «Атлас генома рака», протеомные базы данных ProteomeXchange и PRIDE, база данных метаболома человека, проект «Токсом человека» и другие аналогичные проекты. Для чего они нужны? Упомянутый выше «Атлас генома рака» систематизирует и каталогизирует всю доступную информацию о генетических мутациях, ассоциированных с развитием онкологических заболеваний. Благодаря этим данным можно определять подтипы опухолей, классифицировать пациентов и подбирать наиболее эффективное лечение, основываясь на известных данных о выживаемости пациентов. Существуют работы, рассматривающие эффективность разных аналитических подходов для данных из «Атласа генома рака» [19].
Исследователи могут сопоставить полученные ими экспериментальные результаты с уже известными фактами из выбранной базы данных, либо запланировать исследование, полностью состоящее из нового анализа известных данных для ответа на тот или иной вопрос. База данных метаболома человека содержит и систематизирует уже известные метаболиты, для которых обозначены не только химические свойства, но и то, с какими белками или нуклеиновыми кислотами это вещество может взаимодействовать и какое значение оно имеет в клинической практике (связь с заболеваниями или лекарствами). Но, как мы обсудим в главе «Сложности МО исследований», объем и наполнение разных баз может существенно отличаться, в зависимости от того, к какому направлению омиксных исследований они относятся.
Мультиомиксные исследования: новое направление или комбинация хорошо известного старого?
Выше мы обсудили базы данных, в которых хранятся результаты омиксных исследований. Но если не соединить эти базы, то мы упустим самое важное — взаимосвязи. Каким образом взаимодействуют между собой белки в клетке? К каким результатам приводят модификации в генах? Почему одна модификация может быть незаметна для организма, но комбинация с другой — летальна? Несвязанные данные не дают нам целостной картины, а заставляют описывать один большой объект (организм) как в присказке про трех слепых и слона: каждый ощупал свою часть (хвост, ногу, хобот), но никто из них не понимал, как выглядит животное в целом.
Именно стремление объединить данные нескольких омик, чтобы «увидеть слона» (полную картину взаимосвязей в организме), и привело к возникновению понятия мультиомиксных (МО) исследований: сюда относят работы, опирающиеся на две и более «омики». Совокупные данные геномных, эпигеномных, транскриптомных, протеомных и метаболомных исследований могли бы позволить создавать почти полные модели всех процессов в организме на молекулярном уровне. МО-исследования могут объединять информацию разных уровней: молекулярного, клеточного (проточная цитометрия или микроскопия) и целого организма (рис. 3). Часто новые экспериментальные данные объединяют с тем, что уже собрано в базах данных других омиксных исследований.
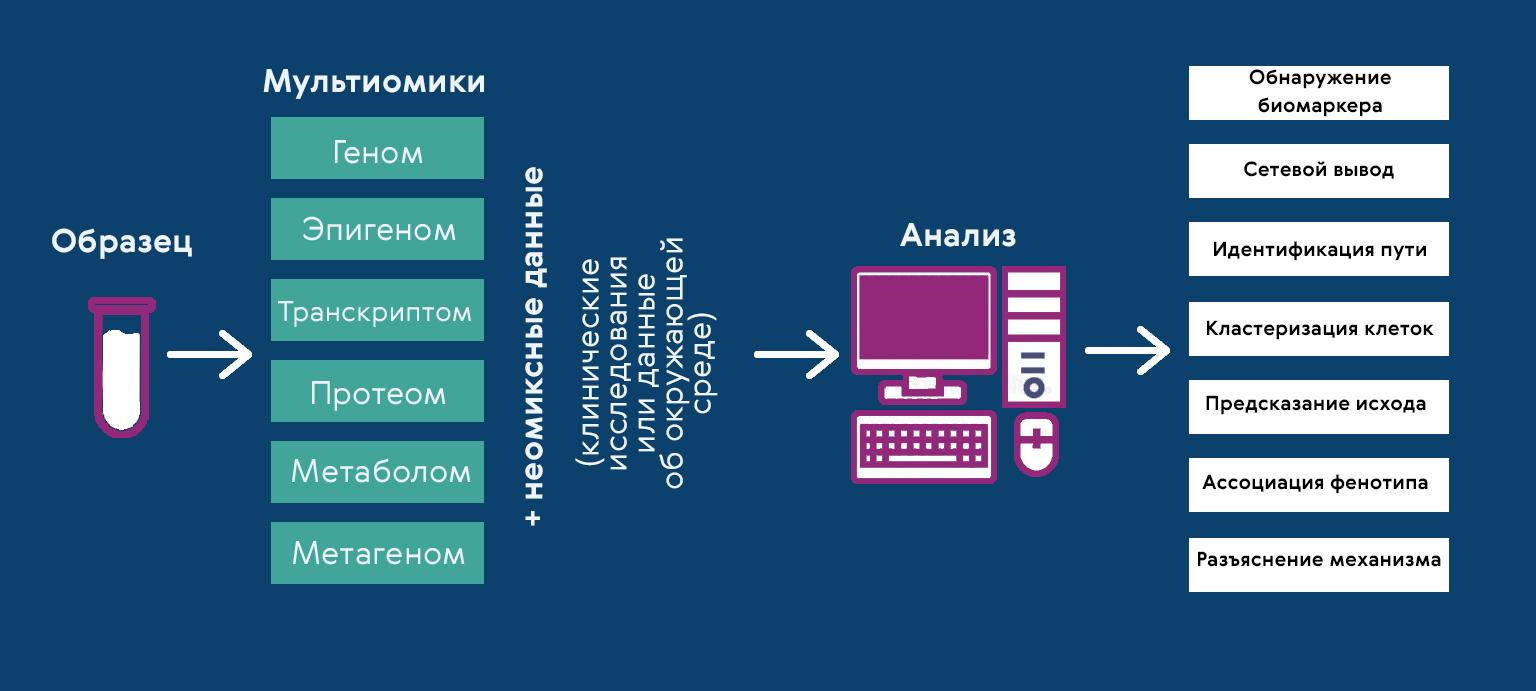
Рисунок 3. Мультиомиксные технологии и их использование. Мультиомиксные данные получают при помощи высокопроизводительных методов — например, секвенированием нуклеиновых кислот (оно позволяет получать геномные и эпигеномные — по ДНК, — а также транскриптомные — по РНК — данные) или масс-спектрометрией (дает информацию о протеоме). Присовокупив к этому и без того уже весьма богатому массиву неомиксные данные (скажем, клиническую информацию о пациентах), можно приступить к анализу — который, по сути, не знает границ. Он может выявить новые биомаркеры и сигнальные пути, объединить клетки в группы по свойствам, понять механизмы интересующих процессов, предсказать состояние здоровья или исходы лечения болезни пациента. В медицине мультиомиксные исследования позволяют объединить персонализированные подходы с системной биологией.
иллюстрация Карины Илиопуло
Рассмотрим несколько мультиомиксных исследований в разных сферах, чтобы приблизиться к пониманию их плюсов и минусов. К примеру, известно, что на фенотип сложного организма в течение всей его жизни оказывает сильное влияние микробиом — здоровье и адаптация к окружающей среде зависят от отношений с собственной микробиотой. Объединение данных (мета)геномики, (мета)транскриптомики, (мета)протеомики и (мета)метаболомики хозяина и его микробов должно способствовать пониманию, можем ли мы использовать микробиом для диагностики или мониторинга эффективности лечения. Впрочем, на данный момент в большинстве работ на эту тему авторы сталкиваются со сложностями сбора и анализа данных, которые пока не позволяют сделать грандиозных выводов [20], [21]. Но для описания исследований, объединяющих омиксные данные хозяина и его микробиома, уже придуман новый термин — холо-омика.
Метагеном — набор генов всех микроорганизмов, находящихся в образце, выделенном из среды (воды, почвы или образца, выделенного из организма). Аналогично для транскриптомики, протеомики и метаболомики, приставка «мета-» говорит о том, что анализируется совокупность транскриптов, белков или метаболитов для всех микроорганизмов, которые есть в образце.
МО-исследования находят применение в поиске терапевтических свойств растительных препаратов. С 2013 года было известно, что анатабин (алкалоид растений семейства пасленовых — например, зеленых помидоров, перца и табака) у мышей улучшает течение болезни Альцгеймера и тиреоидита; может быть эффективен при лечении рассеянного склероза и розацеа; и даже уменьшать хронические боли в суставах. Между тем, несмотря на попытки связать его действие с активностью факторов транскрипции или уровнями противовоспалительных цитокинов, молекулярные механизмы ясны не были.
Проведенное командой научно-исследовательского центра PMI Science исследование, в котором изучали концентрационную зависимость эффектов анатабина по транскриптому, протеому и фосфопротеому, показало, что механизм действия основан на активации транскрипционного фактора NRF2 и сигнального пути протеин-киназы MAPK [22]. Но хотя МО-подход позволил выявить эту связь, авторы исследования называют вопрос наличия других противовоспалительных механизмов анатабина открытым.
В биомедицине мультиомиксные подходы могут спасать жизни, повышая эффективность лечения. Серьезная проблема онкологии — гетерогенность раковых опухолей, состоящих из смеси нормальных и злокачественных клеток, отличающихся морфологически и функционально. Раковые клетки могут быть гетерогенны фенотипически, генетически и эпигенетически — а потому единый план лечения, скорее всего, не будет эффективен: оно может убить 99% раковых клеток, но оставшийся вариабельный 1% не только выживет, но и даст начало новой опухоли, устойчивой к первоначальному лечению. Мультиомиксные подходы могли бы помочь в анализе микроокружения опухолевых клеток и выборе наиболее подходящего лечения. На данный момент чаще всего применяют комбинации геномного и транскриптомного анализа на базе «Атласа генома рака» [19]. В обзоре 2020 года сравнивается эффективность разных методов и алгоритмов на примере 58 исследований для классификации подтипов рака и анализа выживаемости пациентов [19].
Метаболомные, геномные и клинические данные помогают персонализировать медицину не только в сфере онкологии, но и при лечении хронических заболеваний [23]. Пациенты с хронической болезнью почек нередко страдают от некорректного лечения из-за гетерогенности симптомов и причин данного заболевания [24]. Правильный диагноз ставят им слишком поздно. Но что, если при постановке диагноза опираться на объединенные данные, включающие в себя разные параметры, от анализов мочи и крови до биопсии?
Аналогичные исследования проводятся и для других многофакторных заболеваний, таких как остеоартрит, волчанка, диабет 2 типа и болезнь Альцгеймера (рис. 4) [25]. Почему эти заболевания особенно интересны? Мы до сих пор не понимаем до конца, что их запускает, как именно они развиваются, и можем лечить их во многом симптоматически. Например, GWAS (полногеномный поиск ассоциаций) выявил 150 локусов генетического риска развития остеоартрита. Но какие гены и генетические варианты отвечают за начало этой болезни и ее прогрессирование? Почему остеоартрит, который раньше считался заболеванием суставного хряща, на самом деле поражает все ткани суставов? На эти вопросы и пытаются ответить мультиомиксные исследования, занимающиеся поиском механизмов развития заболевания на разных уровнях. На практике это означает, что исследователи выбирают наиболее типичные ткани — для остеоартрита это хрящ и синовиальная оболочка сустава — и делают различные омиксные исследования (например, для хряща анализ метилирования ДНК, транскриптомный и протеомный анализы, оценка степени открытости хроматина). После чего совмещают эти данные между собой и с GWAS и пытаются найти закономерности. В одной из работ авторы, объединив геномные, транскриптомные и протеомные данные, полученные на 115 пациентах с остеоартритом, смогли даже предложить новые молекулярные мишени для терапии этого заболевания [26].
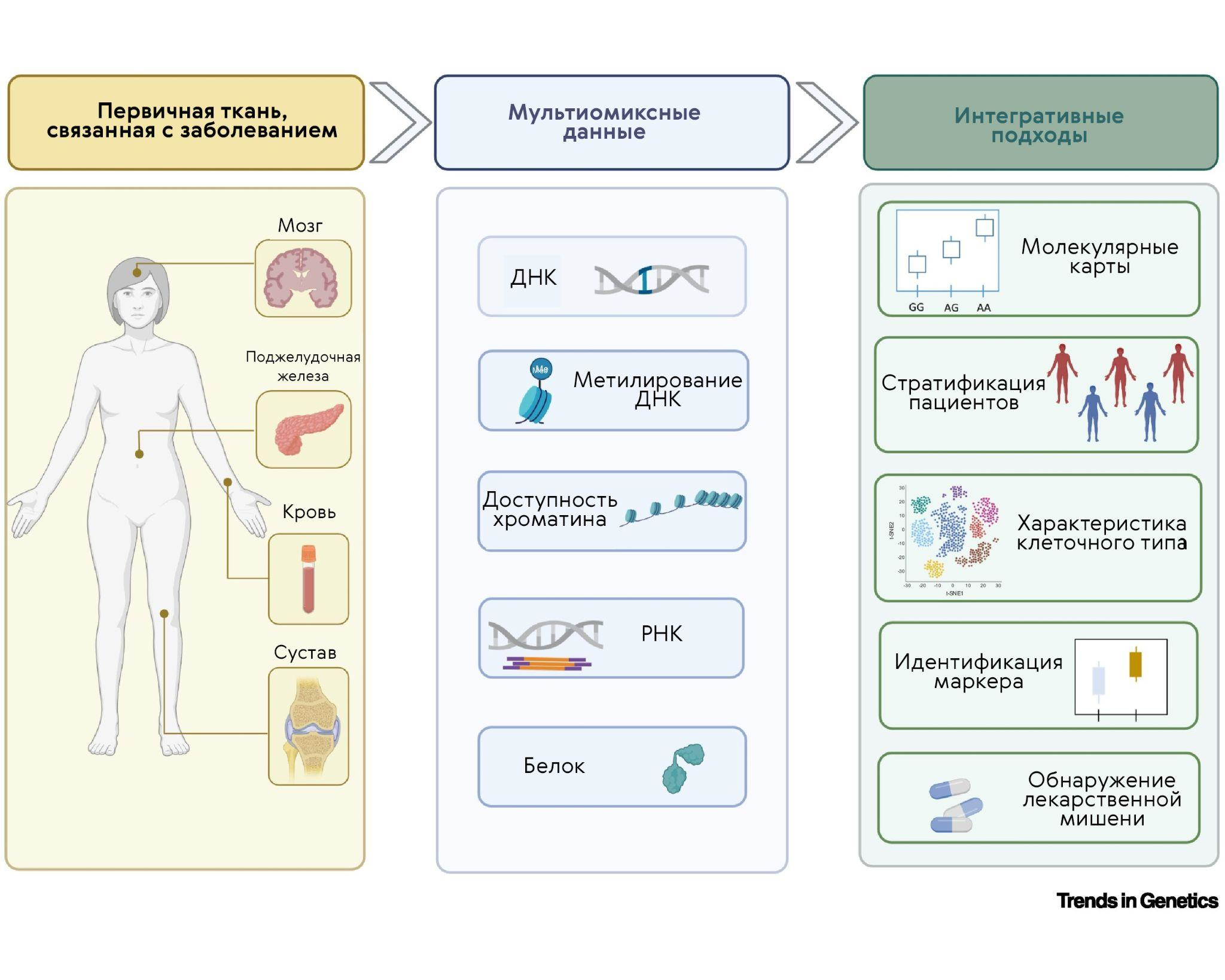
Рисунок 4. Пример мультиомиксных подходов и результатов в биомедицине: у пациентов берут образцы тканей органов, пораженных заболеванием, и делают несколько омиксных анализов. Это, в зависимости от дизайна исследования, может быть секвенирование ДНК или РНК, исследование хроматина, протеомные исследования, и так далее. Полученные данные после объединения и анализа могут привести к созданию молекулярной карты заболевания, разделения пациентов на группы для выбора наиболее эффективного лечения, идентификации новых диагностических маркеров или мишеней для лекарств.
Не следует думать, будто бы МО-подходы нашли свое применение только в биологии и медицине. Вовсе нет, более того — они всё более незаменимы в биоочистке [27]: например, с их помощью можно методами геномного редактирования попытаться создать эффективные микроводоросли и цианобактерии для переработки углекислого газа или очистки сточных вод [27].
Встречаются и работы, посвященные использованию МО-подходов в «зеленой» энергетике [27], развитии сельскохозяйственных культур [28], изучении устойчивости растений [29], аллергенности [30] различных продуктов питания... Об этих сферах МО исследований мы расскажем в следующих статьях спецпроекта. А пока перейдем к небольшой ложке дегтя, которая пока что существует в любой статье или обзоре, посвященных МО-анализу.
Сложности мультиомиксных исследований
Читая научные публикации про мультиомику, несложно заметить, что, несмотря на захватывающие дух перспективы и огромную экспериментальную и аналитическую работу, исследования в основном концентрируются на проблеме анализа данных, либо на том, что полученные данные сложно интерпретировать однозначно.
В этом и заключается одна из основных сложностей МО-исследований (кроме стоимости используемых методов, конечно, которая с годами всё же уменьшается). Планирование и проведение мультиомиксных исследований предполагает чрезвычайно четкие дизайн эксперимента и план обработки результатов. Требуется определить список исследуемых маркеров, предусмотреть условия, по которым могут разделяться образцы и данные, учесть уже известные взаимодействия на разных уровнях или между уровнями, подготовить доступ к вычислительным мощностям, необходимым для обработки данных. Кроме того, нужна серьезная доля критического мышления и специальные методы биостатистики: ведь из-за больших объемов вместе с ограничениями и погрешностями каждого из использованных методов данных риск найти случайные корреляции сильно возрастает.
Дизайн исследований
Продуманный дизайн экспериментов до начала работы увеличивает успех сложного и трудоемкого мультиомиксного исследования — впрочем, как и любого другого. Но тут возникает первая проблема. Хотя для некоторых омик (например, геномики [31] и метаболомики [32]) и описаны кое-какие стандарты дизайна и работы с данными, для мультиомиксных исследований универсальных рекомендаций пока не существует. Создание такого всеобъемлющего мультиомиксного руководства будет непростой задачей, ведь у каждой омики свои проблемы и возможности. Плохой дизайн мультиомиксного исследования обязательно приведет к неинформативности выводов и кризису воспроизводимости результатов. Пока можно дать лишь общие рекомендации относительно того, что должно быть в МО-исследовании [33]:
- тщательная оценка статистической мощности и размера эффекта, соответствующих плану эксперимента;
- выявление факторов, препятствующих получению данных (например, исходные материалы или доступность пациентов, соответствующих критериям исследований) и анализ присущих получаемым данным искажений;
- оценка необходимого количества повторов (биологических и технических) и источников вариаций;
- меры обеспечения и контроля качества;
- меры перекрестной проверки, применяемые в случаях неизбежных искажений.
Вторая проблема: проведение исследований с несколькими омиками означает наличие данных разных модальностей [1], происходящих из различных типов анализов, и c повышенной размерностью. В МО-исследованиях (например, при профилировании РНК, белка или метаболитов) набор данных транскриптомики, основанный на RNA-seq, может генерировать от сотен до тысяч транскриптов и изоформ. Для сравнения, отдельный исследователь может составить профиль только нескольких тысяч белков (и протеоформ) или нескольких сотен идентифицированных метаболитов (и признаков). Таким образом, информационная нагрузка от транскриптома может легко «затмить» более действенные открытия, сделанные из белков или метаболитов, которые ближе к фенотипу: подобные вещи приходится учитывать на стадии анализа данных и их интеграции.
Анализ данных
МО-исследования логично наследуют все проблемы, характерные для отдельных омик. Кроме того, появляются и дополнительные проблемы, связанные с интеграцией и слиянием данных [33], кластеризацией, визуализацией и поиском функциональных взаимосвязей. При интеграции, как правило, оказывается, что для разных омик требуются различные подходы к масштабированию, нормализации и преобразованию данных. А общее количество данных (особенно, если речь идет про популяционные исследования, включающие тысячи отдельных выборов) подразумевает наличие немалых вычислительных мощностей и пространства для хранения.
Для интеграции данных геномики, транскриптомики и протеомики требуется сопоставление идентификаторов генов с ассоциированными транскриптами или белками (что непросто, так как соответствие не обязательно однозначно). Еще больше трудностей возникает при работе с метаболомикой: данные о метаболитах чаще всего получают методом ГХ—МС, реже ВЭЖХ—МС или ЯМР, но полученные спектры трудно идентифицировать ввиду относительно малого количества описанных метаболитов в спектральных базах данных и отличий реальных спектров от расчетных. Количество и качество идентифицированных и аннотированных молекул варьируется в разных омиках, начиная от очень хорошего охвата генома и заканчивая еще не полной картиной фосфопротеома и избирательным охватом метаболома. Что касается статистики и обработки результатов, неконтролируемые методы мультиомики могут усилить любой сигнал, включая систематическую ошибку, если она появилась до проведения количественных измерений, например, во время сбора образцов, транспортировки, логистики обработки и операций. Приборы, используемые для измерений, также могут генерировать ошибки из-за физических или инженерных ограничений. Разумеется, подобные неточности отразятся на интеграции результатов и сделанных выводах.
Будущее мультиомики
Мультиомика стала популярной. Связано это с тем, что при текущем развитии технологий будущее научных открытий и практических достижений — за интегративным подходом. Мы пока мало знаем о взаимосвязи процессов на разных уровнях, зато преуспеваем в высокоскоростных и высокопроизводительных методах анализа. Стоимость технологий, необходимых для омиксных исследований, снижается, а размер грантовой поддержки — увеличивается. Согласно исследованиям 2020 и 2021 годов, каждый четвертый ученый планирует участие в мультиомиксном исследовании, а количество публикаций на эту тему увеличивается ежегодно более чем в полтора раза (рис. 5).
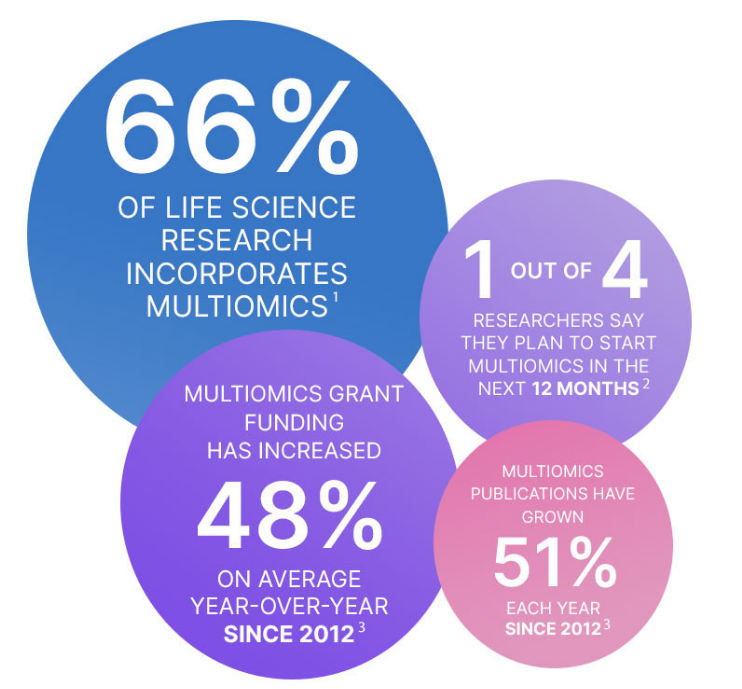
Рисунок 5. Популярность мультиомиксных исследований во всем мире увеличивается: 66% исследований включают мультиомиксные подходы, каждый четвертый исследователь планирует начать мультиомиксное исследование, на 48% в среднем увеличивается финансирование проектов, включающих мультиомиксные подходы, а число публикаций в этой сфере увеличивается ежегодно на 51%.
Термин «мультиомиксные исследования» активно используют и производители высокопроизводительного (а значит, сложного и дорогостоящего) оборудования — конечно, потому что без этого оборудования омиксные исследования не провести, но еще и, вероятно, из-за «волшебного» воздействия самого этого словосочетания. МО-технологии и исследования иногда используются как синоним переднего края науки, который скрывает множество важных открытий — стоит только протянуть руку и нажать кнопку на приборе или компьютере...
Но значит ли это, что любое МО-исследование гарантирует научный прорыв и помогает любому ученому стать звездой с мировым именем? Конечно, нет. В первую очередь, МО-технологии — это возможность найти не самую очевидную закономерность в огромном массиве данных, но они не отменяют необходимости фундаментальных исследований и «обычной» работы по поиску механизмов и причин. Успех МО-исследований зависит и от изначального дизайна исследования, основанного на гипотезе исследователя. Выводы, которые можно сделать из мультиомиксного исследования, существенно зависят от интерпретации данных, а обработка совокупных результатов и поиск достоверных закономерностей может занимать не меньше времени, чем сама экспериментальная работа. Ведь чтобы получить правильный ответ, сначала нужно задать правильный вопрос. Иначе даже большие данные останутся просто данными.
В следующих статьях спецпроекта мы подробно расскажем о применении мультиомиксных исследований в сфере фундаментальной биологии, экологии и геоинжиниринга, в сельском хозяйстве и пищевом производстве, а также биомедицине.
Литература
- Michal Krassowski, Vivek Das, Sangram K. Sahu, Biswapriya B. Misra. (2020). State of the Field in Multi-Omics Research: From Computational Needs to Data Mining and Sharing. Front. Genet.. 11;
- Геном человека: как это было и как это будет;
- «Омики» — эпоха большой биологии;
- Автоматизация омиксных технологий;
- Модельные организмы: нематода;
- Daniel Witvliet, Ben Mulcahy, James K. Mitchell, Yaron Meirovitch, Daniel R. Berger, et. al.. (2021). Connectomes across development reveal principles of brain maturation. Nature. 596, 257-261;
- Prashant Nair. (2013). Connectome. Proc. Natl. Acad. Sci. U.S.A.. 110, 5739-5739;
- Michael Kunst, Eva Laurell, Nouwar Mokayes, Anna Kramer, Fumi Kubo, et. al.. (2019). A Cellular-Resolution Atlas of the Larval Zebrafish Brain. Neuron. 103, 21-38.e5;
- Tasha M. Santiago-Rodriguez, Emily B. Hollister. (2021). Multi ‘omic data integration: A review of concepts, considerations, and approaches. Seminars in Perinatology. 45, 151456;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Нанопоровое секвенирование: на пороге третьей геномной революции;
- Молекулы и эпигеном ;
- Новый взгляд на геном: не просто цепочка генов, а трехмерная сеть, интегрирующая функциональные домены ядра;
- 12 методов в картинках: протеомика;
- Raphael BM Aggio, Arno Mayor, Sophie Reade, Chris SJ Probert, Katya Ruggiero. (2014). Identifying and quantifying metabolites by scoring peaks of GC-MS data. BMC Bioinformatics. 15;
- 12 методов в картинках: структурная биология;
- Фармакогеномика: изучение генов на службе персонализированной медицины;
- Alicia Berard, Andrea Kroeker, Peter McQueen, Kevin M. Coombs. (2018). Methods and approaches to disease mechanisms using systems kinomics. Synthetic and Systems Biotechnology. 3, 34-43;
- Giovanna Nicora, Francesca Vitali, Arianna Dagliati, Nophar Geifman, Riccardo Bellazzi. (2020). Integrated Multi-Omics Analyses in Oncology: A Review of Machine Learning Methods and Tools. Front. Oncol.. 10;
- Lasse Nyholm, Adam Koziol, Sofia Marcos, Amanda Bolt Botnen, Ostaizka Aizpurua, et. al.. (2020). Holo-Omics: Integrated Host-Microbiota Multi-omics for Basic and Applied Biological Research. iScience. 23, 101414;
- Ling Xu, Grady Pierroz, Heidi M.-L. Wipf, Cheng Gao, John W. Taylor, et. al.. (2021). Holo-omics for deciphering plant-microbiome interactions. Microbiome. 9;
- Dimitris E. Messinis, Carine Poussin, Diogo A. R. S. Latino, Yvan Eb-Levadoux, Remi Dulize, et. al.. (2022). Systems biology reveals anatabine to be an NRF2 activator. Front. Pharmacol.. 13;
- Zeeshan Ahmed. (2022). Multi-omics strategies for personalized and predictive medicine: past, current, and future translational opportunities. Emerging Topics in Life Sciences. 6, 215-225;
- Sean Eddy, Laura H. Mariani, Matthias Kretzler. (2020). Integrated multi-omics approaches to improve classification of chronic kidney disease. Nat Rev Nephrol. 16, 657-668;
- Peter Kreitmaier, Georgia Katsoula, Eleftheria Zeggini. (2023). Insights from multi-omics integration in complex disease primary tissues. Trends in Genetics. 39, 46-58;
- Julia Steinberg, Lorraine Southam, Theodoros I. Roumeliotis, Matthew J. Clark, Raveen L. Jayasuriya, et. al.. (2021). A molecular quantitative trait locus map for osteoarthritis. Nat Commun. 12;
- Way-Rong Lin, Shih-I Tan, Chuan-Chieh Hsiang, Po-Kuei Sung, I-Son Ng. (2019). Challenges and opportunity of recent genome editing and multi-omics in cyanobacteria and microalgae for biorefinery. Bioresource Technology. 291, 121932;
- Liumei Jian, Jianbing Yan, Jie Liu. (2022). De Novo Domestication in the Multi-Omics Era. Plant and Cell Physiology. 63, 1592-1606;
- Federico Scossa, Saleh Alseekh, Alisdair R. Fernie. (2021). Integrating multi-omics data for crop improvement. Journal of Plant Physiology. 257, 153352;
- Qiaozhi Zhang, Yanbo Wang, Linglin Fu. (2022). Application of (multi-)omics approaches for advancing food allergy: an updated review. Current Opinion in Food Science. 46, 100854;
- Loren A. Honaas, Naomi S. Altman, Martin Krzywinski. (2016). Study Design for Sequencing Studies. Methods in Molecular Biology. 39-66;
- Su Chu, Mengna Huang, Rachel Kelly, Elisa Benedetti, Jalal Siddiqui, et. al.. (2019). Integration of Metabolomic and Other Omics Data in Population-Based Study Designs: An Epidemiological Perspective. Metabolites. 9, 117;
- Farhana R. Pinu, David J. Beale, Amy M. Paten, Konstantinos Kouremenos, Sanjay Swarup, et. al.. (2019). Systems Biology and Multi-Omics Integration: Viewpoints from the Metabolomics Research Community. Metabolites. 9, 76;
- Bjoern Titz, Raffaella Gadaleta, Giuseppe Lo Sasso, Ashraf Elamin, Kim Ekroos, et. al.. (2018). Proteomics and Lipidomics in Inflammatory Bowel Disease Research: From Mechanistic Insights to Biomarker Identification. IJMS. 19, 2775.







