Охотники за микробами: дело SM254
10 ноября 2025
Охотники за микробами: дело SM254
- 172
- 0
- 3
Streptomyces albidoflavus SM254 против Pseudogymnoascus destructans: на заре справедливости.
Рисунок в полном размере.
-
Авторы
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: Вы когда-нибудь гадали, глядя на бактерию, что там у нее в геноме? Что, если среди множества генов где-то уже записана подсказка к спасению целого вида — и даже экосистемы? Дело SM254 начинается не в лаборатории, а в глубинах шахты, где медь и тьма закалили микроб, способный выживать в экстремальных условиях и, возможно, противостоять возбудителю синдрома белого носа у летучих мышей — грибку, вызвавшему массовую гибель рукокрылых и серьезные экологические последствия. Это рассказ о расследовании, которое пока не дает однозначных ответов, но открывает направление — туда, где микробы могут стать союзниками в восстановлении хрупкого баланса жизни.
Конкурс «Био/Мол/Текст»-2025/2026
Эта работа опубликована в номинации «Своя работа» конкурса «Био/Мол/Текст»-2025/2026.

Эта работа была написана для IV-й Международной конференции «Геномика, метагеномика и молекулярная биология микроорганизмов» 2025 и отобрана для участия в конкурсе «Биомолекулы».
Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Скрытая угроза
Зимой 2006 года в пещерах и шахтах штата Нью-Йорк (США) впервые были замечены случаи массовой гибели зимующих летучих мышей: животные, еще недавно здоровые, лежали мертвыми, с белесым налетом на мордах и крыльях. Так мир узнал о новой болезни — синдроме белого носа рукокрылых [1]. Его возбудителя идентифицировали как гриб Geomyces destructans, однако впоследствии он был переклассифицирован в род Pseudogymnoascus и получил современное имя — Pseudogymnoascus destructans [2]. Это психрофильный (то есть холодолюбивый) патоген, обитающий в холодных пещерах, где температура тела зимующих летучих мышей падает до 2–8 oC; в состоянии гибернации иммунитет подавлен — и это «коварство гибернации» делает животных особенно уязвимыми к грибковой атаке [3], [4].
С тех пор P. destructans стал причиной массовой гибели более 6 миллионов рукокрылых по всему миру (по некоторым оценкам — до 7 миллионов). В зимовках Северной Америки смертность колоний достигает ~90%, а скорость распространения инфекции — от 200 до 900 км в год [5]. К 2023 году синдром белого носа был зарегистрирован уже в 40 штатах США и девяти провинциях Канады [6]. Последствия оказались разрушительными: популяции Myotis lucifugus и Myotis septentrionalis сократились более чем на 90%, а Perimyotis subflavus в некоторых регионах уменьшился на 75% и более [7]. Примечательно, что одни и те же виды, часто рассматриваемые как природные «инкубаторы» вирусов, стали столь уязвимы к грибковой эпидемии — почти как если бы их собственная микробиологическая «броня» дала трещину [8].
Урон от эпидемии выходит далеко за пределы зоологии. Летучие мыши — главные ночные насекомоядные млекопитающие планеты: одна особь среднего размера за ночь уничтожает более 1000 насекомых-вредителей. Когда колонии исчезают, фермеры вынуждены компенсировать потери природного биоконтроля химическими средствами. Экономисты подсчитали: ежегодный ущерб от синдрома белого носа достигает 3,9 млрд долларов для сельского хозяйства США [9] и около 2 млрд долларов для Канады. За пределами североамериканского континента ситуация выглядит несколько иначе: хотя возбудитель болезни обнаружен и в Европе, и в Азии, массовой гибели колоний там не наблюдается. Предполагается, что местные виды рукокрылых выработали частичную устойчивость к грибку — вероятно, благодаря длительной совместной эволюции с ним [10]. В России споры Pseudogymnoascus destructans также выявлены [11], однако масштаб проблемы пока неясен — исследования лишь начинаются.
Более того, последствия эпидемии ощущаются и опосредованно. Недавнее исследование показало, что в округах с сильным спадом численности летучих мышей фермеры компенсировали потерю биологического контроля, увеличив применение инсектицидов на ~31%. Это повышение химического воздействия, как показано статистически, сопутствовало увеличению младенческой смертности в этих же регионах почти на 7,9% [12]. Автор исследования подчеркивает, что речь идет не о прямой причинно-следственной зависимости, а об устойчивой статистической ассоциации, выявленной после учета множества социально-экономических и экологических факторов. Такая связь не доказывает механизм, но указывает на возможную цепочку последствий, когда изменения в экосистемах отражаются на здоровье человека. Справедливости ради, этот вывод подвергался сомнению — другие исследователи отмечают ограничения и смешивающиеся факторы, — но даже в критических статьях признается, что экологические нарушения в экосистемах часто имеют каскадные последствия для здоровья человека и общества [13].
Откуда взялся этот гриб? Версий несколько. Ранее считалось, что Pseudogymnoascus destructans — единый патоген, занесенный в Северную Америку из Евразии, где летучие мыши уже выработали устойчивость [10]. Однако недавние геномные исследования уточнили эту картину. В 2025 году в журнале Nature показано, что гриб на самом деле представлен как минимум двумя криптическими линиями (Pd-1 и Pd-2), различающимися по хозяевам. Североамериканские изоляты относятся к Pd-1, а наиболее вероятным источником заноса названа Восточная Европа [14]. Другая работа, опубликованная в Journal of Fungi, расширила эволюционный контекст: ближайшие к P. destructans виды принадлежат антарктическим грибам, а их расхождение произошло около 141 миллионов лет назад [15] — задолго до появления рукокрылых [16]. Эти данные указывают, что патоген имеет глубокие и сложные эволюционные корни, а североамериканская вспышка — лишь один из недавних эпизодов этой давней истории. На этом фоне усилия ученых сосредоточены не только на мониторинге распространения болезни, но и на поиске естественных антагонистов патогена [17]. Возможно, где-то среди микробов, живущих в тех же экстремальных условиях, уже существует ключ к ограничению P. destructans.
Новая надежда
Мы — научная группа Центра реабилитации рукокрылых Института живых систем Донского государственного технического университета (рис. 1). Наши исследования охватывают всё, что делает летучих мышей уникальными — от особенностей их иммунитета и микробиоты до вирусов и грибов, с которыми они сосуществуют. Мы изучаем, как эти связи формируются, как влияют на здоровье животных и почему именно рукокрылые так часто оказываются в центре эпидемиологических историй.

Рисунок 1. Коллектив Центра реабилитации рукокрылых Института живых систем ДГТУ. Слева направо: Илья Попов: аспирант третьего года, руководитель проекта РНФ; Елизавета Ткачева: ветеринарный врач-реабилитолог; Владислав Горобец: ветеринарный врач-реабилитолог; Игорь Попов, PhD: руководитель молодежной группы.
фото Ильи Попова
Идея этого исследования появилась совершенно спонтанно. В рамках проекта РНФ №25-24-00351 «Эпизоотологический мониторинг синдрома белого носа и других грибковых инфекций рукокрылых в Ростовской области» мы регулярно проводили обзор современной литературы по проблеме. И вот однажды нам попалась на глаза работа с интригующим заголовком — Complete genome sequence of Streptomyces albus SM254, a potent antagonist of Bat White-Nose Syndrome pathogen Pseudogymnoascus destructans, опубликованная в 2016 году в журнале Genome Announcements [18]. В ней сообщалось, что бактериальный штамм SM254 был выделен из подземных вод, богатых медью, в шахте Судан (Миннесота, США; рис. 2) — уникальной среде, где высокая концентрация тяжёлых металлов и дефицит питательных веществ создают естественный отбор микроорганизмов, способных выживать в экстремальных условиях.

Рисунок 2. Приветственная табличка шахты Судан, Миннесота, США.
Позднее, в ходе таксономической ревизии рода Streptomyces, штамм был переименован в S. albidoflavus [19] — под этим названием штамм SM254 и фигурирует в современных базах данных. Публикация же, несмотря на громкое название, была скорее коротким сообщением, чем полноценной исследовательской статьей, поэтому в ней практически не было подробностей. Что? как? почему? — все эти вопросы остались без ответа. Как именно бактериальный штамм S. albidoflavus SM254 подавляет патогенный гриб Pseudogymnoascus destructans? Почему этот штамм оказался таким эффективным антагонистом? А ведь статья вышла почти десять лет назад. В тот момент мы почувствовали себя героями книги Поля де Круи — теми самыми охотниками за микробами, только вооруженными не пипеткой, а Python и R. Так родилась идея нового расследования — «дело SM254». Да, наши возможности ограничены: мы не сможем провести «мокрые» эксперименты, поскольку у нас нет под рукой бактериальной культуры именно этого штамма, но имеется кое-что поинтереснее.
Пангеном наносит ответный удар
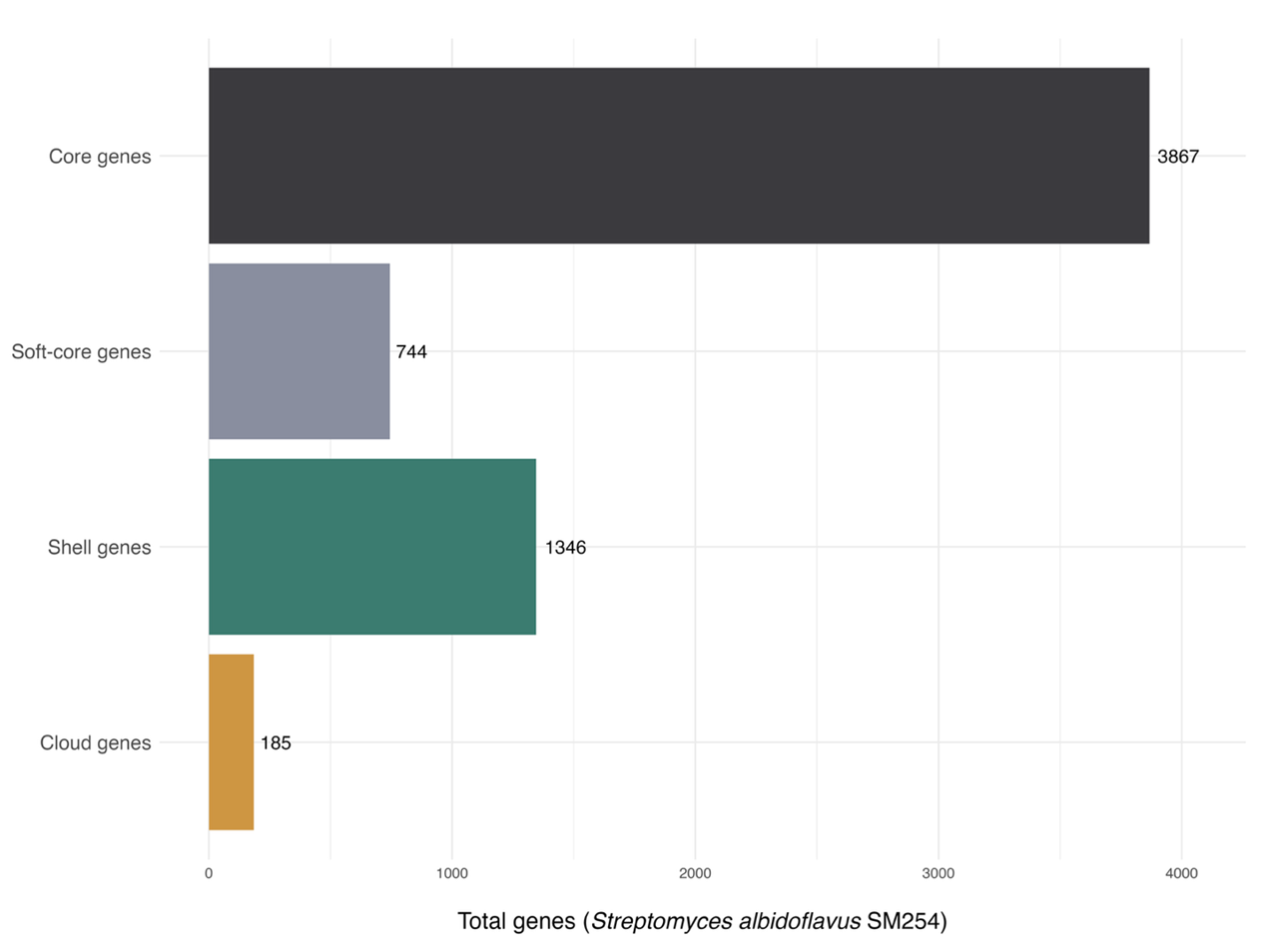
На руку сыграло то, что авторы оригинальной статьи добросовестно выложили собранный геном в открытый доступ. Это стало нашей первой «уликой». Однако одного генома было недостаточно — чтобы понять, чем конкретный штамм отличается от своих родственников, требуется сравнение десятков или сотен геномов одного вида. Такой подход и лежит в основе пангеномного анализа — метода, который позволяет рассмотреть весь генетический репертуар вида целиком: какие гены встречаются у всех штаммов, какие — у большинства, какие — у немногих, а какие уникальны только для одного [20]. На момент исследования в открытых базах данных было доступно 34 полных генома разных штаммов Streptomyces albidoflavus, и для анализа были собраны все эти последовательности, размещенные в базе RefSeq — своего рода «золотом стандарте» среди коллекций геномов, где последовательности тщательно проверены и аннотированы [21]. Несколько команд в терминале (рис. 3А), пара фильтров — и вот результат на рисунке 3Б [22]: в геноме SM254 оказалось 3867 генов, общих для всех штаммов; 744 — встречающихся почти у всех; 1346 — характерных лишь для части; и 185 — уникальных только для SM254.
Сто восемьдесят пять генов, которых нет ни у кого больше… вот оно, зерно разгадки! Но радость быстро сменилась озадаченностью: 185 — слишком много, чтобы сразу понять, где искать главное различие. С чего начать поиски? Как понять, какие из них действительно стоят за загадочной антифунгальной активностью SM254?
Атака клонов
Было принято решение провести филогеномный анализ , опираясь на универсальные гены. Цель простая и прагматичная: восстановить эволюционные связи, выделить ближайшую к SM254 кладу и затем сравнить функциональные профили именно этого «пула» штаммов, чтобы вычленить отличительные признаки нашего подопытного. Следуя лучшим практикам, было собрано объединенное выравнивание 3867 универсальных генов, выбрана модель замещения и построено дерево методом максимального правдоподобия. Дерево было проаннотировано метаданными — страной и годом выделения каждого штамма (важное уточнение: это не «страна происхождения» вида, а место и дата, где/когда материал был выделен и занесен в базу).
Филогеномный анализ — это реконструкция родственных связей на уровне всего генома или значительной его части, обычно путем конкатенации универсальных генов общего происхождения и построения филогенетического дерева; он дает количественную и топологическую картину близости штаммов и служит основой для целевого сравнительного анализа [23].
Так на рисунке 4 была выделена клада из шести штаммов (включая SM254), которая и легла в основу дальнейшего сравнения. Прежде чем переходить к функциональному сопоставлению, мы сопроводили дерево числовой метрикой — Average Nucleotide Identity (ANI) .
ANI — средняя процентная нуклеотидная идентичность по парным выравниваниям между двумя геномами; это стандартный числовой критерий геномной близости [23]: ~95% — порог вида, а значения ≳99% указывают на очень близкие, почти клональные геномы [24].

Рисунок 4. Филогеномная структура 34 геномов Streptomyces albidoflavus. Клада, содержащая штамм SM254, выделена синим цветом, а конец ветви SM254 подсвечен зеленым. Страна изоляции обозначена национальными флагами, а год представлен цветовым градиентом.
Из рисунка 5 видно, что в нашем наборе из шести геномов минимальная ANI составляет ~99,01%, максимальная — 99,38%. На первый взгляд различия крошечны — десятые и сотые доли процента, — но именно они потенциально отражают функционально значимые отличия. Вопрос, который ставит наше расследование далее: можно ли обнаружить биологически интерпретируемые различия на уровне функциональных профилей при такой высокой нуклеотидной схожести? Для ответа переходим к сопоставлению функциональных профилей.
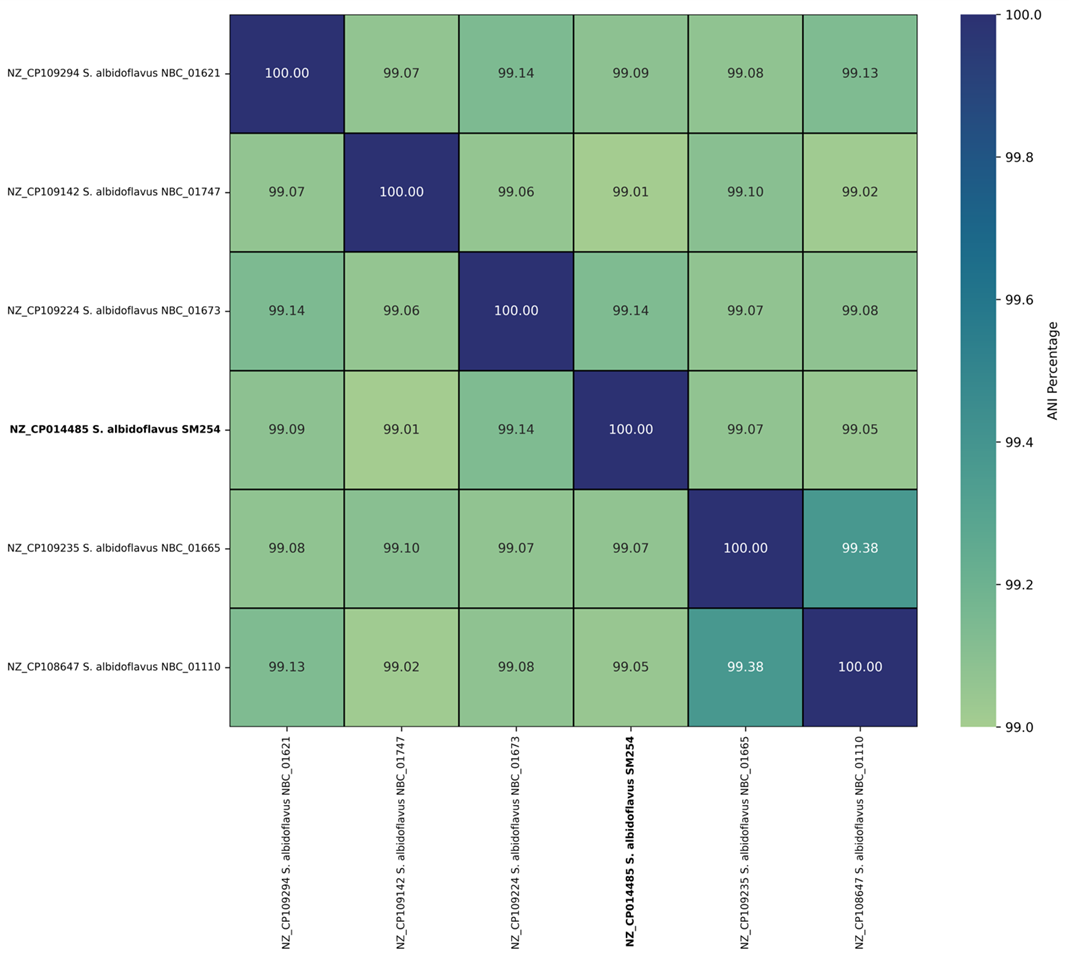
Рисунок 5. Средняя парная идентичность нуклеотидов (ANI) в геномах Streptomyces albidoflavus. Тепловая карта, выделяющая значения ANI среди шести ближайших родственников штамма SM254. Геном SM254 выделен жирным шрифтом для наглядности. Цветовые градиенты представляют процентные значения ANI.
Пробуждение силы
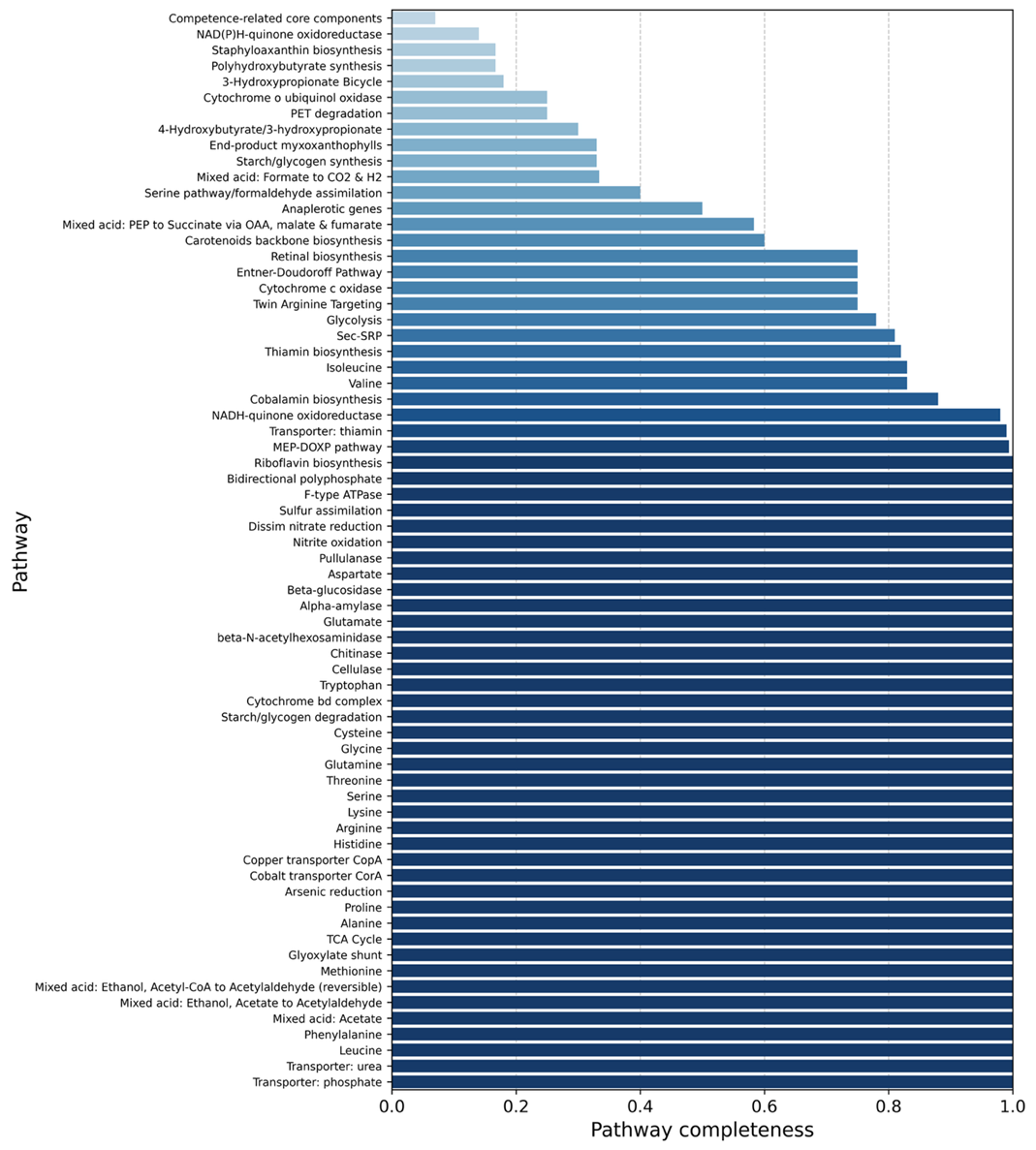
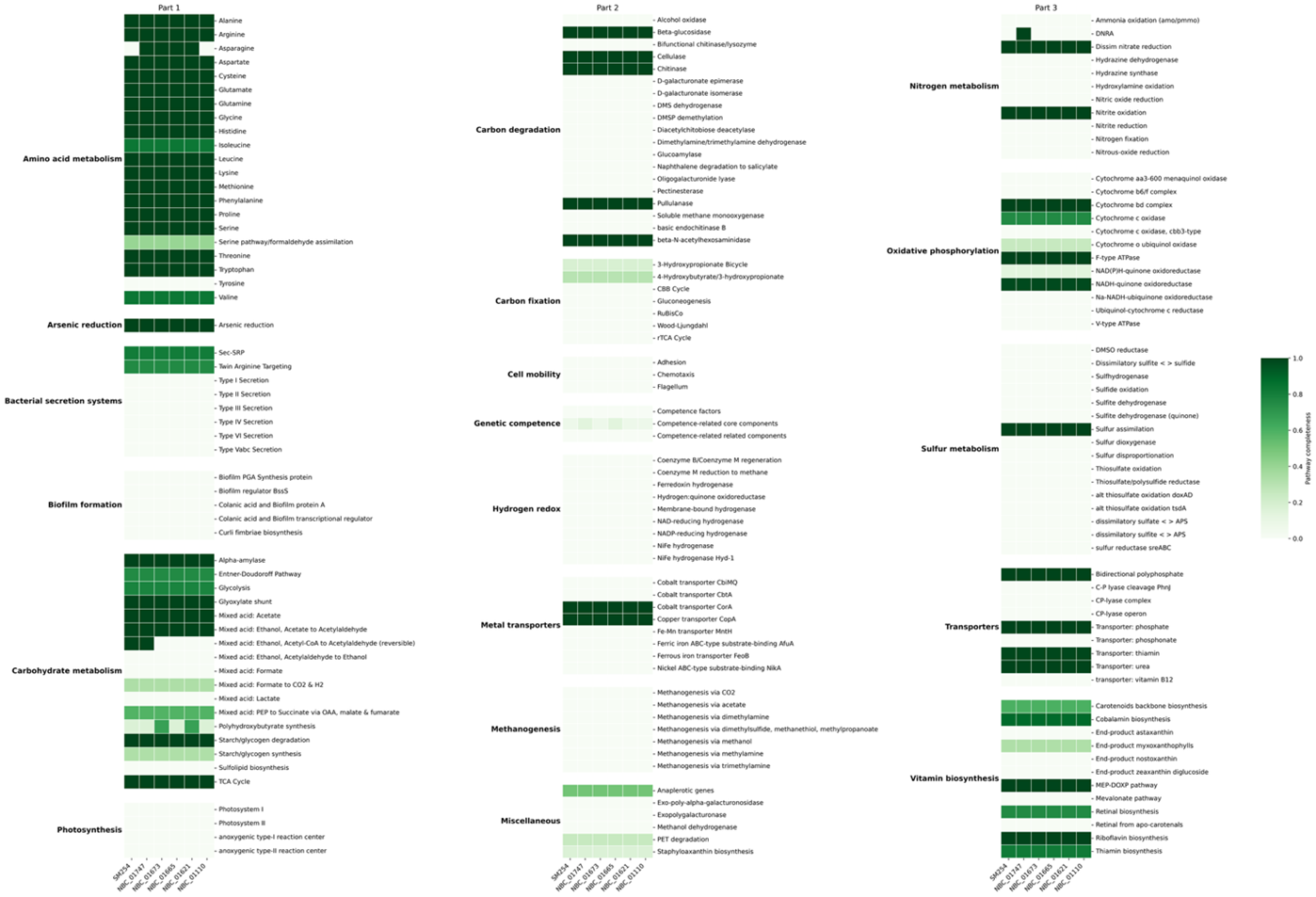
Для следующего шага было решено использовать немного нестандартную схему анализа. И хотя путь был не самым очевидным, результаты оказались более чем показательные. Первым этапом стала аннотация всего набора из шести геномов — с помощью инструмента eggNOG-mapper . Из полученных объемных и, на первый взгляд, почти непроходимых таблиц аннотаций были извлечены KEGG-идентификаторы ферментов и белков из базы Kyoto Encyclopedia of Genes and Genomes (KEGG), которые служат «узлами» метаболических путей. После их расшифровки удалось реконструировать метаболические профили всех шести штаммов S. albidoflavus. Для интерпретации данных была использована домашняя разработка — утилита KEGGaNOG , которая сочетает аннотации eggNOG с расшифровщиком KEGG-Decoder и автоматически формирует визуальные сводки: тепловые карты, диаграммы и корреляционные сети. Давайте посмотрим на результаты. Сначала — функциональный профиль самого SM254 на рисунке 6А, затем — сводная тепловая карта всех шести штаммов на рисунке 6Б и наконец — диаграмма, отражающая ключевые различия между ними на рисунке 6В (в крутилке).
eggNOG-mapper — система функциональной аннотации, основанная на определении ортологов — генов, происходящих от общего предка и сохраняющих схожие функции. Она позволяет сопоставить каждый ген с уже известными семействами и биохимическими путями, а значит — построить функциональный профиль генома [25].
KEGGaNOG была впервые применена именно в этом исследовании и успешно апробирована для анализа метаболических путей, центральных для микробной экологии и биогеохимических циклов. Позже принцип ее работы был описан в отдельной публикации [26].
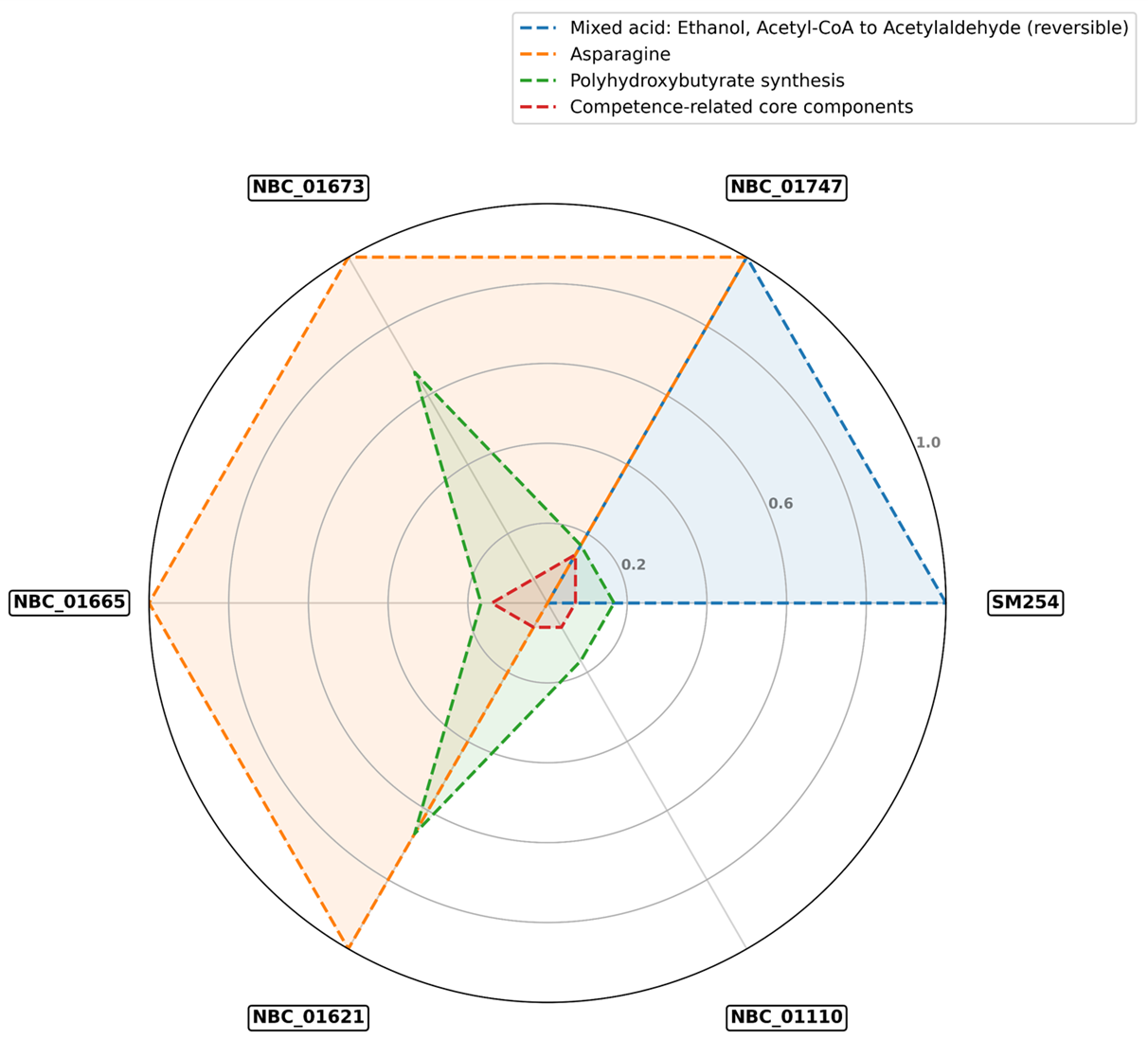
Из визуализаций ясно следует: SM254 — единственный штамм S. albidoflavus, полностью лишенный метаболического пути синтеза аспарагина, но при этом обладающий активным путем mixed acid: ethanol, Acetyl-CoA → Acetaldehyde (reversible) (см. K00132, K04072, K04073, K18366, K04021). Эта комбинация — отсутствие одного из базовых аминокислотных путей при наличии этанольного метаболизма — выглядит нетривиально и, возможно, указывает на специфическую адаптацию SM254 к микросреде его обитания. Дело в том, что аспартат и его производная аспарагин — важные источники азота для многих грибов [27]. Если SM254 не синтезирует аспарагин самостоятельно, он, возможно, активнее потребляет доступный в среде запас этой аминокислоты, снижая ее концентрацию до уровня, при котором рост патогена становится затруднен. Одновременно этанол, который образуется при ферментации, известен как соединение, подавляющее рост и продукцию микотоксинов у целого ряда грибов [28], [29].
Albidoflavus: Восход
Делает ли это S. albidoflavus SM254 тем самым уникальным антагонистом P. destructans? Ответить однозначно пока невозможно: без подтверждающих in vitro экспериментов это остается гипотезой. Тем не менее, полученные результаты показывают, что SM254 действительно отличается рядом функциональных особенностей, которые могут быть связаны с его антагонистической стратегией. Важно отметить, что вид S. albidoflavus в целом известен своей противогрибковой активностью. Многочисленные исследования документируют способность представителей этого вида подавлять широкий спектр патогенных грибов [30–33]. Штамм SM254 лишь усиливает этот интерес: его геном несет черты, которых нет у других представителей вида, и именно они, возможно, лежат в основе его выраженного антагонизма к P. destructans. Это исследование не ставило целью окончательно ответить на вопрос «почему» — скорее, оно открывает новое направление. Его задача — зажечь факел интереса. Быть может, будущие эксперименты in vitro подтвердят, что уникальные функциональные черты SM254 действительно обеспечивают подавление P. destructans, а затем биоинженерные подходы позволят усилить эти свойства. Весь ход анализа, от загрузки данных до визуализации результатов, доступен в открытом репозитории на GitHub, а также визуально отображен на рисунке 7.
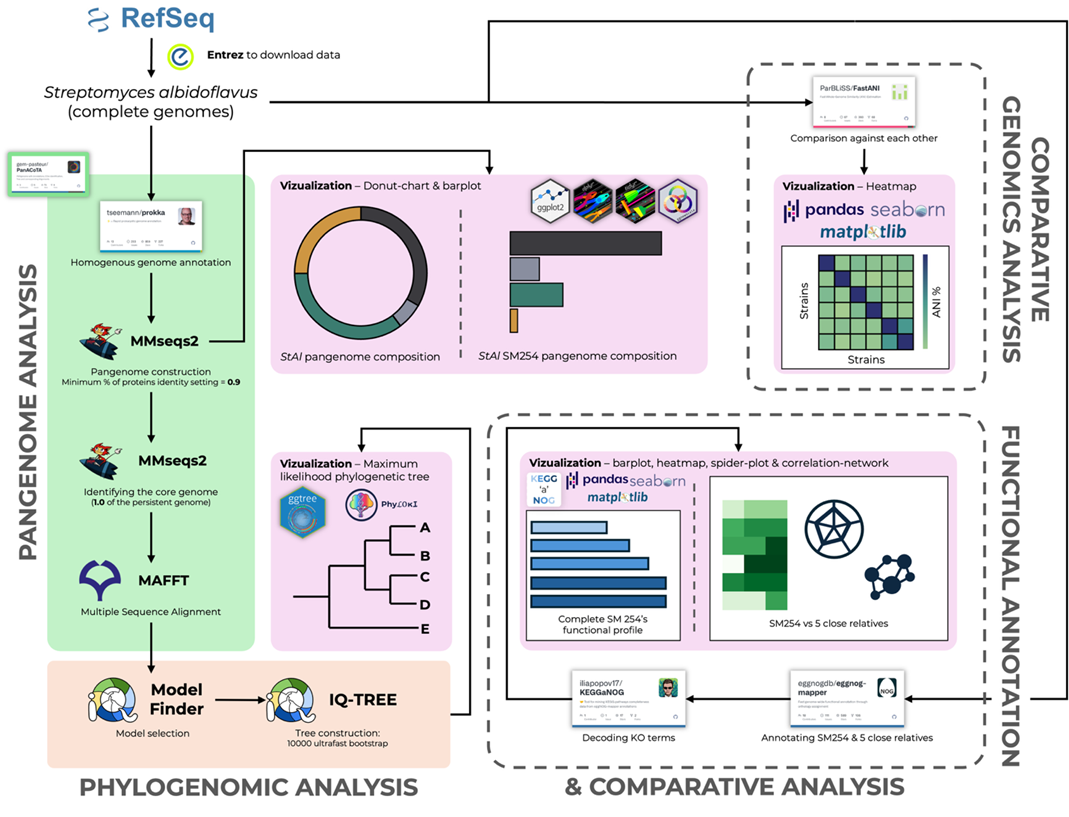
Рисунок 7. Пайплайн биоинформатического анализа, использованный в исследовании.
Работа выполнена на домашнем компьютере Mac mini базовой конфигурации, что подчеркивает доступность современных методов геномного анализа микроорганизмов. Этот проект показывает, что открытые данные и открытые инструменты делают науку по-настоящему доступной. Даже небольшой самостоятельный анализ может стать важным вкладом в общее знание — шагом, который однажды приведет к новым экспериментам и, возможно, к практическому решению проблемы синдрома белого носа у летучих мышей. Для нас это не просто исследование — это часть большой истории о взаимодействии живого и невидимого. Мы изучаем, как бактерии, вирусы и грибы влияют на судьбу рукокрылых, и верим, что понимание этих связей поможет однажды восстановить хрупкое равновесие между ними и окружающим миром.
Работа поддержана грантом РНФ №25-24-00351.
Литература
- David S. Blehert, Alan C. Hicks, Melissa Behr, Carol U. Meteyer, Brenda M. Berlowski-Zier, et. al.. (2009). Bat White-Nose Syndrome: An Emerging Fungal Pathogen?. Science. 323, 227-227;
- Andrew M. Minnis, Daniel L. Lindner. (2013). Phylogenetic evaluation of Geomyces and allies reveals no close relatives of Pseudogymnoascus destructans, comb. nov., in bat hibernacula of eastern North America. Fungal Biology. 117, 638-649;
- Marcos Isidoro-Ayza, Jeffrey M. Lorch, Bruce S. Klein. (2024). The skin I live in: Pathogenesis of white-nose syndrome of bats. PLoS Pathog. 20, e1012342;
- Коварство гибернации в условиях города, или как помочь мыши в холодильнике повеситься;
- Thomas M. Lilley, Jani Anttila, Lasse Ruokolainen. (2018). Landscape structure and ecology influence the spread of a bat fungal disease. Functional Ecology. 32, 2483-2496;
- Joseph R. Hoyt, A. Marm Kilpatrick, Kate E. Langwig. (2021). Ecology and impacts of white-nose syndrome on bats. Nat Rev Microbiol. 19, 196-210;
- Tina L. Cheng, Jonathan D. Reichard, Jeremy T. H. Coleman, Theodore J. Weller, Wayne E. Thogmartin, et. al.. (2021). The scope and severity of white‐nose syndrome on hibernating bats in North America. Conservation Biology. 35, 1586-1597;
- Уроки толерантности от летучей мыши: как ужиться с полчищем смертоносных вирусов;
- Justin G. Boyles, Paul M. Cryan, Gary F. McCracken, Thomas H. Kunz. (2011). Economic Importance of Bats in Agriculture. Science. 332, 41-42;
- V. G. Twort, V. N. Laine, K. A. Field, F. Whiting-Fawcett, F. Ito, et. al.. (2024). Signals of positive selection in genomes of palearctic Myotis-bats coexisting with a fungal pathogen. BMC Genomics. 25;
- Veronika Kovacova, Jan Zukal, Hana Bandouchova, Alexander D. Botvinkin, Markéta Harazim, et. al.. (2018). White-nose syndrome detected in bats over an extensive area of Russia. BMC Vet Res. 14;
- Eyal G. Frank. (2024). The economic impacts of ecosystem disruptions: Costs from substituting biological pest control. Science. 385;
- Philip Marx-Stoelting, Tewes Tralau, Veronika Städele, Stefanie Rotter, Vera Ritz, et. al.. (2025). Bats, bugs, and babies: why stringent guidelines are needed for cause-and-effect interpretation in epidemiological studies. Arch Toxicol. 99, 3443-3448;
- Nicola M. Fischer, Imogen Dumville, Benoit Nabholz, Violeta Zhelyazkova, Ruth-Marie Stecker, et. al.. (2025). Two distinct host-specialized fungal species cause white-nose disease in bats. Nature. 642, 1034-1040;
- Ilia V. Popov, Svetoslav D. Todorov, Michael L. Chikindas, Koen Venema, Alexey M. Ermakov, Igor V. Popov. (2025). Beyond White-Nose Syndrome: Mitochondrial and Functional Genomics of Pseudogymnoascus destructans. JoF. 11, 550;
- Emma C. Teeling, Mark S. Springer, Ole Madsen, Paul Bates, Stephen J. O'Brien, William J. Murphy. (2005). A Molecular Phylogeny for Bats Illuminates Biogeography and the Fossil Record. Science. 307, 580-584;
- Joseph R. Hoyt, Kate E. Langwig, J. Paul White, Heather M. Kaarakka, Jennifer A. Redell, et. al.. (2019). Field trial of a probiotic bacteria to protect bats from white-nose syndrome. Sci Rep. 9;
- Jonathan P. Badalamenti, Joshua D. Erickson, Christine E. Salomon. (2016). Complete Genome Sequence of Streptomyces albus SM254, a Potent Antagonist of Bat White-Nose Syndrome Pathogen Pseudogymnoascus destructans. Genome Announc. 4;
- Eduardo Almeida, Navdeep Kaur, Laurence Jennings, Andrés Felipe Carrillo Rincón, Stephen Jackson, et. al.. (2019). Genome Mining Coupled with OSMAC-Based Cultivation Reveal Differential Production of Surugamide A by the Marine Sponge Isolate Streptomyces sp. SM17 When Compared to Its Terrestrial Relative S. albidoflavus J1074. Microorganisms. 7, 394;
- L. Rouli, V. Merhej, P.-E. Fournier, D. Raoult. (2015). The bacterial pangenome as a new tool for analysing pathogenic bacteria. New Microbes and New Infections. 7, 72-85;
- Tamara Goldfarb, Vamsi K Kodali, Shashikant Pujar, Vyacheslav Brover, Barbara Robbertse, et. al.. (2025). NCBI RefSeq: reference sequence standards through 25 years of curation and annotation. Nucleic Acids Research. 53, D243-D257;
- Ilia V. Popov, Michael L. Chikindas, Igor V. Popov. (2025). Genomic insights into Streptomyces albidoflavus SM254: tracing the putative signs of anti-Pseudogymnoascus destructans properties. Braz J Microbiol. 56, 2121-2131;
- Jesus Lozano-Fernandez. (2022). A Practical Guide to Design and Assess a Phylogenomic Study. Genome Biology and Evolution. 14;
- Luis M. Rodriguez-R, Roth E. Conrad, Tomeu Viver, Dorian J. Feistel, Blake G. Lindner, et. al.. (2024). An ANI gap within bacterial species that advances the definitions of intra-species units. mBio. 15;
- Carlos P Cantalapiedra, Ana Hernández-Plaza, Ivica Letunic, Peer Bork, Jaime Huerta-Cepas. (2021). eggNOG-mapper v2: Functional Annotation, Orthology Assignments, and Domain Prediction at the Metagenomic Scale. Molecular Biology and Evolution. 38, 5825-5829;
- Ilia V. Popov, Michael L. Chikindas, Koen Venema, Alexey M. Ermakov, Igor V. Popov. (2025). KEGGaNOG: A Lightweight Tool for KEGG Module Profiling From Orthology‐Based Annotations. Molecular Nutrition Food Res;
- Dz-Chi Chen, Bi-Dar Wang, Pei-Yin Chou, Tsong-Teh Kuo. (2000). Asparagine as a nitrogen source for improving the secretion of mouse ?-amylase inSaccharomyces cerevisiae protease A-deficient strains. Yeast. 16, 207-217;
- Thien Dao, Philippe Dantigny. (2011). Control of food spoilage fungi by ethanol. Food Control. 22, 360-368;
- Yaoyao Ren, Jing Jin, Mumin Zheng, Qingli Yang, Fuguo Xing. (2020). Ethanol Inhibits Aflatoxin B1 Biosynthesis in Aspergillus flavus by Up-Regulating Oxidative Stress-Related Genes. Front. Microbiol.. 10;
- Ana Luisa Perini Leme Giordano, Marili Villa Nova Rodrigues, Karen Gabriela Araujo dos Santos, Barbara Cipulo Legabão, Lais Pontes, et. al.. (2024). Enhancing Antifungal Drug Discovery Through Co-Culture with Antarctic Streptomyces albidoflavus Strain CBMAI 1855. IJMS. 25, 12744;
- Anna A. Baranova, Alexey A. Chistov, Anton P. Tyurin, Igor A. Prokhorenko, Vladimir A. Korshun, et. al.. (2020). Chemical Ecology of Streptomyces albidoflavus Strain A10 Associated with Carpenter Ant Camponotus vagus. Microorganisms. 8, 1948;
- Celia Bautista-Crescencio, Arturo Casimiro-Ramos, M. Jonathan Fragoso-Vázquez, José Correa-Basurto, Carlos Olano, et. al.. (2023). Streptomyces albidoflavus Q antifungal metabolites inhibit the ergosterol biosynthesis pathway and yeast growth in fluconazole-resistant Candida glabrata : phylogenomic and metabolomic analyses. Microbiol Spectr. 11;
- Jiling Ma, Youhua Long, Weizhen Wang, Wenzhi Li, Xuetang Chen, et. al.. (2025). Biocontrol potential of Streptomyces albidoflavus SC-3 on kiwifruit soft rot caused by Botryosphaeria dothidea. Postharvest Biology and Technology. 222, 113344.