Под шипами: омиксные технологии помогают понять молекулярные механизмы патогенности SARS-CoV-2 и SARS-CoV
07 мая 2021
Под шипами: омиксные технологии помогают понять молекулярные механизмы патогенности SARS-CoV-2 и SARS-CoV
- 1059
- 0
- 2
Мультиомиксное сравнение двух коронавирусов? Для современных технологий это piece of cake!
Рисунок в полном размере.
рисунок автора
-
Автор
-
Редакторы
Что происходит в клетке, когда в нее проникает SARS-CoV-2? Что в этом вирусе уникального, а что общего с другими коронавирусами? Эти вопросы очень важны для поиска эффективных лекарств. Мы расскажем о том, как современные «омиксные» методы (в частности, протеомика) ускоряют поиск ответов на примере мультиомиксного сравнения двух близких родственников — SARS-CoV-2 и SARS-CoV.
Геном коронавируса SARS-CoV-2 — виновника нынешней пандемии — состоит из 14 открытых рамок считывания, кодирующих 29 вирусных белков [1]. В их числе и S-белок, который образует на поверхности вирусной частицы характерные «шипы», давшие коронавирусу его название.

Рисунок 1. Аффинная очистка, совмещенная с масс-спектрометрией.
1. В исследуемый белок («приманку») вносят короткую пептидную последовательность («метку»), которая не меняет его структуру и функции. Белок-приманка трансгенно внедряется в клетку.
2. Аффинная очистка. Антитела, обладающие высоким сродством к «метке», помогают «выудить» помеченный белок из образца. При этом «улов», помимо приманки, содержит другие белки и белковые комплексы, с которыми белок-приманка образует устойчивые связи.
3. Белки, взаимодействующие с приманкой, идентифицируют с помощью масс-спектрометрии [2].
рисунок автора
Многие вирусные белки являются настоящими универсалами и совмещают разнообразные функции, ведь вирусу необходимо закодировать в своем компактном геноме весь непростой жизненный цикл — от попадания внутрь клетки до выпуска наружу новых вирусных частиц, попутно преодолевая защитные механизмы организма-носителя. Понимание механизмов инфекции на молекулярном уровне критически важно для разработки эффективных противовирусных препаратов. При этом функции отдельных белков коронавирусного семейства изучены слабо.
Первый шаг при изучении функций вирусного белка — понять, с какими клеточными белками он взаимодействует. Одна из самых продуктивных технологий для этого — аффинная очистка, совмещенная с масс-спектрометрией (Affinity Purification coupled with Mass Spectrometry, AP/MS) (рис. 1). Вот почему уже в начале февраля прошлого года, когда эпидемия едва распространилась за пределы Китая, многие исследовательские группы, специализирующиеся на изучении вирусов методами масс-спектрометрии, в том числе и наша лаборатория иммунопатологии вирусных инфекций Мюнхенского технического университета, переориентировались на исследование SARS-CoV-2.
Первой, уже в конце марта 2020 года, свой вариант «интерактома» — глобальной карты взаимодействий вирусных и клеточных белков — представила группа Невана Крогана (Nevan Krogan) из Калифорнийского университета (UCSF) [3]. Наша лаборатория опубликовала препринт своей версии в июне 2020. Она содержала интерактомы сразу двух вирусов — SARS-CoV-2 и SARS-CoV, геномы которых совпадают на 79,5% (рис. 2). SARS-CoV был причиной вспышки тяжелого острого респираторного синдрома (SARS) 2002–2003 годов и имел меньшую контагиозность, однако летальность SARS выше, чем у COVID-19. Наша идея состояла в том, что систематическое сравнение клеточных взаимодействий гомологичных белков этих вирусов способно выявить молекулярные механизмы, определяющие различие в течении инфекции. Использование клеточной линии A549, происходящей из клеток рака легкого (в мартовской публикации — HEK293-T из опухоли эмбрионального надпочечника), а также более мощных методов статистического анализа позволило нам обогатить интерактом SARS-CoV-2 сотнями новых белковых взаимодействий.
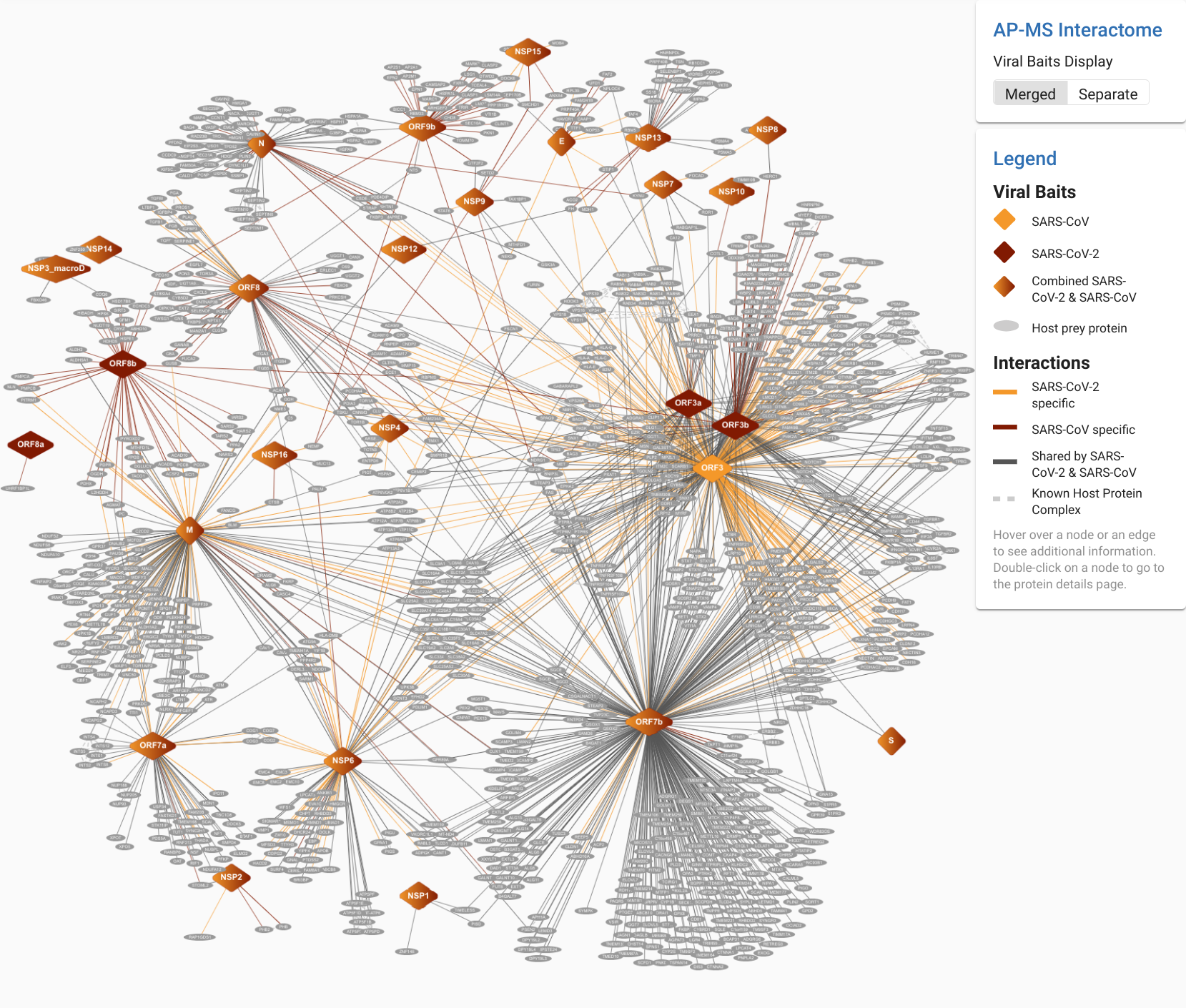
Рисунок 2. Совместный интерактом вирусов SARS-CoV-2 и SARS-CoV.
Граф белок-белковых взаимодействий вирусных (ромбовидные вершины) и клеточных (эллипсы) белков, измеренных методом аффинной очистки, совмещенной с масс-спектрометрией (AP-MS). Гомологичные вирусные белки объединены в общую вершину и имеют градиентную окраску. Белковые взаимодействия, общие для обоих вирусов, обозначены серым, специфичные для SARS-CoV-2 — оранжевым, для SARS-CoV — коричневым. Штриховые линии — известные клеточные белковые комплексы.
На этом мы не остановились и продолжили сравнение двух вирусов методами масс-спектрометрии. Мы измерили, как экспрессия каждого из 54 вирусных белков обоих вирусов, искусственно введенных в качестве трансгенов, воздействует на протеом человеческой клетки-хозяина. Вооружившись базой данных всех клеточных процессов (ReactomeFI), мы биоинформатическими методами попытались ответить на вопрос: в какие биологические процессы вовлечены клеточные белки, с которыми вирусный белок контактирует непосредственно, а также идентифицировать те белки, концентрация которых меняется в присутствии этого вирусного белка.
От отдельных белков мы перешли к изучению того, как меняется экспрессия генов и концентрация белков в клетке, инфицированной SARS-CoV-2 или SARS-CoV, с течением времени. Кроме того, мы оценили влияние инфекции на фосфорилирование и убиквитинирование белков — два ключевых индикатора внутриклеточных процессов. Итогом нашей интенсивной многомесячной работы стала апрельская публикация в Nature [4].
Что же мы обнаружили в таком многослойном массиве данных, который мы назвали COVINET (рис. 3)? Как и следовало ожидать, интерактомы SARS-CoV-2 и SARS-CoV, а также изменения, производимые этими вирусами в клетках, совпали более чем на 60%. Среди прочего, мы выявили общие для двух вирусов и ранее неизвестные взаимодействия вирусного белка ORF7b с ключевыми белками врожденного противовирусного иммунитета — MAVS и UNC93B1. Обнаружили мы и отличия между вирусами. Так, взаимодействие вирусного белка ORF8 с трансформирующим фактором роста бета (TGF-β) было обнаружено только у SARS-CoV-2. Примечательно, что в клетках, зараженных SARS-CoV-2, уровень фибронектина (FN1) и ингибитора активатора плазминогена 1 (SERPINE1) были выше, чем у клеток с SARS-CoV. Все три этих белка связаны с фиброзом и отеком легких — осложнениями, характерными для COVID-19. Также мы выяснили, что белок ORF3 вируса SARS-CoV-2 ингибирует клеточную аутофагию, в то время как у его SARS-CoV гомологов такой эффект не выражен. Вероятно, это связано с взаимодействием белка ORF3 и макромолекулярного комплекса гомотипического клеточного слияния и сортировки белков (HOPS complex), которое мы обнаружили только у SARS-CoV-2.
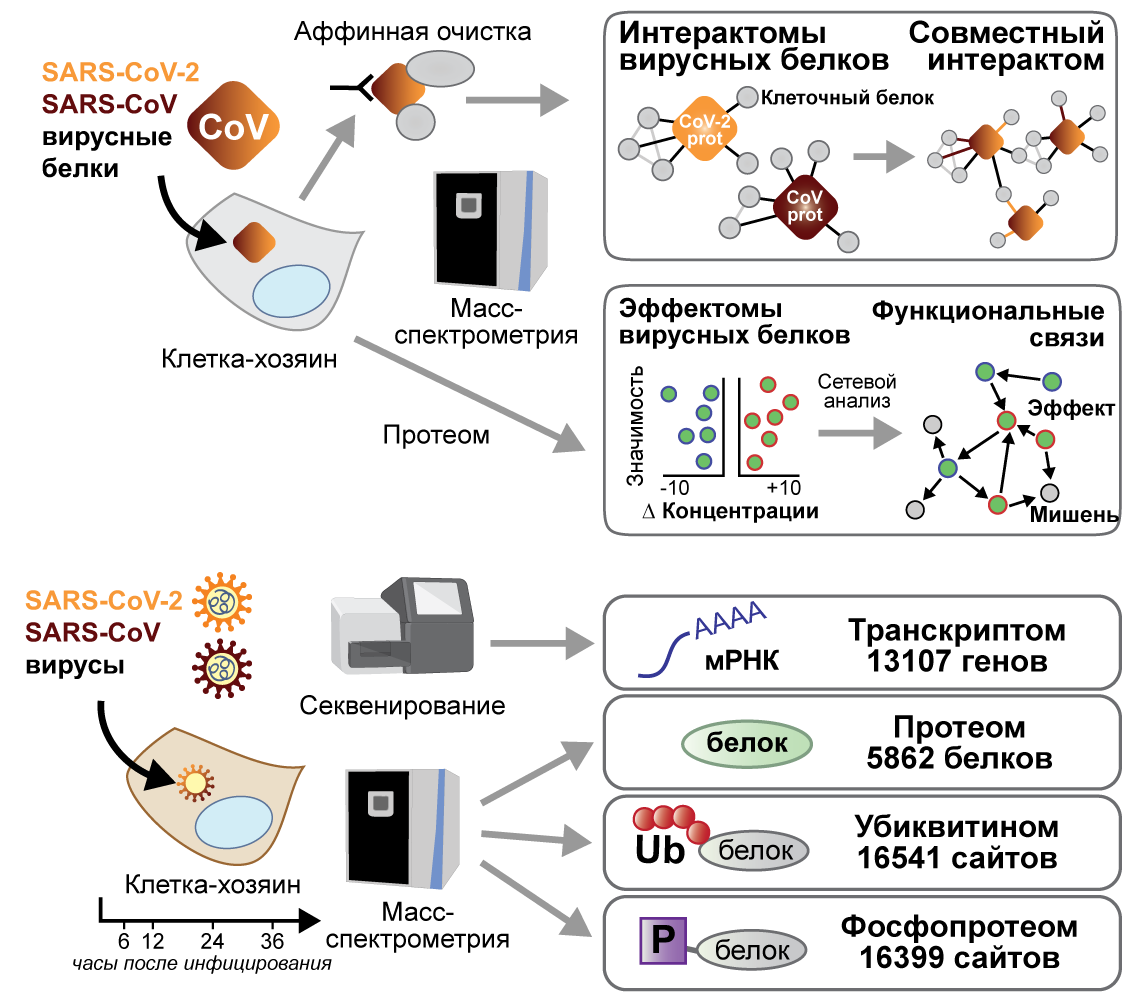
Рисунок 3. COVINET — схема эксперимента. Каждый вирусный белок SARS-CoV-2 и SARS-CoV с помощью продуцирующего их трансгена вводится в клетку-хозяина (A549-ACE2). Полученную модифицированную клеточную линию используют для построения интерактома данного вирусного белка методом аффинной очистки, а также измерения «эффектома» — влияния экспрессии этого белка на концентрацию клеточных белков (верхняя панель). Клетки A549-ACE2 инфицируют вирусом SARS-CoV-2 или SARS-CoV; биологические образцы (в том числе контрольные незараженные клетки) отбирают через 6, 12, 24 и 36 ч после инфекции и анализируют с помощью секвенирования РНК или масс-спектрометрии (нижняя панель).
Разумеется, эти примеры — лишь вершина айсберга. Нашей задачей было получить как можно более качественные и полные данные и сделать их доступными для ученых по всему миру. Дальнейший коллективный анализ данных COVINET должен выявить множество деталей коронавирусной биологии. Чтобы упростить и ускорить работу с ними, мы подготовили специализированный сайт covinet.innatelab.org, где у каждого из тысяч измеренных нами белков есть «персональная страница» с информацией о том, как он ведет себя во время инфекции и как взаимодействует с вирусными белками (рис. 4).
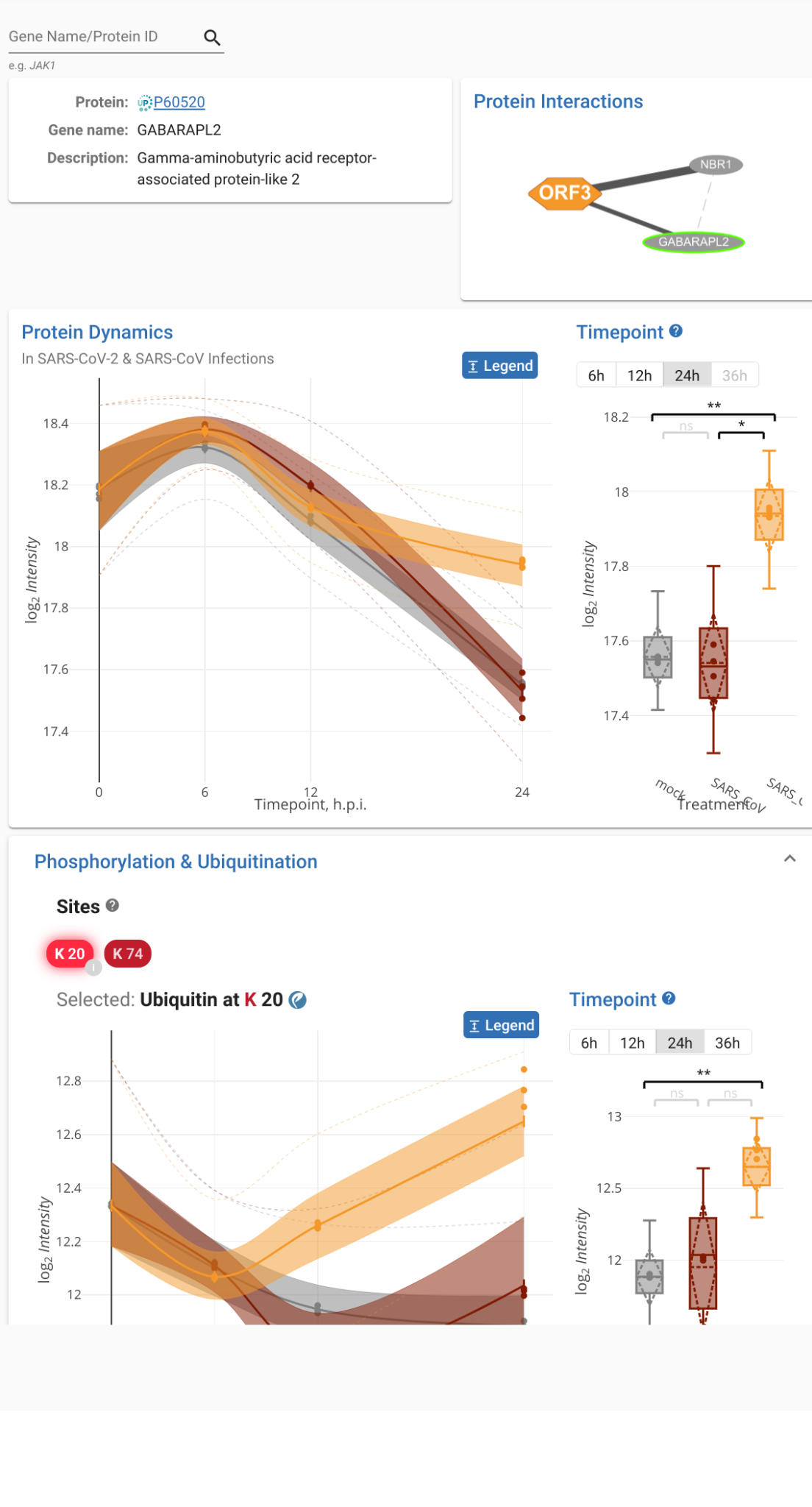
Рисунок 4. «Персональная страница» белка на сайте COVINET на примере GABARAPL2.
Верхняя правая панель — граф всех взаимодействий данного белка с вирусными белками (в данном случае — SARS-CoV2 ORF3) и другими компонентами известных клеточных белковых комплексов. Центральная левая панель — динамика изменения концентрации белка GABARAPL2 в ходе инфекции (серым обозначены контрольные незараженные клетки, оранжевым — SARS-CoV-2, коричневым — SARS-CoV); правая панель — сравнение концентрации белка между SARS-CoV-2, SARS-CoV и контролем в фиксированный момент времени. Нижняя панель — обнаруженные сайты фосфорилирования и убиквитинилирования GABARAPL2, а также динамика их изменений.
Очень перспективным выглядит использование наших результатов для детального компьютерного моделирования инфекции и поиска антивирусных лекарств. Публикация первого интерактома SARS-CoV-2 вызвала целую волну работ, в которых методы сетевого анализа применялись для поиска уже разрешенных к использованию у пациентов препаратов, которые бы нарушали связи между отдельными вирусными и клеточными белками [5], [6]. В то время экспериментальные данные о важности того или иного взаимодействия клеточного и вирусного белков не были доступны и не учитывались. Моделирование инфицированной клетки позволило бы находить уязвимые места SARS-CoV-2 гораздо более эффективно. Часто исходные данные для подобного моделирования компилируются из нескольких публикаций, отличающихся экспериментальными методиками. Влияние этих различий на результаты трудно учесть и компенсировать. Например, в разных клеточных линиях вирус проявляет себя по-разному, и это снижает качество предсказаний. У данных из COVINET подобного источника искажений нет; более того, некоторые данные (например, протеом и профиль убиквитинирования инфицированных клеток) получены из одних и тех же биологических образцов.
Вероятность того, что какой-то вирус, циркулирующий в дикой природе, мутирует и сможет передаваться от человека к человеку, высока [7]. Чтобы не допустить новой пандемии, нужно изучать потенциальных кандидатов уже сейчас. Современные омиксные технологии позволяют поставить такие исследования на поток, а пример SARS-CoV-2 и SARS-CoV показывает, что чем больше вирусов мы сравниваем друг с другом, тем лучше понимаем их молекулярные механизмы и яснее видим уязвимые места.
Литература
- COVID-19: что мы знаем и чего не знаем;
- 12 методов в картинках: протеомика;
- David E. Gordon, Gwendolyn M. Jang, Mehdi Bouhaddou, Jiewei Xu, Kirsten Obernier, et. al.. (2020). A SARS-CoV-2 protein interaction map reveals targets for drug repurposing. Nature. 583, 459-468;
- Alexey Stukalov, Virginie Girault, Vincent Grass, Ozge Karayel, Valter Bergant, et. al.. (2021). Multilevel proteomics reveals host perturbations by SARS-CoV-2 and SARS-CoV. Nature;
- Sepideh Sadegh, Julian Matschinske, David B. Blumenthal, Gihanna Galindez, Tim Kacprowski, et. al.. (2020). Exploring the SARS-CoV-2 virus-host-drug interactome for drug repurposing. Nat Commun. 11;
- Deisy Morselli Gysi, Ítalo do Valle, Marinka Zitnik, Asher Ameli, Xiao Gan, et. al.. (2021). Network medicine framework for identifying drug-repurposing opportunities for COVID-19. Proc Natl Acad Sci USA. 118, e2025581118;
- Zoë L. Grange, Tracey Goldstein, Christine K. Johnson, Simon Anthony, Kirsten Gilardi, et. al.. (2021). Ranking the risk of animal-to-human spillover for newly discovered viruses. Proc Natl Acad Sci USA. 118, e2002324118.