
12 методов в картинках: протеомика
17 ноября 2017
12 методов в картинках: протеомика
- 22901
- 4
- 51
рисунок Ольги Пташник
-
Авторы
-
Редакторы
-
Иллюстратор
Отечественному биологу, особенно, молекулярному, все реже и реже приходится писать на родном языке. Понятно, что бóльшая часть научных статей в нашей области создается на нынешнем языке научного общения — английском. Поэтому вместо вступления хочу поблагодарить главного редактора «Биомолекулы» — это он каким-то образом смог заставить меня написать этот текст, пробудив не то графоманский зуд, не то воспаленное эго, не то просто любовь к русскому языку. Но писать мне было легко: волею судеб я уже более пятнадцати лет занимаюсь одним и тем же — идентификацией и количественным анализом белков. То есть тем, что сегодня называется протеомикой. Практически всё, что я об этом знаю, по возможности доступно изложено в следующих строках.
12 биологических методов в картинках

Генеральный партнер цикла — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
Одна из главных миссий «Биомолекулы» — докопаться до самых корней. Мы не просто рассказываем, какие новые факты обнаружили исследователи — мы говорим о том, как они их обнаружили, стараемся объяснить принципы биологических методик. Как вытащить ген из одного организма и вставить в другой? Как проследить в огромной клетке за судьбой нескольких крошечных молекул? Как возбудить одну крохотную группу нейронов в огромном мозге?
И вот мы решили рассказать о лабораторных методах более системно, собрать воедино в одной рубрике самые главные, самые современные биологические методики. Чтоб было интереснее и нагляднее, мы густо проиллюстрировали статьи и даже кое-где добавили анимации. Мы хотим, чтобы статьи новой рубрики были интересны и понятны даже случайному прохожему. И с другой стороны — чтобы они были так подробны, что даже профессионал мог бы обнаружить в них что-то новое. Мы собрали методики в 12 больших групп и собираемся сделать на их основе биометодический календарь. Ждите обновлений!
Я был доволен, дней мне не хватало, и жизнь моя была полна смысла.
Братья Стругацкие. Понедельник начинается в субботу
— Десять лет, — со смехом проговорил он, жуя. — Вот через столько он что-нибудь напишет.
Джеймс Джойс. Улисс (пер. с англ. В. Хинкиса и С. Хоружего)
Часть 1. До генома. Идентифицировать можно только то, природу чего ты понимаешь
Физики шутят: «В вашем деле все пойдет на лад, когда биология станет химией, а химия — физикой». История протеомики до ее современного состояния немного напоминает эту шутку. Физики создали могучую технику, а когда науки о жизни стали модной тенденцией, попробовали использовать ее для анализа белков. Вначале разработка физических инструментов стимулировала их использование в биологии и медицине, а результаты — демонстрационные — только намекали на какие-то реальные достижения. Сейчас тот самый момент, когда в этой области просматривается серьезный результат, в том числе для медицины. Я попробую здесь рассказать о развитии высокопроизводительного анализа белков — протеомики, — которое наблюдаю давно, и наблюдаю глазами биолога. Может быть, физики опять пошутят, а вот коллегам-биологам и врачам будет немного легче понять суть происходящего.

Рисунок 1. Главные протеомные вехи. 1950 г. — Группа шведа Пера Эдмана предложила химический метод секвенирования пептидов. 1951–1955 гг. — Под началом британца Фредерика Сэнгера определили структуру короткого белка инсулина и доказали, что отдельные белки не аморфны в плане состава, а обладают постоянной последовательностью аминокислотных остатков. 1959 г. — Американцы Розалин Ялоу и Соломон Берсон создали первый иммуноанализ, в том числе для определения белков. 1967 г. — Создали первый автоматический белковый секвенатор, работающий по методу Эдмана. 1970 г. — Швейцарец Ульрихом Лэммли предложил оптимальный метод гель-электрофореза белков в денатурирующих условиях — с использованием додецилсульфата натрия. 1975 г. — Американец Патрик О’Фарелл и немец Йоахим Клозе независимо изобрели 2D-электрофорез белков и получили первые протеомные карты. 1984 г. — Под руководством американца Джона Фенна разработали ионизацию молекул электрораспылением. Впоследствии она позволила осуществлять масс-спектрометрию макромолекул, включая белки, без их разрушения. 1985 г. — Коичи Танака из Японии предложил мягкую ионизацию макромолекул лазером для масс-спектрометрии. Немцы Франц Гилленкамп и Михаэль Карас применили сходный метод для белков и пептидов. Возник метод ионизации MALDI. 1993-1996 гг. — Несколько групп исследователей предложили идентифицировать белки при помощи масс-спектрометрии фрагментов протеолиза и поиска по предсказанной из генома последовательности. Возникла масс-спектрометрическая пептидная карта (пептидный фингерпринт, или дактилоскопия). 1994 г. — Термин «протеом» как белковое дополнение к геному ввел австралийский аспирант Марк Уилкинс. 1994–1999 гг. — Появились первые поисковые программы для идентификации белков масс-спектрометрией по геномным последовальностям. Протеомика стала доступна широкому кругу пользователей. 1999–2001 гг. — Скорострельная (shotgun) протеомика. Несколько научных групп предложили применять для идентификации смеси пептидов совмещение высокоэффективной жидкостной хроматографии и тандемной масс-спектрометрии. Использовали ионизацию электрораспылением. 2000–2005 гг. — Российский физик Александр Макаров, работающий за рубежом, изобрел ионную ловушку нового типа — Orbitrap. Приборы на основе Orbitrap ввели в эксплуатацию. Масс-спектрометрия высокого разрешения демократизировалась и начала широко использоваться в протеомике. 2005 г. — Американцы Кристи Хантер и Ли Андерсон продемонстрировали использование масс-спектрометрического метода мониторинга множественных реакций (MRM) для количественного анализа природных пептидов. Возникла направленная (таргетная) протеомика. 2007 г. — Под руководством американца Стивена Гиги предложили новый метод оценки уровня ложноположительных результатов скорострельной протеомики с использованием «фальшивых» последовательностей (анализ target-decoy). 2012–2014 гг. — Скорострельная протеомика достигла уровня идентификации около 10 тысяч белков человека в одном образце — примерно половины кодируемых в геноме. Под руководством немца Бернхарда Кустера и американца Ахилеша Пандея независимо были опубликованы работы, декларирующие черновые версии полного протеома человека.
рисунок Ольги Пташник
Переместимся в то время, когда матричные процессы передачи информации в клетке уже были вчерне изучены (рис. 1). Было понятно, как in vivo синтезируются белки, из каких аминокислот они состоят. В то же время, в начале 1980-х годов, уже развилась молекулярная иммунология, возникла техника получения моноклональных антител [1]. Начали развиваться методы получения рекомбинантных белков, подпитываемые изобретением полимеразной цепной реакции [2]. Достигли совершенства методы разделения биомолекул — разные виды хроматографии и электрофореза [3].
Ферментативная активность — первые знания о белках
Чтобы поставить задачу «идентификации» белка в биологическом образце, уже было необходимо осознавать существование криковской «догмы» молекулярной биологии, в которой код нуклеиновой кислоты с информационными потерями конвертируется в аминокислотную последовательность. Идентификация некоторого соединения — это установление его структуры, в случае полипептида — полное или частичное определение его последовательности, иными словами, секвенирование. Следующей стадией будет не только идентификация (то есть, качественный анализ белка), но и определение его концентрации — количественный анализ. Интересно, что понятие об определении активности белка развилось еще до того, как установили его химическую природу. Примерно говоря, ферментативная активность свежего мясного фарша (то есть, гомогенизированной мышцы млекопитающего) могла быть установлена простыми спектрофотометрическими методами еще в начале XX века (рис. 2), когда химическая основа жизни оставалась неизвестной. Однако белковый катализатор, осуществляющий эту реакцию, можно было оценить количественно в условных единицах активности. И до сих пор в клинике многие биомаркеры определяют в таких условных единицах — например, аланиниаминотрансферазу и аспартатаминотрансферазу , несмотря на то, что современная техника способна определить их абсолютное количество. В случае многих ферментов определение активности и удобно, и правильно, поскольку часть молекул может не работать вследствие инактивации, при этом присутствуя в образцах.
Это внутриклеточные ферменты, организующие в клеточных митохондриях связь метаболизма углеводов и аминокислот. Их появление в крови свидетельствует о разрушении клеток печени.
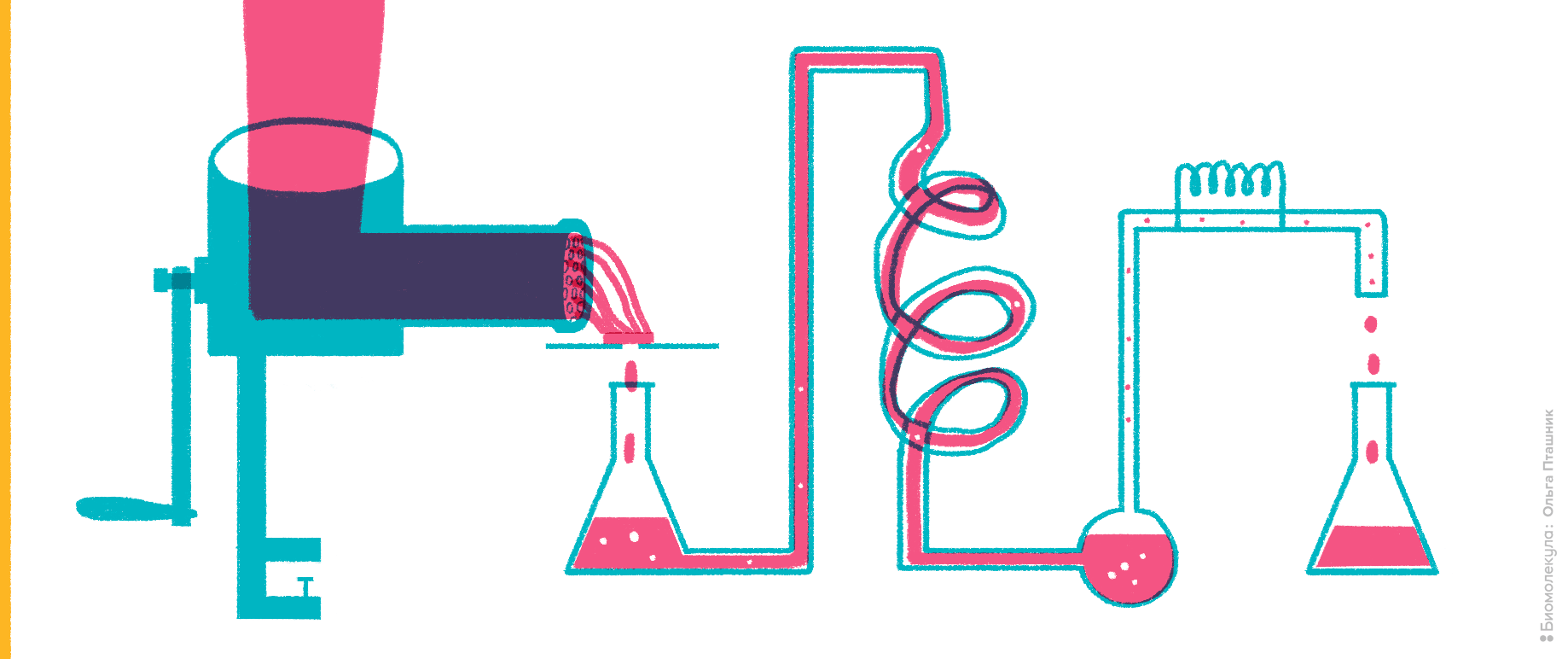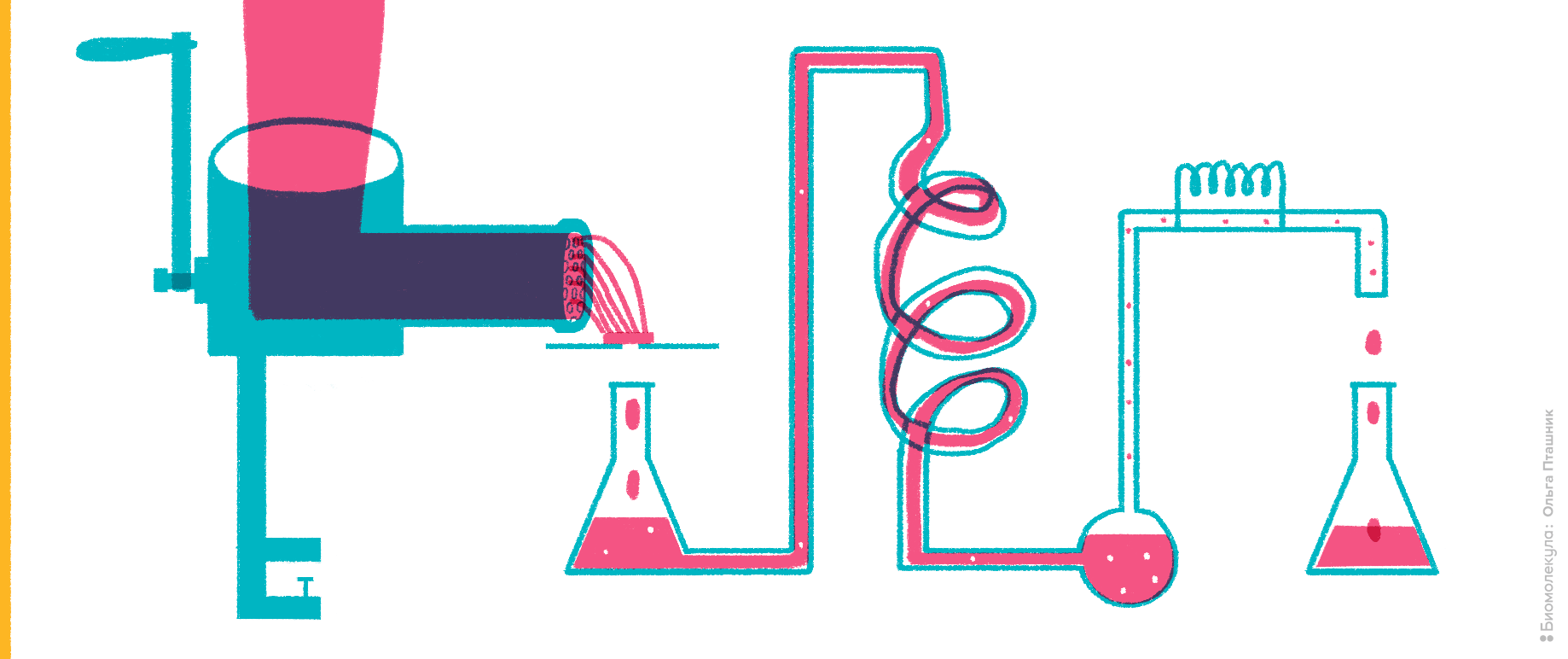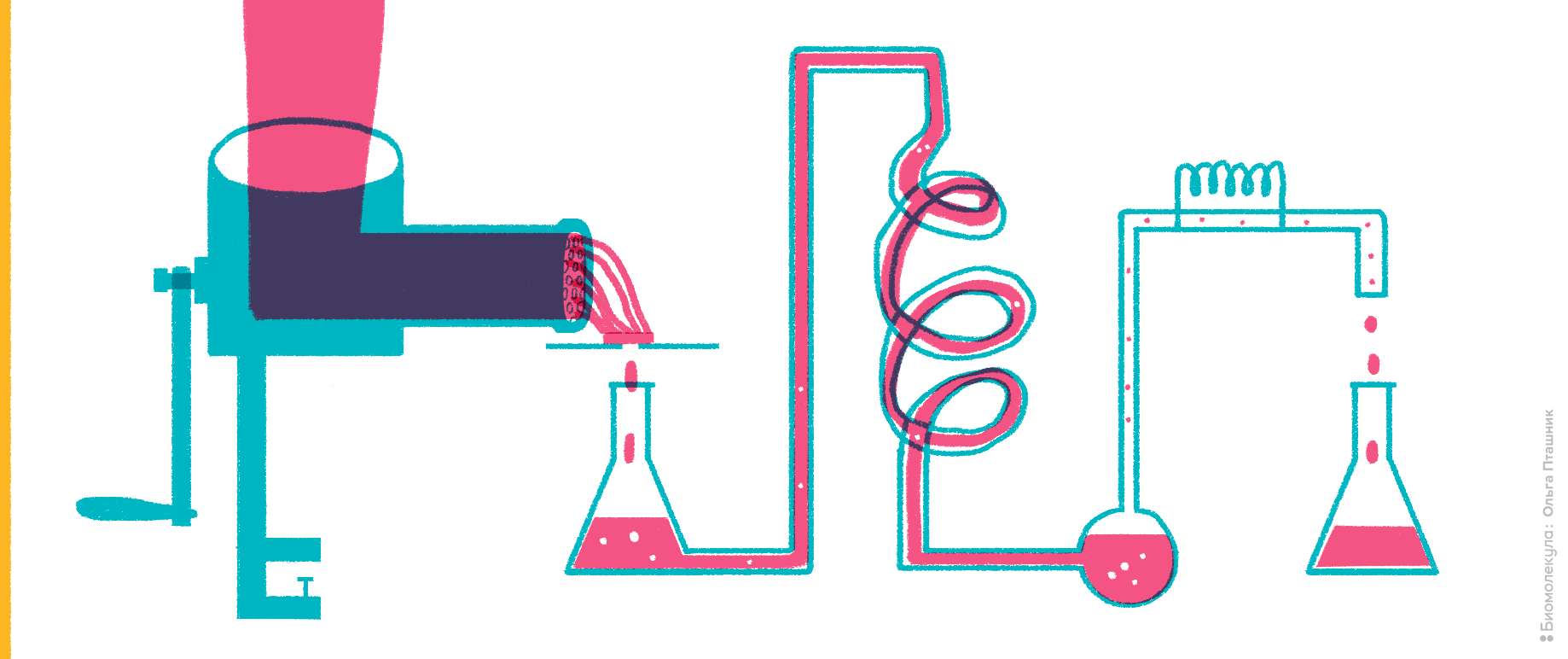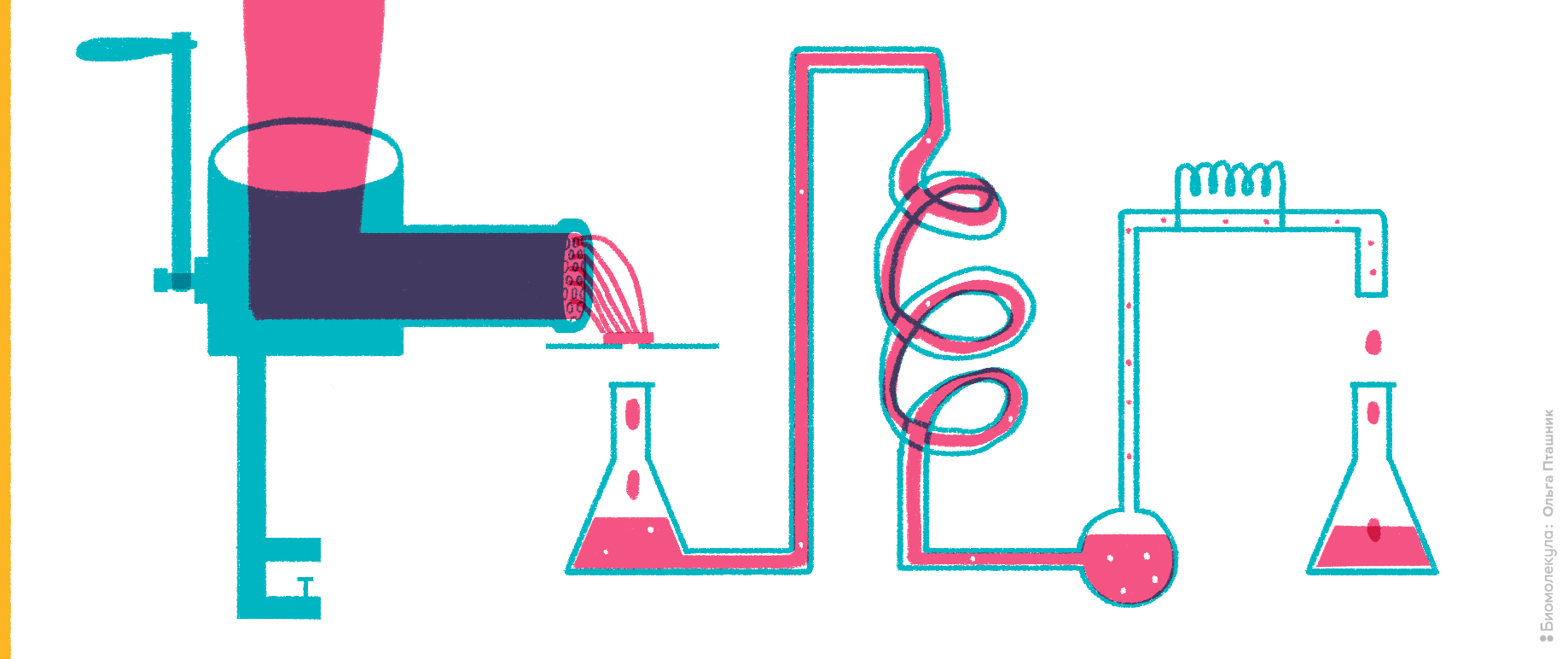
Рисунок 2. Мясной фарш как источник исследования ферментативной активности.
рисунок Ольги Пташник
Антитело как идентификатор от противного
Кроме оценки активности, с 1970-х годов у исследователей появилась еще одна возможность количественно измерять белки, не зная их структуры. Речь идет об использовании антител, особенно моноклональных [1], получение которых изобрели в 1975 г.; подробнее об этом рассказывает статья «12 методов в картинках: иммунологические технологии» [4]. Антитела можно вырабатывать к очищенным компонентам, а можно и к целым тканям, клеткам или фракциям. Причем если они моноклональные, то система их получения и их аналитические характеристики сохраняются без изменений от одной партии к другой. Если мы знаем, против чего были антитела, то есть использовали очищенный и идентифицированный другим, «ортогональным» методом антиген, то полученное связывающее вещество — антитело — может широко использоваться для его повторной идентификации в сложных смесях. Интереснее с антителами, полученными к неизвестным антигенам. Не имея понятия о структуре антигена, такие антитела стали использовать для диагностики злокачественных опухолей. Некоторые из них связывали намного больше своего антигена у больных людей, чем у здоровых. Методику оценки стандартизовали и стали использовать такие моноклональные антитела для диагностики, точно не зная химической структуры антигена. Ярким примером такого подхода служит гликопротеин CA-125, открытый Робертом Бастом с соавторами в виде антитела к нему в далеком 1981 году [5]. Лишь значительно позже идентифицировали ген этого продукта и сам белок — муцин 16.
Выделение чистых белков и секвенирование по Эдману
Тем не менее к 1970-м годам биохимиков перестала удовлетворять работа вслепую, например, измерение активности ферментов и других соединений без представлений об их химической структуре. Появились способы очистки белков, которые сочетали принципы хроматографии, электрофореза, центрифугирования, некоторые из которых исчезли из обращения, а другие используют до сих пор [3]. Отдельной задачей было подтвердить чистоту соединений во фракциях после очистки. Для этого использовали спектральные методы (от простых до сложных), а также визуализацию окрашенных полос при электрофорезе. Получение из биоматериала выделенного хотя бы до 90% чистоты белка без использования антител и других специфических связующих веществ и тогда, и сейчас — длительный трудоемкий процесс. 1970–1980-е годы — золотой век развития методов разделения белков, когда заливали огромные гели для электрофореза, конструировали метровые колонки для ручной и автоматической хроматографии.
Если вам повезло, то после нескольких месяцев или лет кропотливой работы вы убедились, что в пробирке или в геле находится ваш «товар» — белок, функцию которого вы изучаете. Какие варианты его идентификации у вас есть, если вы пока в веке двадцатом? Во-первых, если у вас есть гипотеза по поводу того, что в вашей пробирке, вы можете использовать известные антитела, если они есть в продаже или любезно предоставлены владельцами. Конечно, если сегодня доступны антитела разных видов к большинству белков человека и модельных животных, в то время их ассортимент был гораздо более скромным. Поэтому шанс на окраску интересующей вас молекулы антителами очень мал. Но не отчаивайтесь! Еще в 1950-е годы шведский химик Пер Эдман разработал метод секвенирования пептидов (рис. 3).
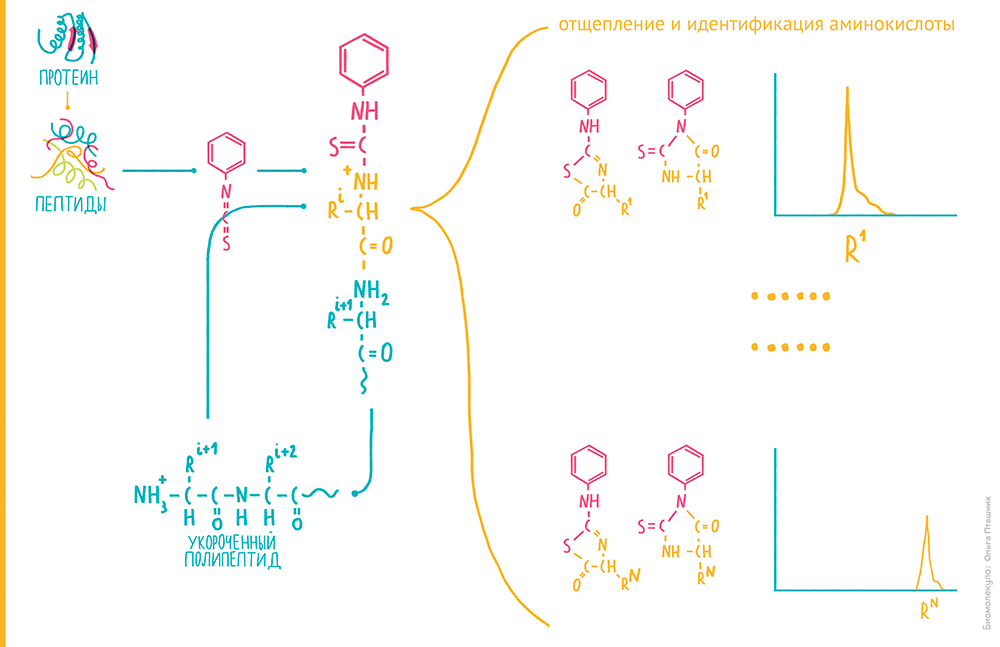
Рисунок 3. Секвенирование белков по Эдману. Если обработать пептид изотиоцианатом фенила (ФИТЦ), электрофильный атом углерода на изотиоцианатном радикале при умеренном подщелачивании взаимодействует с нуклеофильным азотом незаряженной аминогруппы. В итоге на N-конце пептида образуется фенилтиокарбомоильный радикал. Если умеренно закислить реакционную смесь, он отщепляется, увлекая с собой N-концевую аминокислоту, с образованием тиазолинона со специфичным радикалом, характеризующим эту аминокислоту. При этом остальная часть аминокислотной цепи остается неизмененной. Особое производное, которое будет отличаться по присущему аминокислоте радикалу, еще раз преобразуют в кислых условиях — для стабилизации — и анализируют хроматографически. Так можно отличить такие производные для всех аминокислот, поскольку из-за характерного радикала они будут характеризоваться своим временем выхода с обращенной фазы [3]. Если белок или пептид, который мы анализируем, присоединен к твердофазному носителю, производное N-концевой аминокислоты можно смыть и анализировать отдельно, а цикл анализа повторить, выстраивая таким образом аминокислотную последовательность.
рисунок Ольги Пташник
Метод Эдмана был по тем временам очень прогрессивен. Он с высокой точностью предоставлял последовательность до 30 аминокислотных остатков. Характеризовался достаточно высокой чувствительностью, будучи способным секвенировать пептиды в количестве менее 0,1 нмоль с 99% точностью. Более того, в конце 1960-х его автоматизировали в виде пептидного секвенатора, где робот-раскапыватель поочередно снимал N-концевые производные с полипептидов, закрепленных на специальной бумаге, направляя их затем в хроматограф. Но исследователям опять хотелось большего — их не устраивала необходимость в очистке пептидов и белков перед секвенированием, а также некоторые другие ограничения эдмановского метода, в частности, его неспособность секвенировать продукты с модифицированным N-концом.
Небольшой интерес к методу Эдмана существует до сих пор, в особенности, для белков и пептидов тех организмов, последовательность которых нельзя предсказать из данных секвенирования нуклеиновых кислот [6]. В этом методе реализуется прямой анализ, где ошибки связаны с технической погрешностью. Последовавшие за ним способы анализа аминокислотной последовательности содержат элементы предсказания, поэтому к техническим ошибкам в них прибавляются алгоритмические (см. ниже).
Двумерный электрофорез — первая карта протеома
Как я уже упоминал выше, при подготовке к качественному и количественному анализу белков использовали обычные методы разделения молекул [3], в том числе электрофорез. В 1970 году в электрофорезе белков произошла методическая революция — швейцарец Ульрих Лэммли предложил оптимальный метод гель-электрофореза в денатурирующих условиях. Белки жестко денатурировали амфифильным веществом, вроде мыла — додецилсульфатом натрия, — за счет чего каждая молекула покрывалась слоем из этого детергента. Суммарный отрицательный заряд такого комплекса оказывался примерно пропорционален молекулярной массе белка. Это позволяло делить белки в полиакриламидном геле хотя и с помощью электрического поля, но по молекулярной массе. По справедливости отметим, что Лэммли не изобрел метод de novo, а лишь оптимизировал его по существующим в литературе предпосылкам. За это, кстати, его работа сейчас входит в пятерку самых цитируемых в мире научных статей [7]. Разработки в этой области публиковали и ранее, в том числе в 1967 г. американец Арнольд Шапиро с соавторами [8].
Хорошо принятый метод Лэммли стал совершенствоваться и комбинироваться с другими видами разделения белков. В 1975 году американец Патрик О’Фарелл и немец Йоахим Клозе независимо предложили комбинировать денатурирующий электрофорез в геле с предварительной электрофокусировкой белков. Фокусировку проводят в относительно тонкой, толщиной с гель (1–2 мм) стеклянной трубке [9]. Трубку заполняют гелем со специальными полимерами — амфолинами, — которые способны создавать в ней неподвижный градиент рН. Таким образом, при движении в электрическом поле нанесенные в эту трубку белки останавливаются в участке, где амфолинами был достигнут рН, равный изоэлектрической точке молекулы белка. Гель в виде тонкого тяжа выдавливают из трубки и приплавляют к готовой гелевой пластине для обычного денатурирующего фореза по Лэммли, после чего проводят разделение в другом направлении. Белки, вначале распределенные по изоэлектрической точке, теперь движутся в зависимости от их молекулярной массы. Полученный метод справедливо называется двумерным (2D) электрофорезом (рис. 4). Как можно предполагать, каждый белок на итоговой пластине геля после окраски выглядит не как полоса (в отличие от обычного денатурирующего геля), а как сфокусированное, округлое пятно. Таким образом, О’Фарелл и Клозе впервые показали белковую карту, где каждое пятно на большой пластине геля (до 40×40 см) представляет одну изоформу белка, а его размер и интенсивность — более или менее пропорциональны его концентрации.
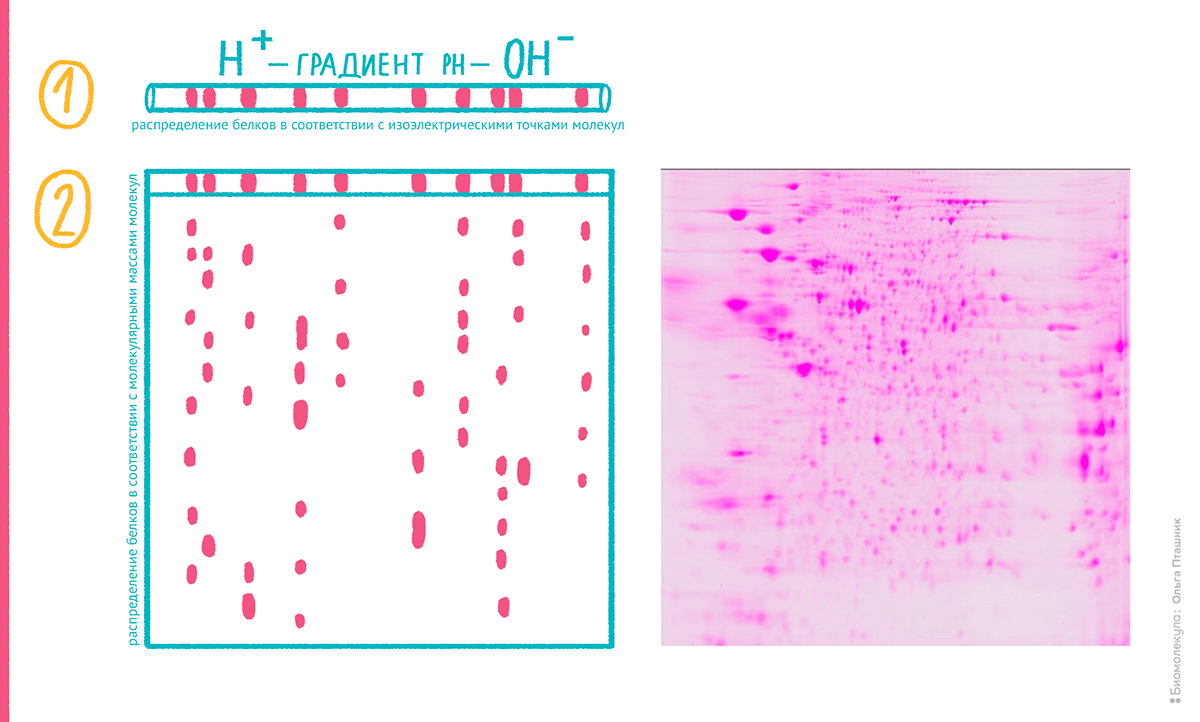
Рисунок 4. Схема двумерного электрофореза белков и реальная электрофореграмма, окрашенная флуоресцентным красителем.
рисунок Ольги Пташник
Искусные руки биохимиков прошлого многократно усовершенствовали метод двумерного электрофореза, который был ведущим в анализе белков до середины 2000-х годов. Вместо заливки трубок амфолины размещали на готовых полосках. Были предложены разные устройства для приготовления гелевых пластин, разных модификаций процесса электрофореза, причем для разных размеров и толщины геля в зависимости от задачи исследования. По чувствительности совершенствовали красители, в том числе, флуоресцентные. Более того, на волне популярности двумерных гелей, некоторые процессы их приготовления и окраски автоматизировали. Поскольку характеристики окрашенных пятен косвенно связаны с количеством белка в пробе, привлекательно сравнивать изображения гелей, полученные из одних и тех же образцов в различных условиях. Процесс обработки изображений гелей также автоматизировали, причем появилось много конкурирующих компьютерных программ, осуществляющих обработку и сравнение сканов двумерных гелей.
В зависимости от интервала концентраций находящихся в биоматериале белков, число отдельных пятен на двумерных гелях достигало 5 тысяч. С сегодняшней точки зрения очевидно, что это не означает, что на геле визуализированы продукты 5 тысяч генов. Изоформы одного генного продукта, которые отличаются по последовательности за счет гетерозиготности или протеолиза или по более тонкой структуре за счет модификаций остатков, будут, как правило, отражаться в виде отдельных пятен. Например, отщепление одного остатка аргинина от небольшого белка альфа-амилоида так существенно меняло изоэлектрическую точку, что на геле пятно перемещалось примерно на 10 см [10].
Тем не менее двумерная электрофореграмма с визуализированными тысячами белков может считаться первым протеомом — то есть, первым видом анализа, в котором определяется всё множество белков, присутствующих в биологическом образце, или существенная их доля. Отмечу, что этот метод был развит существенно задолго до появления термина «протеом», но об этом чуть позже.
Предположим, мы проанализировали путем двумерного электрофореза образцы опыта и контроля, например, клеточные линии после обработки лекарственным веществом и без такой обработки. Получили похожие карты-электрофореграммы, но десять пятен после обработки появились, пять исчезли полностью, и еще какое-то количество изменило свою интенсивность. Что нам можно сделать, если мы находимся в 1990 году? Всё, о чем мы говорили ранее. Применить метод Эдмана. Покрасить электрофореграмму антителами, которые есть в наличии, то есть осуществить вестерн-блоттинг [4]. Для обоих вариантов белки с геля с использованием электрического поля переносят на бумажную или подобную ей мембрану, с которой уже осуществляют дальнейшие манипуляции. Ограничения использования антител понятны — они, хоть и чувствительны, видят только свои мишени. Ограничение эдмановского метода здесь — чувствительность. Он хорошо работает от десятков и сотен пмоль белка, а современные красители «видят» пятна, в которых содержатся 2,5–5 пмоль. Учитывая потери при переносе на мембрану и вероятную потребность в расщеплении белков на пептиды, осознаем, что метод Эдмана сможет справиться с меньшей частью из визуализированных на хорошем геле белковых пятен.
«Звездное небо» двумерного электрофореза — первый и последний способ увидеть протеом воочию. Более того, при качественной постановке методики человеческого глаза вполне хватает, чтобы обнаружить отличия между похожими пластинами с гелем. Последующие методы протеомики, рассказ о которых впереди, образуют «большие данные», невидимые, как божество. Это обстоятельство во многом сохраняет популярность «двумерника», который используют по сей день, хотя и не так часто, как раньше. Тем не менее в продаже до сих пор имеется оборудование и программное обеспечение для полного цикла выполнения этой методики.
По личным впечатлениям, двумерный электрофорез белков — одна из самых трудоемких и сложных для выполнения биохимических процедур, в которой используются десятки стадий, реагентов и несколько типов лабораторного оборудования. В лаборатории мы в шутку называли тех, кто ставит двумерный электрофорез, «протеомными художниками». И действительно, постановка метода длится два—три дня и требует существенной концентрации на всех ее этапах. Малейшая оплошность приводит к существенному искажению «картины» на геле. Метод не автоматизируется целиком, что и было одной из причин снижения его популярности. Однако он получил второе дыхание уже на рубеже веков, когда в науку ворвался полный геном, а в протеомику вслед за ним — масс-спектрометрия.
Часть 2. Постгеном
Протеомика как постгеномная технология
Появление последовательностей геномов множества организмов, начиная с бактерий и завершая большими геномами растений и животных (в том числе человека [11]), уменьшило пространство поиска при идентификации белков. За исключением ситуации с секвенированием общей ДНК сложной смеси организмов (так называемого метагенома почвы, содержимого кишечника, океанских вод и т.д.), биохимики обычно представляют, какой организм они анализируют. И это значит, что белки в исследуемом образце синтезированы при помощи потока информации с кодирующих их генов этого организма. Собственно, так и появился термин «протеом» — в 1994 году Марк Уилкинс, австралийский аспирант, предложил его для обозначения белкового, или протеинового дополнения к геному . Геном — прочитанный геном — породил остальные «-омы» [12], а технологии, позволяющие их анализировать, в конце 1990-х годов почти гипотетические, составили группу постгеномных, или, как их теперь часто называют, омикс-технологий.
Real talk, think about it, господа аспиранты.
Строго говоря, истинные омиксы — это анализ продуктов передачи геномной информации, то есть кодирующих и некодирующих РНК и белков. Остальные омиксы, по сути, косвенные. Они не связаны с генетическим кодом прямым потоком информации и объединяются в группы по химической природе анализируемых соединений. Примечательно, что омикс-технологии производят одновременный анализ тысяч соединений, например, метаболитов, липидов, гликанов и т.д., и называются, соответственно, метаболомикой, липидомикой (частично они перекрываются), гликомикой и т.д. Энтузиасты последнего десятилетия — эпохи слоганов и мемов — придумали невероятное количество «омик», в том числе достаточно комичные. Число разных употреблений суффикса «омика» или «омикс» к 2010 году превзошло две сотни, что позволило пошутить на эту тему даже «гражданскому» Wall Street Journal, обозвавшему процесс «инкризингомикой» [13].
Явление масс-спектрометрии белковому народу
Точное измерение молекулярной массы химического соединения — желанная цель аналитической технологии. Действительно, это знание решает многие проблемы, а иногда, при наличии дополнительной информации, обеспечивает идентификацию искомого вещества. Масс-спектрометрия — набор методов, направленных на измерение молекулярной массы соединений. Этот подход развивался еще с конца 19 века, когда сэру Джозефу Джону Томсону удалось создать масс-спектрограф, состоящий из газоразрядной трубки, разделяющей по траекториям движения заряженные частицы с разной молекулярной массой. Затем Артур Дэмпстер разработал... Но стоп! В этой статье невозможно охватить всю историю масс-спектрометрии, да в этом и нет необходимости, поскольку это много раз делалось профессионалами [14]. Мне, биохимику по специальности, уместно будет предложить обзор этой увлекательной области в биологическом контексте, чтобы навести мосты между сложной физической техникой и ее биомедицинским применением.
Нам, биологам, придется поверить, что нет никакого другого способа измерить молекулярную массу, кроме как заставить молекулы двигаться. И сразу же после этого поверим в то, что заставить молекулы двигаться можно только в заряженном виде, то есть обратив их в ионы. Так что первый этап масс-спектрометрического анализа (рис. 5) — это ионизация. Первые методы ионизации были жесткими, так что макромолекулы в них не сохранялись. Успехи масс-спектрометрии в биологии, как будет ясно из дальнейшего, связаны с возможностью ионизировать биомолекулы, не разрушая их. После ионизации анализируемые соединения под воздействием электрического поля надо переместить в детектор, который поставит особенности движения молекул в электромагнитном поле в соответствие их молекулярной массе, а точнее, отношению молекулярной массы и заряда. Проще говоря, если две разные молекулы несут одинаковый заряд, но отличаются по массе, приложение к ним одинакового электрического поля заставит их летать по-разному. Если обучить, то есть откалибровать детектор с использованием стандартов с заведомо известными массами, можно, оценивая движение неизвестных ионов, определять их отношение массы к заряду. Если заряд равен единице (то есть мы имеем дело с однозарядными ионами), отношение численно равно молекулярной массе.
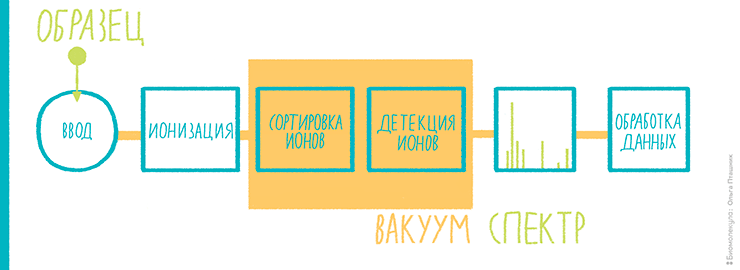
Рисунок 5. Примерная схема масс-спектрометрии для протеомики.
рисунок Ольги Пташник
Масс-спектрометрия сегодня — гигантская область, активно используемая почти во всех областях промышленности, в химии, биологии, медицине, охране окружающей среды. Более того, в Манхэттенском проекте и, вероятно, в советских ядерных проектах при помощи масс-спектрометра обогащали радиоактивный уран, разделяя его на изотопы. На самом крупном форуме по масс-спектрометрии — конференции Американского масс-спектрометрического общества — ежегодно собирается до 15 тысяч участников. Доля биомедицинских методов в масс-спектрометрии продолжает расти вместе с вложениями в биотехнологии в целом.
Проклятие изотопного распределения
Создание масс-спектрометров совпало по времени с обнаружением у химических элементов разных изотопов. Решая химические задачи в школе или проводя различные биологические эксперименты, мы зачастую не задумываемся, что важные элементы, составляющие органические вещества (C, O, N, S), содержат значимую долю стабильных изотопов, отличающихся по массе от номинальных, указанных в таблице Менделеева. Биологи сталкиваются с радиоактивными изотопами, которые еще недавно использовались для мечения биомолекул. Проблему стабильных и радиоактивных изотопов хорошо знают археологи и палеонтологи — с помощью них они датируют свои находки [15]. Но в большинстве молекулярно-биологических экспериментов помнить об этих примесях нет нужды.
Отношение стабильных изотопов для каждого элемента — это некоторое свойство материалов. Интересно, что такие отношения стабильных изотопов различны в разных средах, например, в пресной и морской воде, в горных породах, а также в разные периоды существования Земли и других небесных тел. Поэтому измерение этого параметра в разных условиях вызывает серьезный интерес в разных областях естественных наук [16]. Но для изучения точных масс белков и пептидов в протеомике существование стабильных изотопов — это своего рода проклятие.
Для упрощения предположим, что в составе измеряемого нами соединения имеется примесь только стабильного изотопа углерода — 13С. Его доля в массе общего углерода на планете равна примерно 1%. Таким образом, если в нашей молекуле 10 углеродных атомов, а ее номинальная, по таблице Менделеева, молекулярная масса, скажем, равна 152 атомных единицы массы, лишь каждая десятая молекула будет содержать «тяжелый» атом С. И наша молекула будет иметь молекулярную массу не 152, а около 153 Да. Таким образом, масс-спектрометр от одного соединения зарегистрирует не один пик, а несколько. Первый будет содержать номинальную массу, деленную на заряд (m/z) — при единичном заряде — 152, второй — в 10 раз ниже по интенсивности, которая просто отражает относительное число молекул такой массы, с m/z = 153 Да . Поскольку по статистике будут существовать молекулы с двумя и более «тяжелыми» атомами, их пики также могут находиться в спектре, но из-за малой интенсивности могут не преодолеть чувствительности детектора.
Современные масс-спектрометры способны разрешить пики с разностью молекулярных масс намного меньше 1 Да.
Теперь представим похожее соединение, но оно уже будет включать 100 углеродных атомов. Пусть его номинальная молекулярная масса будет равна 1502 Да. Легко понять, что число молекул, содержащих хотя бы один «тяжелый» атом, в этом случае будет превышать таковое с номинальной молекулярной массой. Из нескольких пиков, которые будут соответствовать разному числу атомов изотопа 13С, самым высоким в этом случае будет уже второй пик, с m/z примерно равным 1503 Да. А что будет, если мы будем снимать масс-спектр соединения размером с небольшой белок, с массой свыше 10 000 Да? Значительное число одинаковых в химическом плане , но разных по изотопному составу молекул образуют целый лес пиков в масс-спектре, причем самый интенсивный из них будет находиться далеко в середине этого набора пиков, а по молекулярной массе он будет значительно отличаться от номинальной, так называемой моноизотопной молекулярной массы соединения (рис. 6). Например, моноизотопная масса сывороточного альбумина крупного рогатого скота, столь любимого биохимиками белкового стандарта, равна 66 389,86 Да, тогда как «средняя» (average) масса, соответствующая самому интенсивному пику на масс-спектре, составляет примерно на 43 Да больше!
Накапливаются сведения о разном поведении стабильных изотопов одного и того же элемента в химических и биологических процессах [16]. Однако в большинстве случаев работает допущение, что свойства соединений одного строения с разным изотопным составом одинаковы.
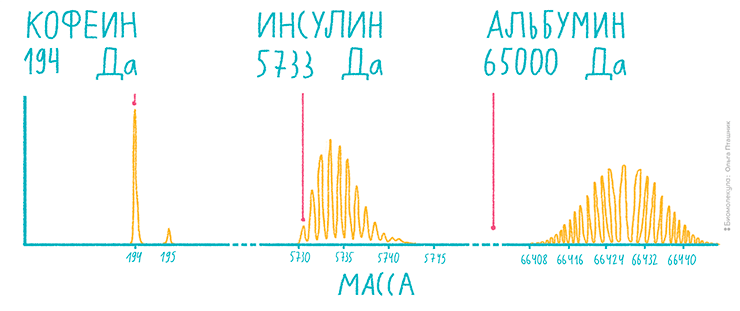
Рисунок 6. Схемы изотопного распределения молекул от низкомолекулярных метаболитов до белка. Чем тяжелее ион, тем ниже интенсивность самого высокого пика. Красной отметкой показана моноизотопная масса — она рассчитывается, как будто в веществе присутствует только основной изотоп.
рисунок Ольги Пташник
Число ионов каждого вещества, перенесенного в детектор масс-спектрометра, очевидно, составляет сумму интенсивностей всех изотопных пиков, относящихся к соединению. Также это количество можно выразить в виде площади под касательной, проходящей по вершинам этих пиков. Представим, что в детектор поступили несколько тысяч ионов аминокислоты массой 150 Да и столько же — белка массой 15 000 Да. Аминокислота даст 2–3 основных, очень высоких пика, причем первый будет самым интенсивным, а белок — несколько десятков, но гораздо более низких, с вершиной этой пологой горки где-то посередине. Ясно, что высокую скалу, стоящую посреди ровной степи, заметить гораздо проще, чем маленький холмик, края которого еще и сливаются с высокой травой — техническим шумом, сопровождающим запись масс-спектра.
Итак, чувствительность масс-спектрометра характеризуется обратной зависимостью от молекулярной массы анализируемого соединения. Чем выше эта масса, тем менее интенсивен максимальный пик среди всех изотопных вариантов соединения. Кроме того, огромное количество этих пиков затрудняет интерпретацию масс-спектра. Вот почему в современной протеомике белки перед анализом чаще всего разлагают на пептиды молекулярной массой 500–2500 Да, обозначая такой подход «протеомикой снизу вверх» (bottom-up). Именно такие пептиды удобно анализировать в масс-спектрометре. Расщепление белков обычно проводят наиболее специфичной из протеаз — трипсином, который с высокой специфичностью осуществляет протеолиз по пептидной связи правее остатков лизина и аргинина. Потребность в расщеплении белков я называю проклятием, поскольку при этом происходит потеря информации. В современных протеомных конвейерах, где такое расщепление производится без предварительного разделения, белки после анализа приходится собирать заново, разумеется, небезошибочно. Ситуация напоминает сборку нуклеотидных последовательностей после секвенирования нового поколения [6], но у последней есть преимущество, поскольку там фрагменты гораздо чаще перекрываются друг с другом.
Несмотря на огромную путаницу в масс-спектрах больших белков, многие исследователи продолжают работу с ними без расщепления. Такой подход называют протеомикой «сверху вниз» (top-down). Для получения качественных масс-спектров целых белков используют мощные детекторы сверхвысокого разрешения. Тем не менее пока не удалось создать метод top-down, надежно и воспроизводимо анализирующий белки в масштабах целого протеома.
Масс-спектрометрия MALDI-TOF и пептидная дактилоскопия
В 1980-е годы в масс-спектрометрии стал развиваться подход к ионизации молекул лазером при их сокристаллизации со светочувствительным органическим веществом — так называемой матрицей. Матрица окружает молекулы анализируемого вещества, а при освещении лазером определенной длины волны она поглощает его энергию, ионизируется сама и способна — по не вполне ясному до сих пор механизму — эффективно ионизировать соседние молекулы вещества. Оказалось, что при некоторых условиях такой вид ионизации — опосредованная матрицей лазерная десорбция-ионизация (matrix-assisted laser desorption ionisation, MALDI) — обеспечивает ионизацию биомолекул без их распада. Как только это стало ясно, метод ворвался в биологию, а одному из его авторов, который первым показал MALDI для белков, — японцу Коити Танаке — в 2002 году присудили Нобелевскую премию по химии . Ионизацию MALDI совместили с простым масс-спектрометрическим детектором — времяпролетным (time-of-flight, TOF), в котором ионы летят в вакуумной трубе, достигая детектора в виде чувствительной к ионам пластины (фотоэлектронного умножителя) (рис. 7). Время, за которое ионы одного заряда преодолеют длину трубки, будет обратно пропорционально их молекулярной массе.
Обычно матрицы представляют собой низкомолекулярные органические кислоты, производные коричной, бензойной и других кислот.
Совместно с Джоном Фенном, применившим для биомолекул ионизацию электрораспылением, и Куртом Вютрихом (вообще за ЯМР). Примечательно, что исследование К. Танаки было обнародовано в виде патента, а его основная статья опубликована в скромном специализированном журнале Rapid Communications of Mass Spectrometry. Сам он — инженер-исследователь в частной компании — не имел ученой степени. Как и во многих других случаях, в присуждении премии Танаке было противоречие. Одновременно большой вклад в применения MALDI для белков внесли немцы Франц Гилленкамп и Михаэль Карас.
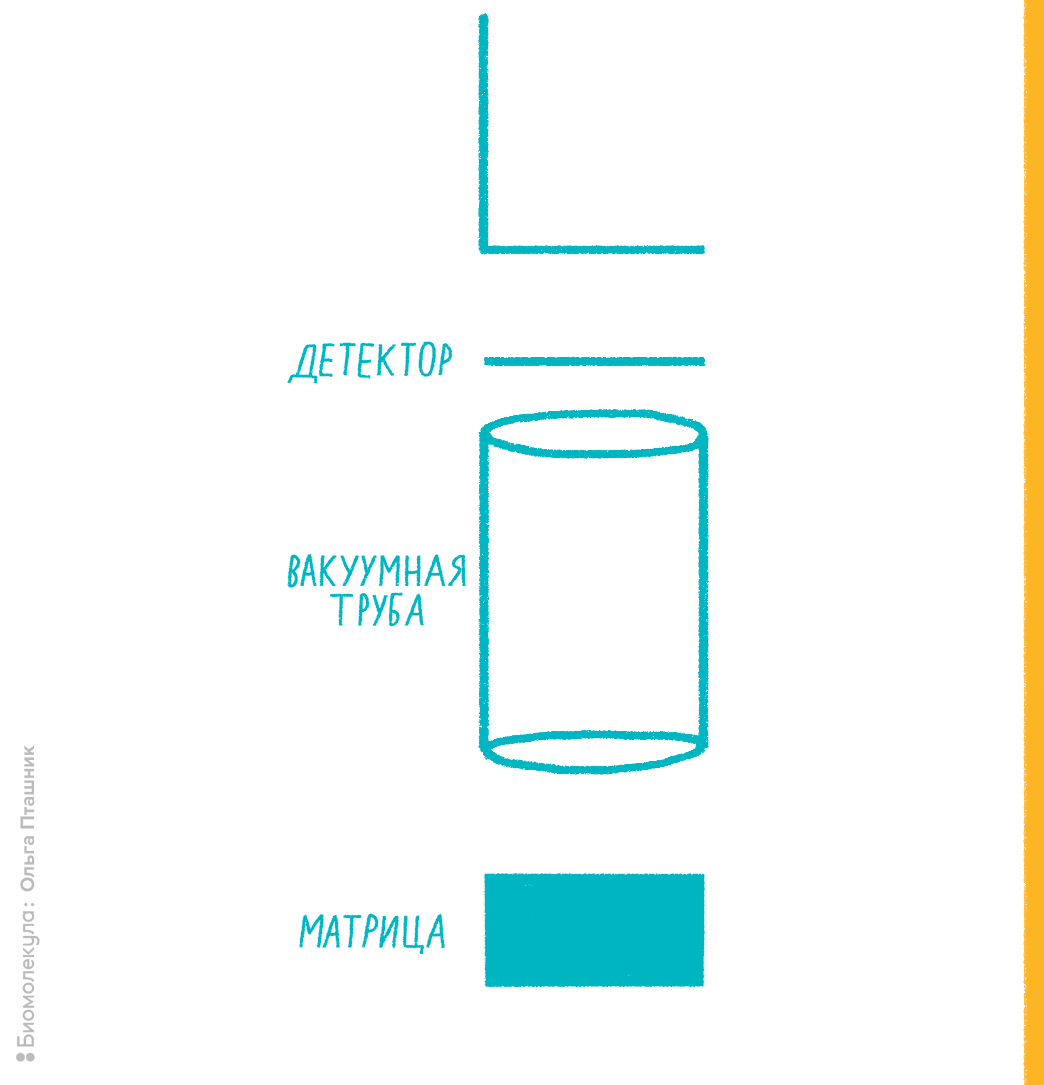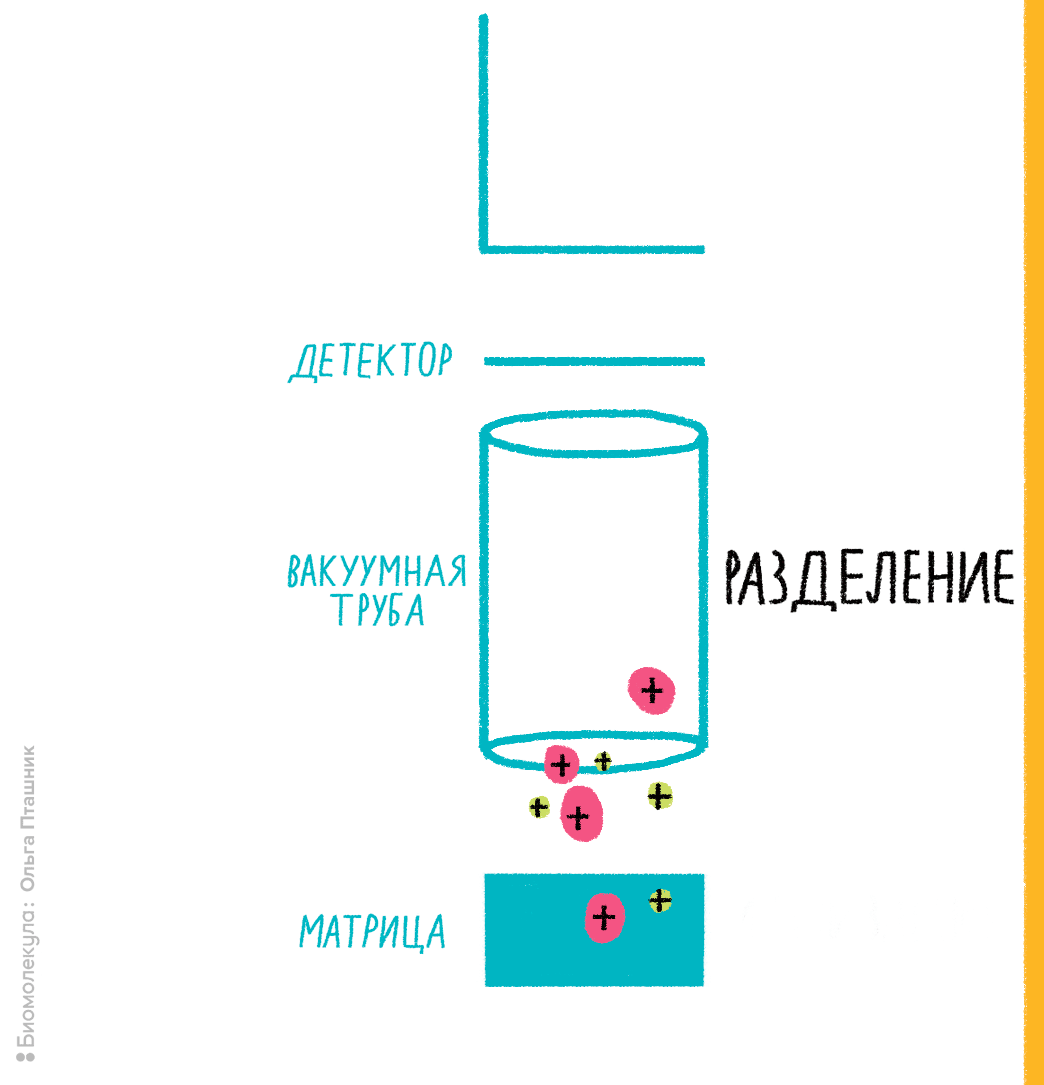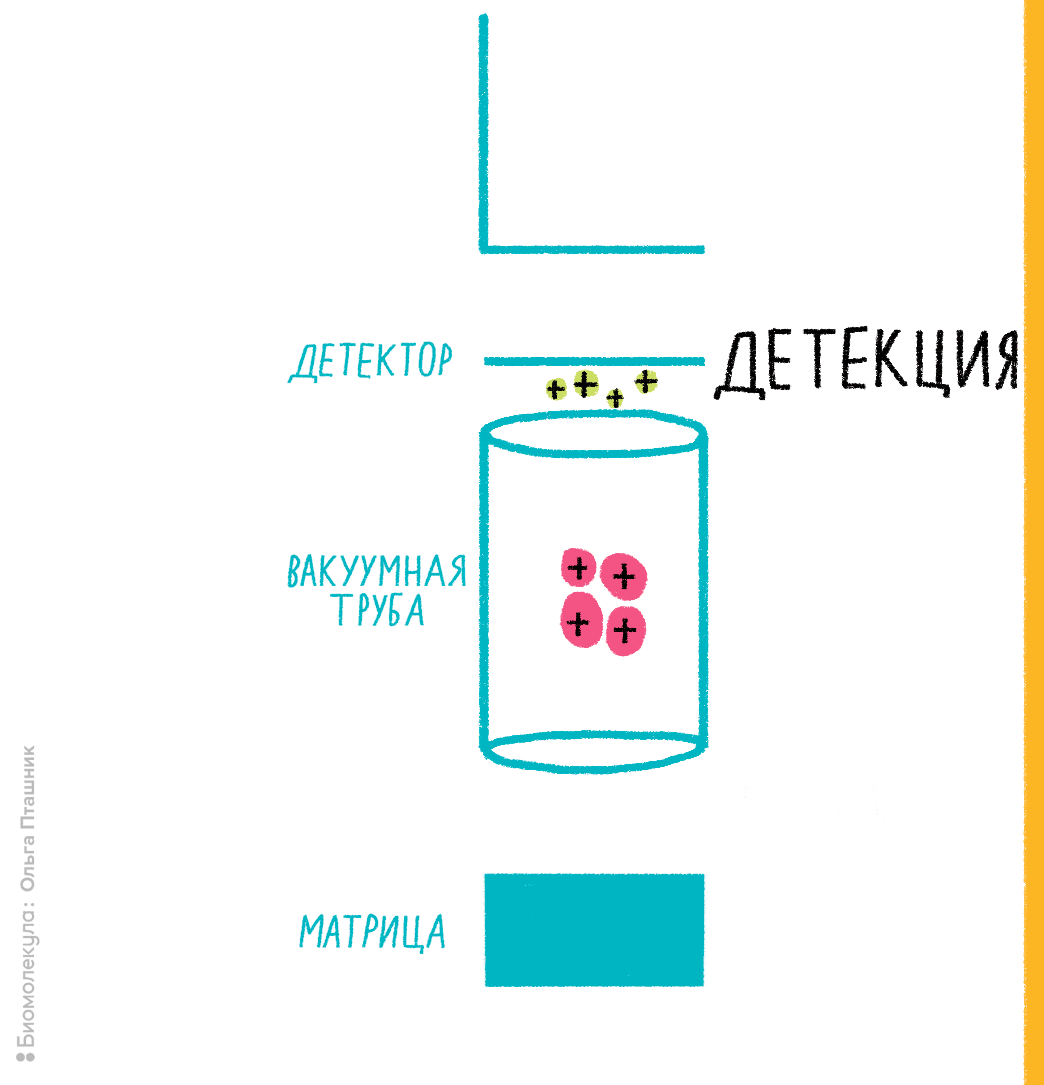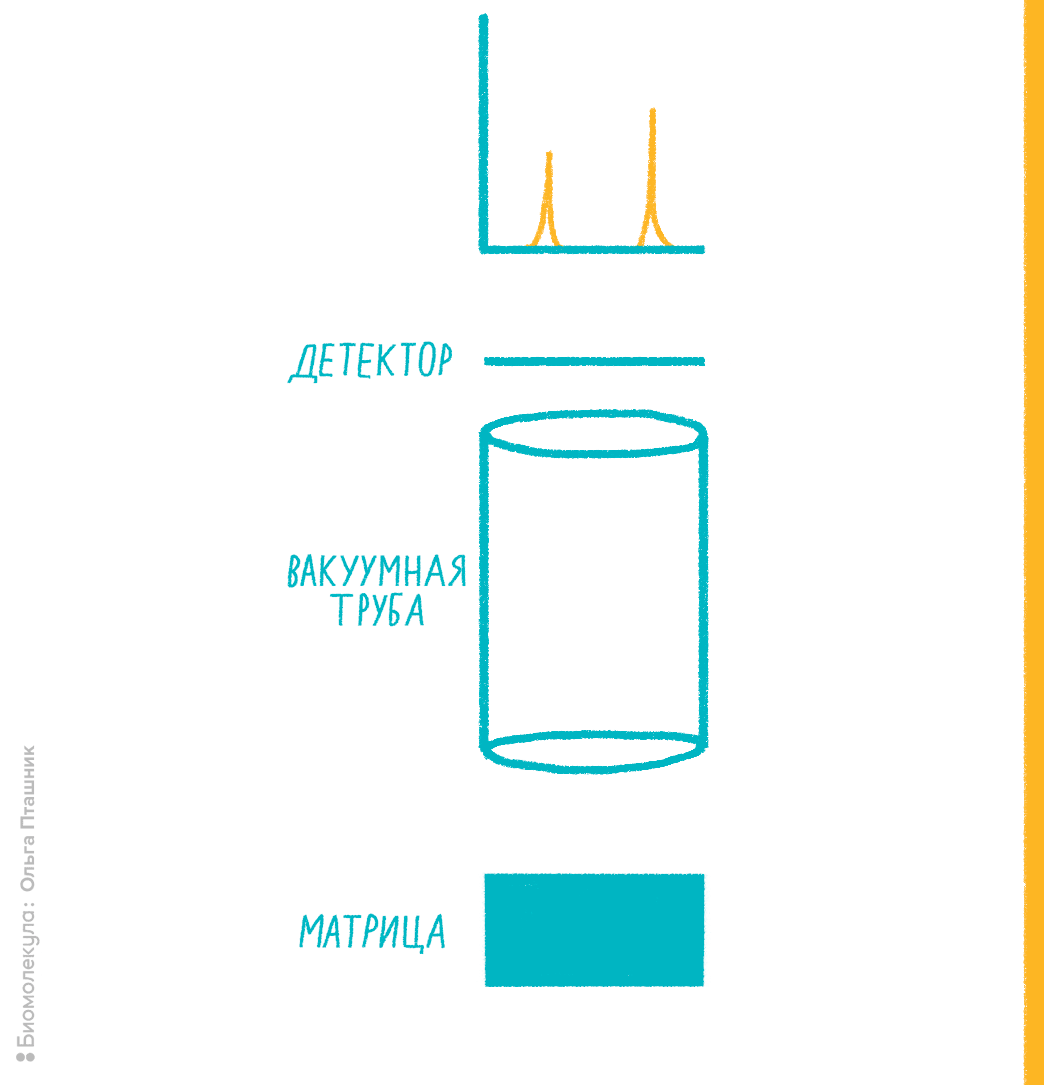
Рисунок 7. Примерная схема MALDI-TOF-масс-спектрометрии.
рисунок Ольги Пташник
В 1990-е и в начале 2000-х простой и надежный MALDI-TOF-масс-спектрометр стал одной из рабочих лошадок протеомики. Как было сказано выше, основным методом разделения белков в масштабах протеома в то время был двумерный электрофорез. Если вырезать из геля пятно с белком некоторой степени очистки и расщепить связанный с гелем денатурированный белок трипсином, совокупность пептидов этого изолированного белка составит более или менее уникальный набор молекулярных масс — по крайней мере, в пределах отдельно взятого протеома. Это происходит, в первую очередь, из-за высокой специфичности трипсина и уникального распределения лизина и аргинина, по которым происходит расщепление, в разных последовательностях. Набор масс пептидов каждого белка напомнил исследователям об использовании для идентификации личности отпечатков пальцев, поэтому новый подход получил название масс-спектрометрического пептидного картирования, пептидного фингерпринта, или, как лучше сказать по-русски, пептидной дактилоскопии (рис. 8) [17].
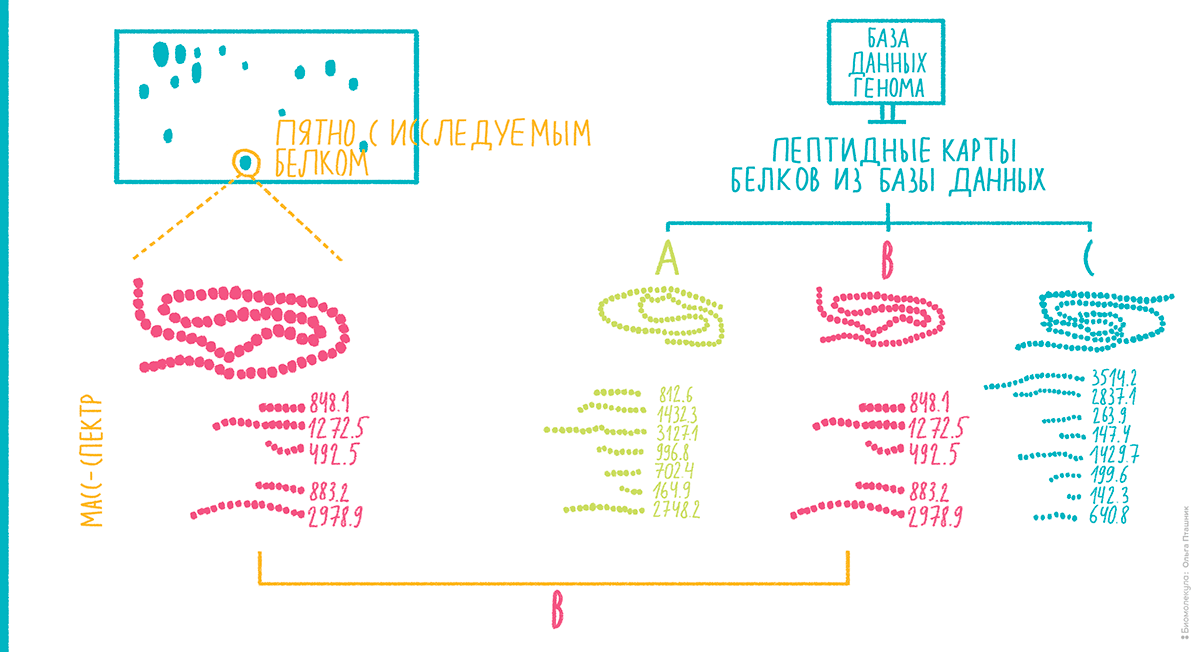
Рисунок 8. Принцип масс-спектрометрического пептидного картирования.
рисунок Ольги Пташник
Идея пептидной карты белка пришла к масс-спектрометрии через развившуюся к тому времени высокоэффективную жидкостную хроматографию [3]. Очищенные белки могли расщепляться протеазой на пептиды, а их анализ на хроматографе давал уникальную пептидную карту. Если определить стандартизированное время выхода каждого из пептидов с хроматографической колонки, по такой хроматографической пептидной карте можно идентифицировать белок [18]. Теперь такой параметр, как время выхода, решено было заменить на более точный и легко формализуемый показатель — определяемое в масс-спектрометре отношения массы к заряду пептидного иона.
Как же формально оценить соответствие наблюдаемого масс-спектра и теоретических представлений о последовательностях белков? Вначале необходимо «расщепить» все белки трипсином виртуально и составить из них базу данных для сравнения со спектрами. Вот тут протеомика и становится постгеномной — ведь без теоретических последовательностей комбинаций станет слишком много, и прогнозирование совпадения перестанет быть возможным. Далее необходим метод оценки вероятности того, что наблюдаемый набор пептидов относится к конкретному белку. В первом воплощении для решения этой задачи использовали подсчет вероятности того, что набор пиков не случаен. Если кто-то помнит из курса статистики, похожие вероятности высчитывают при изъятии из мешка вслепую шаров разных цветов. В нашем случае нужно ответить на вопрос, случайным ли образом насыпались в наш спектр из большого мешка всех возможностей шары-пептиды определенных масс? Если совпадение сильно не случайное, система присваивает нашему спектру высокий вероятностный коэффициент (score).
Одним из первых алгоритмов для пептидной дактилоскопии стал MOWSE [19], который лег в основу широко известной специалистам программы Mascot. Хочу обратить внимание на важный момент в развитии протеомного анализа. С момента введения в обращение метода пептидной дактилоскопии идентификация белков и пептидов превратилась из измерения в предсказание. Таким образом, каждый идентифицированный таким методом белок характеризуется расчетным параметром вероятности того, что это действительно он. Когда мы красим гель антителами, наличие пятнышек на вестерн-блоте ничего подобного нам не скажет. Так протеомика вступила в эпоху «поисковых машин» — программ, которые сравнивают взятые из генома теоретические последовательности с наблюдаемыми масс-спектрами и возвращают вероятность того, что эти спектры получены из соответствующих белков и пептидов.
Пептидная дактилоскопия представляет собой метод анализа расщепленных белков. Параллельно MALDI-TOF стали использовать для исследования целых белков в сложных смесях — в анализе «сверху вниз». Белковые профили крови пациентов с разными заболеваниями, различные бактериальные и эукариотические клетки анализировали целиком и сравнивали полученные масс-спектры в разных группах для целей клинической диагностики и идентификации различных состояний. Масс-спектр использовали как образ, обучая алгоритмы известным случаям, и эффективно распознавая новые случаи. Если применение такого подхода для анализа белков крови в диагностике злокачественных опухолей оказалось недостаточно надежным для внедрения, метод анализа целых бактериальных клеток стал более успешным и сейчас используется в клиниках [20]. Способы, реализуемые на очень простом масс-спектрометре, и прилагаемые к нему специально обученные алгоритмы способны идентифицировать до видов и родов патогенные микроорганизмы, причем анализ MALDI-TOF применяется к целым клеткам бактерий. Их наносят на металлическую мишень масс-спектрометра, покрывают матрицей и облучают лазером для получения специфичных профилей, распознаваемых алгоритмом по характерным массам.
Тандемные масс-спектры и протеомный поиск
Вытаскивание разноцветных шариков из пыльного мешка продолжилось, когда масс-спектрометры научились в мягких условиях фрагментировать пептиды. Внутри некоторых детекторов ионы пептидов и других соединений подвергаются специальным воздействиям, например, столкновению с незаряженными молекулами инертных газов, в результате чего эти ионы диссоциируют, образуя набор фрагментов (подробнее см. на рис. 9). После диссоциации массы фрагментов также можно измерить. Теперь, когда мы научились осуществлять тандемный анализ, или MS-MS, каждый пептид характеризуется массой иона-предшественника, который иногда называют «родительским» ионом, и набором масс ионов-фрагментов («дочерних» ионов).
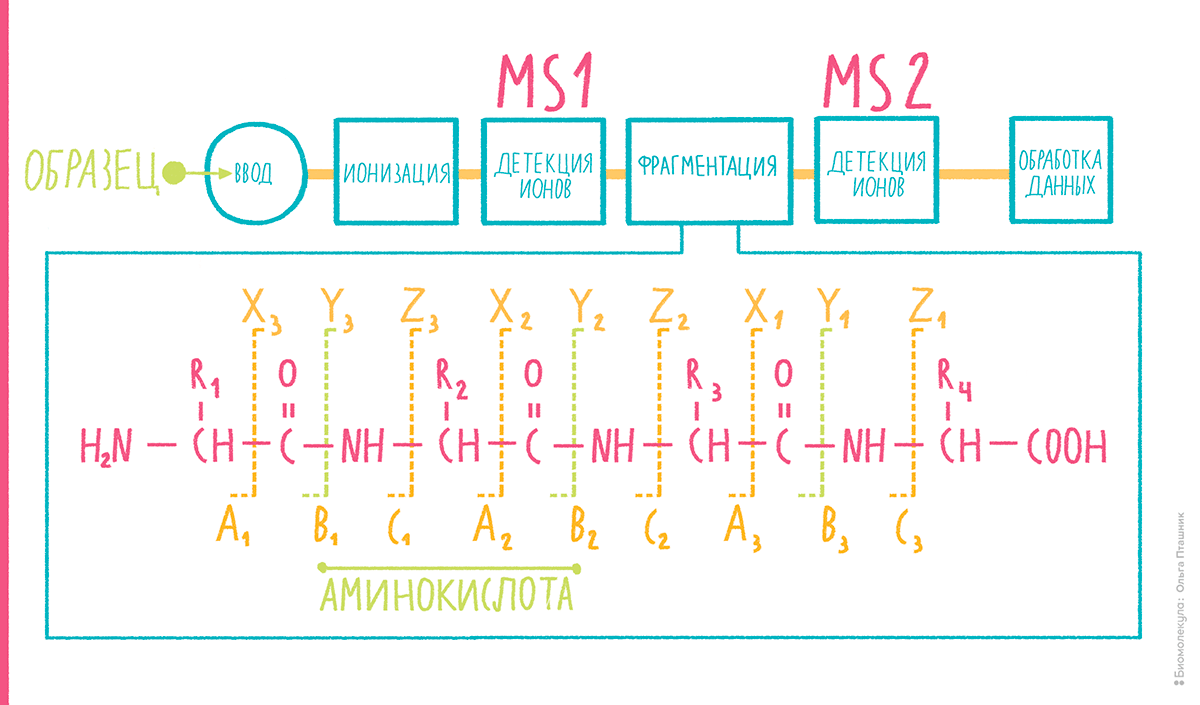
Рисунок 9. Тандемная масс-спектрометрия. Схема метода — вверху. Основные типы фрагментов, которые образуются при диссоциации пептидов внутри масс-спектрометра — внизу. Поскольку пептиды имеют однотипную структуру, при подобранных условиях столкновительной диссоциации они разрушаются по определенным связям, в предсказуемой манере. Пептидная цепь может распадаться по связи между первым и вторым (альфа) атомами углерода в аминокислоте, образуя a- и x-ионы справа и слева от разорванной связи. Аналогично, при разрыве пептидной связи возникают b- и y-ионы, а при разрыве связи между атомом азота и α-атомом углерода — соответственно, с- и z-ионы.
рисунок Ольги Пташник
Итак, из последовательности пептида можно предположить молекулярные массы образующихся из него при диссоциации в масс-спектрометре фрагментов основных типов. Подобно тому, как при пептидной дактилоскопии последовательность белка делится на пептиды, и массы теоретических пептидов сопоставляются с наблюдаемыми в спектре, здесь можно сопоставить теоретические фрагменты каждого виртуального пептида с наблюдаемыми пиками тандемного масс-спектра. Иначе говоря, весь кодирующий геном in silico делится на пептиды с помощью, например, трипсина, для каждого из них по известным эмпирическим правилам строится теоретический спектр фрагментации. Теперь такие теоретические масс-спектры можно сопоставить с настоящими и рассчитать каким-либо образом вероятность того, что это именно тот пептид. Единицей предсказания последовательностей из масс-спектров теперь становится пара теоретический пептид — настоящий спектр (peptide-spectrum match, PSM). Очевидно, что многие спектры, особенно в случае большого протеома (например человеческого), могут образовывать пары с несколькими теоретическими пептидами, из которых надо выбрать лучшие.
Создание поисковых машин для тандемной масс-спектрометрии — огромная область, и таких инструментов разработаны десятки. Среди них, к счастью, есть программы с открытым кодом, и я сторонник использования в науке именно такого софта . Разбираться в тонкостях отсева правильных PSM в этой статье мы вряд ли сможем. Скажу только, что существенным достижением протеомного поиска по тандемным масс-спектрам стало изобретение в 2007 году подхода target-decoy (рис. 10) [21], в котором к настоящим, геномным теоретическим пептидам (целевые — target) при интерпретации масс-спектров стали добавлять равное количество специально образованных бессмысленных, поддельных пептидов (фальшивые — decoy). Когда среди лучших PSM алгоритм начинает выдавать совпадение с заведомо несуществующими пептидами, мы можем остановить процесс и определить уровень ложноположительных результатов (FDR) в наших протеомных данных. То есть в наших предсказаниях всегда есть небольшая примесь вранья, что неизбежно при протеомных поисках такого типа. Нормально то, что мы хотя бы можем оценить долю ложных идентификаций.
Не хотелось бы очернять чей-то бизнес, но использовать в науке дорогой инструмент, не зная, как он работает, на мой взгляд, противоречит самой идее развития научной мысли.
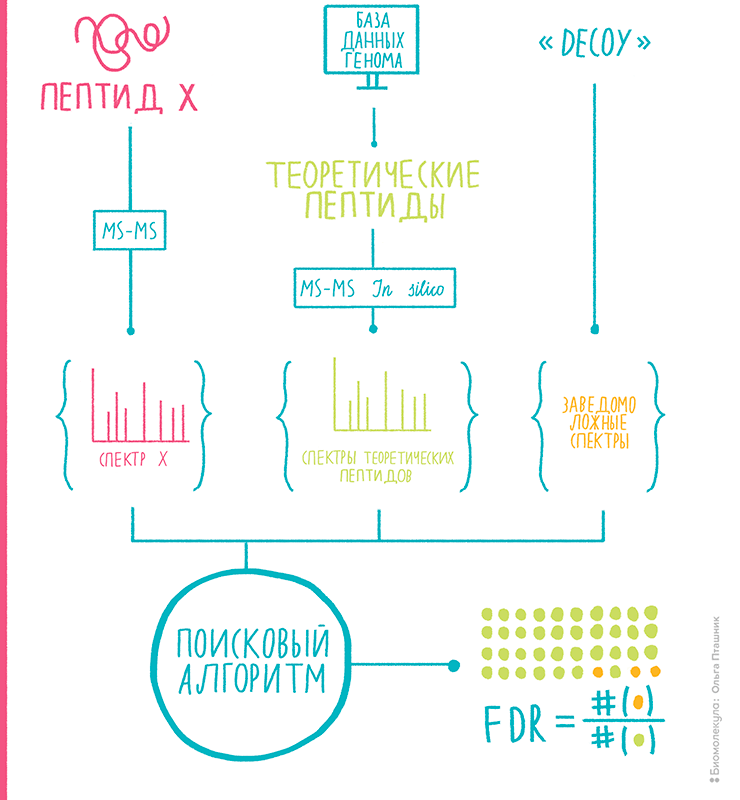
Рисунок 10. Принцип подтверждения результатов протеомного поиска target-decoy. Проверка гипотез о совпадении настоящего спектра с теоретическим приводит к образованию пар спектр—пептид (PSM). Алгоритм поиска приписывает каждому реальному спектру лучший, по его мнению, пептид. Но авторы метода схитрили — они добавили к теоретическим спектрам настоящих пептидов (target) фальшивые, заведомо неподходящие (decoy). И вот когда спектрам начинают соответствовать decoy-пептиды, это явные ошибки, то есть ложноположительные результаты. Мы ждем, когда доля PSM с этими decoy — так называемый уровень ложноположительных результатов (FDR) — достигает определенной величины (обычно, это 1%), и поиск прекращаем. Теперь мы примерно знаем, сколько ошибок у нас среди «правильных» идентификаций PSM, ведь вероятность ошибиться в сторону target равно таковой в сторону decoy.
рисунок Ольги Пташник
Изобретение приемлемого MS/MS пептидов и появление способов обработки таких данных дала возможность для доставки в масс-спектрометр смесей пептидов без разделения целых белков. То есть появилась возможность расщепить все белки в образце протеазой и оперировать уже совокупностью пептидов, а не белков. Появилась протеомика-«дробовик» (shotgun proteomics), которую для благозвучия, в ущерб точности, переводят на русский язык как скорострельную или панорамную.
Ионизация электрораспылением и скорострельная протеомика
Одним из обладателей «нобелевки» 2002 года, о которой я упоминал выше, стал американский химик Джон Фенн. Ранее он предложил использовать в масс-спектрометрии метод ионизации электрораспылением, или, как его еще называют, электроспреем (electrospray ionisation, ESI). При приложении высокого напряжения к выходящей из конического капилляра жидкости она превращается в аэрозоль, а при испарении жидкости из частиц аэрозоля (например, в потоке инертного газа) электрический заряд может переходить на растворенные в этом аэрозоле биомолекулы. Так обеспечивается мягкая ионизация при атмосферном давлении, которая почти не фрагментирует высокомолекулярные соединения, в отличие от многих бытовавших ранее способов ионизации. Не лишенный британского чувства юмора Фенн в своих статьях и лекциях аллегорически сравнивал биомолекулы, которые он заставил воспарить с помощью своего метода, с летающими слонами (рис. 11).

Рисунок 11. Молекулярный слон, притаившийся в конусе для ионизации электрораспылением.
рисунок Ольги Пташник
Ионизация электрораспылением оказалась необычайно удобной для совмещения двух важных методов аналитической биохимии — высокоэффективной жидкостной хроматографии и масс-спектрометрии [3]. Теперь поток хроматографической фазы с аналитической колонки можно было направить в конус для электрораспыления, или организовать такой конус на конце колонки, и применять масс-спектрометр как анализатор разделяемых в колонке молекул. Возможность осуществлять тандемную масс-спектрометрию вместе с развитием протеомного поиска с середины 2000-х сделали комбинацию методов под многоэтажной аббревиатурой HPLC-ESI-MS/MS, или просто LC-MS/MS, предпочтительным способом исследования протеома. Это и есть та самая скорострельная протеомика (рис. 12). Небольшое огорчение доставило то, что для ее осуществления, как правило, приходится расщеплять целый протеом или его фракции на трипсиновые пептиды с потерей при этом информации о целостных белках. Однако бонусов от введения этого подхода оказалось намного больше.
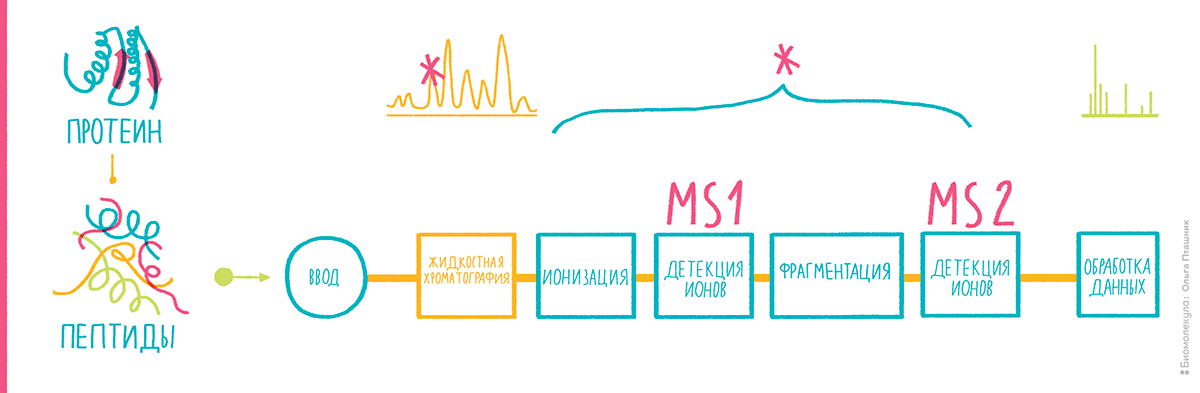
Рисунок 12. Схема скорострельной протеомики.
рисунок Ольги Пташник
Большим подспорьем для роста информативности скорострельной протеомики оказалась «демократизация» масс-спектрометров с высоким разрешением. Ранее для особо высокого разрешения и точности определения требовалось построение приборов ионно-циклотронного резонанса с преобразованием Фурье, в которых использовали могучие сверхпроводящие магниты с индукцией магнитного поля свыше 7 Тесла. В последнее десятилетие детекторы других типов достигли сопоставимых показателей. Примерами таких приборов служат гибридные детекторы разных производителей, например, квадрупольно-времяпролетные масс-спектрометры (Q-TOF). Главенствующее положение среди доступных масс-спектрометров занимает появившаяся на рынке в 2005 году ионная ловушка особого типа — Orbitrap (рис. 13). Приятную гордость пробуждает тот факт, что создатель этой ловушки — российский физик, работающий в компании Thermo, выпускник МИФИ Александр Макаров [22].
Действие патента на Orbitrap, принадлежащего компании Thermo, в ближайшее время истечет, так что мы можем ожидать дальнейшего снижения цены на детекторы этого типа.
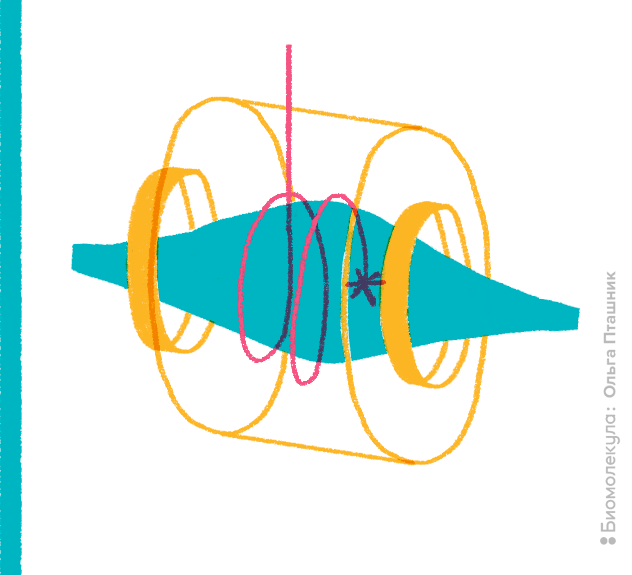
Рисунок 13. Ионная ловушка Orbitrap.
рисунок Ольги Пташник
Точность определения молекулярной массы в запусках рутинного протеомного анализа достигла показателя 5 частей на миллион (то есть 0,0005%) и ниже. Это привело к значительному прогрессу в числе идентифицируемых таким образом белков протеома. Сегодня лучшие научные группы сообщают об идентификации методом скорострельной протеомики в клеточных линиях и тканях человека белковых продуктов 9–10 тысяч генов, то есть почти половину всего кодирующего генома [23]. Справедливо указать, что эти цифры достигаются на уровне 1% ложноположительных результатов.
Количественный анализ и изотопные метки
Просто идентифицировать белки протеомным методом в большинстве случаев явно недостаточно. Для познания механизмов биологических процессов необходимы количественные данные, обеспечивающие сравнение протеомов клеток и тканей в разных состояниях. Проще всего проанализировать некоторые показатели получившихся в ходе запусков системы LC-MS/MS хроматограмм, снабженных спектральными данными. Такой подход называется безметковым (label-free quantitation, LFQ), поскольку не требует специальных модификаций метода пробоподготовки. Например, в контрольном образце от всех пептидов некоторого белка зарегистрировано 200 спектров, а в опытном — 400. Можно предположить, что число записанных спектров пропорционально концентрации белка в образце. Для сравнения используют и другие параметры спектров, например, нормированные значения интенсивности сигнала. Безметковый количественный анализ по протеомным данным привлекателен своей простотой, и для него выработано большое количество программных решений, в том числе бесплатных программ с открытым доступом, среди которых сегодня наиболее популярен разработанный группой Матиаса Манна из Германии пакет MaxQuant. Безметковые методы неточны и полуколичественны, а находки, сделанные с их помощью, требуется проверять другим способом, например, с использованием вестерн-блоттинга [4].
Иное дело, когда один из анализируемых образцов или все они подвергаются мечению с помощью тех же стабильных изотопов, о которых уже шла речь выше. Тогда в масс-спектре пики одной и той же химической природы, но содержащие разное количество стабильных изотопов, будут стоять в спектрах рядом, разделенные расстоянием по оси m/z в зависимости от использованной метки. Мы можем сравнить интенсивность стоящих рядом пиков опыта и контроля и точно рассчитать отношение концентраций соответствующих пептидов и белков.
Разработано большое количество технических решений, позволяющих проводить изотопное мечение. В случае, когда возможно культивирование клеток в искусственных средах, можно пометить все клетки одной группы анализа, используя изотопно меченные среды. В ряде случаев метки вводят при расщеплении трипсином. Существуют метки, проявляющие себя на ионах-предшественниках, а также на ионах фрагментов. Последние зачастую позволяют проводить количественный анализ в мультиплексном режиме, например, набор TMT-меток от компании Thermo предоставляет одновременную обработку разными метками 11 образцов! Использование изотопных меток существенно повышает аналитическую точность количественного анализа, который в ряде случаев может стать абсолютным, то есть определять точные концентрации анализируемых соединений. Однако существенный недостаток в этом случае — цена анализа .
Стоимость набора для мечения нескольких образцов может примерно составлять средний размер гранта РФФИ (!) — для тех, кто знает.
Направленный анализ пептидов — мониторинг множественных реакций
Наконец стоит упомянуть о методе протеомного анализа, который по своей функции составляет конкуренцию методам определения белков при помощи антител. Когда мы знаем, какой пептид, характеризующий целый белок, хотим измерить, можно настроить масс-спектрометр так, чтобы он видел, по сути, только этот пептид. Таким образом, работу проводят в направленном (таргетном) режиме. Для этого используют прибор с детектором типа тройного квадруполя. По сути, это три одинаковых масс-спектрометра, стоящих друг за другом и передающих друг другу ионы. В первом отфильтровывается нужный ион-предшественник, то есть интересующий нас пептид, во втором — куда попал только наш «товар» — он проходит фрагментацию, а третий регистрирует 3–5 выбранных нами заранее фрагментов. По интенсивности фрагментов осуществляется количественный анализ.
Подход известен из анализа низкомолекулярных соединений и начал широко использоваться для пептидов в середине 2000-х под названием «мониторинг множественных реакций» (multiple reaction monitoring, MRM), или «мониторинг выбранных реакций» (selected reaction monitoring, SRM) (рис. 14) [24]. Такой метод не подходит для обнаружения новых явлений в протеоме, но зато обеспечивает надежный количественный анализ, особенно с использованием синтетических изотопно меченных стандартов для интересующих пептидов. MRM позволяет осуществлять анализ нескольких пептидов за один запуск LC-MS/MS. Он позиционируется как «масс-спектрометрический иммунный анализ» и в настоящее время ищет свое место не только в науке, но и в клинической и биотехнологической практике.
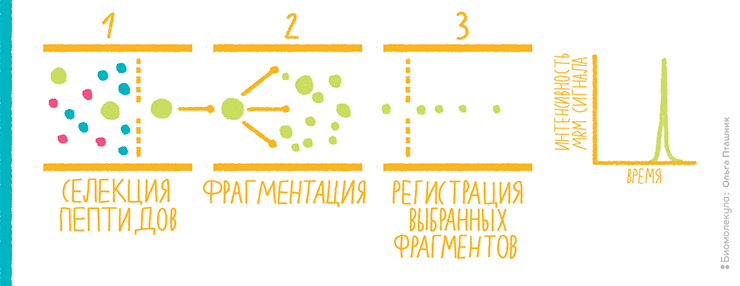
Рисунок 14. Мониторинг множественных реакций в приборе типа тройного квадруполя.
рисунок Ольги Пташник
Протеомика с помощью антител и других связующих молекул
С совершенствованием точного нанесения жидкости на подложку, иными словами, печати микрочипов различных типов, иммунные тесты на белки смогли стать миниатюрными. Одновременно с чипами для гибридизации нуклеиновых кислот возникло множество технических решений размещения на твердых подложках сотен и более антител к различным белкам. Такое умножение известных иммунных тестов в сочетании с различными остроумными методами визуализации связывания белка-мишени перевело известный до этого анализ в режим протеомного. Детектирование белков в множественном режиме с помощью специфичных связывающих молекул (например, антител и их фрагментов) к настоящему моменту приобрело столь разнообразные формы, что для их описания, наверное, требуется создать отдельный материал. Я не специалист в области белковых микрочипов, поэтому предоставлю сделать это кому-нибудь еще.
Следует упомянуть несколько технических решений в этой области. В нашей стране группа под руководством А.Д. Мирзабекова около 20 лет назад создала микрочипы на основе гидрогеля, в том числе для анализа белков [25], причем эта технология до сих пор развивается в Институте молекулярной биологии РАН. Альтернативой антителам для мультиплексного анализа белков служат аптамеры — связующие олигонуклеотиды. На основе химически модифицированных аптамеров американская компания Somalogic создала микрочипы для анализа более тысячи белков человека. Такие чипы всё чаще используют для поиска биомаркеров, как альтернатива масс-спектрометрической протеомике.
Если говорить об антителах для анализа белков в масштабе целого генома, то нельзя не упомянуть мощный по объему шведский проект под руководством Матиаса Улена — «Атлас белков человека». В ходе этого проекта к большинству человеческих белков получили антипептидные поликлональные антитела, которыми затем покрасили большое количество тканей и клеток. Ценой существенных усилий создали большую базу данных, содержащую диаграммы и изображения, иллюстрирующие синтез большинства белков генома в различных органах и тканях [26].
Протеомика и практическая медицина
Из всего сказанного выше понятно, что как технология идентификации белков протеомика за пару десятилетий впечатляюще продвинулась. Однако запросы общества в молекулярной биологии связаны с появлением какой-то полезной продукции. Что же произошло на этом поле? Ранние работы по прямому анализу компонентов крови для диагностики заболеваний, в особенности, злокачественных опухолей, не привели к впечатляющим результатам. Однако, судя по последним открытиям в биологии рака, под сомнением оказалась сама возможность эффективного использования биохимических маркеров для его первичной, ранней диагностики. Но это, в общем, другая история. Выше я упоминал об успехе масс-спектрометрии MALDI-TOF для распознавания бактериальных клеток по профилю анализа их целыми, без предварительной обработки. Несколько альтернативных решений для идентификации патогенных бактерий в этой сфере сегодня используют в клинике (рис. 15).
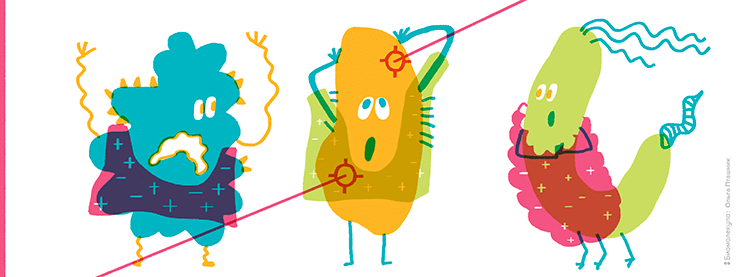
Рисунок 15. Бактериальные клетки, освещенные лучами лазера и покрытые «шубой» из кристаллов фоточувствительной матрицы, распознаются по масс-спектру различных белков в их составе.
рисунок Ольги Пташник
Что касается диагностики опухолей, то протеомика ищет себя в обнаружении биомаркеров для прогностических тестов. Имеется в виду ситуация, когда опухоль уже обнаружена, и требуется определить степень ее злокачественности для того, чтобы правильно выбрать стратегию лечения. Так, ранние попытки анализа MALDI-TOF компонентов крови без их обработки внесли свой вклад в появление разрешенного американскими регуляторами мультиплексного теста OVA1 для прогноза злокачественности опухоли яичника. По справедливости отметим, что сам тест осуществляется уже не с помощью масс-спектрометрии, а иммунными методами. В Северной Америке также разрешен тест Xpresys Lung, который уже использует в своей работе таргетную масс-спектрометрию нескольких белков плазмы крови. Этот тест малоинвазивным путем оценивает степень злокачественности найденных методами визуализации узелков в легких.
Здесь я не ставлю цель оценить, насколько широко используются эти вспомогательные тесты, успешны ли они коммерчески. Обращают на себя внимание факты доведения протеомных экспериментов до продуктов медицинского назначения.
И конечно, не нужно забывать тот вклад в биологию и медицину, который внесли протеомные методы, использованные в многочисленных фундаментальных исследованиях. Настороженность, связанная с недоверием к новой технологии, давно осталась в прошлом, и масс-спектрометрия белков или белковые микрочипы давно стали завсегдатаями страниц самых цитируемых научных журналов. Конечно, методы сами по себе, как бы они ни были хороши, не могут заменить научную мысль. Но когда биологи, способные ее сгенерировать, сочли протеомные методы достаточно надежными, последние начали приносить очевидную пользу в решении сложных фундаментальных задач [27–29].
Что дальше?
В конце полагается очертить перспективы того направления, о котором пишешь. Сивилла из меня не очень — что будет дальше, на самом деле, я не знаю. Но скажу. Все омиксы ждет демократизация — техника станет работать еще лучше, а стоимость анализа будет снижаться [12]. Конечно, с масс-спектрометрией дело не дойдет до решений, похожих на секвенатор нуклеиновых кислот на основе нанопоры, который уже стоит совсем небольших денег. Все-таки там требуется вакуум, который создают насосом. Ну и прочая электроника.
Пробы будут подвергаться сразу нескольким видам омиксного анализа. Даже сегодня в некоторых областях, например при молекулярной характеристике опухолей, образцы стремятся обследовать всесторонне [30], в режиме так называемой протеогеномики. Это нужно для улучшенной классификации образцов, которая может обеспечить более эффективное управление заболеванием.
Снятие молекулярных показателей с гомогенизированных тканей, содержащих миллионы или, как минимум, тысячи клеток, напоминает оценку средней температуры по больнице. Если среди тысяч клеток десятки содержат уникальные белки, важные мишени для лекарств, биомаркеры или другой функционал, при таком анализе сигнал от них просто потеряется. Поэтому должна развиваться протеомика единичных клеток. Надо отметить, что ей гораздо сложнее это сделать, чем, например, транскриптомике, поскольку сигнал от белков нельзя амплифицировать, как нуклеиновые кислоты в полимеразных цепных реакциях.
Что ещё важно: уже сейчас данные, которые получает масс-спектрометр, очень велики — это, похоже, Big Data. И они явно недостаточно интерпретированы. Тенденция ближайшего времени — повышение информационного содержания протеомных данных. В немного переосмысленном виде будет действовать пословица: «Двое с сошкой (люди, проводящие эксперименты и получающие данные), семеро с ложкой (компьютерщики, которые эти данные обрабатывают)». А первых двоих лучше вообще убрать, пусть за них работают роботы. Айтишники будут лежать на пляже с ноутбуками (они это любят) и посылать мне обработанные данные, а я — сидеть на завалинке где-нибудь в русской деревне (это уже люблю я) и писать о протеомах.
И последнее. Исследователи — народ строгий. Потому ожидаю некоторые критические замечания к этому тексту, который ни в коем случае не всеобъемлющ. Возможно, в статье также притаились фактические и технические ошибки. Прошу всех высказывать свое мнение в комментариях и лично, мы открыты к сотрудничеству и обязательно поправим текст в случае обоснованной критики.
Календарь
На основе статей спецпроекта мы решили сделать календарь «12 методов биологии» на 2019 год. Эта статья представляет июнь.
Литература
- Моноклональные антитела;
- 12 методов в картинках: полимеразная цепная реакция;
- 12 методов в картинках: очистка молекул и разделение смесей;
- 12 методов в картинках: иммунологические технологии;
- Bast R.C. Jr., Feeney M., Lazarus H., Nadler L.M., Colvin R.B., Knapp R.C. (1981). Reactivity of a monoclonal antibody with human ovarian carcinoma. J. Clin. Invest. 68, 1331–1337;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- van Noorden R., Maher B., Nuzzo R. (2014). The top 100 papers. Nature News;
- Arnold L. Shapiro, Eladio Viñuela, Jacob V. Maizel. (1967). Molecular weight estimation of polypeptide chains by electrophoresis in SDS-polyacrylamide gels. Biochemical and Biophysical Research Communications. 28, 815-820;
- O'Farrell P.H. (1975). High resolution two-dimensional electrophoresis of proteins. J. Biol. Chem. 250, 4007–4021;
- Sergei A. Moshkovskii, Marina V. Serebryakova, Konstantin B. Kuteykin-Teplyakov, Olga V. Tikhonova, Eugene I. Goufman, et. al.. (2005). Ovarian cancer marker of 11.7 kDa detected by proteomics is a serum amyloid A1. Proteomics. 5, 3790-3797;
- Геном человека: как это было и как это будет;
- «Омики» — эпоха большой биологии;
- Hotz R.L. (2012). Here's an omical tale: scientists discover spreading suffix. Wall Street Journal;
- Лебедев А.Т. Масс-спектрометрия в органической химии. М.: «БИНОМ», 2003. — 493 с.;
- Кузьмин Я.В. (2011). Радиоуглеродный метод и его применение в современной науке. «Антропогенез.ру»;
- Xueshu Xie, Roman A. Zubarev. (2017). On the Effect of Planetary Stable Isotope Compositions on Growth and Survival of Terrestrial Organisms. PLoS ONE. 12, e0169296;
- Shevchenko A., Jensen O.N., Podtelejnikov A.V., Sagliocco F., Wilm M., Vorm O. et al. (1996). Linking genome and proteome by mass spectrometry: large-scale identification of yeast proteins from two dimensional gels. Proc. Natl. Acad. Sci. USA. 93, 14440–14445;
- Kathryn L. Stone, James I. Elliott, Glenn Peterson, Walter McMurray, Kenneth R. Williams. (1990). [21] Reversed-phase high-performance liquid chromatography for fractionation of enzymatic digests and chemical cleavage products of proteins. Mass Spectrometry. 389-412;
- D.J.C. Pappin, P. Hojrup, A.J. Bleasby. (1993). Rapid identification of proteins by peptide-mass fingerprinting. Current Biology. 3, 327-332;
- Elena N. Ilina, Alexandra D. Borovskaya, Maja M. Malakhova, Vladimir A. Vereshchagin, Anna A. Kubanova, et. al.. (2009). Direct Bacterial Profiling by Matrix-Assisted Laser Desorption−Ionization Time-of-Flight Mass Spectrometry for Identification of Pathogenic Neisseria. The Journal of Molecular Diagnostics. 11, 75-86;
- Joshua E Elias, Steven P Gygi. (2007). Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Meth. 4, 207-214;
- Alexander Makarov. (2000). Electrostatic Axially Harmonic Orbital Trapping: A High-Performance Technique of Mass Analysis. Anal. Chem.. 72, 1156-1162;
- Stefka Tyanova, Reidar Albrechtsen, Pauliina Kronqvist, Juergen Cox, Matthias Mann, Tamar Geiger. (2016). Proteomic maps of breast cancer subtypes. Nat Comms. 7, 10259;
- Leigh Anderson, Christie L. Hunter. (2006). Quantitative Mass Spectrometric Multiple Reaction Monitoring Assays for Major Plasma Proteins. Mol Cell Proteomics. 5, 573-588;
- Rubina A.Y., Dementieva E.I., Stomakhin A.A., Darii E.L., Pan'kov S.V., Barsky V.E. et al. (2003). Hydrogel-based protein microchips: manufacturing, properties, and applications. Biotechniques. 34, 1008–1014, 1016–1020, 1022;
- M. Uhlen, L. Fagerberg, B. M. Hallstrom, C. Lindskog, P. Oksvold, et. al.. (2015). Tissue-based map of the human proteome. Science. 347, 1260419-1260419;
- Bing Zhang, Jing Wang, Xiaojing Wang, Jing Zhu, Qi Liu, et. al.. (2014). Proteogenomic characterization of human colon and rectal cancer. Nature. 513, 382-387;
- M. Raschle, G. Smeenk, R. K. Hansen, T. Temu, Y. Oka, et. al.. (2015). Proteomics reveals dynamic assembly of repair complexes during bypass of DNA cross-links. Science. 348, 1253671-1253671;
- Johannes Stadlmann, Jasmin Taubenschmid, Daniel Wenzel, Anna Gattinger, Gerhard Dürnberger, et. al.. (2017). Comparative glycoproteomics of stem cells identifies new players in ricin toxicity. Nature. 549, 538-542;
- Philipp Mertins, D. R. Mani, Kelly V. Ruggles, Michael A. Gillette, Karl R. Clauser, et. al.. (2016). Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534, 55-62.







