
Рыбалка на пришельцев: ДНК-баркодинг помог идентифицировать инвазивные виды рыб в Волжском бассейне
25 февраля 2022
Рыбалка на пришельцев: ДНК-баркодинг помог идентифицировать инвазивные виды рыб в Волжском бассейне
- 607
- 0
- 1
— Вы рыбов продаете?
— По ДНК идентифицируем!
иллюстрация авторов статьи
-
Авторы
-
Редакторы
Слова «мигрант» и «захватчик» надежно вошли в лексикон современного человека, и нам кажется, что Атилла, Чингисхан и Тамерлан — уникальные элементы человеческой культуры. Однако, и в мире животных подобные «захватчики» — совсем не редкость, а расселение видов за пределы исторических ареалов — естественный процесс. Правда, если последовательное расселение животных может занять тысячи лет (как, например, расселение по Земле самого человека), то сейчас вселенцы могут очень быстро попасть в новые места, «воспользовавшись» транспортными потоками цивилизации (случайный завоз колорадского жука из Америки в Старый Свет) или антропогенной трансформацией среды обитания (создание системы водохранилищ на Волге, приведшее к изменению речной системы и расселению черноморско-каспийской тюльки или бычковых рыб на север). Ключевая задача для мониторинга процессов биологических инвазий — быстро и точно определить вселенца. Для успешной идентификации чужеродных видов рыб Волжско-Камского региона мы предлагаем использовать простой и дешевый протокол ДНК-идентификации, оптимизированный именно для российских пресноводных рыб.
«О, кто, скажи ты мне, кто ты...»
Денис Давыдов (1834)
Процессы расселения видов за пределы исторических ареалов, по-видимому, происходили с самого начала возникновения жизни. Однако в естественных условиях расселение животных лимитируется радиусом их индивидуальной активности, который для большинства видов относительно невелик. Например, чтобы расселиться по Северной Евразии мамонтам, в отличие от милых персонажей мультфильма «Ледниковый период», пришлось пройти своими ногами много тысяч километров, а весь процесс растянулся на тысячи лет. Для летающих динозавров и их более поздней ветви — птиц, преодолевать большие расстояния было проще. При этом в воздушной среде есть свои опасности и ограничения, и, конечно, везде имеются хищники и паразиты, готовые полакомиться незадачливыми переселенцами, усложняя и без того их непростой путь. Поэтому многие животные и растения предпочитают осваивать новые местообитания, пользуясь «общественным транспортом», например, прикрепляя свои семена или покоящиеся яйца на других животных. Таким способом для сухопутных обитателей довольно несложно выбраться из «повседневной рутины» в новые условия. Другое дело — жители вод, гидробионты. Для расселения им приходится либо пользоваться водными путями, либо формировать покоящиеся стадии, достаточно защищенные для сухопутного путешествия. При этом для пресноводных гидробионтов что суша, что морская гладь — почти непреодолимая преграда.
Однако все сильно изменилось с появлением человеческой цивилизации. Если расселение палеолитического человека несильно повлияло на местные сообщества, то возникновение транспорта и регулярные грузовые перевозки новейшей истории кардинально изменили биогеографический ландшафт целых континентов.
Обобщив огромное число подобных примеров, более 60 лет назад английский ученый Чарльз Элтон (Charles Elton) издал книгу «Экология нашествий животных и растений» [1], с которой можно начать отсчет отдельной науки о биологических инвазиях. С той поры накоплен такой большой объем знаний, что никто не оспаривает глобальную, планетарную роль антропогенного фактора (вектора) в расселении животных. Поэтому большинство усилий ученых сосредоточено на изучении, предотвращении или контроле биологических инвазий. Изучению биологических инвазий в самом сердце российского государства — Волго-Камском регионе посвящена публикация в международном журнале Water [2], выполненная сотрудниками Института биологии внутренних вод РАН и Институт проблем экологии и эволюции РАН. В состав коллектива исследователей входили авторы этого материала.
Река Волга была вовлечена в колоссальную программу гидростроительства и сейчас здесь имеется девять крупных водохранилищ, а также большой участок, не зарегулированный гидротехническими сооружениями. В этом плане Кама пострадала меньше — здесь всего три крупных водохранилища. Это закономерно отражается в доле чужеродных видов (рис. 1): в настоящее время в обловах на мелководье доля чужеродных видов рыб в водохранилищах Волги составляет от 8% до 32%. Для водохранилищ Камы их значительно меньше — от 2 до 16%.
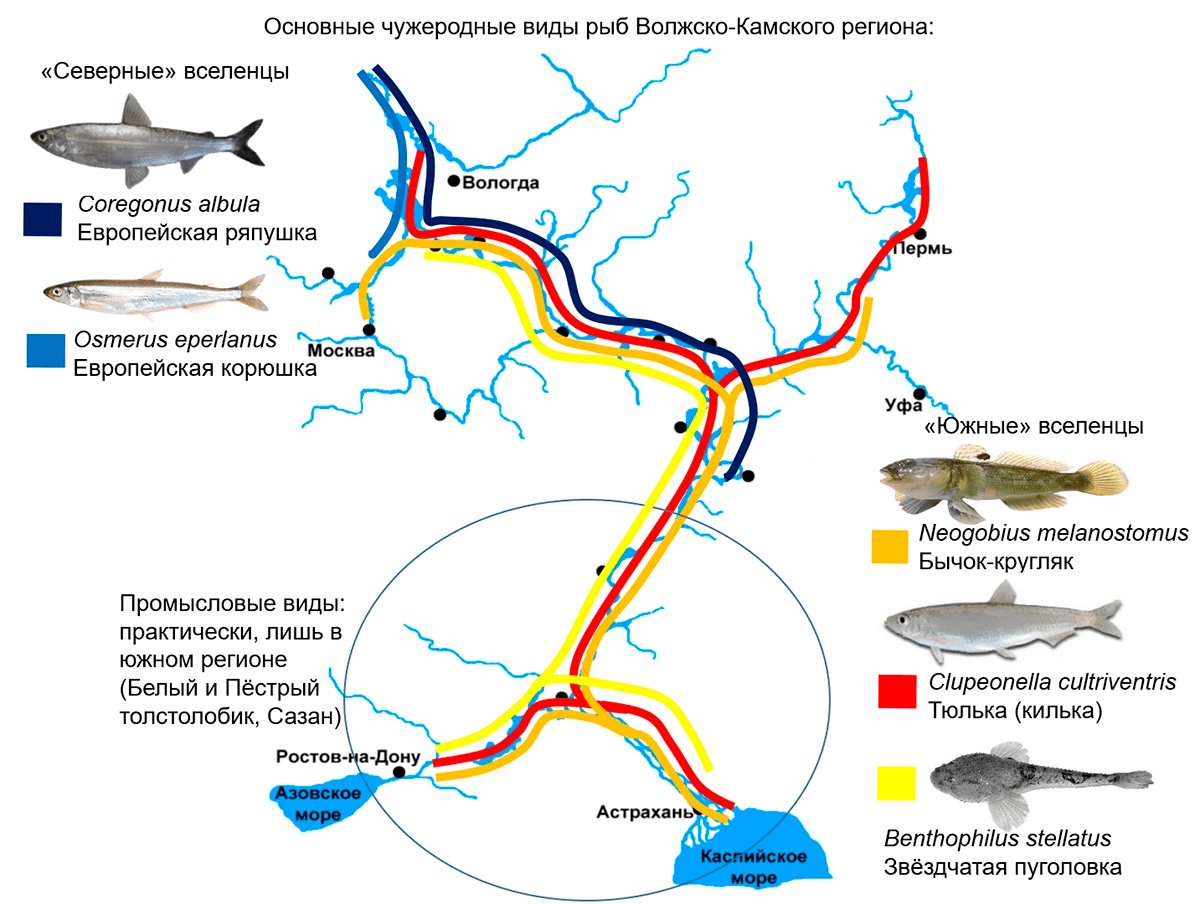
Рисунок 1. Распространение массовых чужеродных видов рыб в Волжско-Камском регионе
Большее число видов-вселенцев относится к бычковым рыбам. Так, среди инвазивных видов бассейна Волги мы обнаружили шесть представителей этого семейства, тогда как в Каме — только два. Это известные нам по консервам «Бычки в томате» относительно крупные бычки — кругляк (Neogobius melanostomus) и головач (Ponticola gorlap), а также их более мелкие родственники, включая такой удивительный вид, как звездчатая пуголовка (Benthophilus stellatus) (рис. 2). Еще один из необычных волжских видов рыб-вселенцев — игла-рыба (Syngnathus abaster), родственница морских коньков, известных нам по диснеевской «Русалочке». Эти рыбы часто встречаются в прибрежных зарослях на Средней и Нижней Волге.

Рисунок 2. Звездчатая пуголовка Benthophilus stellatus — одна из самых необычных видов рыб Волги. Саратовское водохранилище.
фото Дмитрия Павлова
Однако самым массовым по численности вселенцем в обследованных водоемах оказалась мелкая пелагическая сельдевая рыбка — черноморско-каспийская тюлька (Clupeonella cultriventris) (рис. 3). На море ее называют «килька» по причине «киля» из видоизмененных чешуек на брюшке рыбы. Из этих рыбок готовятся замечательные консервированные шпроты. Здесь мы не ошиблись: шпроты — это способ приготовления, и эти консервы не обязательно получать из рыб вида балтийский шпрот, численность которого на Балтике сильно подорвана активным промыслом. Хотя пока массового промысла тюльки в волжских водохранилищах не ведется, аборигенные хищные рыбы, такие как судак, налим, крупный окунь, успешно «распробовали» этого вселенца, и тюлька стала одним из важных компонентов трофической сети в водохранилищах. Однако кроме рыб-вселенцев, «идущих на север», есть и, наоборот, виды, стремящиеся «погреться на юге». Среди таких две сиговых рыбы — европейская корюшка (Osmerus eperlanus), любимая рыба петербуржцев, и не менее вкусная европейская ряпушка (Coregonus albula), встречающаяся вплоть до Куйбышевского водохранилища (рис. 1). В настоящее время численность этих видов невелика, хотя они являются важным компонентом пелагического сообщества в северных водохранилищах Волги.

Рисунок 3. Обычный улов на Волге — два пуда отборной тюльки! Авторы статьи в журнале Water — Дмитрий Карабанов и Дмитрий Павлов — в рейсе на научно-исследовательском судне «Академик Топчиев».
Ключевая задача при мониторинге биологических инвазий — своевременная и точная идентификация вселенца. Это позволяет не только спрогнозировать возможные воздействия вселенцев на нативные экосистемы, но и в случае необходимости как можно скорее провести мероприятия по локализации или уничтожению опасных или вредных видов. К сожалению, традиционные морфологические методы определения видов рыб в некоторых группах осложнены не только объективными трудностями идентификации, но и дефицитом профессиональных ихтиологов. И если дефицит кадров не относится к научным проблемам, то для точного определения животных имеется отличный современный метод — ДНК-идентификация.
Почти 20 лет назад канадец Поль Эбер (Paul Hebert) предложил использовать в качестве единого универсального маркера для видовой идентификации небольшой участок митохондриального гена цитохромоксидазы-c (COI) [3]. Это послужило началом эры ДНК-баркодинга, или штрихкодирования на основе ДНК. Этот способ определения базируется на простом наблюдении: последовательность нуклеотидов в выбранном регионе ДНК позволяет отнести образец к известному или к новому виду. В принципе, штрихкодировать можно практически любые объекты, обладающие некоторым числом альтернативных признаков, как это происходит с товарами на кассе магазинов. Технология ДНК-баркодинга в настоящее время является стандартной процедурой при изучении биологического разнообразия, однако, как и везде, здесь имеются свои нюансы. В частности, при проведении таких работ для амплификации методом полимеразной цепной реакции интересующего нас гена требуется использовать праймеры — небольшие «затравки» из одноцепочечной ДНК, комплементарные определенным участкам ДНК гена-мишени. Выбранный для ДНК-баркодинга животных ген COI относится к достаточно консервативным по последовательности генам, что позволяет использовать универсальные праймеры для большого числа близкородственных видов. Однако чем выше универсальность — тем ниже специфичность, поэтому исследователям постоянно приходится искать компромисс между подходом «много, но плохо» и «хорошо, но мало». Для более точного определения все большую популярность приобретает подбор праймеров, специфичных для конкретной группы животных. Так, например, при исследовании пресноводных рыб Волги нам совершенно не нужны знания о последовательностях ДНК рыб коралловых рифов Австралии. Поэтому мы имели возможность повысить специфичность определения пресноводных рыб, игнорируя другие группы. В результате «волжские» праймеры оказались гораздо эффективнее для конкретной задачи мониторинга видов-вселенцев в этом регионе.
Подробнее о методе ПЦР можно узнать в материале «Биомолекулы» «12 методов в картинках: полимеразная цепная реакция» [4].
Именно по такому пути пошел наш коллектив при ДНК-баркодинге чужеродных видов рыб Волги и Камы. Разработанные нами праймеры позволили почти на четверть улучшить эффективность стандартного ДНК-баркодинга. В случае фрагментарных и сильно деградировавших проб мы предлагаем использовать праймеры для амплификации более коротких последовательностей, что без потери точности позволяет проводить идентификацию «сложных» образцов, недоступных другим традиционным методам. Также эти «короткие» праймеры кроме традиционной амплификации можно использовать в системах высокопроизводительного секвенирования как в случае тотальных проб, так и для eDNA (environmental DNA, «природной» ДНК, в данном случае растворенной в воде) [5]. Однако работы только с одним локусом неминуемо могут приводить к ошибкам, например, к контаминации проб или неверным прочтениям. По оценкам разных исследователей, в международной базе данных NCBI GenBank существенная доля депонированных последовательностей имеют те или иные ошибки или вовсе относятся к другим видам [6], [7]. Для повышения репрезентативности мы разработали и предлагаем к использованию дополнительный набор праймеров для большой субъединицы митохондриальной (16S) и малой субъединицы ядерной (18S) рибосомной ДНК (рДНК). Такой набор имеет дополнительные преимущества. Так, идентификация по митохондриальной 16S рДНК, как правило, совпадает с COI, и по этому локусу накоплен большой набор данных, позволяющий исследовать изменчивость на внутривидовом уровне. Для ядерного гена 18S единственный недостаток — невысокая видовая изменчивость и слабая представленность в международных базах данных. Однако наличие консервативных участков обеспечивает эффективное выравнивание данных последовательностей, а гипервариабельные участки обеспечивают достаточно высокий уровень изменчивости. Также результаты исследований по этому локусу показывают хорошую сходимость с митохондриальными генами и 18S может успешно применяться при филогеографических реконструкциях. В общем, исследование рДНК может быть удачным дополнением для «классического» ДНК-баркодинга, в том числе и при использовании систем высокопроизводительного секвенирования.
При тестировании набора праймеров на участки генов COI, 16S и 18S мы изучили генетическую изменчивость у 31 вида пресноводных рыб. Для Волжско-Камского региона впервые сформирована библиотека «эталонных» последовательностей практически всех массовых чужеродных видов рыб, в том числе подтверждены находки нескольких новых видов. Предложенные нами наборы праймеров демонстрируют высокую эффективность при амплификации и высокую специфичность для области штрих-кода ДНК не только чужеродных видов, но и других таксонов пресноводных рыб (рис. 4).
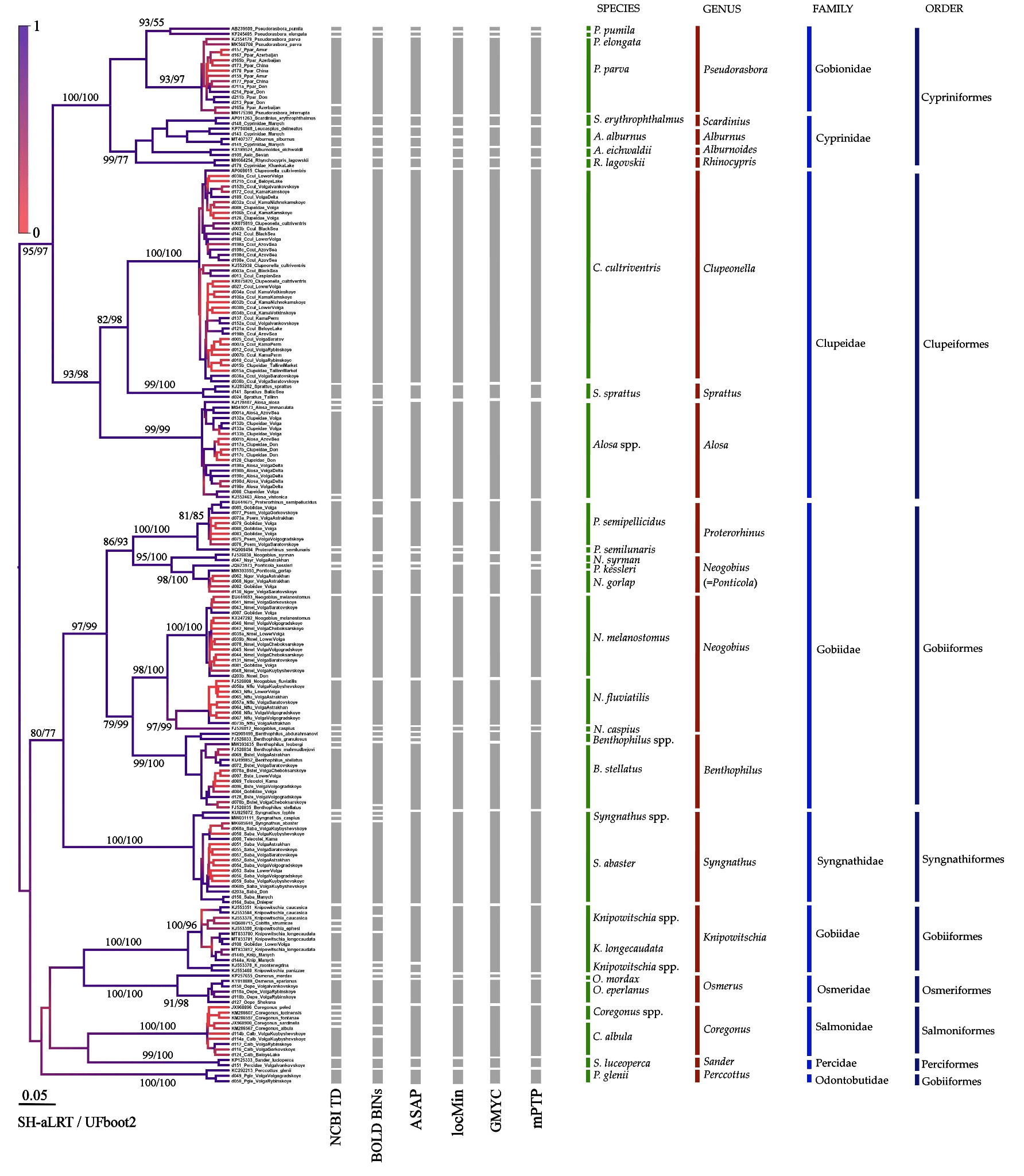
Рисунок 4. Филогенетическое дерево по локусу COI. Серые столбики показывают «молекулярные операционные таксономические единицы» (mOTU) [2]. Выделенные mOTU могут соответствовать целиком биологическим видам, либо разным филогенетическим линиям внутри полиморфного вида. Обсуждение места mOTU в системе «традиционной» таксономии началось практически одновременно с широким внедрением генетических методов [8], и этот вопрос до сих пор обсуждается [9].
Отдельным блоком работ проходила оптимизация протоколов рутинного выделения ДНК, проведения ПЦР и очистки продукта. Конечно, современные коммерческие наборы позволяют максимально упростить и оптимизировать лабораторную работу. Однако тут имеется один недостаток — очень высокая стоимость и часто потребность в специфическом оборудовании. Предлагаемая нами методика позволяет проводить работу от выделения ДНК до секвенирования практически «дома на кухне» — в любой биологической лаборатории, используя самый распространенный набор оборудования, пластика и реактивов. Мы принципиально старались максимально использовать отечественное оборудование и реагенты — благо, в настоящее время в России налажено производство всего необходимого оборудования и расходных материалов, а реактивы и ферменты по качеству ничуть не уступают именитым импортным аналогам. Таким образом нам удалось уложиться в стоимость «классического» секвенирования одной последовательности из интересующих — в сумму менее двух долларов США, что сравнимо с самыми современными системами высокопроизводительного секвенирования. При этом анализ не требует дорогостоящего оборудования, а техника проведения лабораторных работ и обработка результатов доступны любому исследователю.
Подробнее о разных методах секвенирования можно узнать в материале «Биомолекулы» «12 методов в картинках: секвенирование нуклеиновых кислот» [10].
Мы надеемся, что широкое внедрение ДНК-идентификации в классическую работу ихтиологов позволит более точно и быстро следить за проникновением новых видов, контролировать и предотвращать нежелательные инвазии.
Общеизвестно, что фундаментальные исследования — удовольствие недешевое. Благодаря многолетнему финансированию Минобрнауки России на базе Центра коллективного пользования «Научный флот ИБВВ РАН» удалось провести множество экспедиций и собрать уникальный материал по видам-вселенцам Волги и Камы, а также материал сравнения из других водоемов Европы. Генетические работы были выполнены при поддержке гранта РФФИ 20-34-70020 «Чужеродные виды гидробионтов разных таксономических групп в Европейской России: сравнительный анализ полиморфизма в пределах нативных и инвазионных ареалов, истории и особенностей инвазионого процесса».
Литература
- Elton C.S. The ecology of invasions by animals and plants: New edition. Chicago, London: University of Chicago Press, 2000;
- Dmitry P. Karabanov, Eugeniya I. Bekker, Dmitry D. Pavlov, Elena A. Borovikova, Yulia V. Kodukhova, Alexey A. Kotov. (2022). New Sets of Primers for DNA Identification of Non-Indigenous Fish Species in the Volga-Kama Basin (European Russia). Water. 14, 437;
- Paul D. N. Hebert, Alina Cywinska, Shelley L. Ball, Jeremy R. deWaard. (2003). Biological identifications through DNA barcodes. Proc. R. Soc. Lond. B. 270, 313-321;
- 12 методов в картинках: полимеразная цепная реакция;
- Matthieu Leray, Joy Y Yang, Christopher P Meyer, Suzanne C Mills, Natalia Agudelo, et. al.. (2013). A new versatile primer set targeting a short fragment of the mitochondrial COI region for metabarcoding metazoan diversity: application for characterizing coral reef fish gut contents. Front Zool. 10, 34;
- D. James Harris. (2003). Can you bank on GenBank?. Trends in Ecology & Evolution. 18, 317-319;
- Mikko Pentinsaari, Sujeevan Ratnasingham, Scott E. Miller, Paul D. N. Hebert. (2020). BOLD and GenBank revisited – Do identification errors arise in the lab or in the sequence libraries?. PLoS ONE. 15, e0231814;
- Amanda Ciprandi Pires, Luciane Marinoni. (2010). DNA barcoding and traditional taxonomy unified through Integrative Taxonomy: a view that challenges the debate questioning both methodologies. Biota Neotrop.. 10, 339-346;
- Maxim V. VINARSKI. (2020). Roots of the taxonomic impediment: Is the “integrativeness” a remedy?. Integrative Zoology. 15, 2-15;
- 12 методов в картинках: секвенирование нуклеиновых кислот.





