Т9 для транскриптов: что такое редактирование РНК и зачем оно нужно?
17 февраля 2023
Т9 для транскриптов: что такое редактирование РНК и зачем оно нужно?
- 556
- 2
- 2
Рисунок в полном размере.
иллюстрация автора статьи
-
Автор
-
Редакторы
Постер на конкурс «Био/Мол/Текст»: Центральная догма молекулярной биологии гласит, что с гена должна считаться РНК, а с нее 一 белок. Однако не все так просто. Есть альтернативный сплайсинг, когда из одного гена получается несколько вариантов белка. А еще в клетке присутствует целый ряд посттранскрипционных модификаций РНК, которые меняют аминокислотную последовательность будущих пептидов. Например, удаление аминогруппы азотистых оснований в молекуле РНК или включение в нее нуклеотидов, также известное как РНК-редактирование (RNA-editing). Процесс редактирования несколько похож на автозамены T9, потому что результат порой оказывается немного неожиданным. И на клетке это может сказываться неблагоприятно. Например, замена проходит с целью получения нового полезного белка, а в результате получается опасный онкогенный продукт. Феномен редактирования РНК уже давно не дает покоя эволюционным и молекулярным биологам. Его понимание поможет найти ключ к ответу на множество вопросов: от белкового разнообразия позвоночных до механизмов химиорезистентности злокачественных опухолей.
Конкурс «Био/Мол/Текст»-2022/2023
Эта работа опубликована в номинации «Наглядно о ненаглядном» конкурса «Био/Мол/Текст»-2022/2023.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
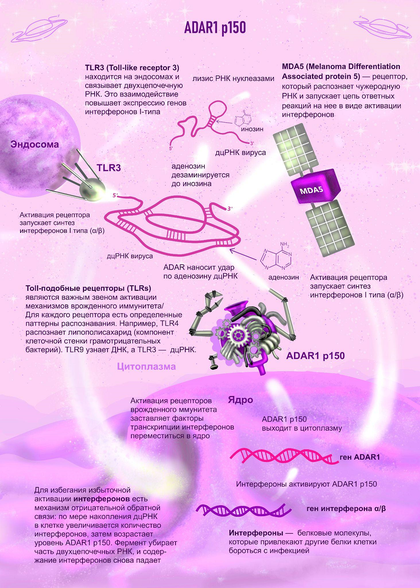
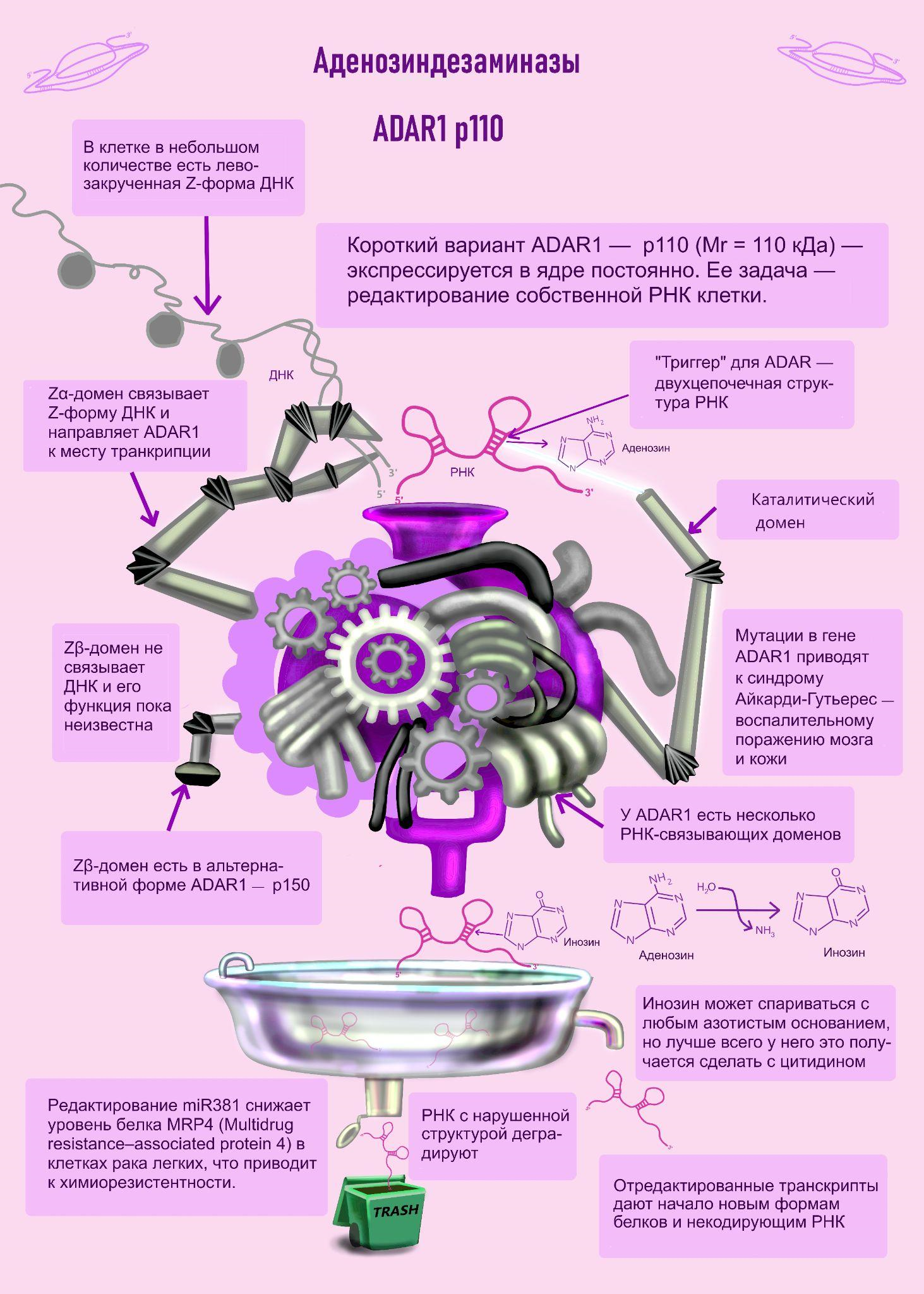
РНК-зависимая аденозиндезаминаза ADAR (adenosine desaminase acting on RNA) удаляет аминогруппу аденозина в свежих транскриптах. В результате чего образуется инозин, который при трансляции распознается как гуанозин. Это приводит к перекодированию белка. Также изменение последовательности РНК влияет на ход сплайсинга и структуру молекулы. У человека есть ADAR1, ADAR2 и ADAR3 [1]. Первые два фермента активно дезаминируют РНК. При этом ADAR1 имеет два варианта — длинный (p150) и короткий (p110). Помимо матричных РНК, ADAR вносит корректировки в молекулы микроРНК. МикроРНК (miR) [2] — класс некодирующих РНК, регулирующих экспрессию генов путем комплементарного спаривания с другими РНК. Кроме того, многие из них вырабатываются в клетках злокачественных опухолей.
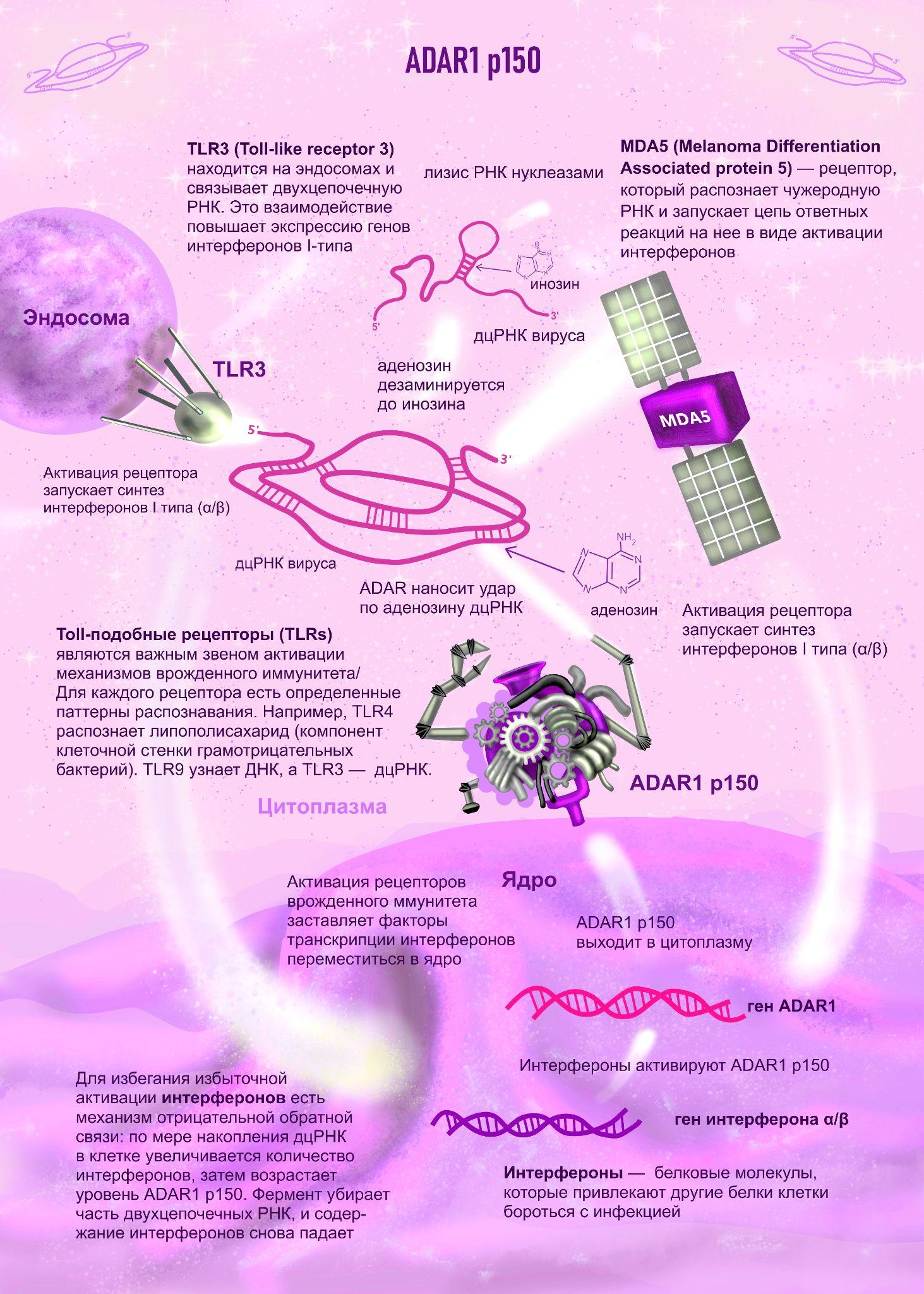
Длинный вариант ADAR1 — p150 (Mr = 150 кДа) — экспрессируется в цитоплазме индуцибельно. Если в клетку попадает чужеродная двухцепочечная РНК вируса, она тут же узнается рецепторами врожденного иммунитете TLR3 и MDA5. Затем активируется синтез интерферонов I типа. Вслед за интерферонами, ADAR1 p150 редактирует геном вируса таким образом, что его РНК меняет структуру и деградирует.
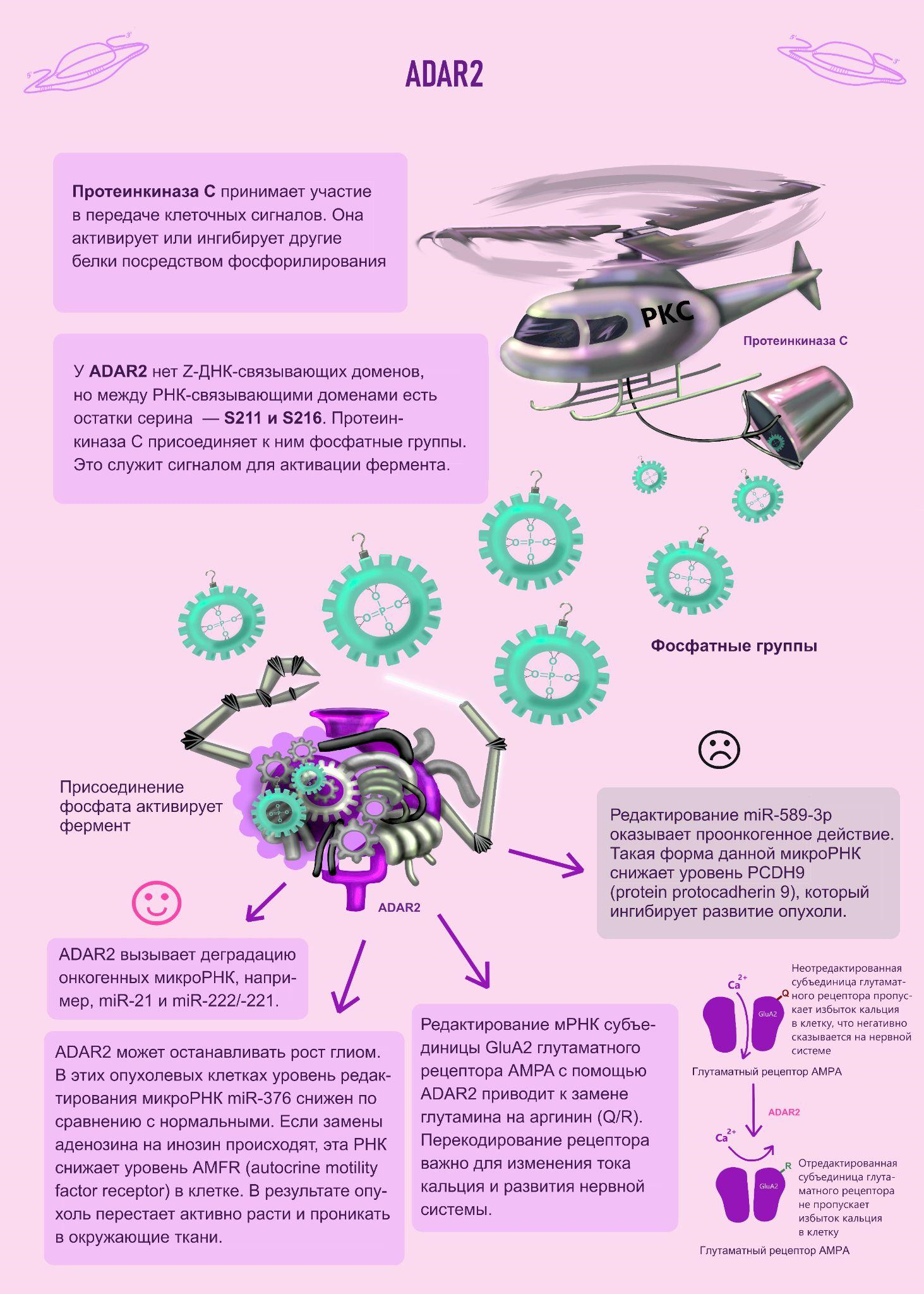
ADAR2 схож по функциям с ADAR1 p110, но немного отличается строением и имеет свои мишени редактирования. ADAR2 играет ключевую роль в онтогенезе нервной системы млекопитающих [3]. Редактирование в мРНК субъединицы GluA2 глутаматного рецептора AMPA приводит к замене глутамина на аргинин (Q/R). Рецептор одновременно является и кальциевым каналом. Благодаря замене проницаемость канала для кальция изменяется. Если бы этого не происходило, нейроны бы попросту погибали из-за избытка кальция.
В отличие от предыдущих ферментов, ADAR3 не обладает ферментативной активностью, но тоже имеет интересные функции [4], [5].
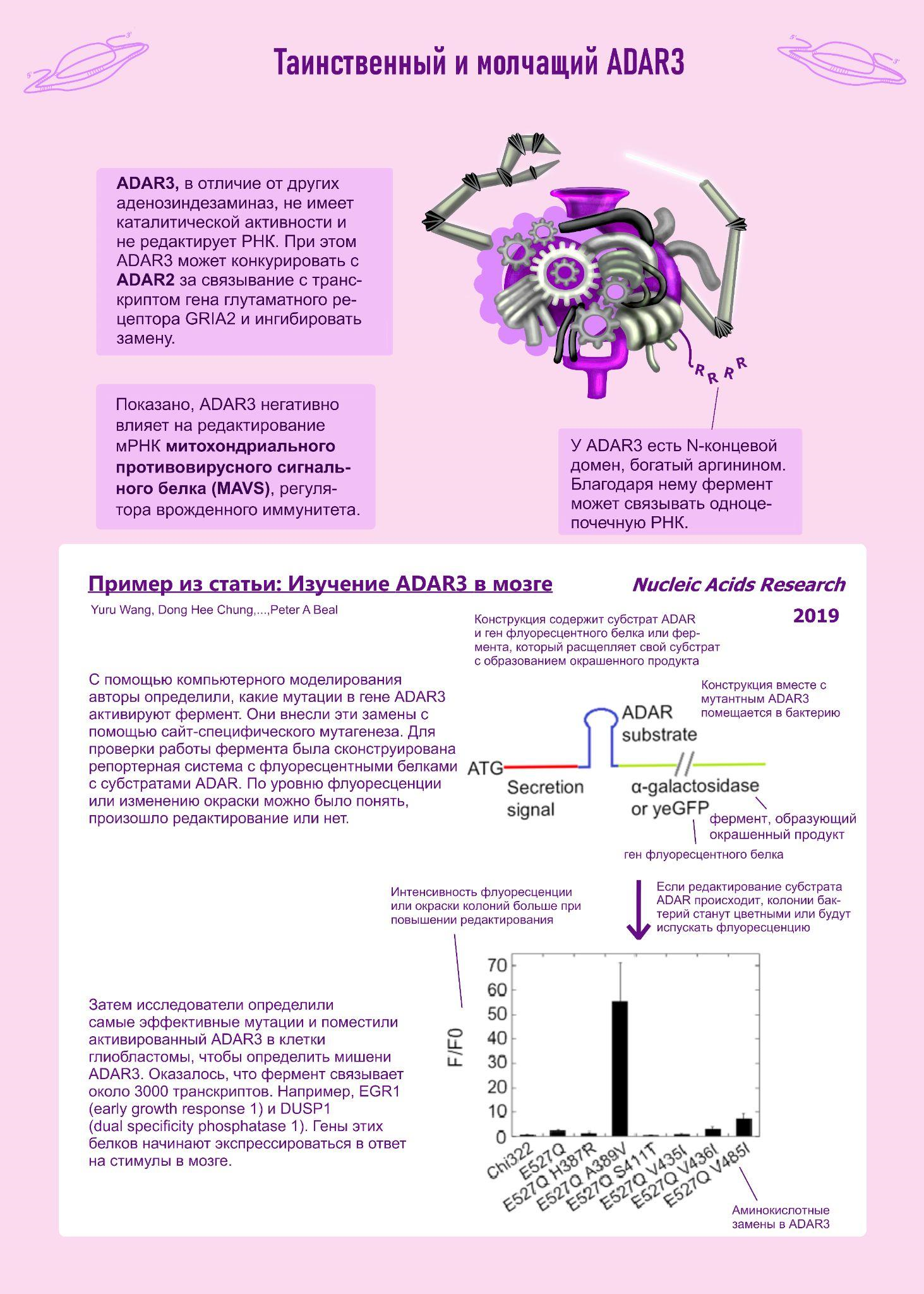
Кроме того, существует фермент, который может редактировать транспортную РНК [6].

Цитидиндезаминазы семейства APOBEC/AID — ферменты, которые образуют урацил из цитозина в молекулах РНК и ДНК [7], [8]. Название APOBEC (Apolipoprotein B mRNA Editing Catalytic Polypeptide-like) происходит от субстрата этого белка — аполипротеина B. В печени синтезируется его полноценная форма, в то время как в тонком кишечнике, благодаря редактированию урацила в стоп-кодон, образуется в середине мРНК, и белок считывается только на 48%. Он получил название apoB48.
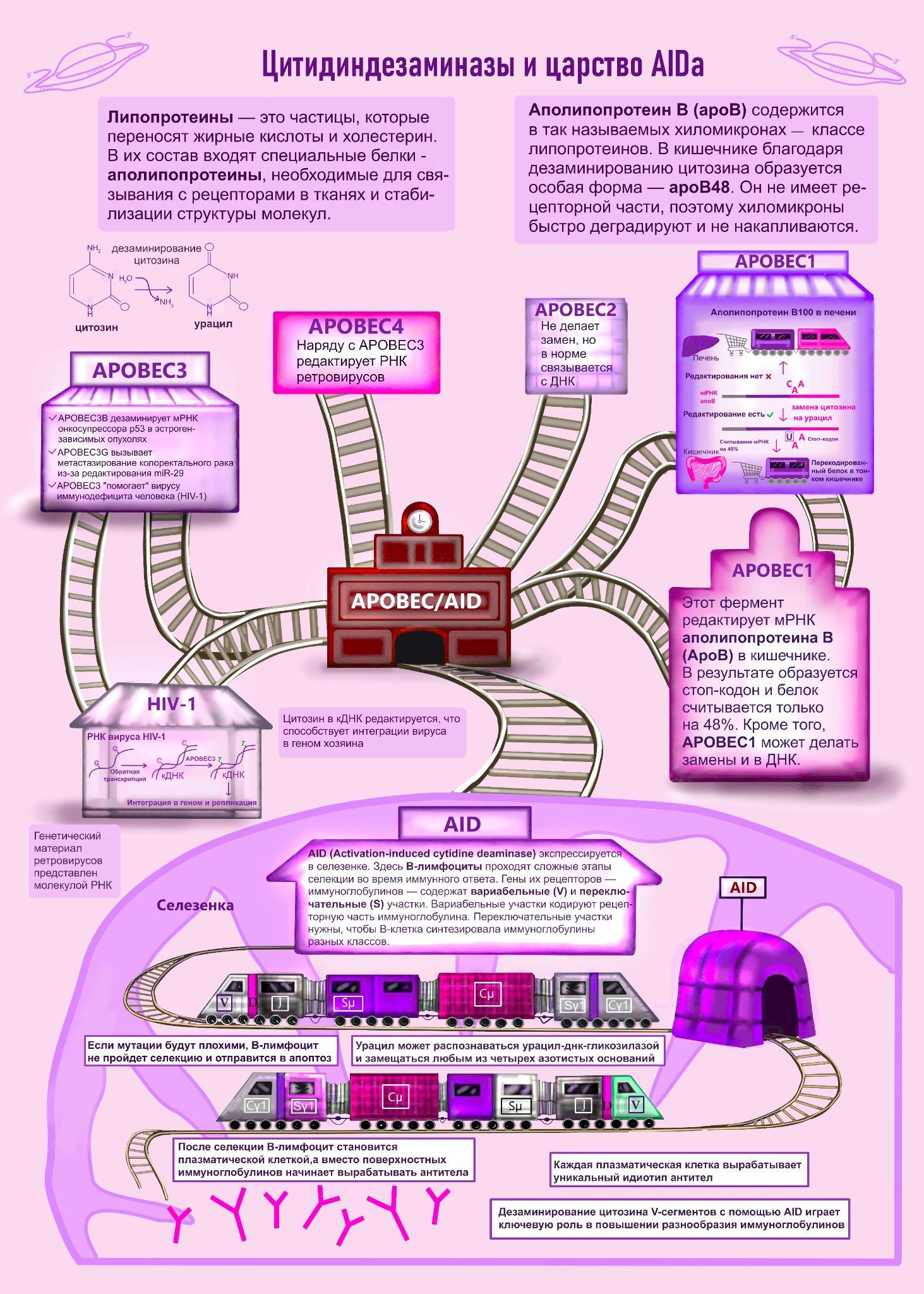
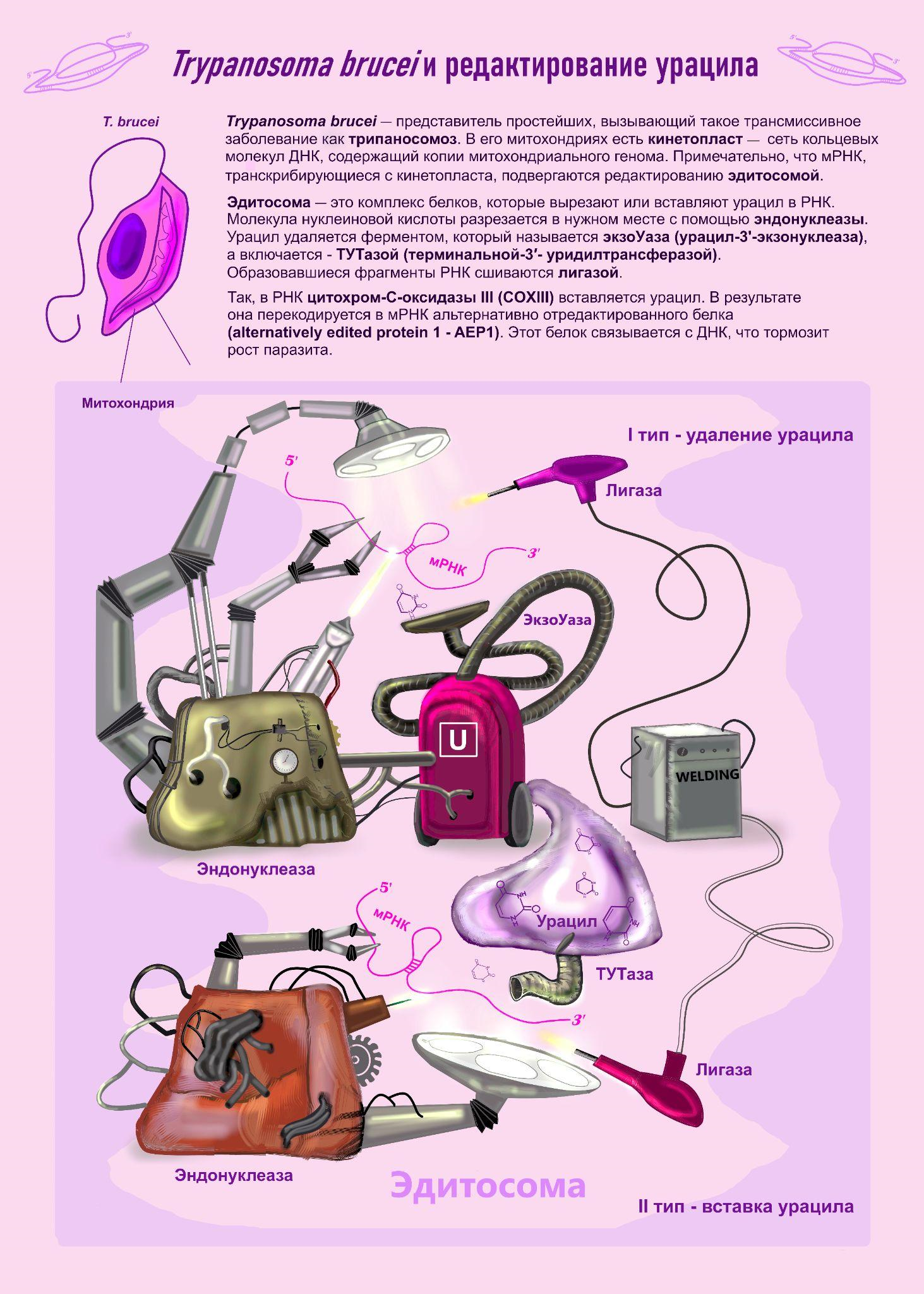
У T.brucei есть необычный механизм редактирования путем вырезания или вставки нуклеотидов [9], [10].
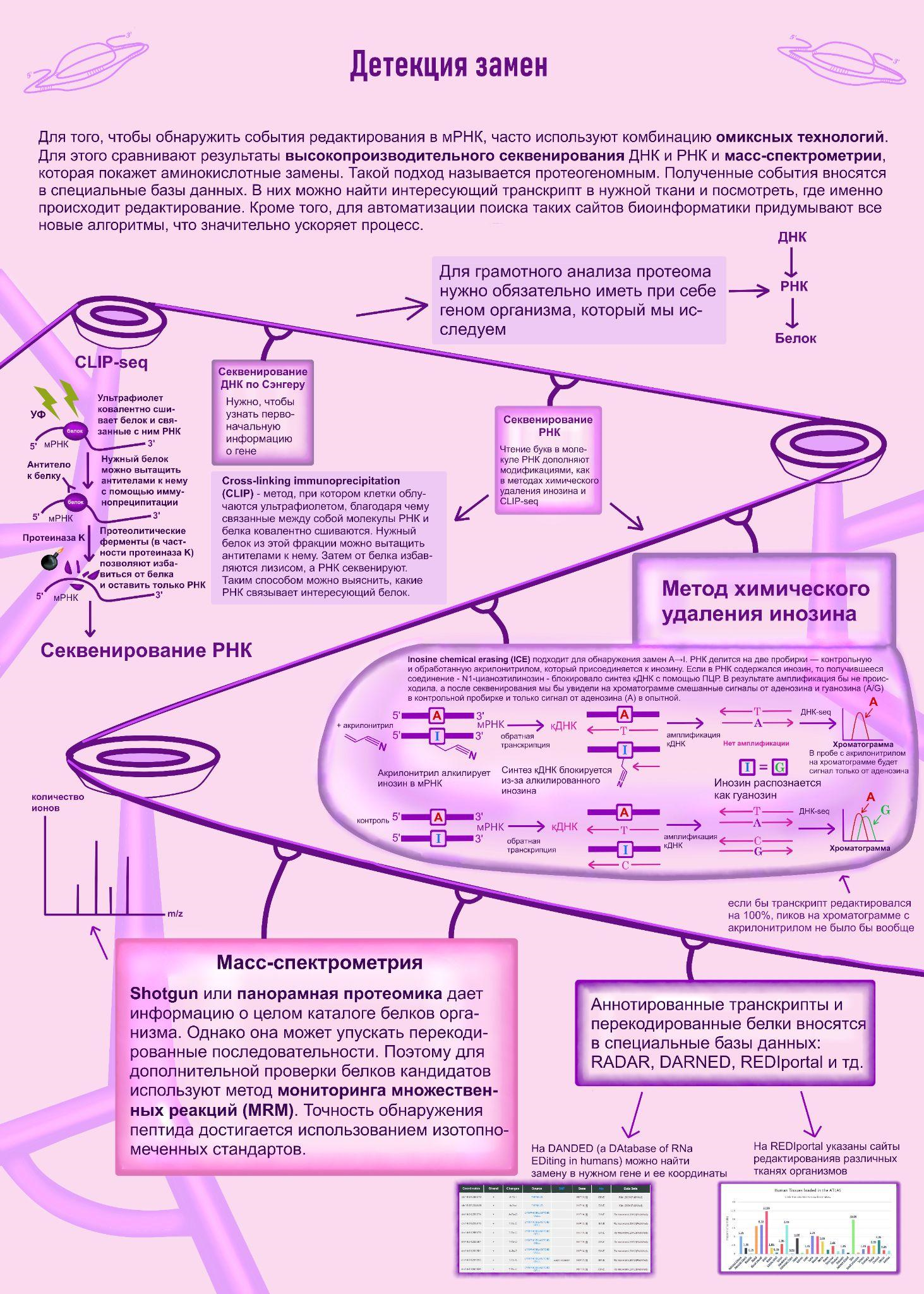
Важный вопрос заключается в том, как эти замены можно обнаружить. И нужно позаботиться об автоматизации процессов поиска [11].

Литература
- Theodoulakis Christofi, Apostolos Zaravinos. (2019). RNA editing in the forefront of epitranscriptomics and human health. J Transl Med. 17;
- Обо всех РНК на свете, больших и малых;
- Yue Zhang, Huizhu Qian, Jing Xu, Wen Gao. (2019). ADAR, the carcinogenesis mechanisms of ADAR and related clinical applications. Ann. Transl. Med.. 7, 686-686;
- Yuru Wang, Dong hee Chung, Leanna R Monteleone, Jie Li, Yao Chiang, et. al.. (2019). RNA binding candidates for human ADAR3 from substrates of a gain of function mutant expressed in neuronal cells. Nucleic Acids Research. 47, 10801-10814;
- Reshma Raghava Kurup, Eimile K. Oakes, Aidan C. Manning, Priyanka Mukherjee, Pranathi Vadlamani, Heather A. Hundley. (2022). RNA binding by ADAR3 inhibits adenosine-to-inosine editing and promotes expression of immune response protein MAVS. Journal of Biological Chemistry. 298, 102267;
- Helena Roura Frigolé, Noelia Camacho, Maria Castellví Coma, Carla Fernández-Lozano, Jorge García-Lema, et. al.. (2019). tRNA deamination by ADAT requires substrate-specific recognition mechanisms and can be inhibited by tRFs. RNA. 25, 607-619;
- Riccardo Pecori, Salvatore Di Giorgio, J. Paulo Lorenzo, F. Nina Papavasiliou. (2022). Functions and consequences of AID/APOBEC-mediated DNA and RNA deamination. Nat Rev Genet. 23, 505-518;
- Yaoyao Xie, Lipeng Yao, Xiuchong Yu, Yao Ruan, Zhe Li, Junming Guo. (2020). Action mechanisms and research methods of tRNA-derived small RNAs. Sig Transduct Target Ther. 5;
- Cristian Del Campo, Wolf-Matthias Leeder, Paul Reißig, H Ulrich Göringer. (2020). Analyzing editosome function in high-throughput. Nucleic Acids Research. 48, e99-e99;
- Suzanne M. McDermott, Jie Luo, Jason Carnes, Jeff A. Ranish, Kenneth Stuart. (2016). The Architecture of Trypanosoma brucei editosomes. Proc. Natl. Acad. Sci. U.S.A.. 113;
- Masayuki Sakurai, Tsutomu Suzuki. (2011). Biochemical Identification of A-to-I RNA Editing Sites by the Inosine Chemical Erasing (ICE) Method. RNA and DNA Editing. 89-99.