
Секвенирование единичных клеток (версия — Metazoa)
05 ноября 2013
Секвенирование единичных клеток (версия — Metazoa)
- 3626
- 0
- 6
Кит секвенирования стремительно плывет в страну Единичной клетки
-
Автор
-
Редакторы
Темы
Статья на конкурс «био/мол/текст»: Ученые исследовали единичные клетки со времен изобретения микроскопа. Но такие методы-киты молекулярной биологии, как анализ на микрочипах, секвенирование нуклеиновых кислот и масс-спектрометрия подразумевают использование некоторой массы клеток. Однако если взять среднее большого числа клеток, то на выходе получим усредненный результат. Есть области, где усредненный результат — это уже неинтересно; есть важные фундаментальные вопросы, на которые вам ответит только она — Единичная клетка. Например, этот подход может стать одним из ключей к познанию тайн эмбрионального развития, закономерностей дифференциации стволовых клеток, функционирования нейронов и выработке стратегий лечения опухолевых заболеваний.
Конкурс «био/мол/текст»-2013
Эта статья представлена на конкурс научно-популярных работ «био/мол/текст»-2013 в номинации «Лучший обзор».
Спонсор конкурса — дальновидная компания Thermo Fisher Scientific. Спонсор приза зрительских симпатий — фирма Helicon.
Существует множество факторов, которые обусловливают гетерогенность генетически идентичных клеток. В частности, это эпигенетика , определяющая тканеспецифическую экспрессию генов, ее зависимость от половой принадлежности, от стадии развития организма. В клетках одного типа процессы транскрипции генов определяются активностью или ингибированием тех или иных сигнальных путей. После транскрипции РНК могут процессироваться по-разному, где-то образуется один сплайс-вариант, а где-то — другой. Далее накладываются различия уровней трансляции и посттрансляционные модификации. В итоге, даже находясь в одинаковом микроокружении, клетки одного типа гетерогенны относительно реализации генетического материала. Это называется стохастической экспрессией генов, «шумом» экспрессии генов [1]. Становится очень интересно: как клетки, живя в этом «шуме», действуют в итоге слаженно? Анализ транскриптома отдельных клеток в комбинации с оверэкспрессией, нокдауном или нокаутом интересующих генов может позволить изучить генетические регуляторные сети прямо и неискаженно [2]. Особенно этот подход актуален для анализа клеток во время раннего эмбрионального развития и для стволовых клеток, когда число клеток, подходящих для анализа, ограниченно, так как они динамично развиваются, и субпопуляции клеток очень гетерогенны [3], [4].
Об эпигенетике и ее роли в реализации генетической программы клетки читайте в статье «Развитие и эпигенетика, или история о минотавре». — Ред.
Чужой среди чужих
Если нормальные клетки организма в большинстве своем имеют идентичную генетическую информацию, то опухолевые клетки по определению «чужие». Более того, клетки одной опухоли (за редким исключением) различаются между собой по набору генетических аберраций. Поэтому в результате исследования образцов злокачественной ткани мы получаем результат, характеризующий большинство клеток. Применяем терапию, направленную на обнаруженные мишени, и убиваем в лучшем случае это большинство. А тем временем меньшая часть клеток, не обладающая этими мутациями, может выжить [5]. В этом случае высок риск рецидива. Вот здесь могло бы прийти на помощь секвенирование ДНК единичных клеток. Кстати, исследуя единичные клетки опухоли, можно узнать ее эволюцию. Так, в результате секвенирования ста геномов единичных клеток двух образцов рака молочной железы подтвердилось, что метастазирующие клетки возникают на поздних этапах развития опухоли [6]. Кроме того, был обнаружен паттерн роста опухоли, который они назвали «точечной клональной эволюцией», заимствовав термин из теории эволюции видов. При этом опухолевые популяции отличались от их предка, без очевидного промежуточного ветвления. То есть скорость геномной эволюции значительно превышала эффективный рост популяции.
Единичных клеток будет достаточно для неинвазивного мониторинга за заболеванием (циркулирующие опухолевые клетки, воздействие химиотерапии) [7]. В будущем это может быть расширено, например, на вирусные заболевания и процесс их мутагенеза в зараженных популяциях. Интересно применение методов секвенирования единичных клеток для изучения редких образцов, таких как палеонтологические, — например, из животных, заключенных в янтарь, которые обычно очень маленького размера.
Суммируя вышесказанное, цели секвенирования единичных клеток многоклеточных организмов разнообразны и затрагивают многие актуальные вопросы биологии. Каковы технические особенности этой процедуры?
Достать любой ценой
Секвенирование нуклеиновых кислот — это определение последовательности нуклеотидов в молекулах ДНК или РНК. Общая схема исследования единичных клеток секвенированием выглядит так: получение единичных клеток, выделение нуклеиновых кислот, подготовка библиотеки, собственно секвенирование и биоинформатический анализ. Кроме того, нужно не забывать про систему контролей [8]. Несмотря на то, что каждая клетка уникальна и полученные результаты не воспроизвести, исследования необходимо проводить параллельно с отрицательным контрольным образцом без клетки и положительным контрольным образцом нуклеиновых кислот с концентрацией, сопоставимой с таковой в экспериментальном образце.
О стремительном развитии технологий секвенирования ДНК и их роли в науке и обществе можно прочесть в статьях «454-секвенирование (высокопроизводительное пиросеквенирование ДНК)», «Важнейшие методы молекулярной биологии и генной инженерии», «Код жизни: прочесть не значит понять», «Перевалило за тысячу: третья фаза геномики человека» и «Огурцы-убийцы, или как встретились Джим Уотсон и Гордон Мур». — Ред.
Получить единичные клетки можно разными методами. Не потребует особого инструментария метод лимитирующих разведений. Однако при работе с тканями будет необходимо подбирать оптимальные условия для диссоциации клеток. При наличии микроскопа можно научиться захватывать единичные клетки пипеткой. Этот прием использовал пионер секвенирования единичных клеток Джеймс Эбервайн (James Eberwine) [9]. Он подойдет для исследования крупных клеток, — например, нейронов [10] или яйцеклеток. Можно попытаться даже исследовать отдельные компартменты клетки (в случае нейрона — дендриты). Именно так был обнаружен новый класс транскриптов — цитоплазматические транскрипты, сохраняющие интронную последовательность (cytoplasmic intron-sequence retaining transcripts), которые, по-видимому, участвуют в посттранскрипционной регуляции экспрессии генов [11].
При наличии инструментов, опыта и мастерства микроскописта лазерная микродиссекция открывает возможность получения единичных клеток и их компартментов из любых тканей [12]. Для выделения единичных клеток (а также ядер или даже отдельных хромосом) из жидких тканей или суспензий клеток отлично подойдет проточная цитометрия [13]. Одним из перспективных методов автоматизации получения единичных клеток является микрофлуидика. Из введенной в чип массы клеток с помощью специальных ловушек отсеиваются отдельные клетки. Так, на основе технологии микрофлуидики разработано устройство для сепарации хромосом [14]. Это позволяет получить информацию о гаплотипе без исследования семейного трио. (Гаплотип — это совокупность материнских и отцовских аллелей на локусах одной хромосомы; его необходимо знать для интерпретации выявленных мутаций.) Есть возможность на микрофлуидном чипе провести операции выделения РНК, обратной транскрипции и преамплификации. Это упрощает, ускоряет и унифицирует процесс, предшествующий приготовлению библиотек.
Таким образом, методов получения единичных клеток много, и остается подобрать оптимальный в соответствии с целями, умениями и инструментами.
Приумножаем богатство
Дальше из полученных единичных клеток нужно выделить нуклеиновые кислоты. Типичная клетка содержит меньше сотни пикограмм ДНК и около 10 пг тотальной РНК (мРНК обычно составляет примерно 1–5% от тотальной РНК). Этого количества недостаточно для приготовления библиотек с помощью коммерческих наборов, где в среднем требуется 100 нг ДНК для полногеномного секвенирования, 100 нг РНК для секвенирования транскриптома (в англоязычной литературе — RNA-seq). Отсюда два вывода. Во-первых, при выделении нуклеиновых кислот, как отдельной процедуре, можно запросто потерять весь материал. Поэтому чаще всего проводят просто лизис клетки в пробирке и в ней же проводят последующие процедуры. Во-вторых, это означает, что придется иметь дело с полногеномной амплификацией (whole genome amplification — WGA) или амплификацией транскриптома. Рассмотрим сначала методы амплификации ДНК.
Более 10 лет самым надежным методом WGA считался метод амплификации со множественным замещением цепи (multiple displacement amplification — MDA) [15]. В этой реакции случайные праймеры удлиняются с помощью фермента с выраженной способностью к замещению цепи — phi29 ДНК-полимеразой. В отличие от амплификации с помощью ПЦР, точность прочтения с помощью MDA на 3–4 порядка выше, и ампликоны имеют среднюю длину >12 т.п.н. Несмотря на высокую точность этой полимеразы, применение MDA для амплификации ДНК единичных клеток показало, что часто не удается определить аллель: гетерозигота ошибочно считается гомозиготой. Но теперь разработан новый метод полногеномной амплификации, подходящий для единичных клеток — MALBAC (Multiple annealing and looping-based amplification cycles) [16]. Он соединяет в себе черты ПЦР и MDA, что приводит к снижению ошибки амплификации (рис. 1). В этом методе используются две ДНК-полимеразы — Bst и Taq. Однако и MALBAC не лишен недостатков: при генотипировании вариантов единичных нуклеотидов этот метод дает много ложноположительных результатов. После амплификации можно приступать к подготовке полногеномной или экзомной библиотек с помощью обычных коммерческих наборов.
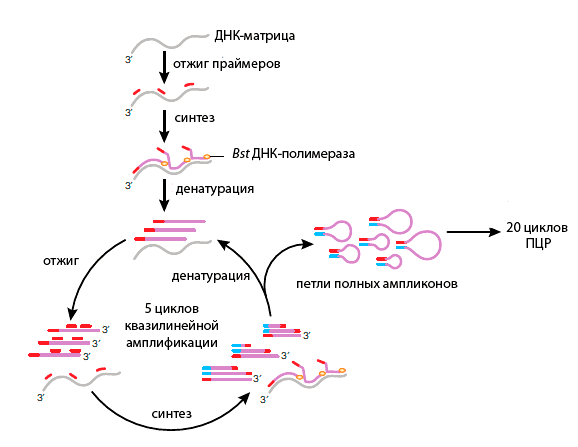
Рисунок 1. Упрощенная схема реакции MALBAC. Праймеры MALBAC (красные) имеют общую последовательность и 8 случайных нуклеотидов, которые отжигаются на ДНК-матрице. Синтез с замещением цепи приводит к образованию частичных ампликонов, которые затем денатурируют. Отжиг на новых позициях ДНК-матрицы увеличивает покрытие генома, а ошибок амплификации в результате немного. Отжиг праймеров на частичных ампликонах с последующим синтезом приводит к образованию полных ампликонов. Они имеют на 5′-конце последовательность праймера MALBAC (красная), а на 3′-конце комплиментарную ему последовательность (голубая). Денатурация приводит к регенерации исходной матрицы и образованию еще более разнообразного набора частичных ампликонов. Полные ампликоны образуют петли, которые могут быть устойчивы к последующей амплификации и гибридизации. Полные ампликоны генерируются в течение пяти циклов и затем амплифицируются экспоненциально в ходе 20 циклов ПЦР с использованием праймеров к общему участку праймеров MALBAC. Bst — Bacillus stearothermophilus.
Теперь обратимся к амплификации транскриптома единичной клетки. Не будем забывать, что РНК плавает в лизате. Нацеленно воздействовать на нее в таком окружении пока возможно только через поли-А хвост, который является характерным признаком зрелых мРНК. Следовательно, пока на уровне транскриптома единичных клеток нам недоступен весь остальной мир РНК. Итак, олиго-dT праймер на поли-А хвосте, синтезируем первую цепь кДНК. А далее есть несколько путей, как получить универсальный «якорь», за который можно будет манипулировать кДНК во всех последующих операциях. Можно выделить два основных приема: poly-A tailing и template switching (рис. 2).
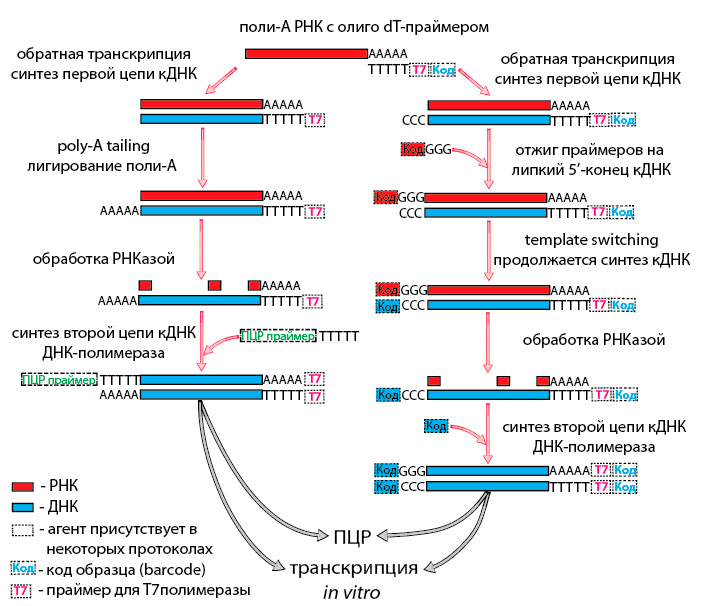
Рисунок 2. Общая схема методов RNA-Seq (пояснения в тексте)
В первом случае к первой цепи кДНК лигируют поли-А хвост — он будет «якорем» [18], [19]. А template switching — переключение матрицы — это свойство РНК-зависимой ДНК-полимеразы: дойдя до 5′-конца матрицы, она присоединяет к кДНК еще несколько оснований без нее, и получается «липкий» конец. К этому концу (как правило, 3C) присоединяются специальные праймеры с 3G на 5′-конце. РНК-зависимая ДНК-полимераза переключается, и теперь праймер служит ей матрицей для синтеза кДНК. Комплементарный праймеру участок первой цепи кДНК и будет «якорем» [20]. После создания «якоря» синтезируют вторую цепь кДНК и приступают к амплификации. Это можно сделать с помощью ПЦР [19], [20] или с помощью транскрипции в пробирке (in vitro transcription — IVT); ее также называют синтезом аРНК [21], [22]. Преимуществом ПЦР является высокая степень амплификации (106) и высокая скорость реакции, но надо учитывать наличие характерных для этой реакции ошибок и побочных неспецифических продуктов.
Методы, в которых РНК нарабатывается с помощью IVT, имеют высокую точность, но они длительные. Кроме того, степень амплификации при IVT довольно низка (103), что требует нескольких раундов этого процесса. Существует также возможность объединить несколько образцов в одной реакционной пробирке для приготовления библиотеки. Для этого при синтезе кДНК каждому образцу присваивается уникальный код (barcode) в составе одного из праймеров [21], [23]. Однако при таком подходе теряется возможность прочесть всю последовательность транскриптов, так как на стадии приготовления библиотек кДНК или РНК фрагментируют, и с кодом остаются лишь 3′- или 5′-концы транскриптов. Вообще методов амплификации транскриптома много и они плавно перетекают друг в друга. Полученную после амплификации кДНК или РНК используют для приготовления библиотеки и последующего секвенирования с помощью коммерчески доступных наборов [24].
Опасные последствия

Рисунок 3. Образное изображение захлестывающего количества информации, генерируемой в результате секвенирования нового поколения (next generation sequencing — NGS).
иллюстрация Артура Рэкама к книге Л. Кэрролла «Алиса в стране чудес»; автор идеи Бабенко В.В.
Когда мы думаем о секвенировании, нельзя ни в коем случае забывать о биоинформатическом анализе получаемой на выходе сырой информации, иначе можно не получить ожидаемой отдачи от этого метода исследования (рис. 3). Здесь сложно выделить универсальный рецепт, каждое исследование будет иметь свои особенности и трудности. Например, необходимо учитывать стохастическую экспрессию генов в генетически идентичных клетках [25], [26].
Взгляд в будущее
Одной из главных целей биологии является понимание связи между генетическим материалом, генетическими регуляторными сетями и фенотипом индивидуальной клетки (в физиологическом или патологическом состоянии). Исходя из этого, ясны пути дальнейшего развития технологии секвенирования единичных клеток. Во-первых, научиться на уровне одной клетки изучать все разнообразие РНК, а не только поли-А РНК; сохранять информацию о принадлежности к цепи (смысловой или несмысловой); секвенировать транслирующиеся РНК. Во-вторых, иметь возможность одновременно секвенировать геном, эпигеном и транскриптом одной клетки. Наконец, чтобы приблизиться к объяснению того, как клетка выбирает путь развития, как регулируется дифференциация, нужно научиться комбинировать секвенирование РНК и ДНК с визуализацией живых клеток с мечеными белками [27].
Кит секвенирования единичных клеток еще только набирает мили, так что сейчас есть возможность запрыгнуть ему на спину и проложить маршрут в неизведанные океаны. Чего вам и желаю!
Литература
- J. M. Raser. (2005). Noise in Gene Expression: Origins, Consequences, and Control. Science. 309, 2010-2013;
- Fuchou Tang, Kaiqin Lao, M Azim Surani. (2011). Development and applications of single-cell transcriptome analysis. Nat Methods. 8, S6-S11;
- Yosef Buganim, Dina A. Faddah, Albert W. Cheng, Elena Itskovich, Styliani Markoulaki, et. al.. (2012). Single-Cell Expression Analyses during Cellular Reprogramming Reveal an Early Stochastic and a Late Hierarchic Phase. Cell. 150, 1209-1222;
- Victoria Moignard, Iain C. Macaulay, Gemma Swiers, Florian Buettner, Judith Schütte, et. al.. (2013). Characterization of transcriptional networks in blood stem and progenitor cells using high-throughput single-cell gene expression analysis. Nat Cell Biol. 15, 363-372;
- Douglas Hanahan, Robert A. Weinberg. (2011). Hallmarks of Cancer: The Next Generation. Cell. 144, 646-674;
- Nicholas Navin, Jude Kendall, Jennifer Troge, Peter Andrews, Linda Rodgers, et. al.. (2011). Tumour evolution inferred by single-cell sequencing. Nature. 472, 90-94;
- Nicholas Navin, James Hicks. (2011). Future medical applications of single-cell sequencing in cancer. Genome Medicine. 3, 31;
- Shenfeng Qiu, Shujun Luo, Oleg Evgrafov, Robin Li, Gary P. Schroth, et. al.. (2012). Single-neuron RNA-Seq: technical feasibility and reproducibility. Front. Gene.. 3;
- Scott A. Mackler, Brian P. Brooks, James H. Eberwine. (1992). Stimulus-induced coordinate changes in mRNA abundance in single postsynaptic hippocampal CA1 neurons. Neuron. 9, 539-548;
- Leonid L. Moroz, Andrea B. Kohn. (2013). Single-Neuron Transcriptome and Methylome Sequencing for Epigenomic Analysis of Aging. Methods in Molecular Biology. 323-352;
- Mugdha Khaladkar, Peter T. Buckley, Miler T. Lee, Chantal Francis, Mitra M. Eghbal, et. al.. (2013). Subcellular RNA Sequencing Reveals Broad Presence of Cytoplasmic Intron-Sequence Retaining Transcripts in Mouse and Rat Neurons. PLoS ONE. 8, e76194;
- Maxwell B. Zeigler, Daniel T. Chiu. (2013). Single-Cell Nanosurgery. Methods in Molecular Biology. 139-148;
- Alex K. Shalek, Rahul Satija, Xian Adiconis, Rona S. Gertner, Jellert T. Gaublomme, et. al.. (2013). Single-cell transcriptomics reveals bimodality in expression and splicing in immune cells. Nature. 498, 236-240;
- Vikas Bansal, Ryan Tewhey, Eric J Topol, Nicholas J Schork. (2011). The next phase in human genetics. Nat Biotechnol. 29, 38-39;
- F. B. Dean. (2001). Rapid Amplification of Plasmid and Phage DNA Using Phi29 DNA Polymerase and Multiply-Primed Rolling Circle Amplification. Genome Research. 11, 1095-1099;
- C. Zong, S. Lu, A. R. Chapman, X. S. Xie. (2012). Genome-Wide Detection of Single-Nucleotide and Copy-Number Variations of a Single Human Cell. Science. 338, 1622-1626;
- Roger S Lasken. (2013). Single-cell sequencing in its prime. Nat Biotechnol. 31, 211-212;
- Fuchou Tang, Catalin Barbacioru, Ellen Nordman, Bin Li, Nanlan Xu, et. al.. (2010). RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535;
- Yohei Sasagawa, Itoshi Nikaido, Tetsutaro Hayashi, Hiroki Danno, Kenichiro D Uno, et. al.. (2013). Quartz-Seq: a highly reproducible and sensitive single-cell RNA sequencing method, reveals non-genetic gene-expression heterogeneity. Genome Biol. 14;
- Simone Picelli, Åsa K Björklund, Omid R Faridani, Sven Sagasser, Gösta Winberg, Rickard Sandberg. (2013). Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 10, 1096-1098;
- Tamar Hashimshony, Florian Wagner, Noa Sher, Itai Yanai. (2012). CEL-Seq: Single-Cell RNA-Seq by Multiplexed Linear Amplification. Cell Reports. 2, 666-673;
- Jacqueline Morris, Jennifer M. Singh, James H. Eberwine. (2011). Transcriptome Analysis of Single Cells. JoVE;
- Saiful Islam, Una Kjällquist, Annalena Moliner, Pawel Zajac, Jian-Bing Fan, et. al.. (2012). Highly multiplexed and strand-specific single-cell RNA 5′ end sequencing. Nat Protoc. 7, 813-828;
- Andrea B. Kohn, Tatiana P. Moroz, Jeffrey P. Barnes, Mandy Netherton, Leonid L. Moroz. (2013). Single-Cell Semiconductor Sequencing. Methods in Molecular Biology. 247-284;
- Philip Brennecke, Simon Anders, Jong Kyoung Kim, Aleksandra A Kołodziejczyk, Xiuwei Zhang, et. al.. (2013). Accounting for technical noise in single-cell RNA-seq experiments. Nat Methods. 10, 1093-1095;
- B. Munsky, G. Neuert, A. van Oudenaarden. (2012). Using Gene Expression Noise to Understand Gene Regulation. Science. 336, 183-187;
- Daniel T. Chiu, Robert M. Lorenz. (2009). Chemistry and Biology in Femtoliter and Picoliter Volume Droplets. Acc. Chem. Res.. 42, 649-658.






