
Разработан метод анализа экспрессии генов на уровне индивидуальных клеток
14 февраля 2015
Разработан метод анализа экспрессии генов на уровне индивидуальных клеток
- 1668
- 0
- 6
Структура популяции моноядерных клеток периферической крови человека. Получена с помощью нового метода цитометрии CytoSeq.
Рисунок в полном размере.
-
Автор
-
Редактор
Благодаря новой технологии, за один эксперимент будет возможно изучить активности генов тысяч индивидуальных клеток. Метод CytoSeq позволяет оценить экспрессию произвольного числа генов, вплоть до всего транскриптома клетки.
Мы изучаем жизнь на все более тонких уровнях, однако по-прежнему существует не так много данных, которые можно получить для индивидуальных клеток, а не для клеточной популяции в целом. Анализировать содержание некоторых белков в единичных клетках позволяет метод проточной цитометрии. С помощью него можно разделять клеточные суспензии на специальных аппаратах, ориентируясь на флуоресценцию введенных в клетки меток . Если есть возможность получить антитела, связывающие определенный белок клетки, то, снабдив такие антитела флуоресцентными метками и обработав ими клетки, можно будет оценить яркость свечения каждой отдельной клетки, а значит, и количество целевого белка в ней. Однако флуоресцирующие антитела можно использовать только к белкам, находящимся на поверхности клетки, так как внутрь живых клеток антитела попасть не могут. Поэтому в анализе содержания белков на уровне отдельных клеток есть определенные ограничения.
Новейшие «навороты» метода подробно обсуждались на семинаре Совета молодых ученых, посвященном современным технологиям в проточной цитометрии (два замечательных видео из ИБХ) — Ред.
Что же касается активности генов, то есть содержания РНК в отдельных клетках, то до недавнего времени методов их анализа просто не было. Чаще всего РНК выделяется из больших количеств клеток, и данные по активности генов, полученные классическими методами — это «средняя температура по больнице». Если и было показано увеличение активности определенного гена, таким методом невозможно определить, увеличилась ли активность во всех клетках равномерно или же лишь в малой доле клеток, но более резко. Также невозможно получить точную информацию об исходном количестве клеточной РНК: для регистрации молекул, присутствующих в малых количествах, приходится амплифицировать («умножать») их число с помощью ПЦР, а она может протекать с разной эффективностью. Чтобы перейти к анализу содержания РНК в отдельных клетках, разрабатываются модификации классических методов, но их использование очень трудоемко, и стоимость знаний о нуклеиновых кислотах из каждой клетки, если их получать такими способами, очень высока [1]. Кроме того, традиционными методами за один эксперимент можно получить информацию о работе лишь ограниченного набора генов. Поэтому важной выглядит новая разработка американских ученых — метод CytoSeq, который позволяет получить информацию об активностях всех генов нескольких тысяч индивидуальных клеток за один эксперимент [2]. Новый метод цитометрии не обходится без этапа секвенирования [3–5].
Основной принцип новой технологии — присвоение каждой клетке и каждой молекуле РНК индивидуального «штрихкода». Для этого клетки сначала разделяют нехитрым способом: размазывают суспензию по плашке с лунками определенного размера, при этом клеток должно быть в 10 раз меньше, чем лунок, чтобы уменьшить вероятность попадания двух клеток в одну лунку. Затем в каждую лунку к клетке добавляют шарик, к которому прикреплено несколько десятков миллионов фрагментов ДНК (рис. 1). Каждый фрагмент включает в себя последовательность для захвата матричных РНК. Матричные РНК, как известно, несут на 3’-конце регуляторную последовательность из нескольких адениновых нуклеотидов — поли(А)-хвост [6]. Поэтому их можно «выловить» из смеси молекул, используя комплементарную последовательность из нескольких тимидинов, которой и снабжены фрагменты ДНК на шарике. Кроме того, в каждом фрагменте содержится универсальный праймер, с которого вся смесь молекул впоследствии амплифицируется, а затем секвенируется. А чтобы после этого понять, какая последовательность из какой клетки происходит, а также определить, сколько в ней изначально было копий каждой молекулы, в каждом фрагменте имеются маркеры — нуклеотиды, индивидуальные для шарика, и нуклеотиды, индивидуальные для фрагмента. После добавления шарика с метками в лунку с клеткой капают лизирующий буфер, который растворяет мембрану и высвобождает клеточное содержимое.
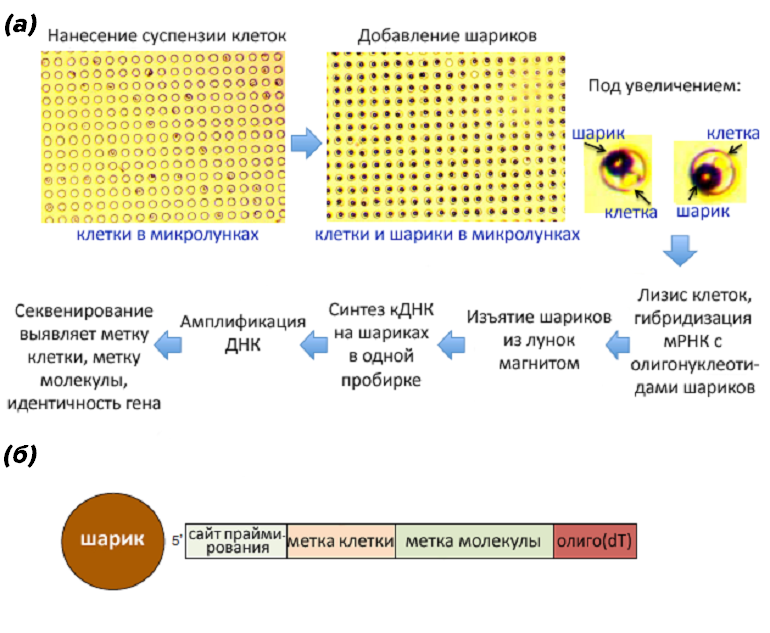
Рисунок 1. CytoSeq — метод оценки экспрессии генов индивидуальной клетки. а — Схема метода. В плашке 100 000 лунок, диаметр лунки 30 мкм, диаметр шарика 20 мкм, количество клеточной суспензии таково, что клетка попадает лишь в каждую десятую лунку. Процедура подробно описана в тексте. Секвенирование — «считывание штрихкода» индивидуальных мРНК. б — Устройство олигонуклеотидов, прикрепленных к шарикам. Концевые фрагменты — сайт праймирования для ПЦР и тимидиновый хвостик — одинаковы у всех олигонуклеотидов всех шариков; метка клетки — последовательность, одинаковая у всех молекул одного шарика (одной клетки); метка молекулы — последовательность, специфичная для каждого олигонуклеотида шарика (индивидуальной мРНК).
адаптировано из [2]
В каждой клетке содержится около 100 000 индивидуальных молекул РНК, так что каждая из них получает шанс связаться с одной из меток на поверхности шарика. Благодаря такому связыванию к каждой молекуле добавляется случайная метка, по которой ее можно отличить, а также метка своего шарика — одна и та же у всех молекул, происходящих из данной клетки. Таким образом и осуществляется «штрихкодирование». После этого все шарики извлекаются из лунок, и на всей пойманной РНК достраивается комплементарная цепочка ДНК, чтобы было удобнее работать дальше: ДНК более стабильна, чем РНК. После этого потенциально возможно определить последовательность всех молекул РНК, которые были во всех исследованных индивидуальных клетках. Правда, обойдется это недешево — по подсчетам авторов, в $15 000 на 1000 клеток.
Тем не менее со временем производительность секвенирования, как ожидается, будет расти, а стоимость его — падать, что характерно для большинства технологий [7]. Так что процедура анализа всего транскриптома индивидуальных клеток когда-нибудь станет по карману большему числу лабораторий. На данный момент авторы рекомендуют ограничиваться анализом работы определенных генов, интересующих исследователей. Ведь даже знание активностей лишь ограниченного их набора в тысячах индивидуальных клеток даст много новых возможностей. Тем более, взрывной рост объемов баз данных, сопровождающий развитие многочисленных -омик [8], уже сейчас порождает проблемы c обработкой и хранением информации [9].
Для апробации своего метода ученые посмотрели уровни активностей генов цитокинов в Т-клетках после стимуляции рецепторов на их поверхностях [2]. Оказалось, что уровни цитокинов после стимуляции резко повышаются лишь у небольшого числа клеток, причем у каждой из среагировавших клеток растет активность определенного набора генов цитокинов. С помощью своего метода исследователи обнаружили единичные отреагировавшие на стимуляцию клетки среди нескольких тысяч! Кроме того, стало возможным проследить индивидуальные различия в реакциях этих клеток. Данные о вкладе отдельных клеток в жизнь популяции помогут нам больше узнать о самых базовых принципах клеточных взаимодействий в организме, механизмах дифференцировки и онкогенеза [1], [9].
Постепенно в научной среде растет доля «сухих» лабораторий, не проводящих экспериментальной работы (это удел лабораторий «мокрых»), а анализирующих и сопоставляющих разные виды данных из биомедицинских баз. Ведь именно биоинформатика позволяет разгрести авгиевы конюшни документированных по всему миру симптомов, побочных и прямых действий лекарств, всевозможных мутаций, интегрировать данные миллионов пациентов и экспериментов... И получить порой очень неожиданные результаты [10]. Возможно, метод CytoSeq окажет неоценимую услугу «белоручкам» и ускорит «пришествие» Интегративной Биологии, которая будет ворочать огромными массивами разнородных «омических» данных и поспособствует развитию доказательной и становлению персональной медицины.
Литература
- Секвенирование единичных клеток (версия — Metazoa);
- H. Christina Fan, Glenn K. Fu, Stephen P. A. Fodor. (2015). Combinatorial labeling of single cells for gene expression cytometry. Science. 347;
- Код жизни: прочесть не значит понять;
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК);
- Огурцы-убийцы, или Как встретились Джим Уотсон и Гордон Мур;
- мРНКаааауу;
- Технология: $1000 за геном;
- «Омики» — эпоха большой биологии;
- Биоинформатика: большие БД против «большого Р»;
- Вычислительное будущее биологии.






