
Аптамеры: графический гайд
30 января 2021
Аптамеры: графический гайд
- 2914
- 2
- 8
-
Автор
-
Редакторы
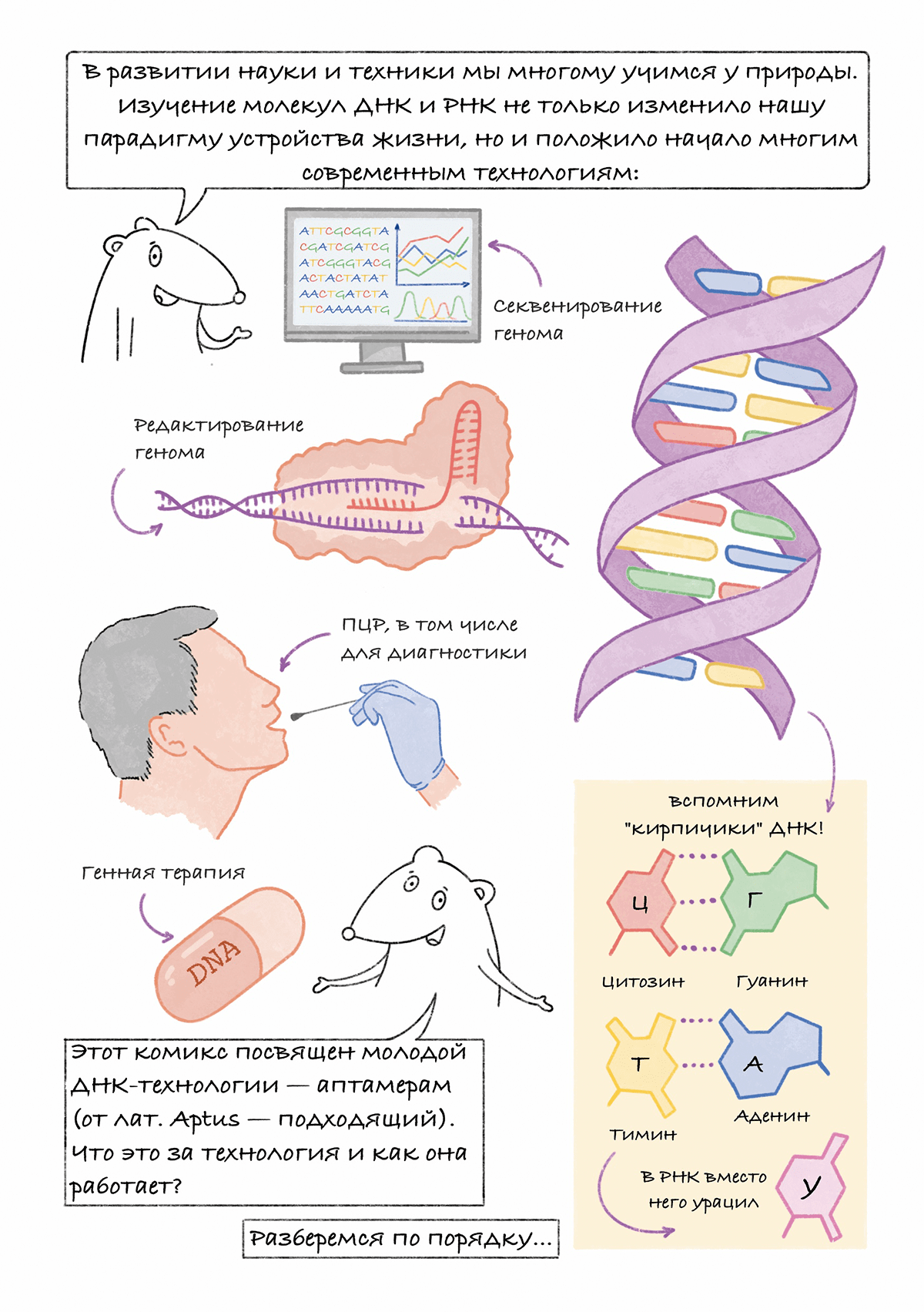
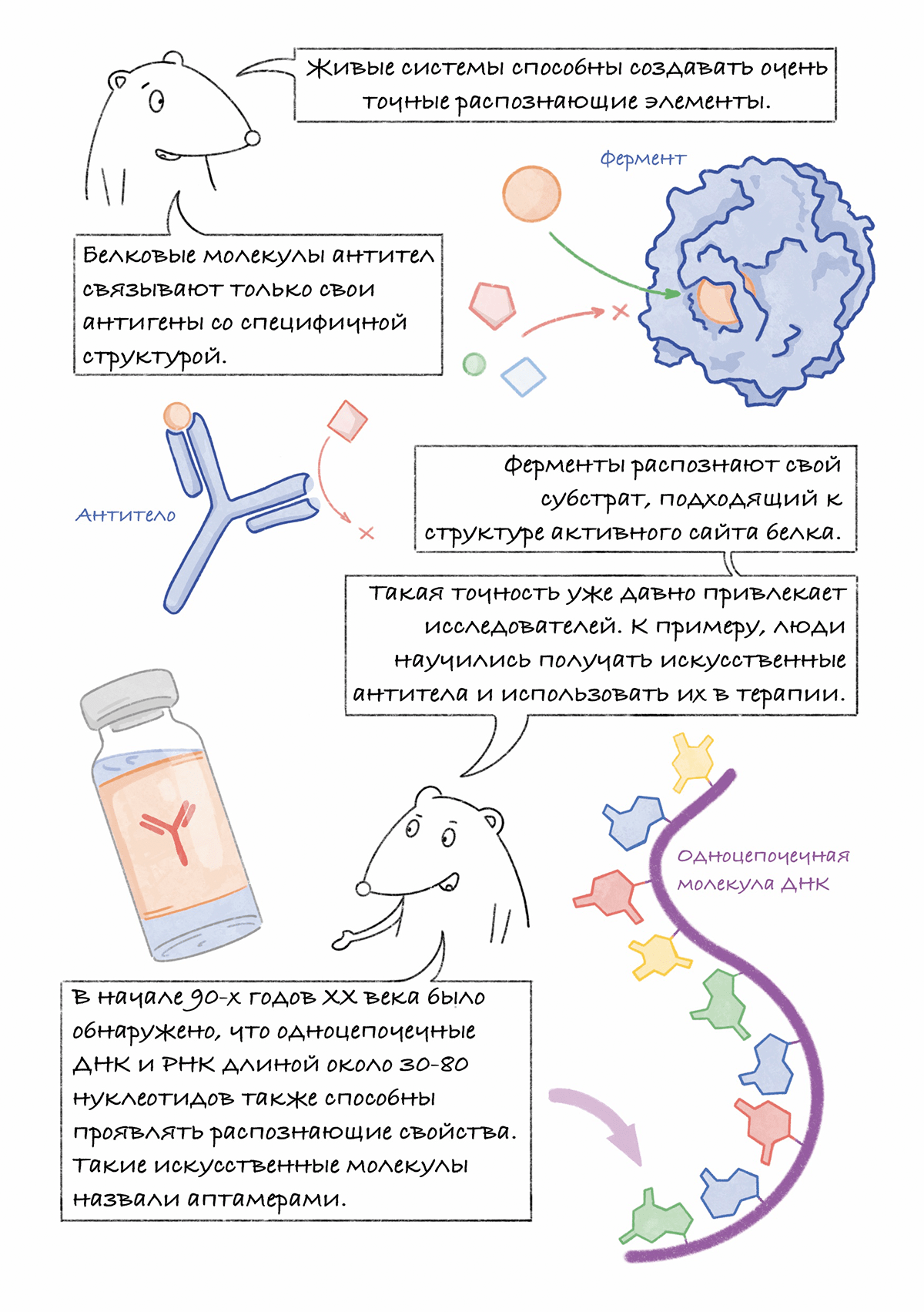
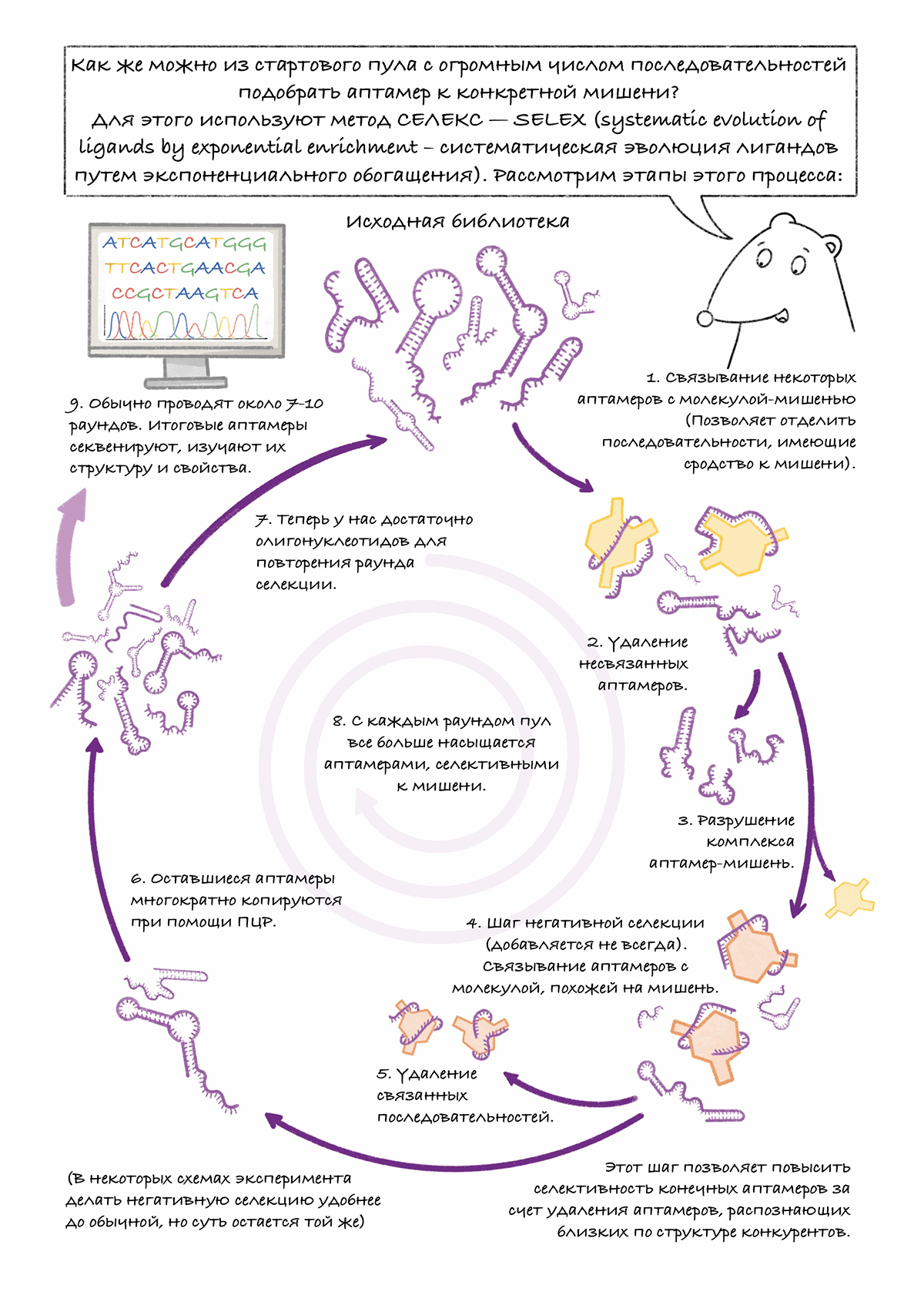
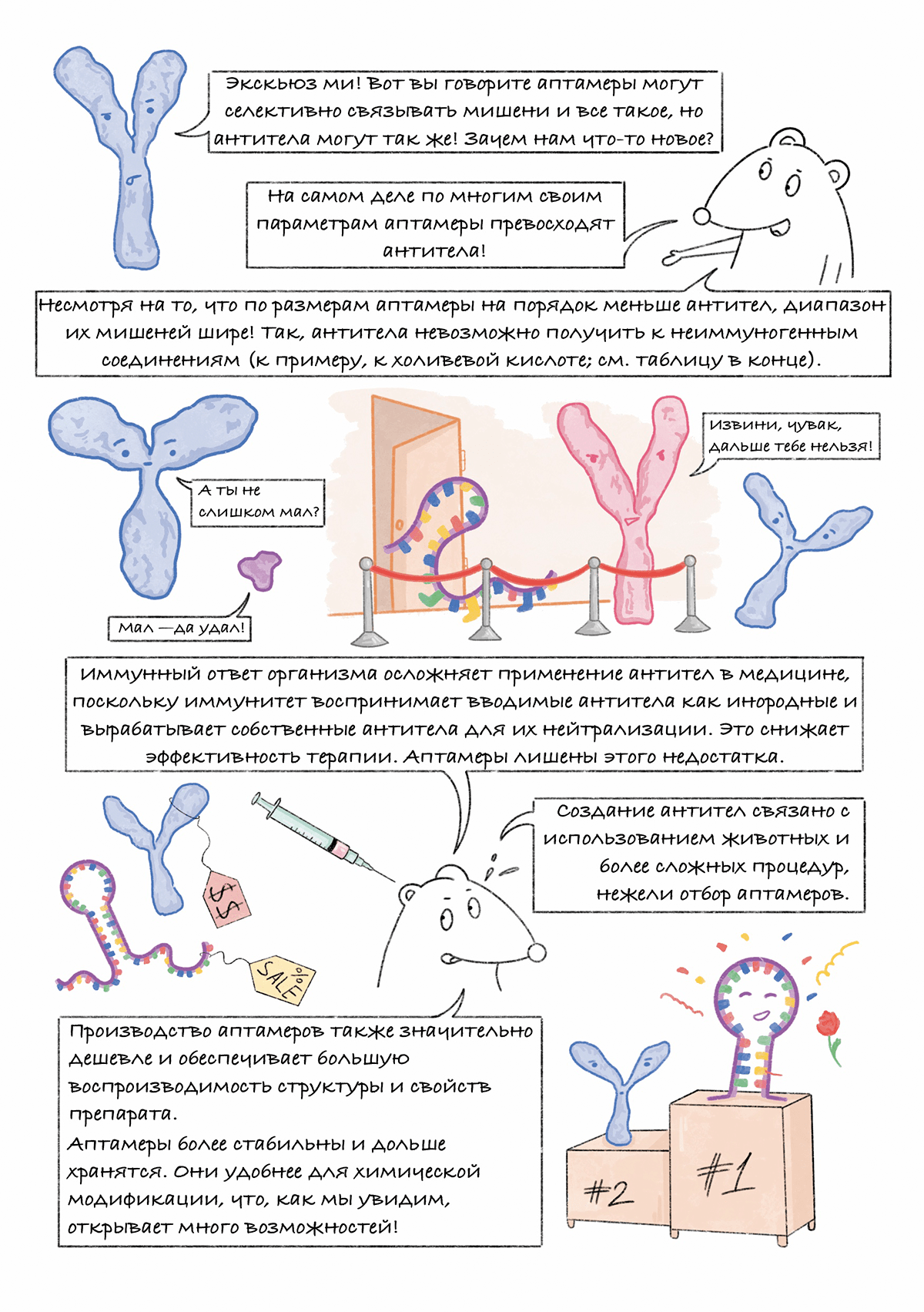
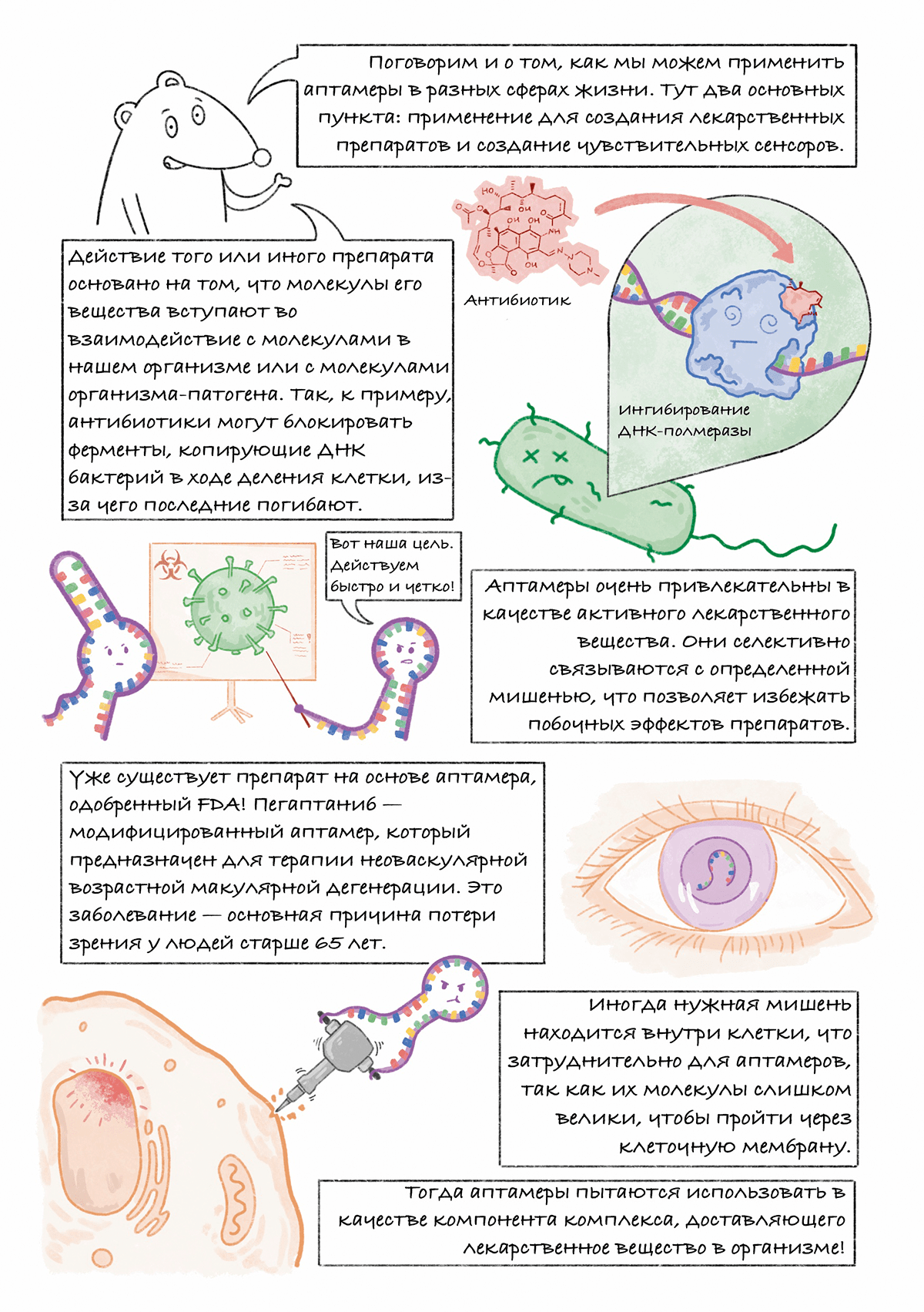
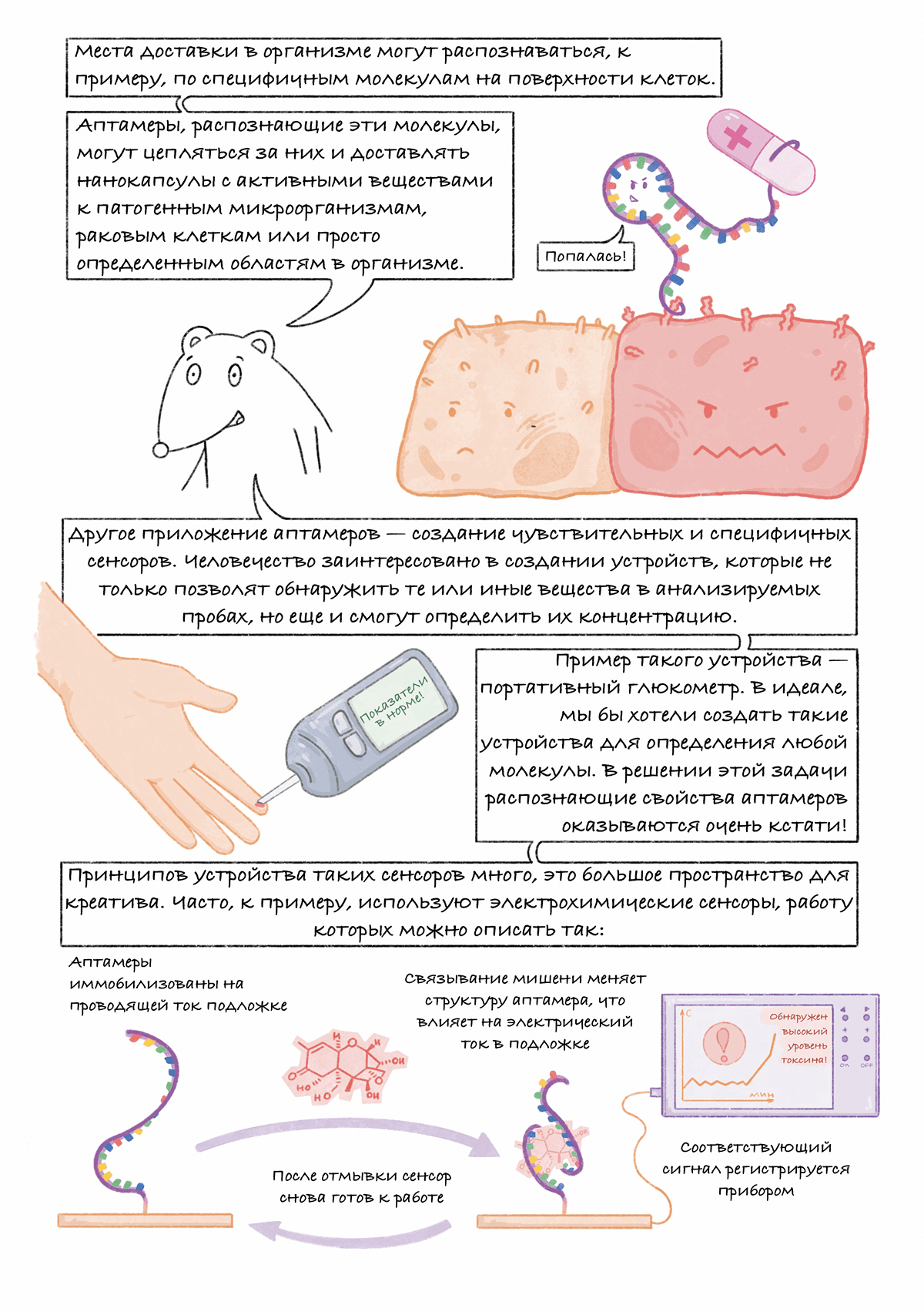
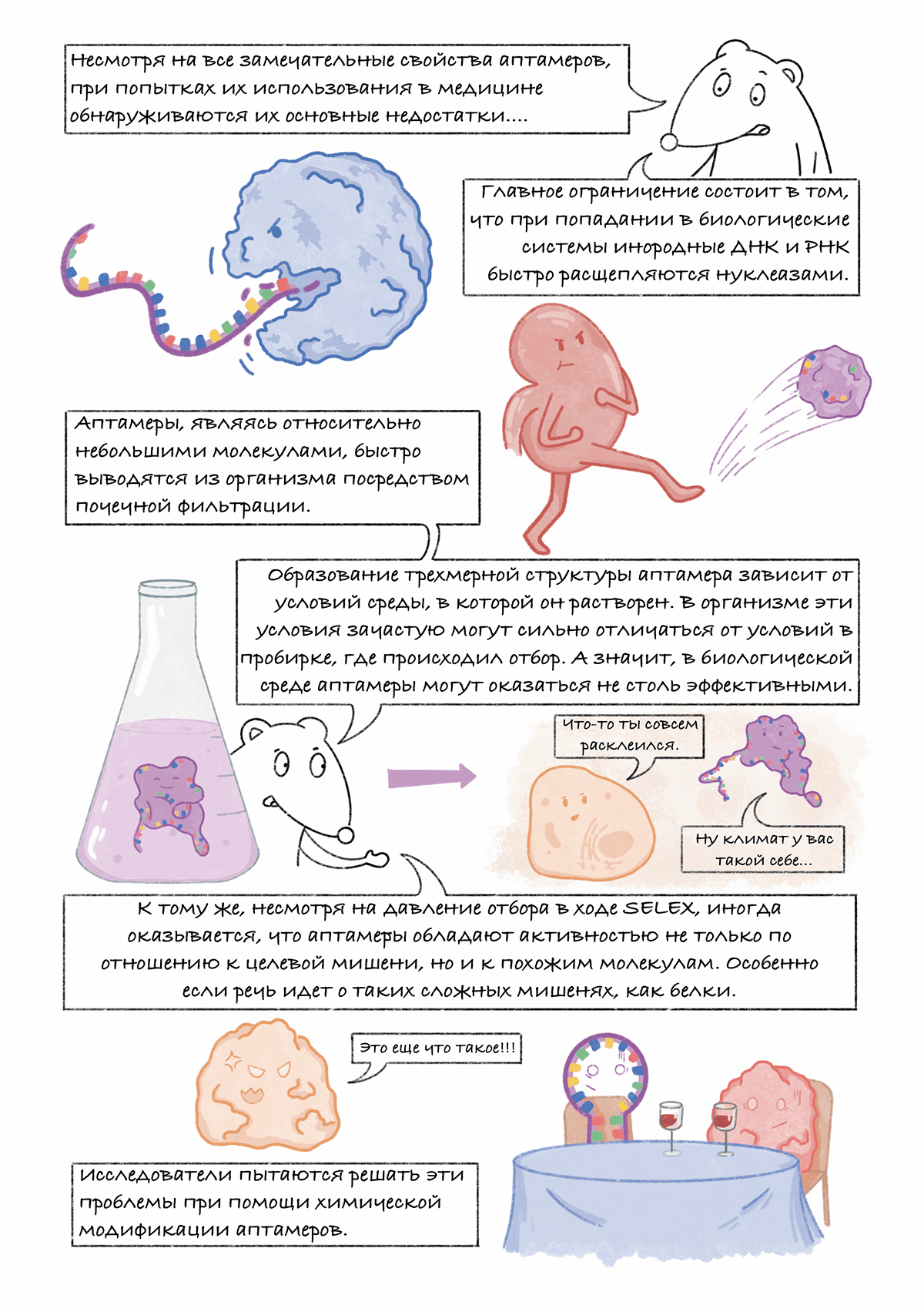
Комикс на конкурс «Био/Мол/Текст»: ДНК-технологии, такие как ПЦР и секвенирование генома, изменили мир, но наука не останавливается на достигнутом и постоянно создает новое. Аптамеры — это молодая ДНК-технология, появившаяся в 90-х годах ХХ века и быстро заинтересовавшая химиков, биологов и многих междисциплинарных специалистов.
Конкурс «Био/Мол/Текст»-2020/2021
 Эта работа заняла первое место в номинации «Наглядно о ненаглядном» конкурса «Био/Мол/Текст»-2020/2021.
Эта работа заняла первое место в номинации «Наглядно о ненаглядном» конкурса «Био/Мол/Текст»-2020/2021.

Генеральный партнер конкурса — ежегодная биотехнологическая конференция BiotechClub, организованная международной инновационной биотехнологической компанией BIOCAD.

Спонсор конкурса — компания SkyGen: передовой дистрибьютор продукции для life science на российском рынке.

Спонсор конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
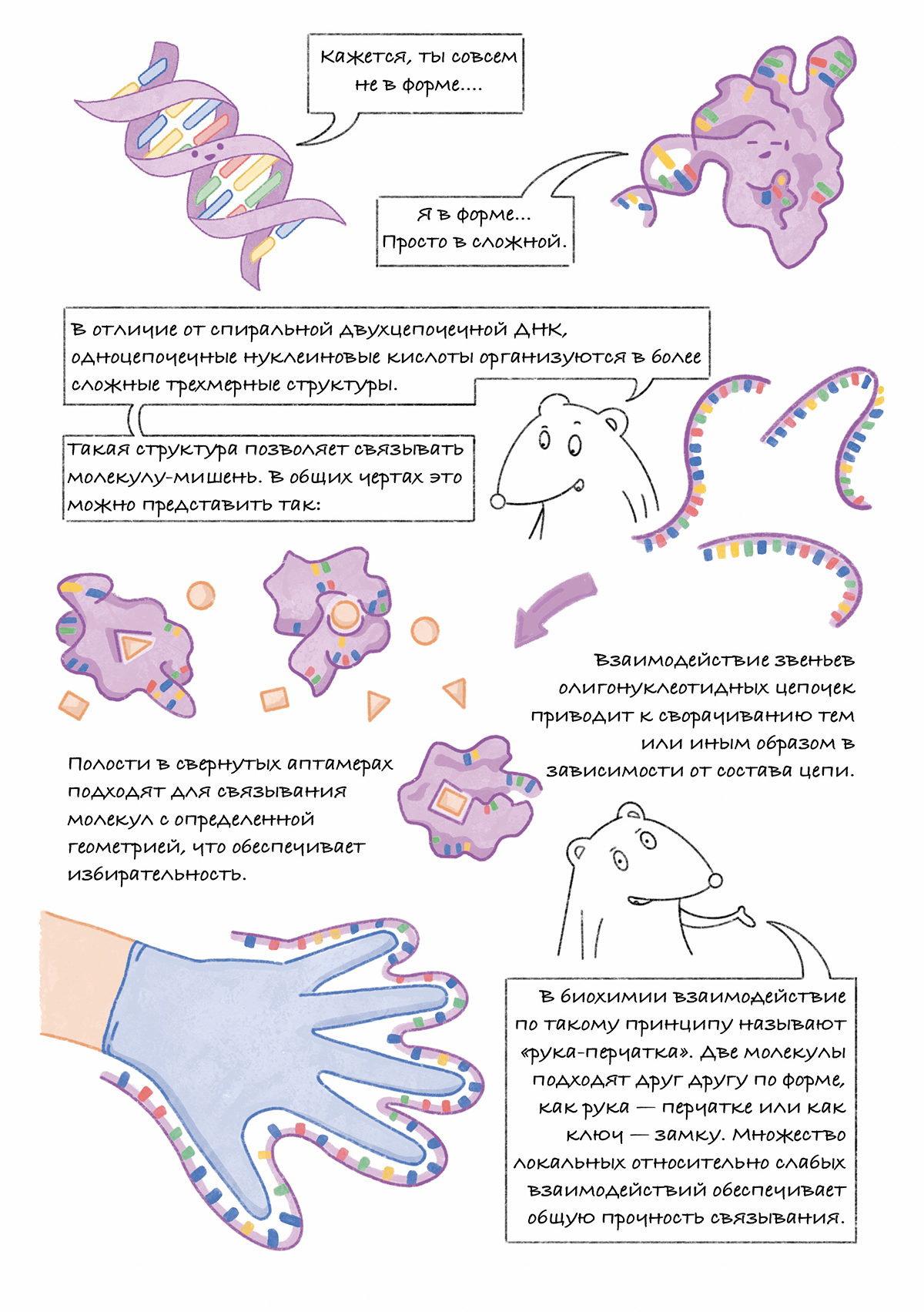
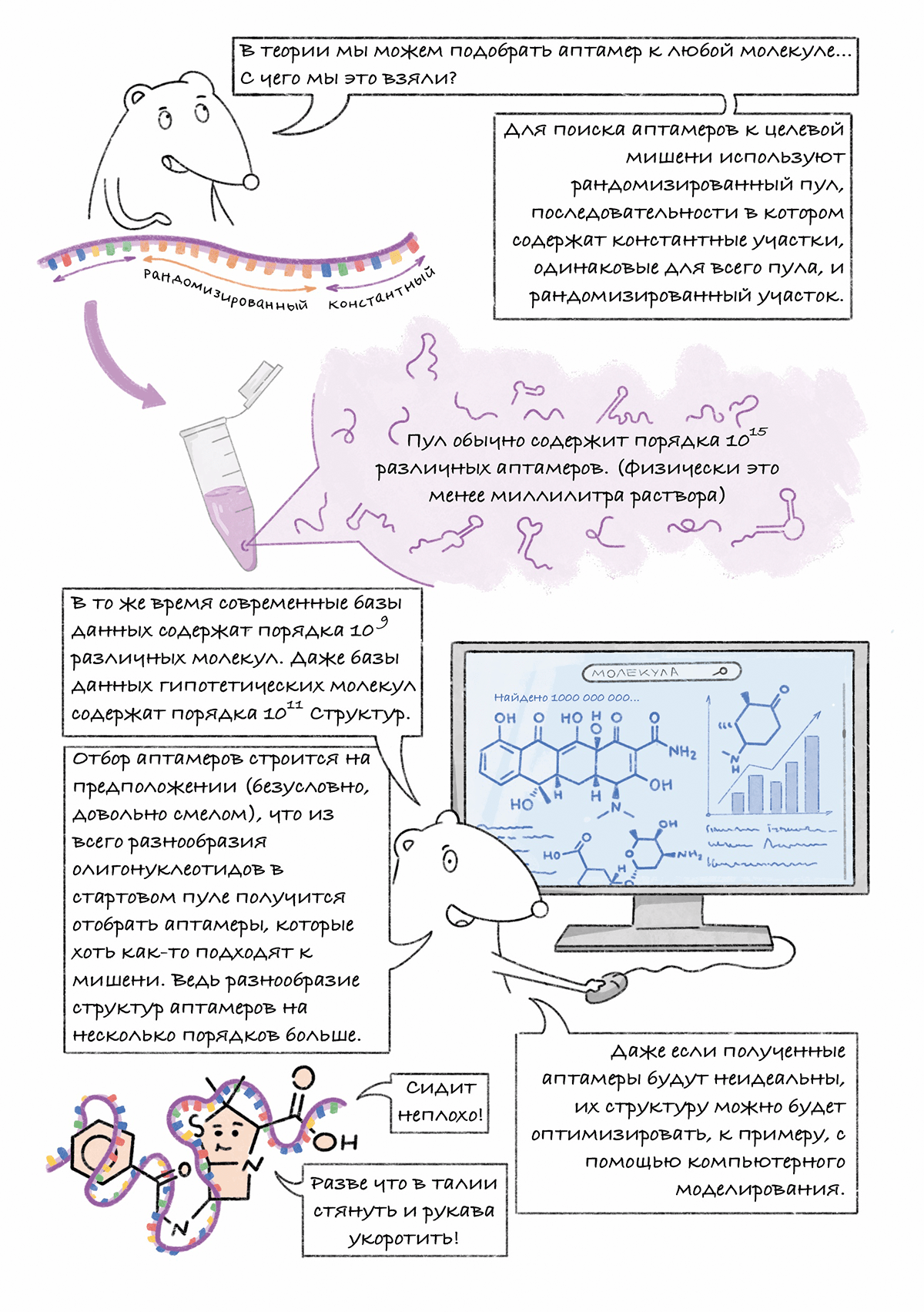
Что такое аптамеры? Как это работает? Почему их называют синтетическими антителами? Экскурс в эти вопросы вы найдете на страницах комикса. Иллюстративный гайд рассказывает об основных идеях технологии аптамеров, направлениях исследований и развития в этой области. Комикс будет интересен как специалистам, работающим в смежных областях, так и читателям, более далеким от молекулярной биологии.
Комикс в формате pdf.
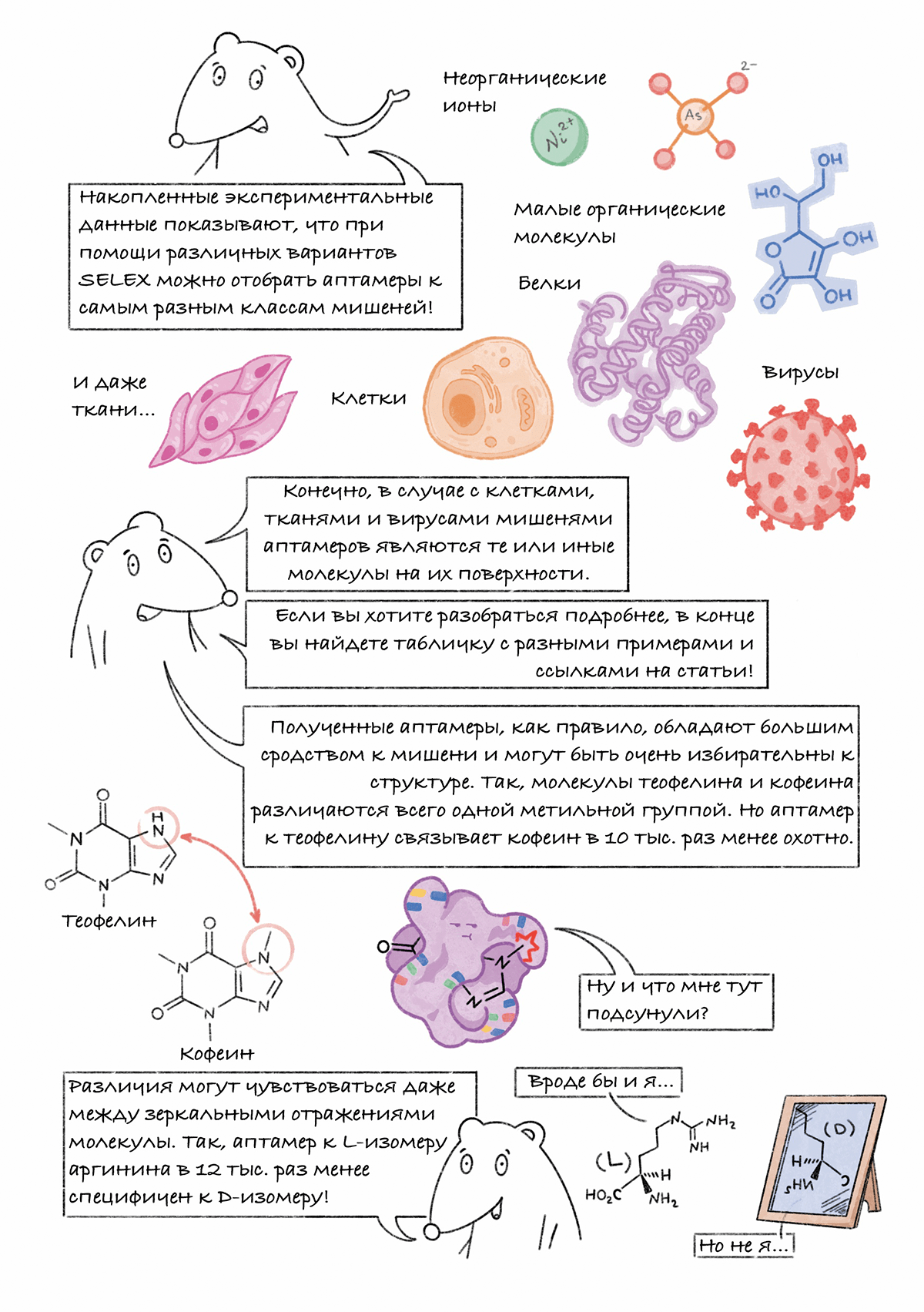
| Класс мишени | Пример | Источник |
|---|---|---|
| Белки | Фибриноген | [8] |
| Белок Tat вируса иммунодефицита человека (HIV-1) | [9] | |
| Рицин | [10] | |
| Пептиды | Аргинин-вазопрессин | [11] |
| Нейропептид Y | [12] | |
| Пептидогликан | [13] | |
| Неорганические ионы | Никель(II) | [14] |
| Арсенат- и арсенит-анионы | [15] | |
| Кадмий(II) | [16] | |
| Малые органические молекулы | Холевая кислота | [17] |
| Бисфенол А | [18] | |
| Кокаин | [19] | |
| Полисахариды | (1→3)-β-D-глюкан (курдлан) | [20] |
| Липополисахарид (ЛПС) | [21] | |
| Целлобиоза | [22] | |
| Вирусы | Вирус гепатита С (неструктурный белок 3) | [23] |
| Вирус человеческого гриппа (белок гемагглютинин) | [24] | |
| Норовирусы человека (HuNoV) | [25] | |
| Клетки | Клетки гепатоцеллюлярной карциномы человека (LH86) | [26] |
| Мелкоклеточный рак легкого | [27] | |
| Lactobacillum acidophilius | [28] | |
| Ткани | Ткани рака груди и клетки MCF7 | [29] |
| Метастазы рака кишечника в мышах (p68, РНК-хеликаза) | [30] |
Литература
- Kyung-Mi Song, Seonghwan Lee, Changill Ban. (2012). Aptamers and Their Biological Applications. Sensors. 12, 612-631;
- Sergey A. Lapa, Alexander V. Chudinov, Edward N. Timofeev. (2016). The Toolbox for Modified Aptamers. Mol Biotechnol. 58, 79-92;
- Jiehua Zhou, John Rossi. (2017). Aptamers as targeted therapeutics: current potential and challenges. Nat Rev Drug Discov. 16, 181-202;
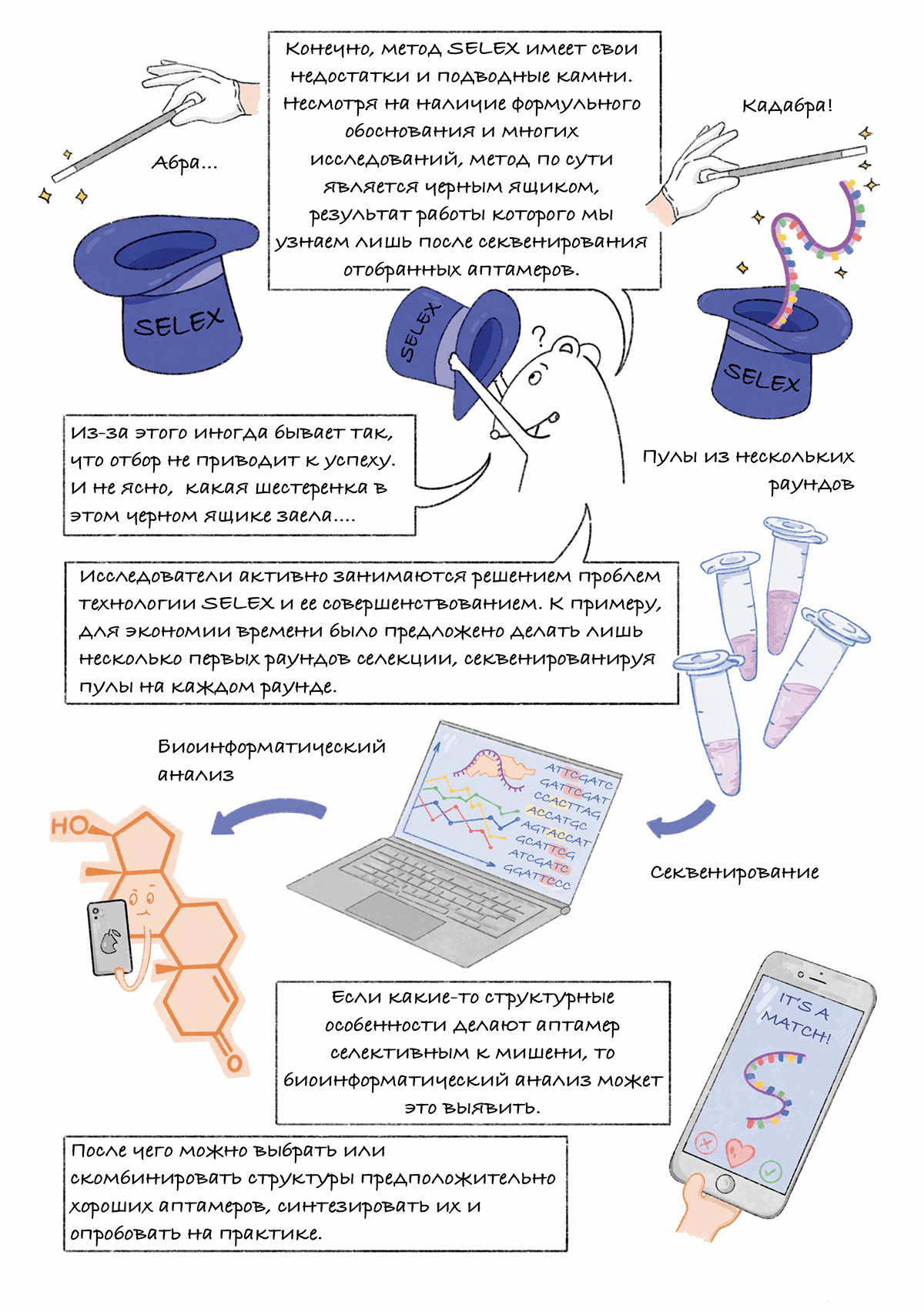
- Shunxiang Gao, Xin Zheng, Binghua Jiao, Lianghua Wang. (2016). Post-SELEX optimization of aptamers. Anal Bioanal Chem. 408, 4567-4573;
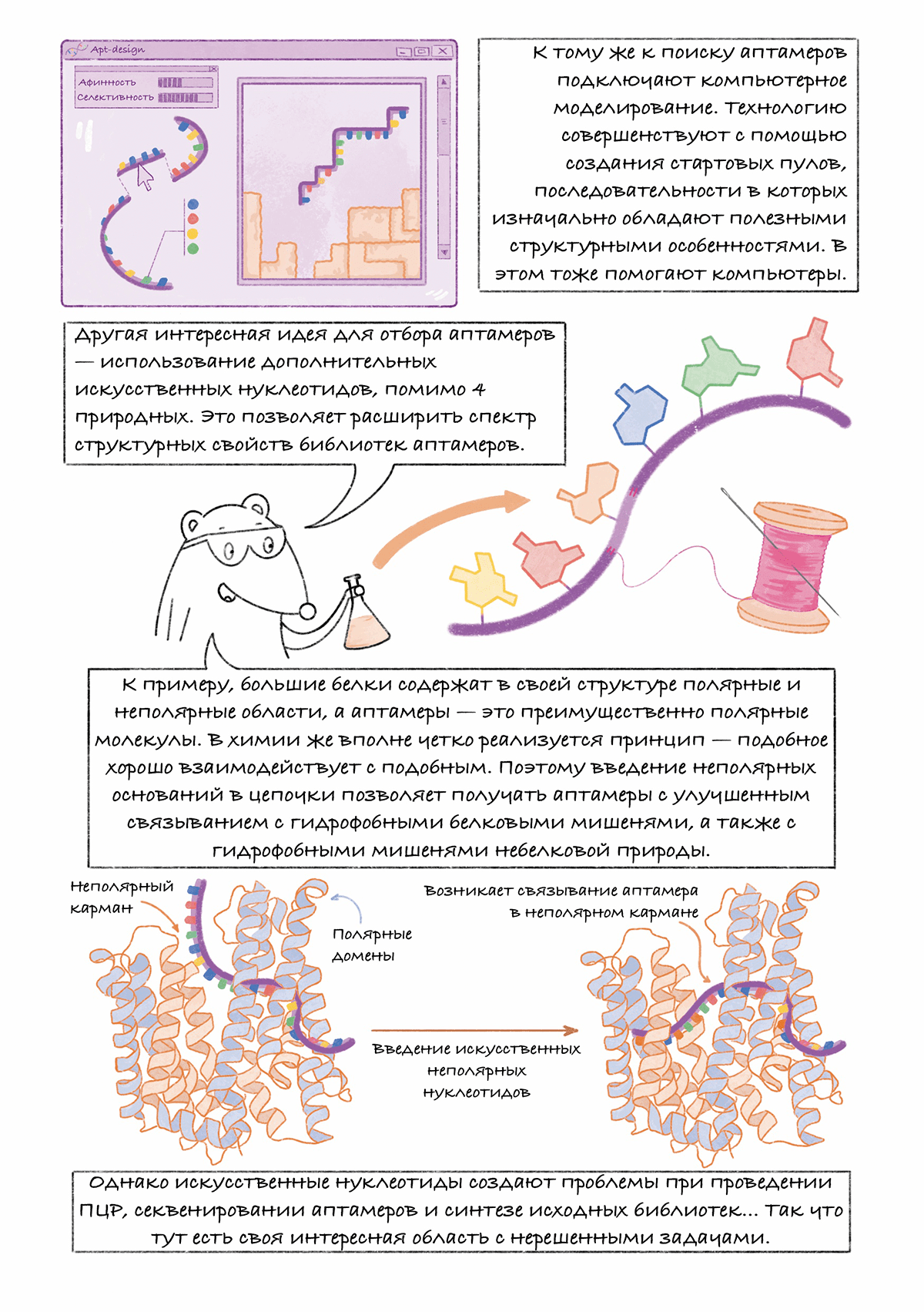
- Andrew Kinghorn, Lewis Fraser, Shaolin Liang, Simon Shiu, Julian Tanner. (2017). Aptamer Bioinformatics. IJMS. 18, 2516;
- Наступает эра стереоизомеров;
- Язык программирования: «ДНК»;
- Minyong Li, Na Lin, Zhen Huang, Lupei Du, Craig Altier, et. al.. (2008). Selecting Aptamers for a Glycoprotein through the Incorporation of the Boronic Acid Moiety. J. Am. Chem. Soc.. 130, 12636-12638;
- Rika Yamamoto, Masato Katahira, Satoshi Nishikawa, Tadashi Baba, Kazunari Taira, Penmetcha K. R. Kumar. (2000). A novel RNA motif that binds efficiently and specifically to the Tat protein of HIV and inhibits the trans -activation by Tat of transcription in vitro and in vivo. Genes to Cells. 5, 371-388;
- Jijun Tang, Jianwei Xie, Ningsheng Shao, Yan Yan. (2006). The DNA aptamers that specifically recognize ricin toxin are selected by twoin vitro selection methods. Electrophoresis. 27, 1303-1311;
- K. P. Williams, X.-H. Liu, T. N. M. Schumacher, H. Y. Lin, D. A. Ausiello, et. al.. (1997). Bioactive and nuclease-resistant L-DNA ligand of vasopressin. Proceedings of the National Academy of Sciences. 94, 11285-11290;
- Daniela Proske, Martin Höfliger, Richard M. Söll, Annette G. Beck-Sickinger, Michael Famulok. (2002). A Y2 Receptor Mimetic Aptamer Directed against Neuropeptide Y. Journal of Biological Chemistry. 277, 11416-11422;
- Iêda Mendes Ferreira, Camila Maria de Souza Lacerda, Lígia Santana de Faria, Cristiane Rodrigues Corrêa, Antero Silva Ribeiro de Andrade. (2014). Selection of Peptidoglycan-Specific Aptamers for Bacterial Cells Identification. Appl Biochem Biotechnol. 174, 2548-2556;
- Hofmann H. P., Limmer S., Hornung V., Sprinzl M. (1997). Ni2+-binding RNA motifs with an asymmetric purine-rich internal loop and a G-A base pair. RNA (New York, N.Y.). 3(11), 1289–1300;
- Mina Kim, Hyun-Ju Um, SunBaek Bang, Sang-Hee Lee, Suk-Jung Oh, et. al.. (2009). Arsenic Removal from Vietnamese Groundwater Using the Arsenic-Binding DNA Aptamer. Environ. Sci. Technol.. 43, 9335-9340;
- Yuangen Wu, Shenshan Zhan, Lumei Wang, Pei Zhou. (2014). Selection of a DNA aptamer for cadmium detection based on cationic polymer mediated aggregation of gold nanoparticles. Analyst. 139, 1550-1561;
- Teru Kato, Taro Takemura, Kazuyoshi Yano, Kazunori Ikebukuro, Isao Karube. (2000). In vitro selection of DNA aptamers which bind to cholic acid. Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression. 1493, 12-18;
- Minjoung Jo, Ji-Young Ahn, Joohyung Lee, Seram Lee, Sun Woo Hong, et. al.. (2011). Development of Single-Stranded DNA Aptamers for Specific Bisphenol A Detection. Oligonucleotides. 21, 85-91;
- Milan N. Stojanovic, Paloma de Prada, Donald W. Landry. (2001). Aptamer-Based Folding Fluorescent Sensor for Cocaine. J. Am. Chem. Soc.. 123, 4928-4931;
- Swee Yang Low, Jane E. Hill, Jordan Peccia. (2009). DNA aptamers bind specifically and selectively to (1→3)-β-d-glucans. Biochemical and Biophysical Research Communications. 378, 701-705;
- J.L. Ding, S.T. Gan, B. Ho. (2009). Single-Stranded DNA Oligoaptamers: Molecular Recognition and LPS Antagonism Are Length- and Secondary Structure-Dependent. J Innate Immun. 1, 46-58;
- Q. Yang, I. J. Goldstein, H.-Y. Mei, D. R. Engelke. (1998). DNA ligands that bind tightly and selectively to cellobiose. Proceedings of the National Academy of Sciences. 95, 5462-5467;
- Kotaro Fukuda, Daesety Vishnuvardhan, Satoru Sekiya, Joonsung Hwang, Nobuko Kakiuchi, et. al.. (2000). Isolation and characterization of RNA aptamers specific for the hepatitis C virus nonstructural protein 3 protease. European Journal of Biochemistry. 267, 3685-3694;
- Tomoko S. Misono, Penmetcha K.R. Kumar. (2005). Selection of RNA aptamers against human influenza virus hemagglutinin using surface plasmon resonance. Analytical Biochemistry. 342, 312-317;
- Blanca I. Escudero-Abarca, Soo Hwan Suh, Matthew D. Moore, Hari P. Dwivedi, Lee-Ann Jaykus. (2014). Selection, Characterization and Application of Nucleic Acid Aptamers for the Capture and Detection of Human Norovirus Strains. PLoS ONE. 9, e106805;
- Ling Meng, Liu Yang, Xiangxuan Zhao, Lucy Zhang, Haizhen Zhu, et. al.. (2012). Targeted Delivery of Chemotherapy Agents Using a Liver Cancer-Specific Aptamer. PLoS ONE. 7, e33434;
- Hui William Chen, Colin D. Medley, Kwame Sefah, Dihua Shangguan, Zhiwen Tang, et. al.. (2008). Molecular Recognition of Small-Cell Lung Cancer Cells Using Aptamers. ChemMedChem. 3, 991-1001;
- Camille L. A. Hamula, Hongquan Zhang, Le Luo Guan, Xing-Fang Li, X. Chris Le. (2008). Selection of Aptamers against Live Bacterial Cells. Anal. Chem.. 80, 7812-7819;
- Shaohua Li, Hua Xu, Hongmei Ding, Yanping Huang, Xiaoxiao Cao, et. al.. (2009). Identification of an aptamer targeting hnRNP A1 by tissue slide‐based SELEX. J. Pathol.. 218, 327-336;
- Jing Mi, Yingmiao Liu, Zahid N Rabbani, Zhongguang Yang, Johannes H Urban, et. al.. (2010). In vivo selection of tumor-targeting RNA motifs. Nat Chem Biol. 6, 22-24.







