Язык программирования: «ДНК»
27 октября 2018
Язык программирования: «ДНК»
- 2765
- 0
- 11
От строчек кода до программируемых роботов!
-
Автор
-
Редакторы
Темы
Статья на конкурс «био/мол/текст»: Точная последовательность нуклеотидов в цепях РНК и ДНК, единственная ошибка в которой может привести к отказу в работе всей молекулы, невольно заставляет задуматься о схожести устройства нуклеиновых кислот с компьютерной программой, которая может «вылететь», если в ее коде всего в одном месте проставлена неверная буква. Данная статья посвящена необычным функциям, которые могут выполнять нуклеиновые кислоты. Это рассказ о различных молекулярных механизмах, логических схемах и даже роботах, созданных на основе ДНК, на которые мы предлагаем взглянуть почти как на блок-схемы и строчки программного кода.
Конкурс «био/мол/текст»-2018
Эта работа опубликована в номинации «Свободная тема» конкурса «био/мол/текст»-2018.

Генеральный спонсор конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

Спонсором приза зрительских симпатий выступил медико-генетический центр Genotek.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
В основу базовых знаний человека о биохимии его организма положена центральная догма молекулярной биологии, которая гласит, что переход генетической информации осуществляется последовательно, в одном направлении: от ДНК к РНК, а затем от РНК к белку. Разумеется, из этой догмы существуют свои исключения, как, например, подробно описанные на сайте ретровирусное направление передачи от РНК к ДНК, или прионное направление белок—белок. Тем не менее для подавляющего большинства организмов, правило выполняется.
Однако понятие переноса информации является термином весьма обтекаемым, и в данной статье внимание уделяется тому, как можно интерпретировать данный термин на базе известных нам этапов выполнения некоторых биологических процессов, и какие достижения прогнозируются или уже сделаны на основании этих интерпретаций. Иными словами, давайте взглянем на мир ДНК и РНК с точки зрения синтетического биолога: как это работает и как это можно изменить таким образом, чтобы оно работало именно так, как мы того пожелаем [1], [2].
Рассмотрим в качестве примера трансляцию по матрице мРНК — процесс синтеза белка с учетом заложенной в РНК информации. Процесс укладывается в повторяющиеся друг за другом три стадии, а именно инициацию, элонгацию и терминацию, и представляет собой типичный многоуровневый цикл. Подобные циклы легко укладываются в алгоритмы, а соответственно, могут быть записаны на большинстве современных языков программирования.
Например, для представленной на рисунке 1 схемы трансляции в примитивном виде можно записать следующее. ЕСЛИ рибосома встречает при движении вдоль мРНК стартовый кодон (триплет АУГ), ТО прикрепить к большой субъединице рибосомы аминокислоту метионин И затем НАЧАТЬ цикл. Сместить рибосому вдоль мРНК, И ЕСЛИ рибосома встречает при движении вдоль мРНК кодон, соответствующий определенной аминокислоте, ТО прикрепить к большой субъединице рибосомы эту аминокислоту И, ИНАЧЕ ЕСЛИ рибосома встречает при движении вдоль мРНК стоп-кодон, ТО открепить цепь белка от рибосомы и ЗАВЕРШИТЬ цикл.
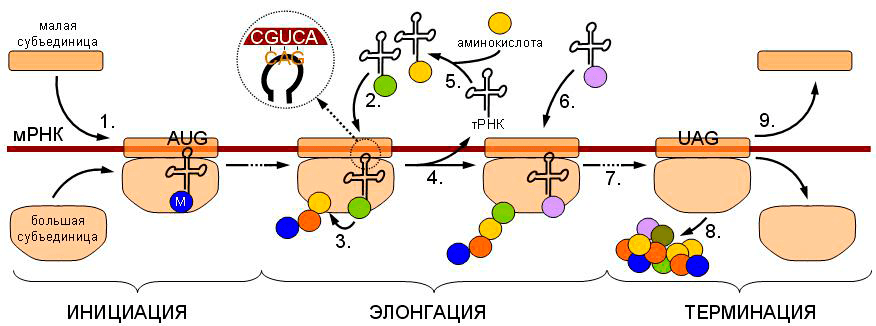
Рисунок 1. Схематичное изображение процесса трансляции. Рибосома (две ее субъединицы изображены на картинке) движется вдоль нити мРНК, и, когда встречает на своем пути стартовый кодон метионина, воспринимает его как сигнал к началу цикла. Цикл заключается в наращивании белковой цепочки вплоть до момента, когда встречается стоп-кодон (УАГ на рисунке), воспринимаемый рибосомой как сигнал к завершению цикла.
Таким образом, операция, схематично представленная на рисунке 1, описывается простейшими операторами, присутствующими практически в любом языке программирования. Задавшись целью наиболее точно и верно описать весь процесс трансляции, при написании кода можно учесть в качестве начала цикла не стартовый кодон, а последовательность Шайна-Дальгарно, или Козак, или 5′-кэп; в теле цикла будет необходимо подробно описать сравнение антикодона текущей тРНК с текущим кодоном мРНК; описать диссоциацию рибосомных субъединиц и что делать в случае, если стоп-кодон так и не повстречался; задать возможность прохождения по одной мРНК нескольких рибосом одновременно — на самом деле, пунктов очень большое количество, однако все они могут быть включены в код программы посредством вложенных циклов с условием.
С подобной же идеей можно «разложить» в программный код и другие явления: репликацию ДНК или транскрипцию, да и не только матричные процессы. Например, и процесс прикрепления аминокислот к тРНК, и расщепляющие функции малых интерферирующих РНК, и работа R-плазмид или систем CRISPR-Cas основаны на принципах начала выполнения процедуры при встрече на пути исполнителя необходимого набора нуклеотидов. Этапы процедуры, а также ее завершение, если она является сложной, также определяются исключительно информацией, заложенной в коде соответствующих РНК и ДНК.
Иными словами, последовательность нуклеотидов в нуклеиновых кислотах, как и последовательности символов в языках программирования, складывается в определенные «слова», которые воспринимаются исполнителями как команды проделать ту или другую операцию. С такого ракурса буквенную запись нуклеотидов (А, Т (У), Г, Ц) можно рассматривать как программный код, команды которого написаны с помощью четырехбуквенного алфавита. А сами молекулы нуклеиновых кислот в таком случае являются физическим воплощением подобного языка — реально существующим языком программирования.
Если исполнителями виртуальных языков программирования являются ЭВМ, то исполнителями реального языка ДНК в большинстве случаев можно назвать ферменты. Тем не менее при более детальном взгляде можно обнаружить, что некоторые реакции, выполняемые по принципу узнавания последовательностей РНК и ДНК, катализируются самими же молекулами РНК и ДНК. В частности, речь идет о каталитической активности особых последовательностей РНК, называемых рибозимами. Благодаря их существованию, нуклеиновые кислоты являются не только реальным, но еще и способным к самоисполнению программным кодом.
Макросы
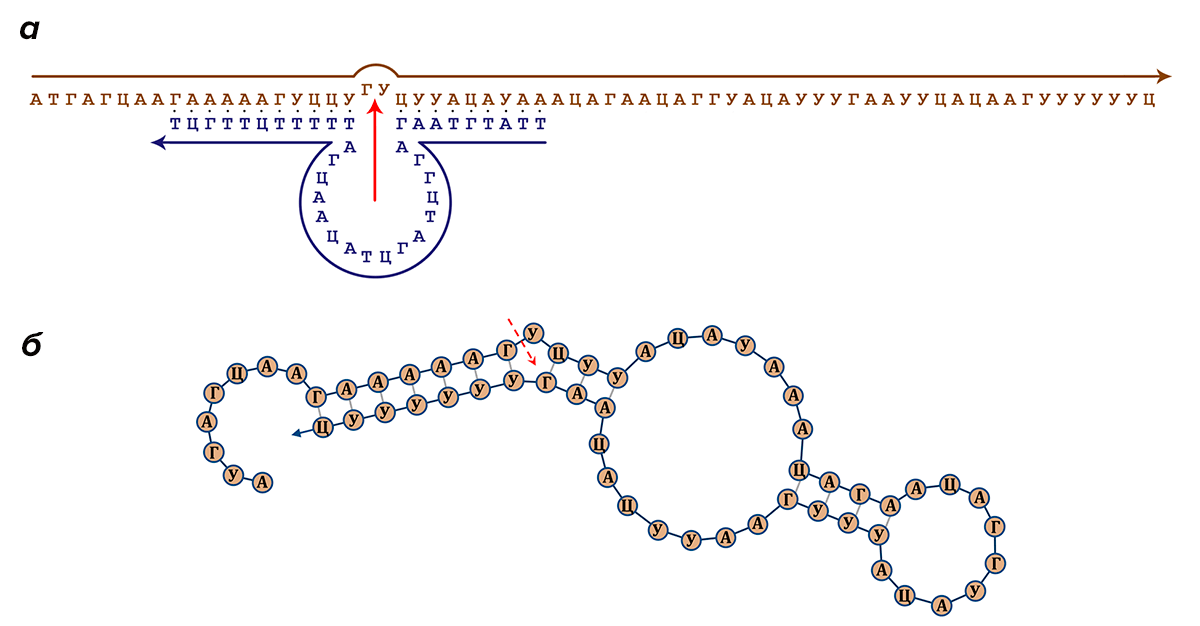
Главнейшую роль в исполнении команд, связанных с поиском и распознаванием соответствующих участков кода, играет известный принцип комплементарности. Однако и в двуцепочечной структуре ДНК, и, гораздо чаще, в структуре одноцепочечных и гибридизованных (соединившихся) РНК встречаются участки, на которых нуклеотиды не подходят друг другу для образования пары оснований. В таком случае образуются шпильки, петли, узлы и другие структуры (рис. 2). Иногда они не несут в себе никакого принципиального значения, но в других случаях их образование — причем в строго определенном месте и составленное из строго определенного набора нуклеотидов — является ключом для выполнения той или иной операции [6].
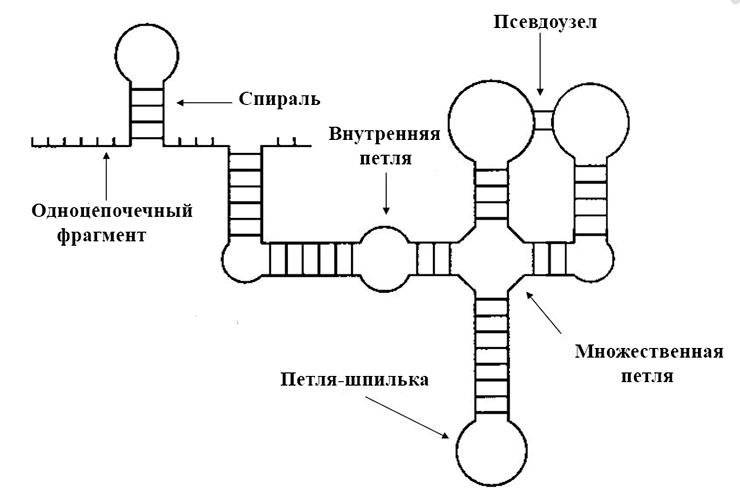
Рисунок 2. Схематичное изображение возможных образований во вторичной структуре одноцепочечных молекул нуклеиновых кислот
Операции, выполняемые подобными участками, могут быть самыми различными, и ниже мы поговорим о некоторых из них. Но перед тем укажем, что подобные структуры могут образовываться не только в пределах одной молекулы, но и при неполной комплементарности двух и более цепей РНК или ДНК [7]. Представим себе два коротких фрагмента ДНК: один вида 5′-ААГТАТЦ(X)ТАГЦЦГА и другой вида ТТЦАТАГАТЦГГЦТ-5′, где (X) — это какая-то недлинная последовательность нуклеотидов, которая как раз и будет выполнять некоторую операцию. Второй фрагмент ДНК комплементарен первому по краям от (X), и потому эти два фрагмента могут гибридизоваться друг с другом, образуя двуцепочечную систему, у которой посередине будет наблюдаться «выпячивание», благодаря неспаренному участку (X) первого фрагмента.
Таким образом, если (X) выполняет какую-то функцию — например, расщепляет второй фрагмент ДНК в строго определенном месте, — то весь фрагмент 5′-ААГТАТЦ(X)ТАГЦЦГА целиком выполняет уже две следующие друг за другом операции: распознавание строго определенной комплементарной ему последовательности и, если распознание выполнено успешно, ее расщепление. Итого, мы получаем фрагмент программного кода, независимый в исполнении своих функций от других элементов программы (от генетического кода организма или других фрагментов поблизости). Подобные структуры в среде программистов иногда называют макросами; воспользуемся этим термином и мы.
Рибозимы
Уже упоминаемые выше молекулы РНК, проявляющие ферментативные свойства и способные разрезать другие последовательности ДНК и РНК, являются именно такими «макросами». Рибозимы — это короткие последовательности РНК, условно состоящие из двух частей. Каталитический участок, последовательность которого строго предопределена, выполняет функцию расщепления другой молекулы ДНК или РНК в определенном сайте. Гибридизационный участок, последовательности которого могут быть различны, окружает каталитический с двух сторон, и отвечает за распознавание того участка другой молекулы ДНК или РНК, которую необходимо разрезать, и присоединение непосредственно к ней.
Схематично процесс работы рибозимного «макроса» представлен на рисунке 3.
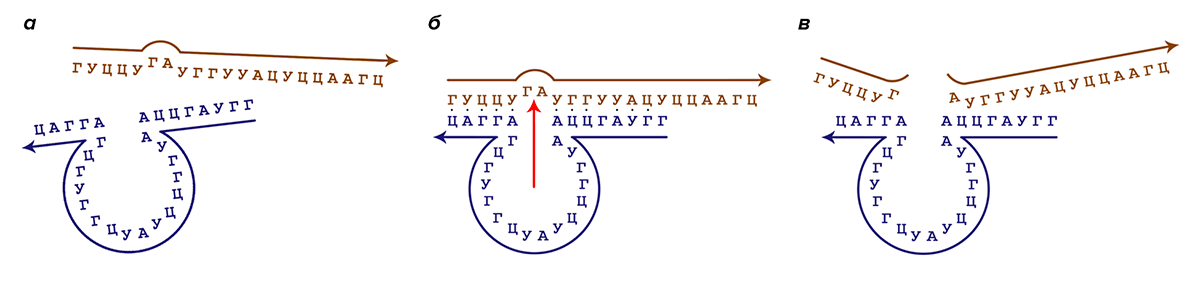
Рисунок 3. Принцип работы рибозимов. Синим цветом выделена последовательность рибозимного «макроса», коричневым цветом — целевая для него последовательность РНК, которую он призван расщеплять. Стрелками указано направление от 5′- к 3′-концу молекул. а — Молекулы «макроса» и целевой РНК встречаются в растворе, происходит операция логического сравнения. б — Если последовательности комплементарны и сравнение дало положительный ответ, молекулы гибридизуются, и выполняется операция расщепления РНК в строго определенном месте (указано красной стрелкой) рибозимной частью «макроса». в — Фрагменты расщепленной молекулы РНК отщепляются от рибозимного «макроса».
[6], рисунок адаптирован
Стоит отметить, что помимо рибозимов существуют и дезоксирибозимы — молекулы ДНК, также проявляющие каталитическую активность и при этом имеющие большую стабильность самих молекул [8], [9].
Аптамеры
По несколько иному принципу работают аптамерные «макросы». Нуклеиновые аптамеры — это короткие последовательности ДНК или РНК, которые способны распознавать присутствие других молекул (обычно сложных органических веществ) поблизости и, если рядом находится молекула определенного вещества, то захватывать ее, образуя кольцо или шпильку вокруг цели. Подобный захват может приводить к различным итогам [10]. В частности, некоторые органические вещества при аптамерном захвате немного изменяют свою пространственную структуру, что может приводить, например, к изменению их фотохимических свойств [11], [12].
Аптамерный участок цепи ДНК или РНК, продолженный участками, комплементарными к какой-либо целевой нуклеиновой кислоте, может служить способом обнаружения присутствия в системе этой кислоты. Таким образом, аптамерный «макрос» выполняет две последовательные функции: обнаружить целевую РНК или ДНК и затем подать сигнал в случае ее обнаружения.
Подобные аптамерные конструкции могут использоваться в качестве сенсоров [13], [14], когда захват молекулы-флуорофора «макросом» происходит только в присутствии целевой последовательности ДНК. Схематично процесс работы подобной системы представлен на рисунке 4.
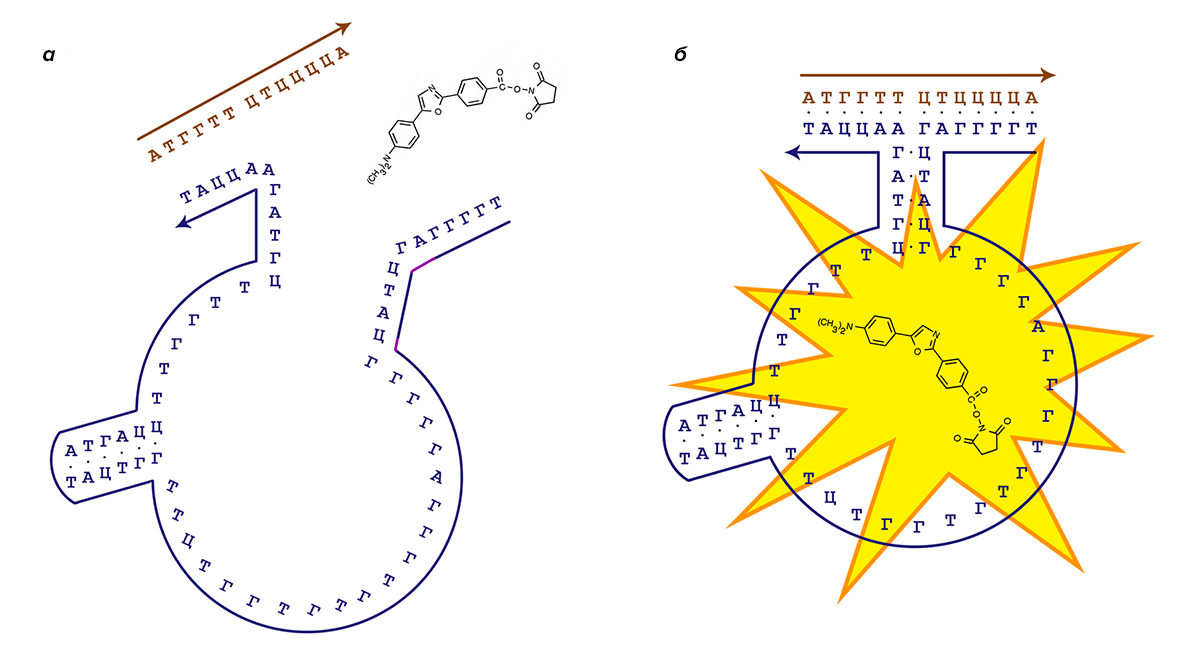
Рисунок 4. Принцип работы аптамерных конструкций. Фиолетовым цветом выделена последовательность аптамерного «макроса», коричневым цветом — целевая для него последовательность ДНК. а — В отсутствии целевой последовательности «макрос» не может соединиться в аптамерный замок из-за недостаточной стабильности такого замка. б — Целевая последовательность ДНК, будучи узнанной, стабилизирует замок, и захваченная «макросом» молекула (в данном случае это модифицированная молекула дапоксила) подает интенсивный флуоресцентный сигнал.
[14], рисунок адаптирован
Молекулярные маяки
Испускание флуоресцентного сигнала «макросом» может программироваться не только путем аптамерного захвата какой-нибудь флуоресцирующей молекулы. Альтернативным вариантом является вид ДНК-зондов [15], называемый молекулярными маяками — короткие самогибридизующиеся фрагменты ДНК, меченные двумя веществами: флуорофором (соединением, способным испускать свет при облучении) и гасителем флуоресценции. Сам по себе такой фрагмент имеет вторичную структуру, при которой флуорофор и гаситель находятся рядом, вследствие чего весь сигнал флуорофора поглощается гасителем, и наблюдателю никакого сигнала не заметно. Однако если по близости от маяка присутствует другая молекула ДНК или РНК, комплементарная последовательности маяка, то маяк раскрывается и гибридизуется с ней, что ведет к удалению на значительное расстояние гасителя флуоресценции от флуорофора, и в результате наблюдатель видит сигнал, более не подавляемый гасителем.
Иными словами, данный «макрос» так же, как и аптамерный, выполняет две функции: обнаружение целевого фрагмента ДНК или РНК и информирование наблюдателя в случае его обнаружения. Схематично принцип его работы представлен на рисунке 5.

Рисунок 5. Принцип работы ДНК-зондов типа молекулярных маяков. Синим цветом выделена последовательность маякового «макроса», на одном из концов которой находится флуорофор (желтый), а на другом — гаситель флуоресценции (серый); коричневым цветом — целевая для него последовательность РНК. а — В отсутствии целевой последовательности «макрос» находится в состоянии шпильки, и из-за близости гасителя к флуорофору сигнала не наблюдается. б — При узнавании целевой последовательности ДНК и связывании с ней, шпилька раскрывается, флуорофор отдаляется от гасителя, и «макрос» подает интенсивный флуоресцентный сигнал.
[15], рисунок адаптирован
Различные ДНК-зонды, в частности молекулярные маяки, уже много лет применяются в полимеразной цепной реакции в режиме реального времени (или количественной ПЦР), благодаря которой количество специфической последовательности ДНК в продукте можно отслеживать непосредственно в процессе самой реакции, не дожидаясь завершения последнего цикла и проведения какого-либо количественного анализа.
Хугстиновые носители
Нуклеотиды могут объединяться друг с другом не только по принципу комплементарности, названному так Джеймсом Уотсоном и Френсисом Криком, но и целым рядом других механизмов. Одним из таких механизмов является хугстиновское взаимодействие (по имени открывшего его Карста Хугстина), которое работает по схожему комплементарному взаимодействию, однако, выражаясь простым языком, нуклеотиды в составе нуклеиновых кислот при хугстиновском взаимодействии соединяются в направлении, значительно повернутом относительно уотсон-криковского (рис. 6).
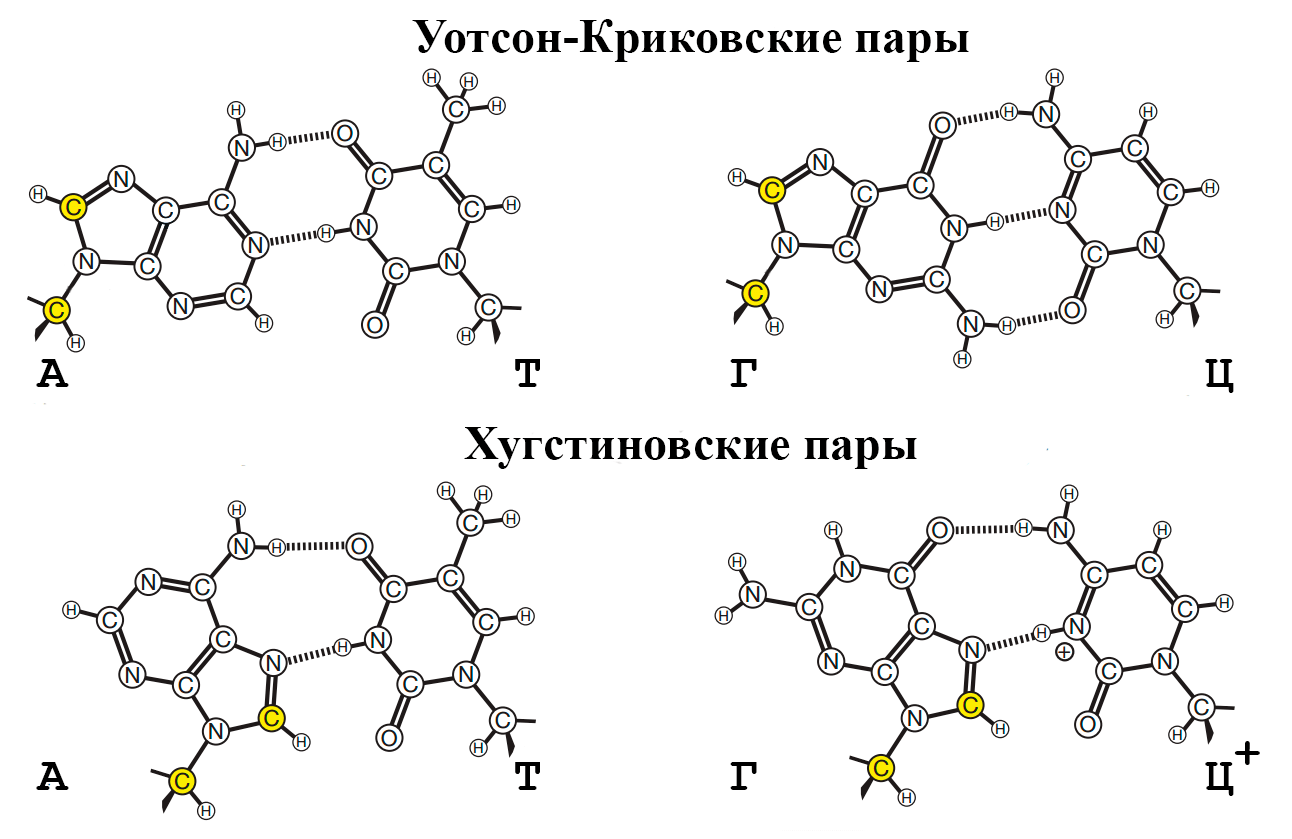
Рисунок 6. Разница в образовании хугстиновской и уотсон-криковской комплементарных нуклеотидных пар. Штриховыми линиями обозначены водородные связи между атомами.
Таким образом, гипотетически один нуклеотид может вступать в комплементарное взаимодействие сразу с двумя другими нуклеотидами. На основе этой возможности ученые синтезировали одноцепочечную молекулу ДНК, один фрагмент которой был комплементарен целевой молекуле нуклеиновой кислоты по принципу Уотсона и Крика, а второй — по принципу Хугстина. В результате этого в присутствии целевой ДНК или РНК создавался комплекс, внутри которого мишень была сохранена от внешних воздействий и в то же время сама не проявляла никакой активности. Иными словами, синтезированная последовательность ДНК выполняла оператор с условием (нахождение рядом мишени) и, в случае положительного ответа, выключала функцию другой молекулы.
Вторым пунктом, которому должен соответствовать подобный «макрос», чтобы называться не замком, а носителем, является функция высвобождения целевого фрагмента именно в том месте организма, где от него требуется. Это условие можно запрограммировать, например, добавив на концы молекулы-носителя небольшие органические фрагменты, воспринимаемые в качестве антигенов различными иммунными тельцами нашего организма [17]. В среде, где присутствуют антитела конкретного типа, они свяжутся с молекулой-носителем и таким образом «раскроют» ее цепь и высвободят содержимое. Принцип работы такого носителя представлен на рисунке 7.
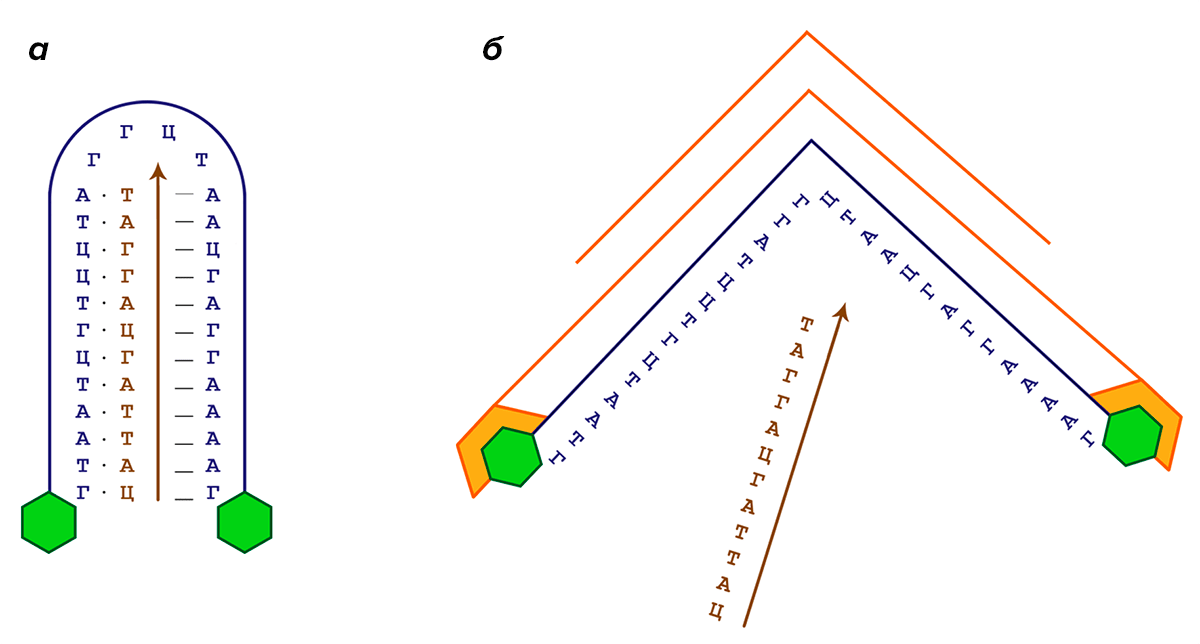
Рисунок 7. Принцип работы хугстиновского носителя на основе отношений антитело—антиген. Синим цветом выделена последовательность «макроса»-носителя, на обоих концах которого находятся молекулы антигена (зеленые); коричневым цветом — последовательность ДНК, переносимая «макросом». а — «Макрос» в связанном состоянии; точками отмечены уотсон-криковские пары, штрихами — хугстиновские. б — Антитело (оранжевое), специфическое к меткам на концах «макроса», связывается с носителем и раскрывает его, тем самым высвобождая содержимое.
[19], рисунок адаптирован
Эти фрагменты по обоим концам могут быть одинаковыми или различными, и второй случай соответствует логическому сравнению: высвобождение будет происходить только тогда, когда вокруг молекулы-носителя имеется и один вид антител, и другой. Возможность встраивания функции сравнения повышает точность подобной адресной доставки.
На самом деле, разнообразие функций, которые могут исполнять различные последовательности ДНК, куда более обширны. Например, есть исследования по созданию «макросов»-детекторов, зависящих от светового [18–20] и радиоактивного излучений или наличия определенных молекул вокруг [21], или «макросов»-носителей, зависящих от электрических импульсов [20], [22].
Логические элементы
Принцип комплементарности, по которому связываются между собой нуклеиновые кислоты, является инструментом для создания практически идеально селективных (не допускающих ложного срабатывания) условий объединения двух молекул. Однако по такому же принципу могут объединяться в одну систему не только две молекулы, но и гораздо большее их число, если различные фрагменты этих молекул комплементарны другим фрагментам других молекул. Примером подобных систем служит неоднократно уже освещавшееся во многих научно-популярных источниках ДНК-оригами [23], в том числе и на «Биомолекуле»: «ДНК-оригами: путь от гравюры до нанороботов длиной в 30 лет» [24] и «Голактеко опасносте: ДНК-роботы в живом организме» [25].
Это создает отличные условия для моделирования на языке ДНК логических операций, гораздо более сложных, чем унарное «ДА». Используя описанные выше молекулярные маяки или другие ДНК-зонды со схожим механизмом, можно создавать системы, способные проводить более сложное логическое сравнение [26], [27]. Одной из таковых является логическая операция «И» (рис. 8), дающая положительный ответ только в случае присутствия двух различных сигналов [28]. Это может помочь в проведении комплексных исследований с участием нескольких целевых фрагментов ДНК или РНК, что очень важно для клинической диагностики многих заболеваний.
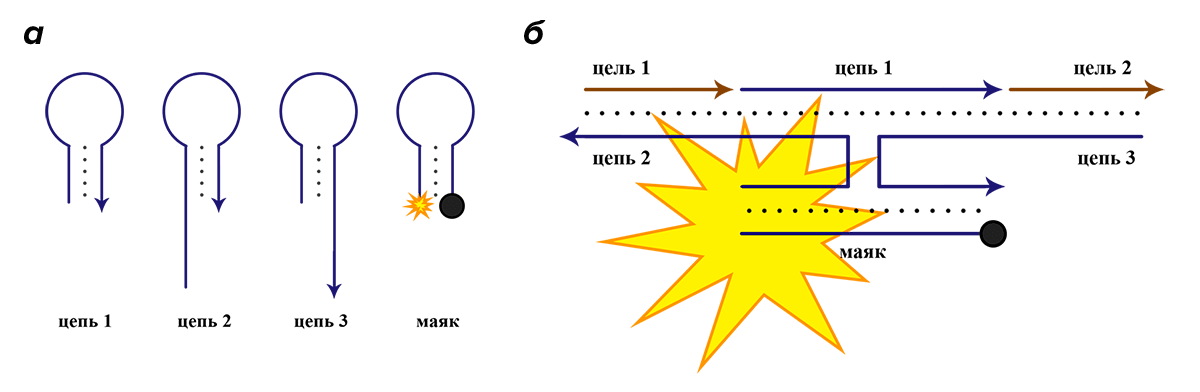
Рисунок 8. Идея логического элемента «И», написанного на языке ДНК. Синим цветом выделены последовательности, принадлежащие коду логического элемента. Коричневым цветом выделены целевые фрагменты ДНК или РНК, которые элемент призван обнаружить вместе. а — Логический элемент состоит из четырех коротких фрагментов ДНК, один из которых является молекулярным маяком. б — Только в присутствии обеих целевых последовательностей части логического элемента гибридизуются полностью и подается флуоресцентный сигнал. Для упрощения схемы нуклеотидный код каждой цепи не указан.
[28], рисунок адаптирован
Напротив, можно создать конструкции «ИЛИ», которые будут давать положительный ответ и в присутствии только одной из двух (или даже более) целевых молекул нуклеиновых кислот (рис. 9). Или же конструкции типа «Исключающее ИЛИ», которые дают сигнал в присутствии только одной из целевых последовательностей, и не дают сигнал, когда присутствуют обе, что очень полезно при исследовании объектов с частыми мутациями в геноме — например, туберкулеза [29].
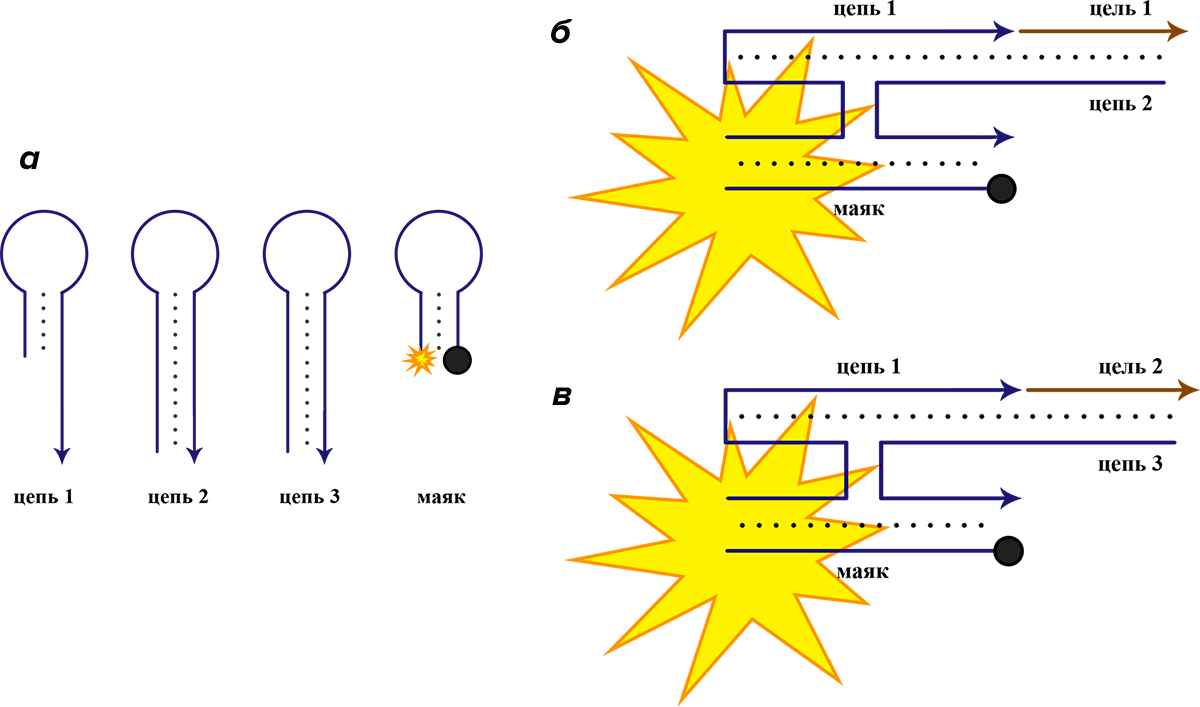
Рисунок 9. Идея логического элемента «ИЛИ», написанного на языке ДНК. Синим цветом выделены последовательности, принадлежащие коду логического элемента. Коричневым цветом выделены целевые фрагменты ДНК или РНК, которые элемент призван обнаруживать. а — Логический элемент состоит из четырех коротких фрагментов ДНК, один из которых является молекулярным маяком. б и в — В присутствии любой из целевых последовательностей три из четырех частей логического элемента, включая маяк, гибридизуются между собой, и подается флуоресцентный сигнал. Для упрощения схемы нуклеотидный код каждой цепи не указан.
[29], рисунок адаптирован
Простейшие «ДА»-элементы, положенные в основу теста на туберкулез и его тип (Xpert MTB/RIF), уже активно используются в ряде стран и пропагандируются Всемирной организацией здравоохранения. В это же время модернизированные логические конструкции, такие как элементы «ИЛИ», «НЕ» или «ИЛИ-НЕ» (он же стрелка Пирса) на основе ДНК позволяют значительно облегчить подобные тесты [30]. Пример такого применения представлен на рисунке 10.
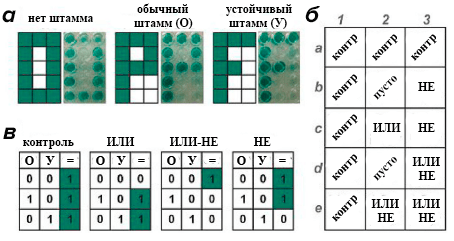
Рисунок 10. Идея экспресс-теста на определение рифампицинустойчивого штамма палочки Коха с помощью логических элементов на основе ДНК. В каждую лунку планшета вносится определенный логический элемент ДНК, а затем в каждую — одна и та же проба. а — Слева: теоретически ожидавшийся результат; справа: окраска лунок в зависимости от того, какая проба была добавлена на планшет. б — Схема нанесения логических элементов на планшет. в — Принцип окрашивания каждого логического элемента в зависимости от того, какая проба вносится в ту же лунку, где он находится.
[30], рисунок адаптирован
Программируемые роботы
Итого, мы имеем язык программирования, записанный в реальности и имеющий возможность взаимодействовать с реальными объектами. В этом языке нам уже известна запись достаточно большого числа команд и операций и даже независимых групп команд («макросов»), которые выполняют широкий спектр функций. Более того, большинство из перечисленных выше «макросов» являются самоисполняемыми: ни рибозимы, ни аптамерные или маяковые сенсоры не нуждаются в стороннем ферментативном исполнении заложенных в них команд, как и логические элементы — достаточно простого удовлетворения команды-запуска «макроса» (в большинстве случаев это просто встреча с целевой последовательностью ДНК или РНК).
Однако проектирование и применение подобных макросов для повседневных задач, будь то детектирование бактериальных штаммов или же лечение каких-либо заболеваний, имеет ряд недостатков. Рассмотрим таковые, задавшись целью использовать дезоксирибозимный «макрос» для расщепления РНК вируса гриппа A, попавшего в клетку. Расщепление ее в определенном месте приведет к неспособности вируса воспроизводить копии себя и, как следствие, распространение вируса по организму прекратится, что будет означать, что, когда вся вирусная РНК окажется расщепленной, человек успешно выздоровеет.
Возьмем короткий шестидесятинуклеотидный фрагмент вирусной РНК, в котором содержится участок, который мы хотим расщепить дезоксирибозимом (воспользуемся базой данных GenBank). Спроектируем ту часть «макроса», что отвечает за распознавание, таким образом, чтобы она была полностью комплементарна нуклеотидам, что окружают целевой участок (рис. 11а). Основу дезоксирибозима выберем из известного исследования [31].
Однако программный код (а это фактически он), представленный на рисунке 11а, является лишь проекцией, виртуальной копией реального кода. Иными словами, необходимо чтобы «макрос» подходил к целевому фрагменту РНК не только с точки зрения логического сравнения, но и с точки зрения физических законов мира: как минимум, термодинамических и стереохимических, а также чтобы окружающая среда (ионный состав, ее кислотность) была оптимальна для работы «макроса».
На рис. 11б представлена плоская проекция вторичной структуры выбранного шестидесятинуклеотидного фрагмента. Как видно, доступ к целевому участку этого фрагмента в естественном состоянии гораздо более затруднен, чем это было изображено в строчке программного кода. Учитывая то, что каждая буква в подобном коде — это даже не массивный атом, а целая группа из двух десятков атомов, трудность подсоединения дезоксирибозимного макроса к подобной конструкции становится понятной даже с точки зрения бытовой логики. Как следствие, результат работы дезоксирибозима уже будет очень далек от стопроцентного.
Наконец, представим проекцию вторичной структуры части РНК вируса гриппа A, ответственную за кодирование одной из субъединиц вирусной полимеразы (так называемой субъединицы PB-1). В ней содержится такое огромное число нуклеотидов (более 2000), что ее вид, даже представленный в виде проекции, здесь бы просто не уместился. Очевидно, что вероятность присоединения дезоксирибозимного «макроса» к данному участку в реальных условиях крайне мала.
Иными словами, просто функции распознавания целевого фрагмента и ее расщепления уже недостаточно для эффективной работы системы и выполнения поставленных перед ней задач. Необходимо включать в ее программный код и другие функции. Первой из таких напрашивается расплетение вторичной структуры РНК вблизи целевого участка для лучшего распознавания и связывания. Второй — стабилизация комплекса РНК—дезоксирибозим. Для повышения эффективности также можно добавить в систему еще несколько дезоксирибозимов, нацеленных на другие, расположенные поблизости участки.
Итоговый комплекс молекул, выполняющих различные функции и соединенных между собой по принципу комплементарности, уже можно будет называть машиной, которая запрограммирована на выполнение множества операций в зависимости от окружающих условий без контроля со стороны оператора. Это полностью отвечает понятию «робот», и потому подобные сложные конструкции и называются ДНК-нанороботами, и подобный термин уже появлялся на сайте [25].
Основы для программных кодов таких ДНК-нанороботов, направленных на различные задачи, приведены на рисунках 12 и 13.
Заключение
Подводя итог всему вышесказанному, хотелось бы отметить, что мир молекул РНК и ДНК, в отрыве от привычной для нас функции «передачи информации», таит в себе множество интересных возможностей и неожиданных проявлений. Более того, многогранность этих возможностей позволяет рассматривать их с совершенно различных сторон — в частности, как и было представлено в этой статье, работа механизмов на основе нуклеиновых кислот может быть представлена даже в виде функционирования реально существующего языка программирования.
Наличие подобных ракурсов, не лишенных изрядной доли смысла, возможно, станет интересным для людей самых различных профессий и специальностей, что станет причиной вовлеченности в изучение основы нашего существования все большего и большего числа людей.
В самом начале данной статьи уже упоминалось, что подобный взгляд, да и сам термин «ДНК-программирование» являются неотъемлемыми составляющими одной большой прикладной науки, носящей название синтетической биологии. Изучить живое, «взломать» его программный код, чтобы понять, как это живое работает, и на основе полученных знаний создать нечто новое — задача целого ряда дисциплин и уровней, начиная от организменного и заканчивая молекулярным и даже атомным. Наука не стоит на месте и, возможно, уже в самом ближайшем времени мы сможем «перепрограммировать» самих себя.
Литература
- Синтетическая биология: от программирования компьютеров к программированию клеток;
- Синтетическая биология: от наблюдения к вмешательству;
- Обо всех РНК на свете, больших и малых;
- Просто о сложном: CRISPR/Cas;
- CRISPR-системы: иммунизация прокариот;
- J. Tang, R. R. Breaker. (2000). Structural diversity of self-cleaving ribozymes. Proceedings of the National Academy of Sciences. 97, 5784-5789;
- Ronald R. Breaker. (2004). Natural and engineered nucleic acids as tools to explore biology. Nature. 432, 838-845;
- Hongzhou Gu, Kazuhiro Furukawa, Zasha Weinberg, Daniel F. Berenson, Ronald R. Breaker. (2013). Small, Highly Active DNAs That Hydrolyze DNA. J. Am. Chem. Soc.. 135, 9121-9129;
- Jiahao Huang, Jueqi Wu, Zhigang Li. (2016). Molecular beacon-based enzyme-free strategy for amplified DNA detection. Biosensors and Bioelectronics. 79, 758-762;
- Eun Jeong Cho, Joo-Woon Lee, Andrew D. Ellington. (2009). Applications of Aptamers as Sensors. Annual Rev. Anal. Chem.. 2, 241-264;
- Jeremy R. Babendure, Stephen R. Adams, Roger Y. Tsien. (2003). Aptamers Switch on Fluorescence of Triphenylmethane Dyes. J. Am. Chem. Soc.. 125, 14716-14717;
- Dmitry M. Kolpashchikov. (2005). Binary Malachite Green Aptamer for Fluorescent Detection of Nucleic Acids. J. Am. Chem. Soc.. 127, 12442-12443;
- Alexander Kuznetsov, Natalia Komarova, Maria Andrianova, Vitaliy Grudtsov, Evgeniy Kuznetsov. (2018). Aptamer based vanillin sensor using an ion-sensitive field-effect transistor. Microchim Acta. 185;
- Teru Kato, Ippei Shimada, Ryota Kimura, Masumi Hyuga. (2016). Light-up fluorophore–DNA aptamer pair for label-free turn-on aptamer sensors. Chem. Commun.. 52, 4041-4044;
- James G. Wetmur. (1991). DNA Probes: Applications of the Principles of Nucleic Acid Hybridization. Critical Reviews in Biochemistry and Molecular Biology. 26, 227-259;
- Рулетка для спектроскописта;
- Simona Ranallo, Carl Prévost-Tremblay, Andrea Idili, Alexis Vallée-Bélisle, Francesco Ricci. (2017). Antibody-powered nucleic acid release using a DNA-based nanomachine. Nat Comms. 8, 15150;
- Yukiko Kamiya, Hiroyuki Asanuma. (2014). Light-Driven DNA Nanomachine with a Photoresponsive Molecular Engine. Acc. Chem. Res.. 47, 1663-1672;
- D. Grate, C. Wilson. (1999). Laser-mediated, site-specific inactivation of RNA transcripts. Proceedings of the National Academy of Sciences. 96, 6131-6136;
- Johanna Moratz, Avik Samanta, Jens Voskuhl, Siva Krishna Mohan Nalluri, Bart Jan Ravoo. (2015). Light-Triggered Capture and Release of DNA and Proteins by Host-Guest Binding and Electrostatic Interaction. Chem. Eur. J.. 21, 3271-3277;
- Yanhua Li, Yuanyuan Chen, Wei Pan, Zhengze Yu, Limin Yang, et. al.. (2017). Nanocarriers with multi-locked DNA valves targeting intracellular tumor-related mRNAs for controlled drug release. Nanoscale. 9, 17318-17324;
- Simona Ranallo, Alessia Amodio, Andrea Idili, Alessandro Porchetta, Francesco Ricci. (2016). Electronic control of DNA-based nanoswitches and nanodevices. Chem. Sci.. 7, 66-71;
- Nadrian C. Seeman. (2010). Nanomaterials Based on DNA. Annu. Rev. Biochem.. 79, 65-87;
- ДНК-оригами: путь от гравюры до нанороботов длиной в 30 лет;
- Голактеко опасносте: ДНК-роботы в живом организме;
- Alex Lake, Stephen Shang, Dmitry M. Kolpashchikov. (2010). Molecular Logic Gates Connected through DNA Four-Way Junctions. Angewandte Chemie International Edition. 49, 4459-4462;
- Melissa Massey, Igor L. Medintz, Mario G. Ancona, W. Russ Algar. (2017). Time-Gated FRET and DNA-Based Photonic Molecular Logic Gates: AND, OR, NAND, and NOR. ACS Sens.. 2, 1205-1214;
- Yulia V. Gerasimova, Dmitry M. Kolpashchikov. (2012). Connectable DNA Logic Gates: OR and XOR Logics. Chem. Asian J.. 7, 534-540;
- Evan M. Cornett, Eleanor A. Campbell, George Gulenay, Evan Peterson, Neha Bhaskar, Dmitry M. Kolpashchikov. (2012). Molecular Logic Gates for DNA Analysis: Detection of Rifampin Resistance in M. tuberculosis DNA. Angew. Chem. Int. Ed.. 51, 9075-9077;
- Ryan P. Connelly, Evgeny S. Morozkin, Yulia V. Gerasimova. (2018). Alphanumerical Visual Display Made of DNA Logic Gates for Drug Susceptibility Testing of Pathogens. ChemBioChem. 19, 203-206;
- Stephen W. Santoro, Gerald F. Joyce. (1998). Mechanism and Utility of an RNA-Cleaving DNA Enzyme†. Biochemistry. 37, 13330-13342;
- Биоинформатика в мире РНК-структур;
- Dmitry M. Kolpashchikov. (2010). Binary Probes for Nucleic Acid Analysis. Chem. Rev.. 110, 4709-4723;
- Dmitry M. Kolpashchikov. (2007). A Binary Deoxyribozyme for Nucleic Acid Analysis. ChemBioChem. 8, 2039-2042;
- A. J. Cox, H. N. Bengtson, K. H. Rohde, D. M. Kolpashchikov. (2016). DNA nanotechnology for nucleic acid analysis: multifunctional molecular DNA machine for RNA detection. Chem. Commun.. 52, 14318-14321.