
Летняя школа по биоинформатике — как это было
18 мая 2018
Летняя школа по биоинформатике — как это было
- 1022
- 0
- 2
-
Автор
-
Редакторы
Как известно, то, что случилось один раз, — случайность, два раза — совпадение, а три раза — уже закономерность. Так, в минувшем 2017 году Институт биоинформатики уже в пятый раз провел летнюю школу по биоинформатике. В этой статье мы хотим поделиться впечатлениями преподавателей и студентов, которые стали непосредственными участниками школы.
Институт биоинформатики начал свое существование в 2010 году в виде курсов на базе Академического университета РАН (Санкт-Петербург). С 2013 года Институт действует как самостоятельная организация. Основная миссия — подготовить молодое поколение высококвалифицированных специалистов в области биоинформатики. Сейчас Институт организует ряд образовательных программ для слушателей из разных областей биологии и IT — от летней школы для начинающих до семинара по системной биологии для тех, кто в теме. Для всех, кто хочет оценить свои силы, ежегодно проводит олимпиаду и хакатон по биоинформатике, а для желающих освоить биоинформатику более полно предусмотрены годовые программы: «Биоинформатика для биологов» и «Алгоритмическая биоинформатика». Если вы задумываетесь о получении высшего образования, а именно магистерской степени, то советуем рассмотреть две программы: «Биоинформатика и системная биология» и «Алгоритмы и анализ данных в биоинформатике». Во всех подробностях об Институте биоинформатики и нескольких его учебных программах на «Биомолекуле» уже писала Марина Слащева: «Come to the bioinformatics side: Институт биоинформатики в Санкт-Петербурге» [1].
А сегодня мы хотим рассказать вам подробнее про летнюю школу. Если вы только приглядываетесь к биоинформатике, летняя школа сможет в короткие сроки показать вам (почти ) всё её разнообразие, включая геномику, транскриптомику, протеомику и многое другое. Однако если биоинформатика — ваша стихия, то мы бы советовали вам обратить внимание на другие программы Института.
Редакция «Биомолекулы» хочет отметить, что, кроме биоинформатики, к «сухой» биологии относятся также молекулярное моделирование, системная биология и т.д. Подробнее об этой классификации можно почитать в нашей статье «12 методов в картинках: „сухая“ биология» [2]. — Ред.
Насыщенная программа, целеустремленные коллеги, замечательные люди.
Летняя школа. Начало
В 2017 году на территории МФТИ в городе Долгопрудный прошла летняя школа, которая собрала 48 информатиков и 51 биолога. Стоит отметить, что деление на биологов и информатиков довольно условно: так, к первым, помимо собственно биологов, относятся медики, химики и биотехнологи, а ко вторым — физики, математики и программисты. Главное, что несомненно объединяет всех студентов школы, — это высокий уровень мотивации и желание погрузиться в биоинформатику.
Несмотря на то, что я тоже была участником, я не попала ни в категорию студентов, ни, тем более, преподавателей. Как так получилось? Сначала я подала заявку на участие в школе и начала проходить курс по программированию на Stepik’е. Однако не успела к объявленному дедлайну набрать необходимых баллов, и мою заявку оргкомитет не рассмотрел. Немного отчаявшись, я решила принести пользу организаторам, с которыми успела познакомиться еще на семинаре по системной биологии, и помочь с трансфером участников к месту проведения школы. Организаторы в свою очередь поверили в мои силы и пригласили на школу. Так началась моя деятельность в качестве волонтера.

Рисунок 1. Это я (в центре, в белой футболке) в окружении организаторов и лекторов летней школы.
В первый день школы участники прослушали вступительную лекцию по биоинформатике от Александра Предеуса (Институт биоинформатики), вспомнили основы статистики и языка программирования R с Германом Демидовым (Centre for Genomic Regulation) и основы молекулярной биологии и генетики с Александром Предеусом. Прекрасным завершением первого дня стал вечер знакомств. Со следующего дня лекции начинались строго по расписанию — в 9 утра. Для сильных духом и тех, кто находил силы вставать еще раньше, в 7:45 Наталья Машьянова (Институт биоинформатики) проводила зарядку на свежем воздухе, после которой ребят ожидал вкусный, полезный завтрак.
Предеус, кстати, рассказывал как-то на «Биомолекуле» о Выездном семинаре по системной биологии: «Как перестать бояться и полюбить системную биологию» [3]. — Ред.
Только второй день на ЛШ биоинформатики закончился, а кажется, что я тут неделю. 8 лекций, 4 практики, вечер стартапов, вечер настолок, куча знакомств с интересными людьми со всей России...
Темой школы 2017 года стал интеллектуальный анализ данных в биоинформатике. Участники прослушали лекции по введению в машинное обучение и deep learning от Григория Сапунова (Intento), узнали больше про глубокое обучение в вычислительной биологии от Дмитрия Фишмана (University of Tartu) и изучили, как методы машинного обучения применяют для поиска потенциально патогенных мутаций в геноме человека, с Анной Ершовой (МФТИ). Полученные знания студенты применили на практических занятиях по машинному обучению вместе с Дмитрием Фишманом и Константином Зайцевым (Институт биоинформатики, ИТМО). Для всех заинтересовавшихся, записи лекций доступны на официальном канале Института биоинформатики на Youtube.

Рисунок 2. Участники летней школы погружены в глубокое обучение.
На третий день школы произошло сразу несколько интересных событий, среди которых конференция для участников летней школы и начало самостоятельной работы над проектами. На конференции выступили 11 участников с докладами на темы своих научных работ. Тезисы и презентации докладов опубликованы на сайте.
Работа над проектами
После окончания основного блока лекций и конференции, началась одна из самых ожидаемых частей школы — работа над научными проектами, или хакатон. Организаторы заранее подготовили пять проектов, непосредственно связанных с главной темой школы — интеллектуальным анализом данных. Над каждым проектом независимо работали по четыре команды. В течение двух дней с применением смекалки, ловкости рук и знаний, полученных на практических занятиях, участники анализировали single-cell RNA-Seq, определяли пол и тип ткани по данным экспрессии генов и предсказывали по данным секвенирования патогенность бактерий. О нескольких проектах мы бы хотели рассказать подробнее.
Для хакатона руководители старались подбирать проекты, связанные с машинным обучением и основанные на недавних статьях в научных журналах. Важным аспектом было дать студентам интересный проект, который при этом можно успеть выполнить за короткий промежуток времени. Один из кураторов, Константин Зайцев, выбрал статью [4], которая легла в основу его проекта по идентификации патогенности бактерий.
Бактерии бывают разные: вызывающие заболевания человека мы называем патогенными, а бактерии, которые в норме заболеваний не вызывают, мы называем непатогенными. К первым можно отнести стрептококки, вызывающие, например, менингит, а ко вторым — бифидобактерии. В рамках проекта командам было необходимо реализовать алгоритм машинного обучения, который бы принимал на вход данные секвенирования (NGS) [5] бактерии — короткие последовательности генома — и предсказывал, является ли бактерия патогенной. Работа над проектом началась с общего собрания, на котором Константин представил проект и постарался объяснить, что примерно ожидает от команд. После вступительной части, команды приступили к решению поставленной задачи.
В проекте было множество деталей, каждая из которых могла стать трудностью — от выбора алгоритма для машинного обучения до составления обучающего датасета. Необходимо было выгрузить геномные данные и данные о патогенности по всем бактериям из специальной базы данных. В течение двух дней работы руководитель проекта активно поддерживал участников: отслеживал прогресс, давал ценные советы командам, а также создал чат в одном из популярных мессенджеров, где участники делились друг с другом полезной информацией.
У каждого члена команды была своя специализация и подзадачи для достижения общей цели. Например, Юрий Барбитов отвечал за организацию труда и работу с базой данных, а именно выкачивание последовательностей из базы данных NCBI. Его команда за два дня справилась со всеми поставленными задачами и в итоге заняла первое место по проекту.

Рисунок 3. Команда победителей проекта по идентификации патогенности бактерий вместе с руководителем проекта — Константином Зайцевым (справа).
Другой проект, руководил которым Алексей Сергушичев (Университет ИТМО), был связан с предсказанием метаданных эксперимента по экспрессии генов. По словам Алексея, идея проекта появилась непосредственно из его основной работы — анализа данных экспрессии генов. Незадолго до начала летней школы вышел препринт в рамках проекта recount2, где авторы показали, что можно хорошо предсказывать некоторые метаданные по данным РНК-секвенирования [6]. В рамках проекта командам требовалось по данным экспрессии генов, полученным с помощью микрочипов, научиться делать три типа предсказаний: пол, ткань и мышиную линию (поскольку использовали данные мышей). Из более мелких задач: предстояло найти больше наборов данных для обучения (несколько штук предоставили на старте), попробовать разные алгоритмы предсказания и, используя имеющиеся биологические знания, интерпретировать полученные предсказательные модели или дополнить их.
Работу Алексей, как и Константин, начал с общего «брифинга» со всеми командами, после которого проводил индивидуальные консультации с участниками команд отдельно. Несмотря на то, что команды получали схожие советы по ходу работы, далеко не все одинаково им следовали. Одним из важных моментов проекта было то, что в датасетах присутствовали разные типы микрочипов. По совету Алексея, стоило сначала рассмотреть датасеты по отдельности, не объединяя их. И наилучшим образом справилась с работой именно та команда, которая прислушалась к этому совету.
Один из участников проекта Алексея — Антон Каразеев — рассказал, как была организована работа его команды: «Роли были в самом начале распределены, и каждый делал свою часть проекта. Я отвечал как раз за саму классификацию данных микрочипов — ментор дал нам наборы данных для обучения и тестирования нашей модели. Они включали в себя разные виды тканей: легочная, ткань печени и какая-то мышечная. Ещё был второй набор данных экспрессии генов — по нему надо было определить пол мышки. Получилось плюс-минус хорошо классифицировать пол мышек, а вот с тканями результат не очень хороший вышел (но лучше, чем просто подкидывать монетку и принимать решение)». Главным лимитирующим фактором Антон отметил недостаток времени: «Всё же за такой срок что-то годное получить трудно, но зато очень много нового удалось узнать благодаря нашим биологам в команде — они нам помогали разбираться с тем, что мы вообще делаем».

Рисунок 4. Алексей Сергушичев на практических занятиях.
В проекте Дмитрия Фишмана команды изучали возможности фреймворка DragoNN и применяли его на геномных данных. Идея проекта появилась благодаря коллеге Дмитрия, который занимается анализом геномных данных. DragoNN — одна из самых простых нейронных сетей, находящихся в открытом доступе, для работы с такими данными. В процессе работы участники тренировали нейронную сеть достоверно предсказывать экспрессию генов исходя из последовательности ДНК. Для представления результатов нужно было оценить эффективность работы сети на тестовых данных. Однако задача оказалась не так проста, и участники в процессе работы столкнулись самыми разными проблемами, включая сложности понимания концепции работы нейронных сетей, принципов регрессионного анализа, использования чужого кода в новом контексте, организации работы команды и многими другими.
Дмитрий старался найти свой подход к каждой команде, но в конце концов все команды слились в единое целое, так как распределение информатиков по командам было неравномерным, а те ребята, которые уже справились со своей задачей, стремились помочь другим, у кого не так хорошо получалось. Для итогового представления результатов участники все-таки разделились на две команды и представили два подхода для решения поставленной задачи.

Рисунок 5. Дмитрий Фишман (по центру, в тёмной футболке) в окружении участников школы.
В целом, руководители отметили, что для них самым сложным оказалась даже не работа с несколькими командами одновременно, а способность достичь баланса, при котором все команды остаются в равных условиях при работе над проектом. Удержать в голове прогресс и план работ каждой из 4–5 команд не так сложно, как отследить возможный сдвиг вектора работы одной из команд в неправильном направлении.
Что касается самих команд, достаточно нетривиальной, но очень жизненной оказалась задача взаимопонимания в коллективе, состоящем из программистов, биологов и биоинформатиков. Так, Ирина Булушева из команды по поиску сайта начала транскрипции (руководитель — Татьяна Татаринова, University of La Verne) поделилась секретом создания, по ее мнению, наиболее гармоничной команды, когда есть несколько биологов, программистов, и человек на стыке двух наук. Таким человеком в своей команде стала сама Ирина: при работе над проектом она обсуждала задачу с биологами и переформировывала ее на алгоритмический язык для программистов.
Главный совет, который хотели бы дать кураторы участникам проектов — это не бояться нового и незнакомого. На практике оно оказывается не таким пугающим, как казалось сначала. Также не стоит бояться задавать вопросы кураторам, лекторам и другим участникам летней школы. Оставайтесь открытыми новым идеям, подходам, проблемам и концепциям, но при этом не забывайте все подвергать сомнению. Участники летней школы 2017 года советуют сохранять хорошее настроение, активно заниматься научным нетворкингом и посещать лекции и практические занятия.
Ознакомиться со всеми проектами, посмотреть результаты работы команд и узнать, кто в итоге победил, можно тут.
Победой нашей команды в хакатоне закончилась успевшая стать почти родной школа. :)
Шесть дней лекций и практик с девяти до девяти, погружение в огромный, удивительный и совершенно неизвестный мне до этого мир биоинформатики, знакомство с интереснейшими людьми из параллельной вселенной, которые умеют собирать молекулы и редактировать геномы, много приятных призов и еще больше машинного обучения.
Было очень круто. Большое спасибо организаторам!
Немного о лекциях
В общей сложности, за шесть дней школы участники познакомились с такими разделами биоинформатики, как геномика, транскриптомика, эпигенетика, протеомика, а также узнали больше об интеграции всех полученных данных с помощью мультиомики (Константин Оконечников, German Cancer Research Center). Многое было сказано и про практическое применение существующих технологий. Геном человека содержит невероятное количество информации о нас, нашей истории и о том, что ожидает нас в будущем. О нашем прошлом — геногеографии — и немного о будущем — предсказании фенотипов по генотипу — рассказала Татьяна Татаринова (University of La Verne). Михаил Пятницкий (НИИ биомедицинской химии им. В.Н. Ореховича) рассказал участникам школы об анализе омиксных данных в онкологии, персонализированной медицине и «ошибке выжившего», с которой может столкнуться каждый биоинформатик. Вадим Назаров (НИУ ВШЭ, ИБХ РАН) познакомил студентов с иммуноинформатикой и анализом данных в иммунологии. Секретами продуктивной работы и моделирования белков поделился в своей лекции Артур Залевский (МГУ им. М. В. Ломоносова). И это только часть лекций, прочитанных на школе. Но организаторы обещали написать более подробно про каждую лекцию, так что следите за обновлениями!
О том, что такое эти «-омики», можно прочесть в статье «„Омики“ — эпоха большой биологии» [7]. — Ред.
Артур как-то написал для нас хорошую статью по применению «облачных» вычислений в биологии: «Наука витает в облаках» [8]. — Ред.

Рисунок 6. Лекция Василия Раменского о предсказании эффекта мутаций.
Вот и кончилась пятая Летняя школа по биоинформатике. Было круто.
И хотя выделенного времени (особенно на практики и проект) было маловато, я очень рада, что поехала. Ведь по большому счету, суть таких мероприятий вовсе не в том, чтобы стать крутым биоинформатиком за неделю, это как бы слегка невозможно. Суть летней школы — показать, что вот такие клевые штуки бывают, и вот такие клевые штуки бывают, а еще вот здесь целая куча клевых штук. А еще посмотреть на знакомые клевые штуки так, как никогда в жизни на них не смотрел.
Послесловие
Летняя школа прошла при поддержке JetBrains — ведущего мирового производителя профессиональных средств разработки, EPAM — глобального разработчика программного обеспечения и поставщика цифровых платформ, а также BIOCAD — международной инновационной биотехнологической компании полного цикла.
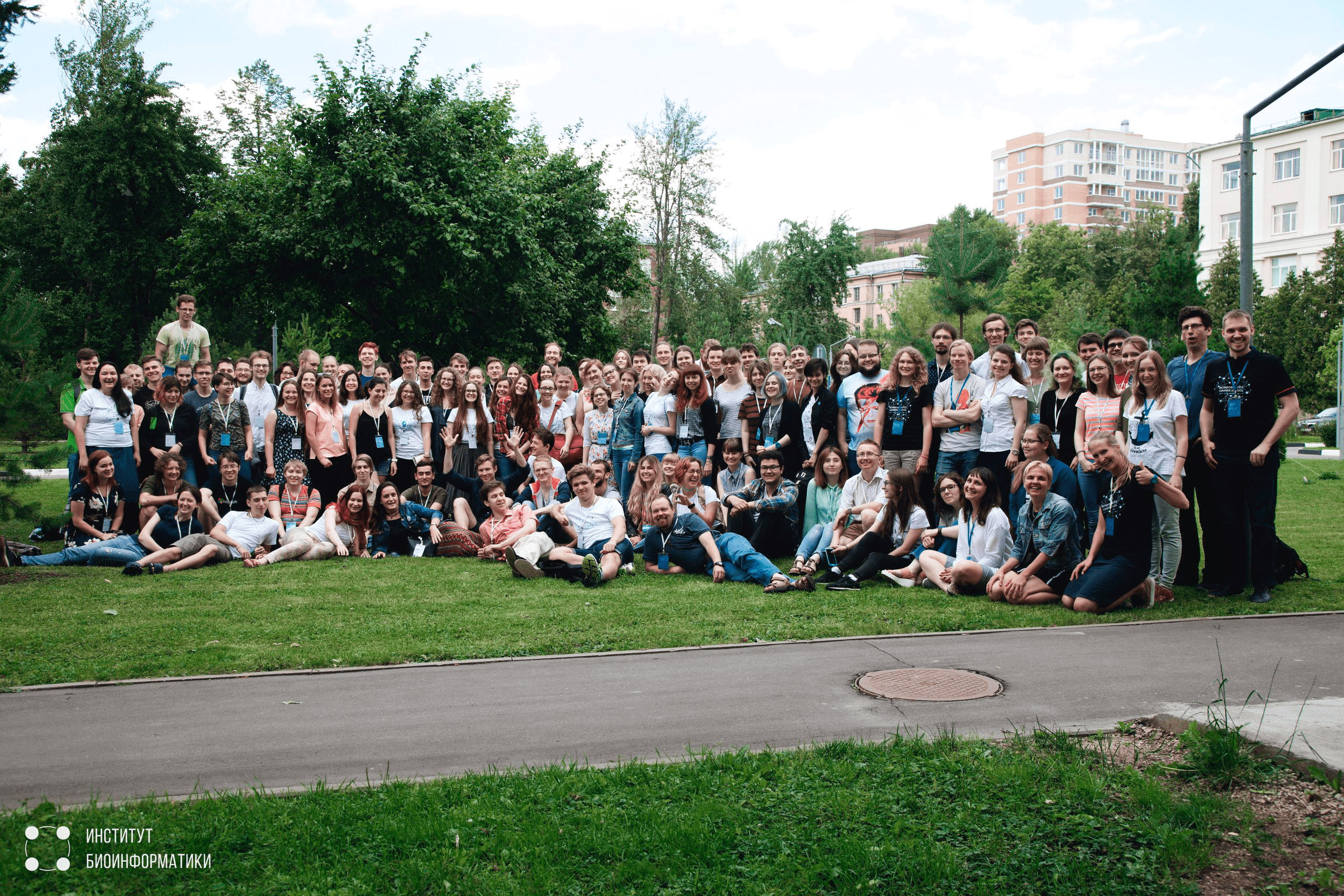
Рисунок 7. Все (или почти все) участники летней школы.
В этом (2018) году Институт биоинформатики снова приглашает всех желающих прикоснуться к биоинформатике и попробовать свои силы в обработке биологических данных. Я с удовольствием отвечу на все ваши вопросы как в комментариях к этой статье, так и на самой школе, поскольку в этом году меня снова выбрали в качестве волонтера. :)
Прием заявок на школу еще открыт, успейте заполнить форму и пройти курсы на Stepik’е! Основной темой этого года станет биоинформатика в исследованиях рака, а сама школа пройдет с 23 по 28 июля в пригороде Санкт-Петербурга. Оставайтесь с нами — будет весело!
Вторая благодарочка, как называет ее Алина, отправляется в сердце института биоинформатики. Ребята-организаторы, спасибо вам за то, что вы делаете. Вы пробудили еще больший интерес к биоинформатике, показали на своем примере, как это круто.
Лекторы, спасибо вам, что пытались объяснить сложное на простом и понятном для биолога языке. После ваших лекций биоинформатический мир стал яснее. :) Лекции, интенсивы, песни, пляски и хакатон — чего только не было за эту неделю. И это правда незабываемо.
Большое спасибо моей хакатоновской команде. Наша команда победила только благодаря тому, что каждый из нас старался как можно больше привнести в решение поставленной задачи, наша степень коммуникации меня поразила!
Большое спасибо всем и желаю каждому хороших ROC кривых, правильных деревьев и чистых данных! Майните дату, а не биткоины. ;)
Литература
- Сome to the bioinformatics side: Институт биоинформатики в Санкт-Петербурге;
- 12 методов в картинках: «сухая» биология;
- Как перестать бояться и полюбить системную биологию;
- Carlus Deneke, Robert Rentzsch, Bernhard Y. Renard. (2017). PaPrBaG: A machine learning approach for the detection of novel pathogens from NGS data. Sci. Rep.. 7, 39194;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Ellis S.E., Torres L.C., Leek J. (2017). Improving the value of public RNA-seq expression data by phenotype prediction. bioRxiv;
- «Омики» — эпоха большой биологии;
- Наука витает в облаках.






